一株大肠杆菌噬菌体的分离鉴定与系统进化分析
2022-08-06赵修报王艳苗立中于金枝张莎莎程立坤
付 强,赵修报,王艳,苗立中,于金枝,张莎莎,程立坤
(1.山东省滨州畜牧兽医研究院,山东滨州 256600;2.山东绿都生物科技有限公司,山东滨州 256600)
噬菌体是侵袭细菌的病毒,是自然界中最为普遍和分布最广的群体(Suttle等,2021),其必须在活菌内寄生,有严格的宿主特异性。噬菌体可作为分子生物学研究的试验工具(Bertoglio等,2020)以及用于检测和控制致病菌,比如噬菌体疗法等(Pirnay等,2021),但在细菌发酵生产中会带来无形的威胁。本试验通过对噬菌体的研究,以了解其生长规律、进化特性,为更好的利用和防治积累经验。
1 材料与方法
1.1 噬菌体样品和菌株 大肠杆菌BL21株,污染噬菌体的发酵液。
1.2 主要试剂 SM 液:NaCl 5.8 g,MgSO4·7H2O 2.0 g,明胶0.1 g,1 mol/L Tris-HCl(pH 7.5)50 mL,加水定容至1 L,121 ℃高压蒸汽灭菌15 min。普通LB 琼脂和0.7%上层半固体LB 琼脂,按常规方法制备。Trans15K DNA Marker,北京全式金生物技术有限公司;常规限制酶EcoR V、EcoR Ⅰ等,宝生物工程有限公司。
1.3 噬菌体的分离与纯化 取10 mL 变清的发酵液8000 r/min 离心10 min,获取上清液后用0.22 μm 滤器过滤除菌后备用。取10 μL 样品液与0.5 mL 对数生长期正常菌液混合,室温放置10 min,将混合物与5 mL 上层半固体琼脂混合后,立即倾倒于LB 固体培养基上。待凝固后,37 ℃培养12 h,挑取单个空斑,纯化3 次。将纯化的噬菌体加入甘油至30%浓度,-80 ℃保存。
1.4 最佳感染复数测定(MOI)参考张琳等(2013)的方法,将宿主菌培养至对数生长前期,调菌液麦氏浓度为0.5,相当于1×108cfu/mL。按照感染复数分别为10、1、0.1、0.01、0.001、0.0001 的比例,加入噬菌体纯培养液和宿主菌,在36 ℃摇床中150 r/min 培养3.5 h后,12000 r/min 离心10 min收集上清液,测定噬菌体的效价。以产生最高噬菌体效价的MOI 为最佳感染复数。试验重复3次,设置不加噬菌体的宿主菌培养液为试验对照。
1.5 一步生长曲线的测定 参考 Koonjan 等(2020)方法,将宿主菌培养至对数生长初期(D600为0.1~0.2),按照最佳感染复数比例接种噬菌体YHphg-1,室温吸附10 min。将混合物1 mL 离心1 min(12000 r/min),沉淀重悬于1 mL LB 培养基,再转入37 ℃条件预热的100 mL 液体培养基,37 ℃、200 r/min 培养。同时开始计时,在0 时刻和每隔10 min 取样一次,连续取样120 min,取上清用双层平板法测定噬菌体效价。以感染时间为横坐标,噬菌体的效价为纵坐标,绘制一步生长曲线。试验重复3次,取其平均值。
1.6 噬菌体的测序与鉴定
1.6.1 噬菌体基因组提取 按鞠磊等(2016)的方法,取固体增殖后效价达109pfu/mL 的噬菌体液1 mL 转移至离心管,12000 g 离心5 min,去沉淀,取上清液,加200 mL 2.5 mol NaCl 20% PEG8000混合,室温15 min。4 ℃、12000 g 离心5 min,弃上清,再离心30 s,弃上清。加100 μL TE(pH 8.0),室温泡30 min,振荡溶解,加入5 μL DNase I(20 mg/μL),5 μL RNase A(20 mg/mL)和10 μL DNase I Buffer,在37 ℃水浴锅中孵育1 h,利用细菌基因组DNA 提取试剂盒提取噬菌体基因组,冷冻保存备用。
1.6.2 噬菌体的限制性酶切 在预试验中,选用了一些限制性内切酶消化噬菌体基因组DNA,以筛选能够切割噬菌体基因组的限制性内切酶。按预试验结果,采用EcoR I、EcoR V 和Sal I 进行酶切噬菌体基因组,经1.0%琼脂糖凝胶120V 电泳25 min,观察酶切情况并拍照留存。
1.6.3 重组质粒的构建 将用EcoR V 酶切后的基因组DNA,用taq 酶72 ℃反应10 min,最后将反应产物电泳并切胶回收2~3 kb 的DNA 片段。按常规操作取回收片段与pBLUE-T 载体连接,将连接产物电转化入感受态细菌DH5a中,涂麦康凯平板(Amp+)37 ℃培养16 h,次日挑取无色菌落,EcoR Ⅰ酶切检测阳性菌,扩大培养后提取质粒送上海生物工程股份有限公司测序。
1.6.4 统计绘图与测序数据分析 利用ggplot2软件包(Ginestet,2011)进行统计绘图。使用在线Blastn 工具比较核酸序列的同源性。使用pDRAW32 软件,选择限制性内切酶对噬菌体基因组进行电子酶切。利用MEGA-X 软件(Version 10.2),根据噬菌体核酸序列测序结果构建进化树,用ggtree(v3.1.3)(Yu,2020)构图,参考ICTV最新公布的2019 病毒分类系统,完成噬菌体种属的分类图。
2 结果与分析
2.1 噬菌体的分离与纯化 从感染的发酵液中成功分离并纯化出1 株噬菌体,命名为YHphg-2019。噬菌斑形态为圆形,直径3~4 mm,有晕环(图1)。据报道,晕环的形成与噬菌体释放的多糖解聚酶有关(Latka等,2017)。
2.2 噬菌体的最佳感染复数 从表1 中可以看出,当MOI=0.01时,噬菌体YHphg-2019 增殖后测定得到的噬菌体效价最高,为6.7×1011pfu/mL。因此噬菌体YHphg-2019 的最佳感染复数为0.01。
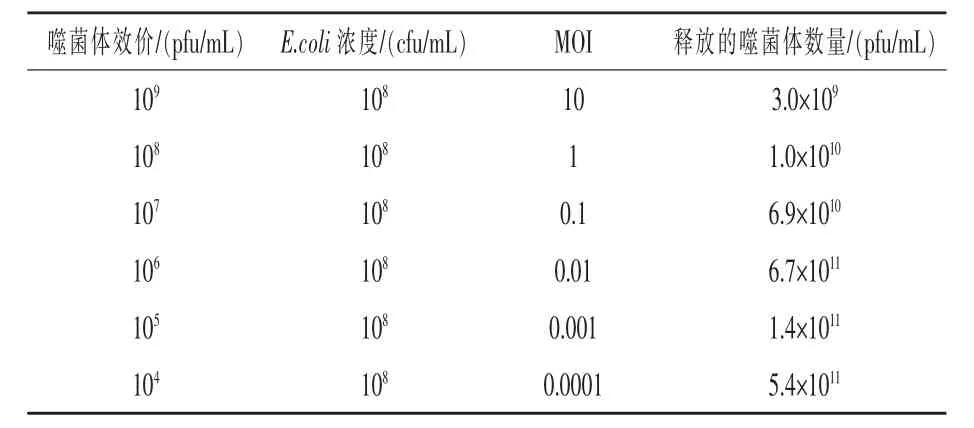
表1 噬菌体YHphg-2019最佳感染复数的测定
2.3 噬菌体的一步生长曲线 由图2 可知,噬菌体YHphg-2019 与生长对数期的宿主菌混合20 min 曲线走势无明显变化,即判断该噬菌体的潜伏期约为20 min。在20~80 min,噬菌体的滴度开始增加,而超过80 min 后曲线平稳,即判断该噬菌体的裂解时间约为80 min,因此,可得裂解量为1.86×109pfu/mL/ 1×107cfu/mL=186 pfu/cell,即噬菌体YHphg-2019 感染宿主菌的裂解量约为186 pfu/cell。

图2 噬菌体YHphg-2019 的一步生长曲线
2.4 噬菌体基因组的酶切图谱 基因组随机文库的构建一般是通过超声波法或酶切法,这里选用酶切法,3 种限制性内切酶酶切效果如图3 所示。
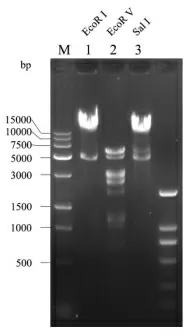
图3 噬菌体YHphg-2019 基因组的酶切图谱
可以看出,EcoR V 酶切后在3 kbp 处有明显条带,为了提高连接的成功率,选用EcoR V 的酶切产物用于噬菌体随机文库的构建。
2.5 噬菌体基因片段的克隆及质粒筛选 切胶产物克隆入pBLUE-T 质粒中,重组质粒经EcoRⅠ酶切后,出现大小不一的条带,这里选择4 号质粒送交测序(图4)。在细菌重组子的筛选方面,由于是单酶切片段连接,假阳性较高,常规方法通常为蓝白斑筛选,本试验采用了麦康凯培养基,阳性菌载体编码的β-半乳糖苷酶氨基端被插入片段打断,无法与宿主菌E.coli DH5a 实现α-β 互补,菌落呈现无色(反之显红色),从而提高阳性检出率。试验证实,虽然无法杜绝,但可有效降低假阳性。
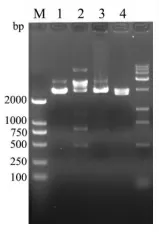
图4 重组质粒酶切鉴定
2.6 测序结果分析 测序数据显示DNA 片段大小为2608 bp,经Blastn 比对分析,与Escherichia phage vB_EcoS_SH2(NC_047828)、Escherichia phage JMPW2(NC_041873)相似度最高,Identities值 为97.13%,与Enterobacteria phage vB_EcoS_IME167(MH051912)、Escherichia virus T1(AY216660)、Shigella virus Sfin-3(NC_049831)等菌株相似度也在90%以上。以EscherichiaphagevB_EcoS_SH2 为参考株,经进一步分析,测序片段主要编码尾丝蛋白,确定属于尾噬菌体目(Caudovirales)。
2.7 噬菌体基因组的电子酶切图谱 使用限制性内切酶来确定基因组大小对噬菌体的分类和相互区分具有重要意义,组间的差异可以通过酶切位点表现出来(González-Villalobos等,2021),选择三株相似度高的噬菌体菌株进行电子酶切,如图5 所示。对比图3、图5 可以看出,在EcoR Ⅰ、EcoR V 酶切条件下,噬菌体YHphg-2019 与Escherichia phage vB_EcoS_SH2(NC_047828)表 现出较一致的酶切图谱,但在Sal Ⅰ酶切条件下又存在明显差异,反应出种间特异性。需要说明的是,通过计算机模拟的酶切图谱和实际效果还是有区别的。

图5 噬菌体基因组的酶切电子图谱
2.8 噬菌体尾丝蛋白系统进化树 国际病毒分类委员会(ICTV)于2020 年3 月批准了最新的2019 病毒分类系统,新准则在前一准则文件的基础上全面采用了十五级分类阶元,分别为:域、亚域、界、亚界、门、亚门、纲、亚纲、目、亚目、科、亚科、属、亚属、种(Adriaenssens等,2020)。大多数噬菌体属于有尾噬菌体目(Caudovirales),具有尾丝蛋白,能特异性识别并结合宿主菌表面的受体(Black等,2012)。较熟悉的有Myoviridae(肌尾噬菌体科)、Siphoviridae(长尾噬菌体科)、Podoviridae(短尾噬菌体科)以及新加的Drexlerviridae(科),资料显示尾丝蛋白具有一定保守性(Koonjan等,2020),根据噬菌体尾丝核酸序列构建进化树,如图6 所示。从进化树可以看出,长尾噬菌体科(HK446 和HK97)作为外群,噬菌体YHphg-2019 与SH2、JMPW2 等处于最近分支,结合最新的2019 病毒分类系统,属于Tunavirus(属)。

图6 根据尾丝蛋白绘制的噬菌体家族进化关系
3 结论
本试验从发酵液中分离到一株烈性噬菌体YHphg-2019,幸运的是其宿主范围较窄,通过轮换其他菌株减少了再次污染几率。试验还证实采用麦康凯抗性板“红白斑”筛选代替“蓝白斑”筛选的可行性。
该噬菌体基因组没有通过二代高通量测序仪测序,仅通过鸟枪法(shot-gun) 获得部分随机序列,来达到菌种鉴定的目的。为了提高准确性,采用物理酶切和电子酶切图谱进行了整体基因组的比较,可看出具有一致性的同时还存在种间差异。实验选择≥95%的DNA 序列同一性作为该属物种划分的标准(Evelien等,2017)。结合系统发育树和Blastn 分析,认为噬菌体分离株YHphg-2019 属于Duplodnaviria(域)、Heunggongvirae(界)、Uroviricota(门)、Caudoviricetes(纲)、Caudovirales(目)、Drexlerviridae(科)、Tunavirinae(亚科)、Tunavirus(属)。
