基于拮抗及ITS 序列分析的河南香菇主产区种质资源鉴定及遗传多样性分析
2022-08-02孔维丽张玉亭胡素娟王彦坡康源春孔维威袁瑞奇
崔 筱,刘 芹,孔维丽,张玉亭,胡素娟,王彦坡,康源春,孔维威,袁瑞奇
(1.河南省农业科学院植物营养与资源环境研究所 郑州 450002; 2.西峡县食用菌生产办公室 河南 西峡 474599)
香菇()隶属于真菌门担子菌纲香菇属,是世界上重要的食用菌栽培品种之一。中国是世界上最大的香菇生产国,2020 年我国香菇总产量为1 188.21 万t,占全国食用菌生产总量(4 061.43 万t)的29.26%。我国香菇生产基地分布于河南、河北、湖北、浙江、福建、贵州、黑龙江等省,其中,河南省2020 年香菇产量为365.08 万t,占河南省食用菌产量(561.85 万t)的64.98%,居全国第一位。河南省香菇栽培主要分布在南阳西峡、南召、桐柏,三门峡卢氏、灵宝,驻马店泌阳、驿城区,平顶山汝州、鲁山等地,栽培规模接近30 亿棒。但生产中由于引种混乱,同名异种、同种异名现象频发,伤农害农事件屡次出现。河南省农业科学院植物营养与资源环境研究所食用菌团队前期采用模糊数学和聚类分析的方法对河南省种质资源库保存的香菇表型性状进行评价,但无法确定品种基因序列遗传差异,厘清菌株之间的差异性、避免香菇菌株的混淆是推动香菇产业健康发展的必要路径。随着分子生物技术的发展,SSR、RAPD、ITS等方法在香菇、平菇、灵芝等食用菌种群的系统发育和遗传多样性鉴定中广泛应用。而拮抗法是传统的菌株鉴定方法,传统鉴定和分子生物学鉴定相结合能够较好地鉴定食用菌种内种质资源的特异性。前期,笔者已通过“拮抗+ITS”序列分析相结合的方法鉴定了54 个平菇菌株,笔者以河南省内香菇生产大县的不同菌种企业和栽培基地收集到的41 份香菇菌株为研究对象,应用“ITS+拮抗”相结合的方法进行菌种鉴定和遗传多样性分析,进而为香菇种质资源鉴定提供技术支撑。
1 材料与方法
1.1 材料
参试菌株为收集的河南省内栽培香菇菌株41个,编号及来源见表1。

表1 供试香菇菌株
母种培养基为PDA 综合培养基,购自北京奥博星生物技术有限责任公司的PDA 基础培养基40 g·L,蛋白胨2 g·L,KHPO1 g·L,MgSO0.5 g·L。主要试剂及引物为PCR Master Mix聚合酶、DNA 纯化试剂盒、DNA 凝胶回收试剂盒、PCR 扩增试剂及4sGelRed 染液,均购自上海生工有限公司;pMD-18T 载体、大肠杆菌DH5α 感受态细胞购自宝生物工程(大连)有限公司;rDNA ITS 分析所用的ITS 通用引物ITS1(5’-TCCGTAGGTGAACCTGCGG-3’)和ITS4(5’-TCCTCCGCTTATTGATATGC-3’)购自上海生工生物工程有限公司。
1.2 方法
1.2.1 拮抗试验 2021 年6—8 月收集河南省科研单位、企业及种植户栽培的香菇品种,2021 年10月,在河南省农业科学院植物营养与资源环境研究所将收集到的41 份香菇菌株在PDA 综合培养基上经活化后,采用直径5 mm 的打孔器打取各菌株种块于直径90 mm 培养皿内对峙培养,每皿放置6个菌块,种块间距1.0 cm,每个菌株3 次重复,于25 ℃培养7 d,按照拮抗试验国家标准的方法观察记录41 份参试菌株两两之间的拮抗线的形态。
1.2.2 DNA提取 将培养的香菇菌丝体置于1.5 mL无菌离心管中,在全自动快速研磨仪中研磨成粉,提取各样品的基因组DNA,基因组DNA 提取采用微波法,采用1.0%的琼脂糖凝胶检测DNA 样品质量和浓度,-20 ℃保存备用。
1.2.3 PCR 扩增 选用引物ITS1(5’-TCCGTAGGTGAACCTGCGG-3’)和ITS4(5’-TCCTCCGCTTATTGATATGC-3’)。PCR 反应体系:DNA 模板1.0 μL,PCR Master Mix 聚合酶12.5 μL,上下游引物(10 μmol·L)各1.0 μL,ddHO 4.5 μL。反应程序:95 ℃5 min,95 ℃1 min,58 ℃30 s,72 ℃50 s,72 ℃10 min,30 个循环。
1.2.4 PCR 产物检测及测序扩增 反应完毕后,取5.0 μL 的PCR 产物与1.0 μL 4sGelRed 染液混合,加样于琼脂糖凝胶点样孔中进行电泳,电泳结束后,全自动凝胶成像分析仪JS-680D 下检测扩增条带。回收正确的扩增条带(700~800 bp)连于pMD-18T 载体上,将连接产物转化于大肠杆菌()DH5α 菌株感受态细胞,涂布在含有Amp 的LB 固体培养基上,37 ℃培养过夜,菌落PCR 法验证单克隆大肠杆菌菌株,将验证正确的大肠杆菌菌株送北京六合华大基因科技有限公司测序。
1.2.5 数据分析 测序获得的序列经DNAMAN 软件多序列比对重排后,于NCBI 数据库上比对,采用MEGA-X 软件计算遗传距离,以Genebank 中平菇()为外类群,构建基于ITS 序列的系统进化树。
2 结果与分析
2.1 拮抗结果分析
41 个香菇菌株共设计拮抗组合861 组,拮抗现象明显的有480 组,占55.75%;拮抗现象不明显的有298 组,占34.61%;无拮抗的有83 组,占9.64%。其中,香9608(LE-1)、香931-2(LE-2)、庆元2 号(LE-10)、香939(LE-13)、香LS-1(LE-14)、灵仙1 号-1(LE-25)、香931(LE-34)、香升龙1-1(LE-37)、香856(LE-38)、香泌阳18-1(LE-39)、泌阳17(LE-40)、香泌阳18-2(LE-41)之间无拮抗,与其他各菌株之间拮抗明显,表明这些菌株亲缘关系较近,初步鉴定为同一菌株;同理,朕迪CGL-10(LE-20)、豫香1 号(LE-31)之间无拮抗,与其他各菌株之间拮抗明显,表明这些菌株亲缘关系较近,为同一菌株;DM-18(LE-24)、灵仙1 号-3 分(LE-27)之间无拮抗,与其他各菌株之间拮抗,明显被鉴定为同一菌株;灵仙1 号(LE-4)、灵仙1 号-5(LE-5)、雨花3 号(LE-6)、申香215(LE-8)、朕迪XZL-3(LE-17)、朕迪ZSL-8(LE-18)之间无拮抗,与其他各菌株间拮抗不明显,鉴定为同一菌株;L18(LE-3)、沪农1 号(LE- 9)、朕 迪CGL- 9(LE- 19)、朕 迪CGL- 11(LE-21)、朕 迪DM-12(LE-22)、朕 迪DM-13(LE-23)、灵仙1 号-2(LE-26)、南山1 号-3(LE-28)、香菇分(LE-29)、卢香(LE-30)、香808(LE-33)、香ZX-4(LE-35)、香939 1-2(LE-36)与其他各菌株之间拮抗明显,表明这些菌株亲缘关系较远;武香1 号-2(LE-7)、香25(LE-11)、香27(LE-12)、香平顶18-04(LE-15)、朕迪XZL-2(LE-16)、夏1(LE-32)与其他各菌株间拮抗不明显,鉴定为疑似同一菌株。拮抗试验结果可将41 个香菇菌株初步分为16 个不同菌株及7 个不同亚种(图1~2)。
图1 41 个香菇菌株拮抗试验结果
2.2 rDNA ITS区段PCR测序及NCBI比对结果
41 株香菇菌株的rDNA ITS 区段PCR 扩增均扩增到了目的条带。经电泳检测,扩增条带在700~800 bp 之间,符合测序要求。经检验条带正确的阳性克隆菌液测序后,将测序正确的核酸序列提交到NCBI 数据库获得Genebank 登录号,并与Genebank 核酸序列数据库进行BLAST 比对,得到与其最相似的物种名、登记号及序列相似性。结果表明,41 个菌株核酸序列在NCBI 上的序列相似性在99.17%~100%(表2),均为香菇.。

表2 供试菌株在NCBI 数据库中比对结果
2.3 各菌株ITS序列比对分析
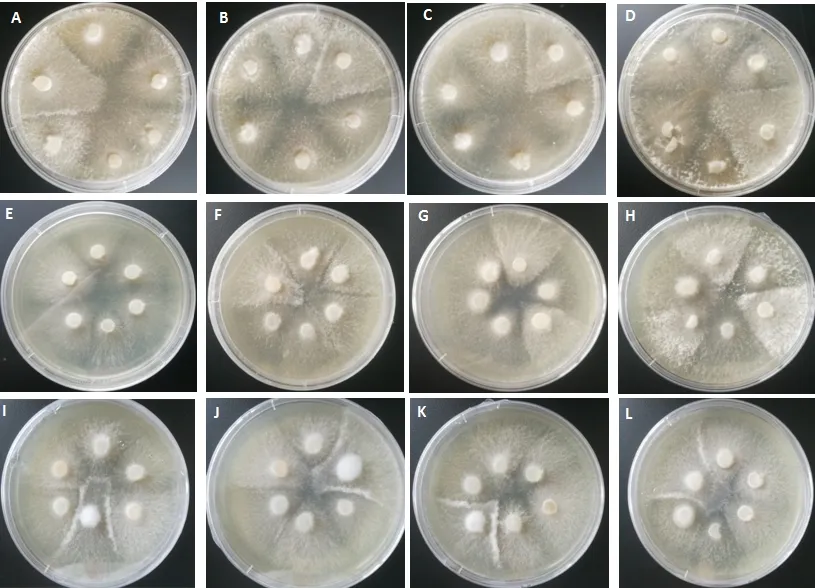
图2 香菇部分拮抗试验结果
将经测序的香菇属的41 个菌株之间进一步进行ITS 序列比对分析,结果表明,沪农1 号(LE-9)与其他各菌株的序列相似性在95.97%~96.4%,为单一菌株;香931-2(LE-2)、灵仙1 号(LE-4)、灵仙1号-5(LE-5)、申香215(LE-8)、香27(LE-12)、香939(LE-13)、香LS-1(LE-14)、香平顶18-04(LE-15)、朕迪XZL-2(LE-16)、朕迪ZSL-8(LE-18)、朕迪CGL-9(LE-19)、朕 迪CGL-10(LE-20)、朕 迪CGL-11(LE-21)、朕迪DM-12(LE-22)、朕迪DM-13(LE-23)、朕 迪DM-18(LE-24)、灵 仙1 号-1(LE-25)、灵仙1 号-2(LE-26)、灵仙1 号-3 分(LE-27)、香菇分(LE-29)、卢香(LE-30)、夏1(LE-32)、香808(LE-33)、香ZX-4(LE-35)、香939 1-2(LE-36)、香升龙1-1(LE-37)、香856(LE-38)、香泌阳18-1(LE-39)、泌阳17(LE-40)、香泌阳18-2(LE-41)与其他菌株之间的序列相似性在99.9%~100%,为同一菌株;L18(LE-3)与南山1 号-3(LE-28)序列相似性为100%,为同一菌株;香9608(LE-1)、雨花3 号(LE-6)、武香1 号-2(LE-7)、庆元2 号(LE-10)、香25(LE-11)、朕迪XZL-3(LE-17)、豫香1 号(LE-31)、香931(LE-34)与其他菌株序列相似性在98.07%~99.86%之间,为不同的亚种;41 个菌株可分为3 个不同的菌株和8 个不同的亚种。
2.4 基于ITS序列的系统发育分析
经测序获得41 株供试菌的ITS 序列长度为718~724 bp,与GeneBank 数据库中的香菇菌种的ITS 序列相似度为99%以上(表2),初步认定41 株供试菌株为香菇菌株。根据Kimura-2 参数的遗传距离模型计算各样本序列间的遗传距离(数据未提供),41 份香菇的遗传距离在0.000 0~0.006 5 之间。其中,沪农1 号与其他菌株的遗传距离在0.000 0~0.004 8 之间,香9608(LE-1)与豫香1 号(LE-31)的遗传距离最远,为0.006 5,且豫香1 号与其他各菌株的遗传距离在0.003 232~0.004 8 之间,遗传距离也较远;香931-2(LE-2)、L18(LE-3)、沪农1 号(LE-9)、庆元2 号(LE-10)、香939(LE-13)、香LS-1(LE-14)、朕迪XZL-2(LE-16)、朕迪DM-13(LE-23)、灵仙1 号-1(LE-25)、夏1(LE-32)、香931(LE-34)、香939 1-2(LE-36)与其他各菌株的遗传距离在0.001 6~0.004 8 之间,遗传距离相对较远。在Genebank 中下载平菇.(KY962509)ITS序列,长度为640 bp,将其作为外类群,与各香菇菌株的遗传距离为0.374,种间遗传距离远大于种内遗传距离。
以平菇.为组外对照,与41 份香菇种质资源的ITS 序列构建NJ 系统进化树(图3),分析系统进化树的数值结果可以发现,41 份香菇品种可以聚为4 个大类,4 个大类遗传关系相对较远、独立进化。其中,朕迪XZL-2(LE-16)、沪农1 号(LE-9)各自独立为一支。香泌阳18-2(LE-41)、香931(LE-34)、泌 阳17(LE-40)、香 泌 阳18-1(LE-39)、香856(LE-38)、香升龙1-1(LE-37)、南山1 号-3(LE-28)、灵 仙1 号-1(LE-25)、香LS-1(LE-14)、L18(LE-3)、香931-2(LE-2)、香939(LE-13)、庆元2 号(LE-10)、香9608(LE-1)聚为一支;香931(LE-34)、香泌阳18-2(LE-41)2 个菌株组成姐妹群,其自展支持强度为39,且与泌阳17(LE-40)亲缘关系较近;香9608(LE-1)、庆元2 号(LE-10)、香939(LE-13)3 个菌株组成一个单系类群,其自展支持强度为50,亲缘关系较近。其余25个品种为第四支,其中,香808(LE-33)、香ZX-4(LE-35)2 个菌株组成姐妹群,其自展支持强度为3;卢香(LE-30)、豫香1 号(LE-31)、香9391-2(LE-36)3 个菌株组成一个单系类群,其自展支持强度为6,亲缘关系较近。

图3 基于41 份香菇菌株ITS 序列构建的系统发育树
3 讨论与结论
香菇在我国人工栽培历史悠久,是我国主要的食用菌栽培品种之一。随着国家精准扶贫策略的推进,香菇短、平、快的生产特性及较高的经济效益,驱动我国的香菇产业进入了快速发展阶段。河南省是香菇生产大省,随着产业发展,菌种作为食用菌产业的核心,同种异名、同名异种、菌种间遗传关系问题一直未得到有效的解决,因此对香菇建立快速准确的鉴定方法是十分必要的。
微生物与微生物之间拮抗现象是一个复杂的反应过程,具有防止遗传上明显不同的个体间融合的作用,以保持个体遗传上的稳定性。拮抗反应试验可用于菌株之间亲缘关系远近的预测,在真菌分类学上被广泛应用。笔者对河南省内香菇主产区栽培的种质资源进行拮抗试验。结果表明,41 个香菇菌株可确定为16 个不同菌株及7 个不同的亚种。其中在西峡、泌阳、卢氏香菇种植基地收集到的香菇菌株虽然命名不同,分别命名为香9608、香931-2、香939、香LS-1、灵仙1 号-1、香931、香升龙1-1、香856、香泌阳18-1、泌阳17、香泌阳18-2,但这些菌株之间并无拮抗现象,可初步认定为同一菌株。在汝州收集到的命名为朕迪CGL-10 的菌株与三门峡市农业科学院保藏菌株豫香1 号无拮抗现象,可初步认定为同一菌株。在汝州收集到的命名为朕迪DM-18 的菌株与在灵宝香菇基地命名为灵仙1号-3 分的菌株之间无拮抗现象,可初步认定为同一菌株。在西峡、驿城区、泌阳、上海市农科院、汝州收集到的命名为灵仙1 号、灵仙1 号-5、雨花3 号、申香215、朕迪XZL-3、朕迪ZSL-8 菌株之间无拮抗现象,可初步认定为同一菌株;认定为同一菌株的菌株之间还需要借鉴分子生物学测序结果进一步比对。此外,虽然三门峡市农科院保存的菌株灵仙1 号-1 与西峡及驿城区的灵仙1 号香菇菌株名称相同,但二者拮抗现象明显,可初步认定为不同菌株。由此可见,虽然菌株间命名不同,可能为同一菌株,命名相同,却不一定是同一菌株。
物种内的遗传距离越小,则其用DNA 条形码进行分类和鉴定的效果越理想,ITS 序列在种间变异大,种内保守性高,其中,ITS2 序列片段较短,容易与单对引物结合,易于测序与扩增,且种间变异度高,在物种和亚种水平上的识别信息最为丰富,作为一种通用的DNA 条形码,在植物中已被用于近缘种的鉴定,在丝状真菌中,也作为菌种鉴定的一种手段。笔者前期对河南省山区部分野生食药用菌种质资源鉴定分析得出,菌株间序列相似性≥99.9%的定义为同一菌株,序列相似性为99.0%~99.9%的定义为不同亚种,序列相似性为95%~99%鉴别为相同属的不同菌株。笔者对本试验中41 份样本进行DNA 提取、PCR 测序,并成功获得ITS 序列,ITS 测序分析表明,41 个菌株可分为3 个不同的菌株和8 个不同的亚种,与前面拮抗试验香931-2(LE-2)、香939(LE-13)、香LS-1(LE-14)、灵仙1 号-1(LE-25)、香升龙1-1(LE-37)、香856(LE-38)、香泌阳18-1(LE-39)、泌阳17(LE-40)、香泌阳18-2(LE-41)为相同菌株,DM-18(LE-24)与灵仙1 号-3 分(LE-27)为相同菌株,灵仙1 号(LE-4)、灵仙1 号-5(LE-5)、申香215(LE-8)为相同菌株及雨花3 号(LE-6)、武香1 号-2(LE-7)、香25(LE-11)、朕迪XZL-3(LE-17)与其他菌株为不同亚种的结果一致,更进一步印证了无拮抗现象的菌株间距离的远近。此结果与前期平菇种质资源的遗传多样性分析结果相似,通过“拮抗+ITS”法,可将41 份香菇菌株确定为26 个不同菌株及4 个不同亚种。本试验结果表明,该方法可以有效地鉴定香菇种类,基本解决了河南省内香菇品种命名混乱的问题。
使用MEGA7.0 软件对41 株种内遗传距离进行计算,种内遗传距离在0.000 0~0.006 5 之间,引用外源种.(KY962509)ITS 序列作为外类群,与各香菇菌株的遗传距离为0.374,种间遗传距离明显大于种内遗传距离,说明可以使用ITS 对供试样本进行分析鉴别。以平菇.为组外对照,与41 份香菇种质资源的ITS 序列构建NJ 系统进化树,41 份香菇品种可以聚为4 个大类,4 个大类遗传关系相对较远、独立进化,同时能够观察到不同种亲缘关系的远近程度。
“ITS+拮抗法”结合可以明确地区分菌株间拮抗、无拮抗及拮抗不明显现象基因序列的相似度,遗传距离的差异及亲缘关系的远近,本研究结果可为厘清河南省香菇品种提供理论支撑。
