应用多重竞争性PCR联合毛细管电泳技术进行脊髓性肌萎缩症携带者筛查
2022-07-27李烨荣吕娟王玉国谭建新邵彬彬张菁菁
李烨荣,吕娟,王玉国,谭建新,邵彬彬,张菁菁
应用多重竞争性PCR联合毛细管电泳技术进行脊髓性肌萎缩症携带者筛查
李烨荣,吕娟,王玉国,谭建新,邵彬彬,张菁菁
南京医科大学附属妇产医院(南京市妇幼保健院)遗传医学中心, 南京 210004
脊髓性肌萎缩症(spinal muscular atrophy, SMA)是一种常染色体隐性遗传、儿童致死性神经系统疾病。SMA致病基因为运动神经元存活基因(survival motor neuron1,)。虽然检测基因拷贝数的方法众多,但目前适于大规模人群筛查的技术较少。为寻求一种快速准确的实验技术可以用于人群中SMA携带者的大规模筛查,了解区域人群携带情况及常见变异的分布,本研究应用多重竞争性PCR联合毛细管电泳技术检测12例SMA患者及其父母基因拷贝数,同时对江苏地区151例健康孕妇人群基因进行拷贝数检测,并通过多重连接依赖探针扩增(multiplex ligation-dependent probe amplification, MLPA)技术验证检测结果。多重竞争性PCR联合毛细管电泳技术结果与MLPA结果一致,显示12例SMA患者均为基因零拷贝,其父母的基因拷贝数均为单拷贝,151例健康人群中检测出基因单拷贝3例(即SMA携带者),占2.0%;基因双拷贝134例,占88.7%;基因大于双拷贝14例,占9.3%。因此,多重竞争性PCR联合毛细管电泳技术作为一种快速、简便和准确的检测技术具有着应用于人群中SMA携带者的大规模筛查的潜力。
脊髓性肌萎缩症;多重竞争性PCR联合毛细管电泳技术;基因;携带者筛查
脊髓性肌萎缩症(spinal muscular atrophy, SMA)指一类由于脊髓运动神经元变性导致的进行性、对称性近端骨骼肌无力和萎缩的一组疾病。其发病率为1/5000~1/10,000,属常染色体隐性遗传病,人群中的携带率为1/35~1/85[1]。
SMA致病基因为运动神经元存活基因(survival motor neuron ,),定位于染色体5q11.2-5q13.3区域,基因有两个高度同源的和基因,两者编码区仅在外显子7上相差1个碱基,同源性>99%。基因编码有功能的全长SMN蛋白,而基因仅能表达少量(大约10%~20%)全长且有正常功能的SMN蛋白[2],所以基因为SMA的致病基因,而基因作为修饰基因与SMA表型的严重程度相关[3]。SMA的发生90%~95%是由于基因的纯合缺失导致,而3%~5%是由于基因点突变和部分缺失的复合杂合突变导致[4]。因此,对基因进行拷贝数检测是SMA分子诊断和筛查的首选策略[5]。
虽然目前检测基因拷贝数的方法众多[6~10],但是适于大规模人群筛查的技术较少,临床上应用较多的是多重连接依赖探针扩增技术和荧光定量PCR技术。本研究拟采用一种多重竞争性PCR联合毛细管电泳技术检测基因的拷贝数,该技术既能检测及基因的拷贝数,同时对基因可能存在部分外显子(如E1~E4,E6~E8)缺失给出提示,以寻求可用于人群中大规模筛查SMA携带者的新途径,预防SMA患儿的出生。
1 对象与方法
1.1 样本来源
选取南京市妇幼保健院遗传医学中心就诊的12例SMA患者及其父母的样本,同时选取2021年1月至2021年6月在南京市妇幼保健院遗传医学中心就诊的表型正常的人群,对其进行SMA携带者筛查的宣教,其中151例自愿接受SMA携带者筛查,所有接受筛查的患者均签署知情同意书。本项研究已获得南京市妇幼保健院伦理委员会批准(宁妇伦字[2017]119号)。
1.2 外周血DNA的提取
采用外周血基因组DNA提取试剂盒(QIAGEN公司,德国)说明书提取外周血DNA,利用Nanodrop分光光度计(ThermoFisher,美国)测定DNA浓度和纯度,260/280比值在1.7~2.0之间,用1×TE (pH 8.0)将DNA浓度调整至10 ng/µL后备用,检测体系内样本DNA的加入量不宜过高或过低,以20 ng为宜。
1.3 多重竞争性PCR扩增
多重竞争性PCR技术联合毛细管电泳技术主要用于多重目标基因片段的拷贝数检测,其主要原理是通过定量合成针对每一个目标基因和每一个参照基因片段相对应的竞争性DNA片段(竞争性DNA片段与各自对应的基因片段仅有2~3 bp差别),然后取一定量的竞争性DNA片段混合物与适量的待测样本DNA混合,利用两者仅有序列上微弱的差异,可以共用同一对引物进行扩增[11,12],因此在同一反应体系对竞争性DNA和待测样本DNA的目标基因和参照基因共同扩增,由此实现在其他反应条件一致下,竞争性DNA和待测样本DNA竞争性扩增,且扩增效率接近一致,扩增产物量的比值就反应初始模板拷贝数差异,扩增产物经荧光毛细管电泳后根据扩增长度差异进行分离,获取不同基因片段的检测样本峰(S)以及竞争性DNA峰(C)的峰高值,通过数据内部标化,最后进行拷贝数分析(原理示意图见图1)。
参照文献[13~16]设计用于检测基因拷贝数的引物:(1)针对外显子7(C)、外显子8(G)及外显子7(T)、外显子8(A)上的特异碱基设计特异扩增引物对;(2)针对和基因分别设计7对和3对特异引物用于检测拷贝数;(3)针对参照区段2p、20q、10p、16p设计对应参照引物;(4)针对待测基因设计带有通用荧光(5/6羧基荧光素,FAM)的引物(表1)。实验包括若干检测样本、1个参照样本和1个空白对照。单个样本需同时进行2个PCR反应(panel1和panel2,具体基因及扩增片段信息见表2),每个反应20 µL,包含2×PCR缓冲液G 10 µL,DNA聚合酶0.3 µL,PCR引物混合液2003 (或2004) 1 µL,1×竞争DNA混合液4003 (或4004) 2 µL(包含的每个竞争性DNA片段的DNA量为20 ng),双蒸水4.7 µL,模板DNA 2.0 µL。反应体系配置完成后,振荡均匀,2000 r/min 离心10 s,再放入基因扩增仪,按以下PCR程序进行扩增:98℃ 5 min→(94℃ 20 s, 64℃每个循环减少0.5℃ 40 s,72℃ 1 min)7 个循环→(94℃ 20 s, 60℃ 30 s, 72℃ 1 min) 28个循环→60 ℃ 30 min→4℃ 保存。
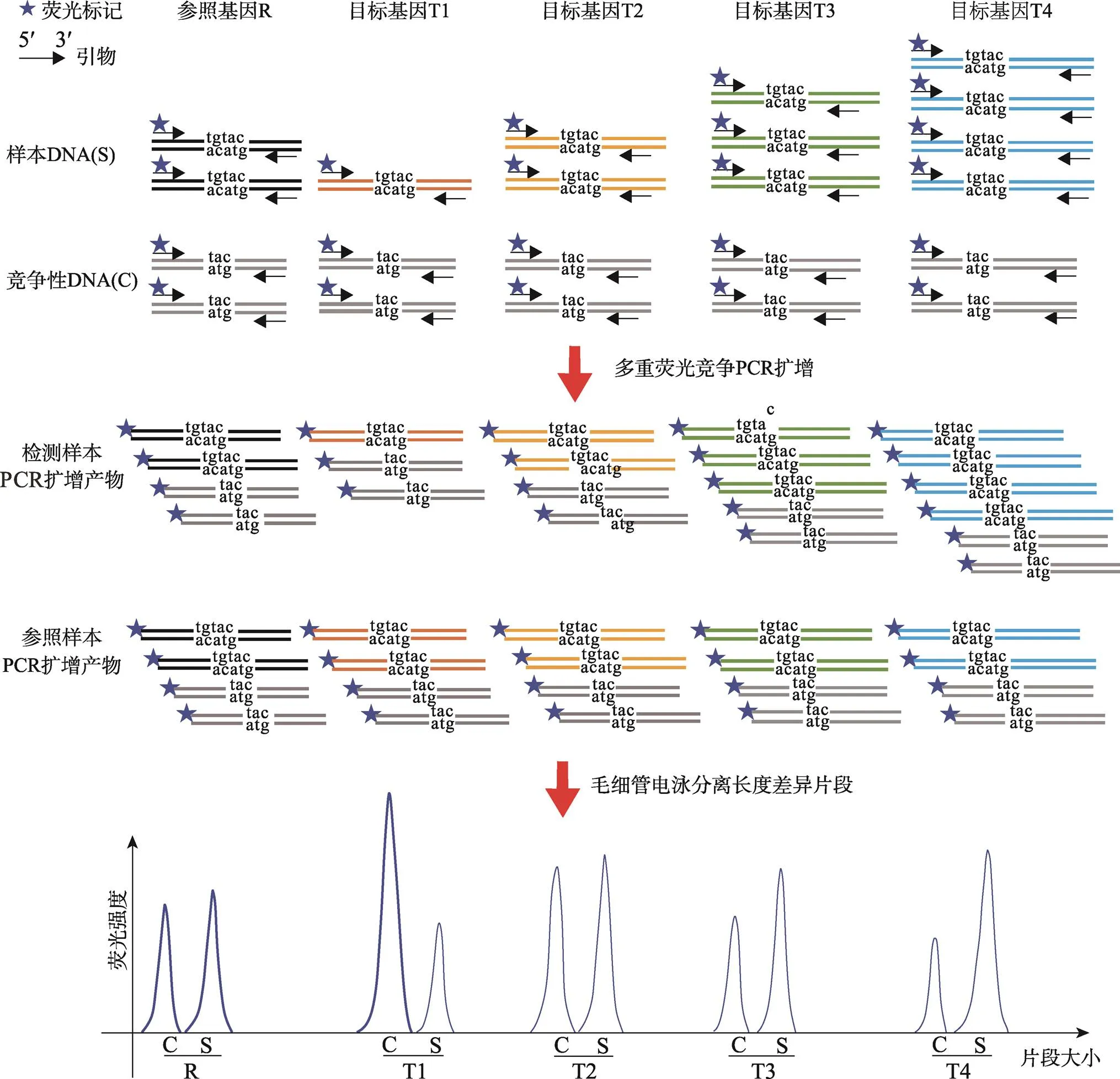
图1 多重竞争性PCR技术联合毛细管电泳技术原理图
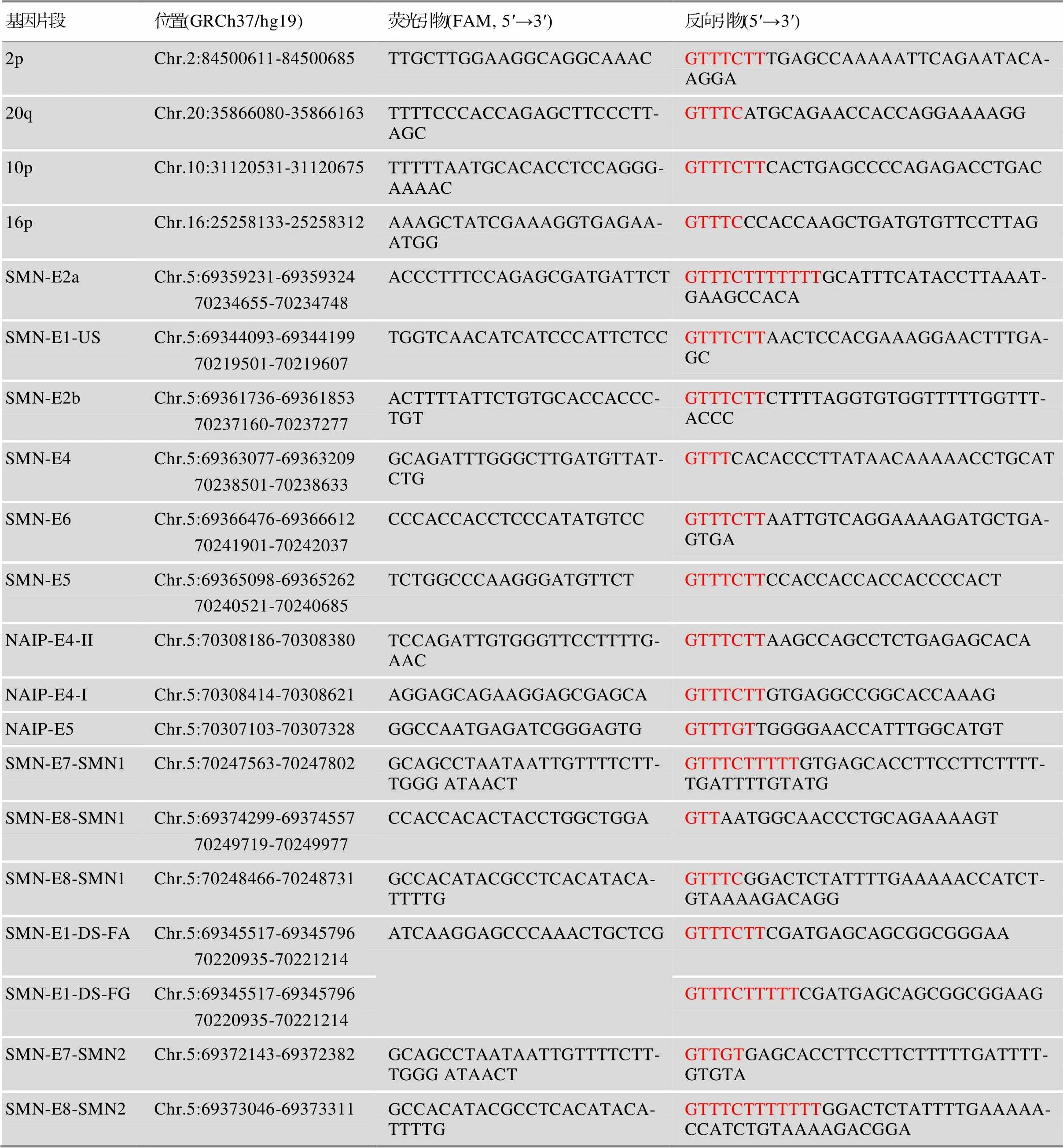
表1 PCR扩增的基因片段及引物序列
红色碱基是额外添加的碱基,其作用为调整PCR产物长度便于毛细管电泳。
1.4 扩增产物荧光毛细管电泳分离
依次吸取每个样本的Panel1和Panel2各0.75 μL PCR产物一同加入9 μL 500LIZ®标准品/Hi-Di混合液(0.1 μL /8.9 μL)中,震荡混匀后,95℃ 5 min变性后放入ABI3500基因分析仪(ThermoFisher,美国)。
1.5 数据读取及分析
通过GeneMapper软件读取原始数据,设置相应参数后(SMAF1008 Panels/SMAF1008_STR_bins/ GS500LIZ(From75)等),核对峰型图并导出位点信息。通过GeneMapper得到参照基因片段和目标基因片段的检测样本峰(S)和与之对应的竞争性DNA峰(C)的峰高值,分析每个基因片段的S/C峰高比值(称R值),然后将目标基因R值除以参照基因R值获得RR值(用于校正不同检测样本DNA用量差异),通过将检测样本的RR值除以参照样本的RR值(用于校正不同基因片段对应竞争性DNA的用量差异)后再乘以参照样本在该目标基因上的拷贝数,即可获得目标基因的精确拷贝数,计算公式如下,对每个样本的拷贝数检测数据,本研究会给出一个数据质量(QC)评估信息:对于0.00~0.20判读为0拷贝,0.80~1.20判读为1拷贝,1.70~2.30判读为2拷贝,2.70~3.30判读为3拷贝,3.60~4.40判读为4拷贝。

1、1:检测样本中目标基因的检测样本峰、竞争性DNA峰;0、0:检测样本中参照基因的检测样本峰、竞争性DNA峰;1-1、1-1:参照样本中目标基因的检测样本峰、竞争性DNA峰;0-0、C0-0:参照样本中参照基因的检测样本峰、竞争性DNA峰。
1.6 多重连接依赖探针扩增技术
本研究采用的是多重连接依赖探针扩增(multiplex ligation-dependent probe amplification, MLPA) P021 试剂盒(MRC-Holland公司,荷兰)对基因E7、E8外显子及基因E7、E8外显子进行拷贝数验证。MLPA的每一个探针都包含2个寡核苷酸片段,分别为一段引物序列和一段特异性序列,这2个寡核苷酸片段与DNA靶序列杂交,通过软件分析扩增峰的降低、升高来判断靶序列的拷贝数缺失和重复。具体操作步骤如下:(1)DNA变性及杂交:稀释样本浓度至20 ng/µL,于98℃变性5 min,降至25℃加入探针后60℃杂交16 h;(2)连接反应:待PCR仪降至54℃时加入32 µL连接酶混合液,54℃孵育15 min,98℃变性5 min后使连接酶失活;(3) PCR扩增反应:取10 µL连接产物,加入已配制好的PCR混合液(荧光引物、DNA聚合酶、缓冲液和双蒸水),反应体系为50 µL,进行PCR反应:95℃变性30 s,62℃退火35 s,72℃延伸60 s,循环35次,再72℃延伸20 min,25℃保温;(4)毛细管电泳:取1.0 µL PCR产物,加入8.5 µL Hi-Di甲酰胺(ThermFisher,美国)及0.5 µL LIZ-500Marker (ThermFisher,美国),产物通过ABI3500基因分析仪(ThermFisher,美国)进行检测;(5)数据分析:得到的数据用Coffalyser V8.0软件分析(http://www. mlpa.com/coffalyser)。应用Excel软件进行Ratio值的均数的计算。Ratio值<0.7时结果判读为缺失,0.7≤Ratio值<1.3时判读为正常,1.3≤Ratio值时判读为重复。
2 结果与分析
2.1 多重竞争性PCR联合毛细管电泳技术数据分析
所有样本经过多重竞争性PCR联合毛细管电泳技术方法检测,通过计算所有样本的基因拷贝数结果,进行统计分析获得基因为0~4拷贝的QC (数据质量)值分布情况(表3,图2),其中单拷贝与双拷贝2个标准偏差之差为0.65,3个标准偏差之差值为0.48。所有QC值组内变异系数均小于10%。所有样本QC值对应的基因拷贝数与MLPA分析结果一致。
2.2 12例SMA患者及其父母的SMN1、SMN2基因型结果
通过多重竞争性PCR联合毛细管电泳检测技术分析检测12例SMA患者及其父母的样本,检测结果显示12例SMA患者基因均为零拷贝(图3,A和E;B为其标准对照),基因为2拷贝的为5例、3拷贝的为7例,其父母的基因拷贝数均为单拷贝(图3,C和F;D为其标准对照),基因为零拷贝有1例、单拷贝的有5例、双拷贝的有13例及3拷贝的有5例。通过多重竞争性PCR联合毛细管电泳技术分析检测结果与MLPA法符合率达100.0%。
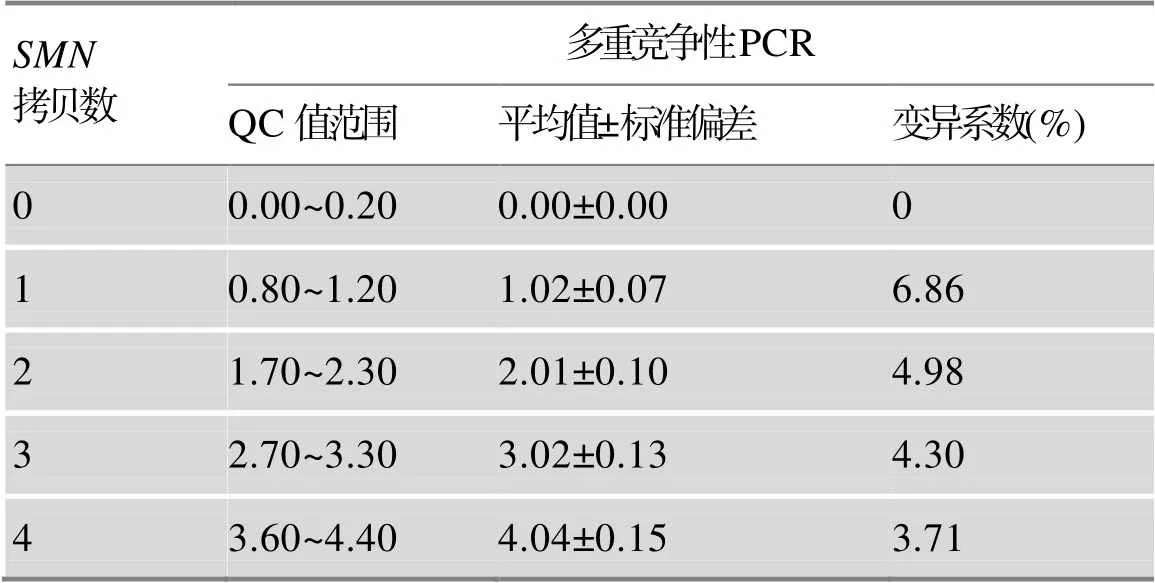
表3 SMN基因拷贝数的QC值分布情况
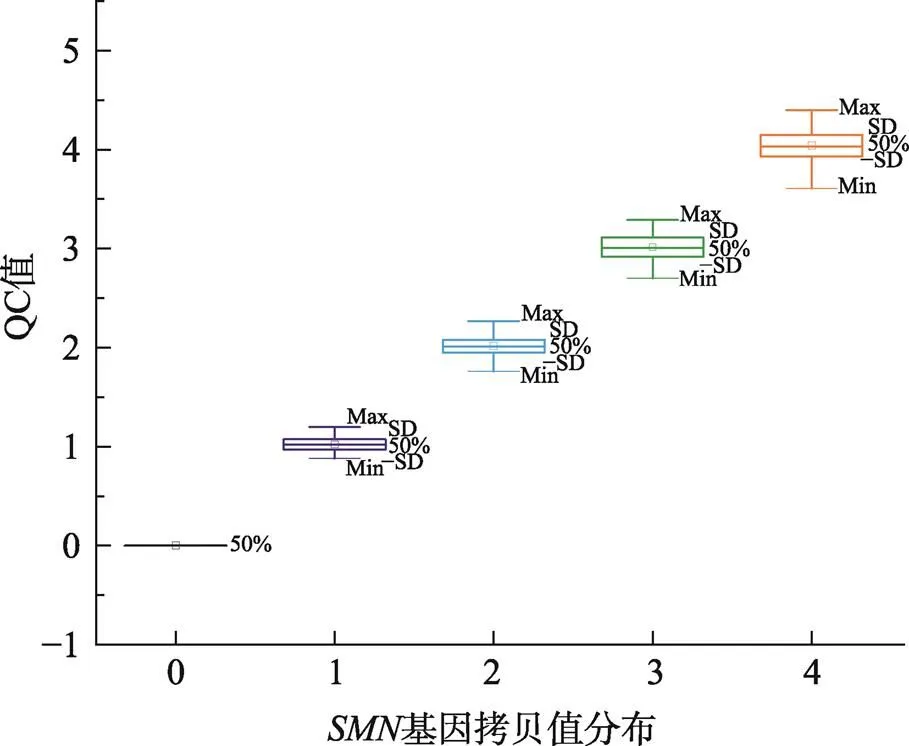
图2 SMN基因QC值分布
3.3 151例本地人群样本SMN1、SMN2基因型结果
在151例本地人群中,基因拷贝数为1的有3例,携带者频率为2.0%;双拷贝的134例,占88.7%;大于双拷贝的14例,占9.3%,不同拷贝数对应的拷贝数结果见表4。其结果与MLPA法检测结果一致。
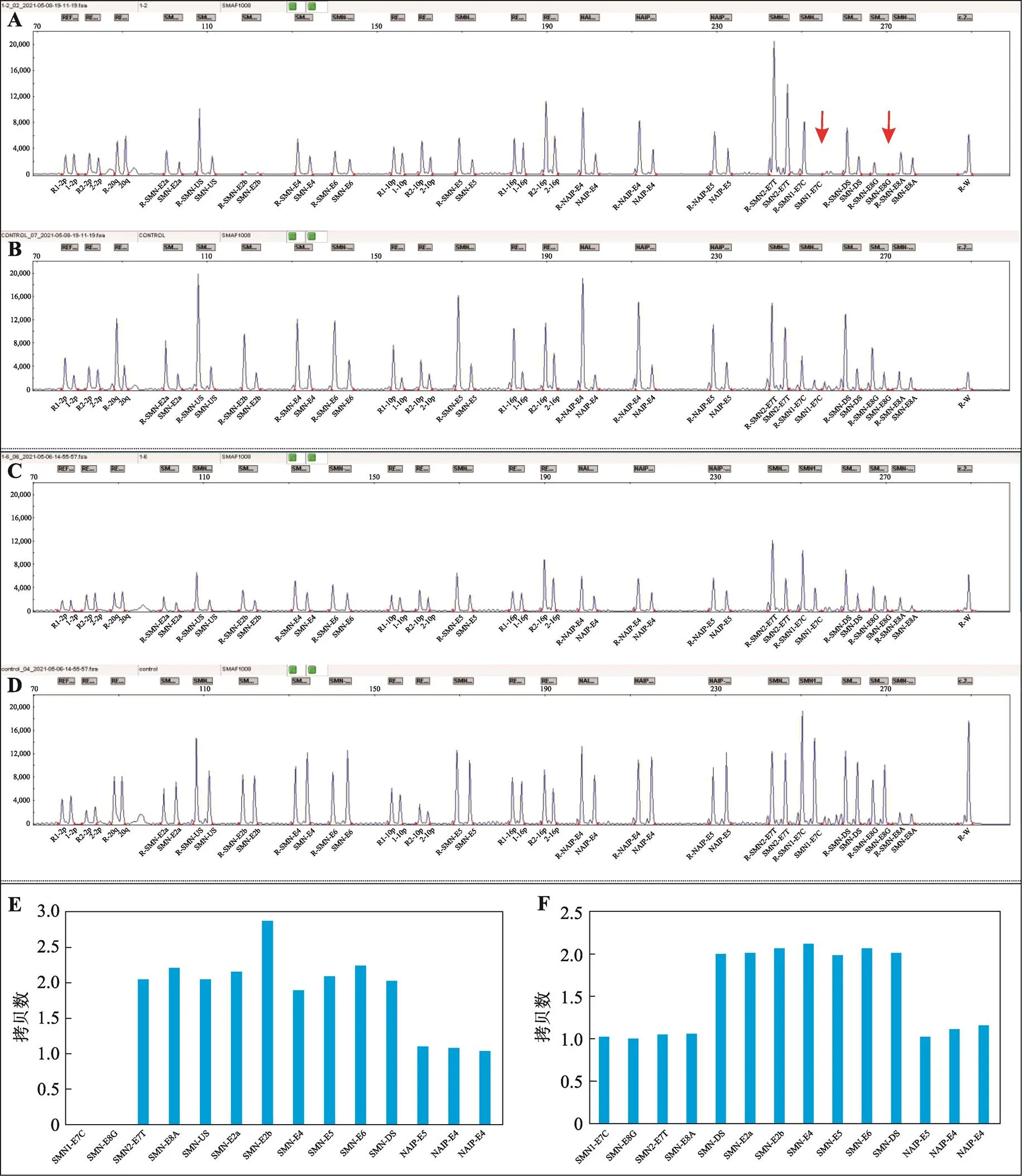
图3 1例SMA患者及1例SMA携带者的基因型
R-:竞争性DNA片段;A,B:1例SMA患者及标准对照的基因型原始峰图;C,D:1例SMA携带者及标准对照的基因型原始峰图;E:该例SMA患者的基因型柱形图:SMA患者的基因型为(E7 E8)=0、(E7 E8)=2、(US E2a E2b E4 E5 E6 DS)=2、(E4I E4II E5)=1;F:该例SMA携带者的基因型柱形图:SMA携带者的基因型为(E7 E8)=1、(E7 E8)=1、(US E2a E2b E4 E5 E6 DS)=2、(E4I E4II E5)=1。

表4 151例人群样本的SMN1及SMN2基因拷贝数
3 讨论
SMA是一种常见的致死性神经肌肉遗传病,根据发病年龄和进程将SMA分为四种类型[17]:I型(婴儿型)、II型(中间型)、III型(少年型)和IV型(成年型)。I型病情最严重,约占总病例的45%,一般于出生后6月内发病,大多数患儿在2岁内死于呼吸衰竭。II型约占40%,于出生后6~18个发病,患儿表现为能坐但无法站立和行走,生存期取决于呼吸肌麻痹程度,一般超过2岁。III型和IV型属于轻型,对生存期无明显影响。SMA病情严重,人群中携带率高且致病基因较明确,根据美国医学遗传学会(American College of Medical Genetics, ACMG)[18]、美国妇产科学会(American College of Obstetricians and Gynecologists, ACOG)[19]建议,应对所有育龄人群进行SMA携带者筛查。早在2011年中国台湾地区已为当地10万余人进行SMA携带者筛查[20],而大陆地区的大样本量筛查起步较晚,但近几年已有多篇文献报道各个省及地区的携带者筛查情况[21~24],SMA筛查已逐步成为临床常规筛查项目。
目前临床常用检测SMA拷贝数的方法包括MLPA和荧光定量PCR技术。MLPA技术是一种高效、特异的基因检测技术,不仅可对基因的拷贝数缺失定性检测,还可对待测靶点拷贝数进行半定量分析,检测结果稳定可靠,但其试剂、仪器等耗材价格昂贵、非国产化,并且操作步骤多、检测时间长、对技术人员的技术能力要求较高等缺点,导致MLPA不适于大规模的人群筛查[25,26]。相对而言,定量PCR平台在国内的普及更为广泛,现已成为分子筛查领域的重要技术。荧光定量PCR技术操作简单,可以较快速准确的检测出基因的第7、8号外显子[27],但无法检测基因以及SMA的其他相关基因,使得临床开展有一定的局限性[25]。鉴于MLPA及荧光定量PCR技术都存在不同的弊端,因此,迫切需要寻求一种适用于中国人群的SMA携带者筛查的技术体系,既能同时检测和基因的拷贝数,又能达到技术简便、快速、有效且成本较低,能适于大规模人群筛查的目的。
本研究采用多重竞争性PCR联合毛细管电泳技术检测基因的拷贝数,因脊髓性肌萎缩症(SMA)的致病基因比较特殊,基因为致病基因,基因为修饰基因,SMA的致病95%都是因为基因外显子7缺失导致,而基因外显子7拷贝数与临床表型相关。和基因高度同源,仅在外显子7上相差1个碱基(c.840C>T),所以本研究在设计引物时都会跨越这个唯一不同的碱基区域以区分基因和基因,这也是目前应用的MLPA和定量PCR引物或探针设计时遵循的原则[28,29]。该方法通过一定量的竞争性DNA片段混合物(每个竞争性DNA片段与各自对应基因片段通常仅有2~3bp差别)与适量的检测样本DNA混合,反应体系通过扩增和毛细管电泳,得到检测样本及竞争性DNA片段的参照基因和目标基因峰高值,通过对检测样本峰与竞争性DNA峰间进行校正、与参照样本比对,使结果更加可靠准确,最后直接得出待检基因相应的拷贝数。该方法不仅检测位点范围更广,相对于荧光定量PCR仅能检测的E7E8位点,还增加了E7E8和(包括E4和E5)位点,同时可以检测其余7个位于同源区的位点,即(USE2aE2bE4E5E6DS),其对应拷贝数理论上为“”,可辅助判读;而且该方法操作简单,仅需进行PCR和毛细管电泳,检测周期短,实验用时远远少于为MLPA技术,分析过程经过多重校正与比较,相对提高其结果的准确性和可重复性,并且其检测结果通过柱形图判读,简便直观(表5)。
本研究对12例SMA患者及其父母,以及151例人群样本进行和基因拷贝数检测,结果均与MLPA技术结果一致,评估该技术对SMA相关基因拷贝数检测的准确性。检测结果显示12例SMA患者的基因均为0拷贝,其父母均为基因单拷贝,即为SMA携带者;对自愿接受SMA携带者筛查的表型正常的人群151例进行检测,结果显示,基因单拷贝的有3例,携带者频率为2.0%,与之前关于中国人群SMA致病基因携带者频率的报道相近[30];双拷贝的134例,占88.7%;大于双拷贝的14例,占9.3%,所有结果与MLPA结果一致。
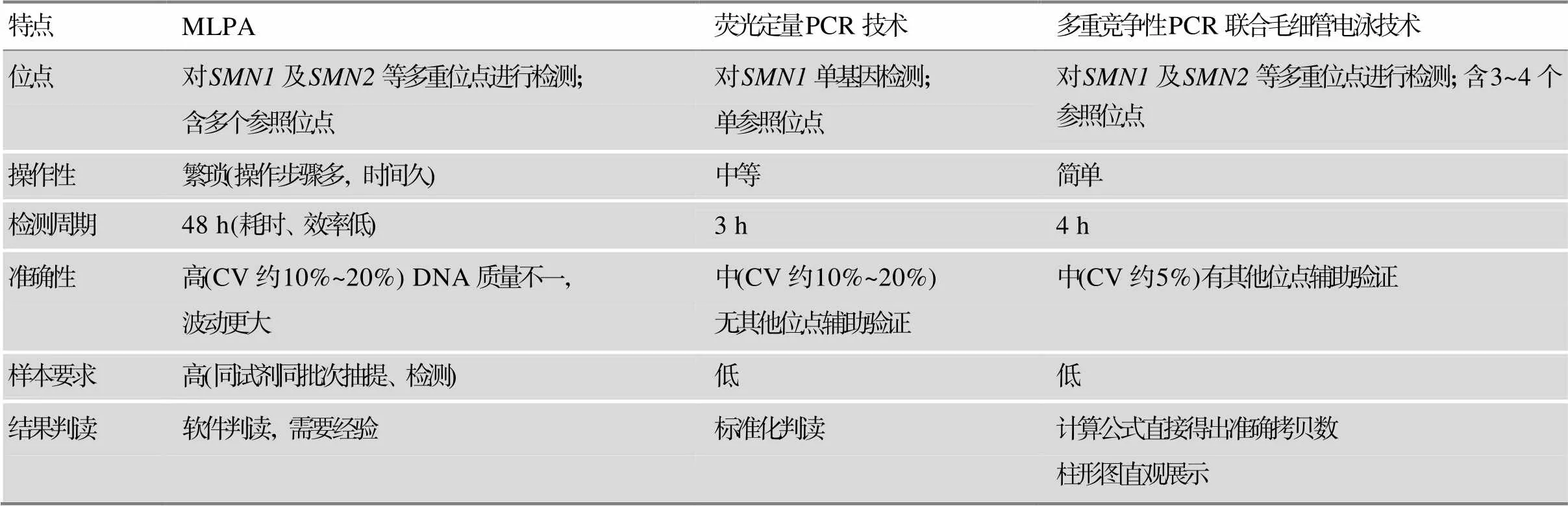
表5 多重竞争性PCR联合毛细管电泳技术、MLPA和荧光定量PCR技术特点对比
本研究旨在采用一种快速准确的实验技术可以用于人群中SMA携带者的大规模筛查,了解区域人群携带情况及常见变异的分布,同时也为构建适合中国人群SMA携带者筛查的技术体系提供研究基础。从以上研究结果看来,该方法不仅具有成本低廉、操作步骤简单、结果判读简便直观等优势,同时其敏感性和特异性高,有潜力应用于人群中SMA的大规模筛查,但仍需进一步扩大样本量以评估该方法在大样本筛查中的应用。此外,该方法也有其局限性,对于人群中少见的“2+0”型携带者及其他点突变患者无法检出,还需进行连锁分析或测序分析等检测确定。选择适合人群中SMA大规模筛查的技术方法并对高危夫妇进行生育指导,及早进行产前诊断或植入前诊断,就可以有效防止SMA患儿的出生,有效降低SMA的发病率,对提高我国人口素质有重要意义。
[1] Verhaart IEC, Robertson A, Wilson IJ, Aartsma-Rus A, Cameron S, Jones CC, Cook SF, Lochmüller H. Prevalence, incidence and carrier frequency of 5q-linked spinal muscular atrophy-a literature review., 2017, 12(1): 124.
[2] Bürglen L, Lefebvre S, Clermont O, Burlet P, Viollet L, Cruaud C, Munnich A, Melki J. Structure and organization of the human survival motor neurone (SMN) gene., 1996, 32(3): 479–482.
[3] Qu YJ, Ge XS, Bai JL, Wang LW, Cao YY, Lu YY, Jin YW, Wang H, Song F. Association of copy numbers of survival motor neuron gene 2 and neuronal apoptosis inhibitory protein gene with the natural history in a Chinese spinal muscular atrophy cohort., 2015, 30(4): 429–436.
[4] Clermont O, Burlet P, Benit P, Chanterau D, Saugier-Veber P, Munnich A, Cusin V. Molecular analysis of SMA patients without homozygous SMN1 deletions using a new strategy for identification of SMN1 subtle mutations., 2004, 24(3): 417–427.
[5] Alías L, Barceló MJ, Bernal S, Martínez-Hernández R, Also-Rallo E, Vázquez C, Santana A, Millán JM, Baiget M, Tizzano EF. Improving detection and genetic counseling in carriers of spinal muscular atrophy with two copies of the SMN1 gene.. 2014, 85(5): 470–475.
[6] Deng KY, Peng JM, Fan HG, Li LL, Xiang YQ. Application of real-time quantitative polymerase chain reaction rapid screening in spinal muscular atrophy smn1 gene carrier., 2016(37): 2983–2984.
邓坤仪, 彭建明, 范汉恭, 李丽莲, 向燕群. 荧光定量PCR快速筛查脊髓性肌肉萎缩症smn1致病基因携带者. 国际检验医学杂志, 2016(37): 2983–2984.
[7] Chen LH, Meng YT, Shu JB, Song L. Genetic diagnosis of spinal muscular atrophy using MLPA., 2012, 40(11): 1095–1098.
陈红苓, 孟英韬, 舒剑波, 宋力. 运用MLPA进行脊肌萎缩症的基因诊断. 天津医药, 2012, 40(11): 1095– 1098.
[8] Kraszewski JN, Kay DM, Stevens CF, Koval C, Haser B, Ortiz V, Albertorio A, Cohen LL, Jain R, Andrew SP, Young SD, LaMarca NM, Vivo DCD, Caggana M, Chung WK. Pilot study of population-based newborn screening for spinal muscular atrophy in New York state., 2018, 20(6): 608–613.
[9] Hwang HS, Shin GW, Jung GY, Jung GY. A simple and precise diagnostic method for spinal muscular atrophy using a quantitative SNP analysis system., 2015, 35(23): 3402–3407.
[10] Zhang XQ, Wang B, Zhang LC, You GL, Palais RA, Zhou LM, Fu QH. Accurate diagnosis of spinal muscular atrophy and 22q11.2 deletion syndrome using limited deoxynucleotide triphosphates and high-resolution melting., 2018, 19(1): 485–492.
[11] Orlando C, Pinzani P, Pazzagli M. Developments in quantitative PCR., 1998, 36(5): 255–269.
[12] Hung CC, Lee CN, Lin CY, Cheng WF, Chen CA, Hsieh ST, Yang CC, Jong YJ, Su YN, Lin WL. Identification of deletion and duplication genotypes of the PMP22 gene using PCR-RFLP, competitive multiplex PCR, and multiplex ligation-dependent probe amplification: a comparison., 2008, 29(3): 618–625.
[13] Brass N, Heckel D, Meese E. Comparative PCR: an improved method to detect gene amplification., 1998, 24(1): 22–24, 26.
[14] Wirth B, Herz M, Wetter A, Moskau S, Hahnen E, Rudnik-Schöneborn S, Wienker T, Zerres K. Quantitative analysis of survival motor neuron copies: identification of subtle SMN1 mutations in patients with spinal muscular atrophy, genotype-phenotype correlation, and implications for genetic counseling., 1999, 64(5): 1340–1356.
[15] Su YN, Hung CC, Li H, Lee CN, Cheng WF, Tsao PN, Chang MC, Yu CL, Hsieh WS, Lin WL, Hsu SM. Quantitative analysis of SMN1 and SMN2 genes based on DHPLC: a highly efficient and reliable carrier-screening test., 2005, 25(5): 460–467.
[16] Du RQ, Lu CC, Jiang ZW, Li SL, Ma RX, An HJ, Xu MF, An Y, Xia YK, Jin L, Wang XR, Zhang F. Efficient typing of copy number variations in a segmental duplication- mediated rearrangement hotspot using multiplex competitive amplification., 2012, 57(8): 545–551.
[17] Munsat TL, Davies KE. International SMA consortium meeting (26–28 June 1992, Bonn, Germany)., 1992, 2(5–6): 423–428.
[18] Prior TW, Professional Practice and Guidelines Committee. Carrier screening for spinal muscular atrophy., 2008, 10(11): 840–842.
[19] Committee Opinion No.691: Carrier Screening for Genetic Conditions., 2017, 129(3): e41–e55.
[20] Su YN, Hung CC, Lin SY, Chen FY, Chern JPS, Tsai C, Chang TS, Yang CC, Li H, Ho HN, Lee CN. Carrier screening for spinal muscular atrophy (SMA) in 107,611 pregnant women during the period 2005–2009: a prospective population-based cohort study., 2011, 6(2): e17067.
[21] Gong B, Zhang L, Hou YP, Hu HY, Li HC, Tan MY, Chen J, Yu J. Carrier screening for spinal muscular atrophy in 4719 pregnant women in Shanghai region.,2013, 30(6): 670–672.
龚波, 章莉, 侯雅萍, 胡荷宇, 李海川, 谭美玉, 陈劲, 俞菁. 中国上海地区4719名孕妇脊髓性肌萎缩症携带者筛查. 中华医学遗传学杂志, 2013, 30(6): 670–672.
[22] Zhang YH, Wang L, He J, Guo JJ, Jin CC, Tang XH, Zhang JM, Chen H, Zhang J, Su J, Zhu BS. Analysis of screening results of spinal muscular atrophy carriers in 3049 people of reproductive age in Yunnan.,2020, 37(4): 384–388.
章印红, 王蕾, 贺静, 郭晶晶, 靳婵婵,唐新华, 章锦曼, 陈红, 张杰, 苏洁, 朱宝生. 云南地区3049名育龄人群脊髓性肌萎缩症携带者筛查结果分析. 中华医学遗传学杂志, 2020, 37(4): 384–388.
[23] Xu Y, Li Y, Song TT, Zhan Y, Guo FF, Chen BL, Zhang JF. Screening of 6616 carriers of spinal muscular atrophy and analysis of prenatal genetic diagnosis in high-risk fetus., 2020, 36(01): 47–52.
徐盈, 黎昱, 宋婷婷, 詹瑛, 郭芬芬, 陈必良, 张建芳. 6616例脊髓性肌萎缩症携带者筛查及高风险胎儿产前诊断分析. 实用妇产科杂志, 2020, 36(1): 47–52.
[24] Tan JQ, Zhang X, Wang YL, Luo SQ, Yang FH, Liu BL, Cai R. Screening and prenatal diagnosis of 4931 pregnant women with spinal muscular atrophy mutation carriers in Liuzhou, Guangxi.,2018, 35(4): 467–470.
谭建强, 张旭, 王远流, 罗世强, 杨芳华, 刘百灵, 蔡稔. 广西柳州地区4931例孕妇脊髓性肌萎缩症突变携带者的筛查及产前诊断. 中华医学遗传学杂志, 2018, 35(4): 467–470.
[25] Huang CH, Chang YY, Chen CH, Kuo YS, Hwu WL, Gerdes T, Ko TM. Copy number analysis of survival motor neuron genes by multiplex ligation-dependent probe amplification.. 2007, 9(4): 241–248.
[26] Cao YY, Cheng MM, Song F, Qu YJ, Bai JL, Jin YW, Wang H. Familial study of spinal muscular atrophy carriers with SMN1 (2+0) genotype.,2021, 43(2): 160–168.
曹延延, 程苗苗, 宋昉, 瞿宇晋, 白晋丽, 金煜炜, 王红. 脊髓性肌萎缩症SMN1基因2+0基因型携带者的家系研究. 遗传, 2021, 43(2): 160–168.
[27] Hu HY, Fu XL, Yu J, Zhang B, Tang ZH, Chen HF, Zhang L, Gong B. SMN1 gene deletion analysis using mid-trimester amniotic fluid cells by real-time PCR., 2016, 39(6): 418–422.
胡荷宇, 傅行礼, 俞菁, 张蓓, 唐振华, 陈慧芬, 章莉, 龚波. 利用实时荧光定量PCR分析孕中期羊水细胞SMN1基因缺失. 中华检验医学杂志, 2016, 39(6): 418–422.
[28] Yoon S, Lee CH, Lee KA. Determination of SMN1 and SMN2 copy numbers in a Korean population using multiplex ligation-dependent probe amplification., 2010, 30(1): 93–96.
[29] Gómez-Curet I, Robinson KG, Funanage VL, Crawford TO, Scavina M, Wang W. Robust quantification of the SMN gene copy number by real-time TaqMan PCR., 2007, 8(4): 271–278.
[30] Zhang JJ, Wang YG, Ma DY, Sun Y, Li YH, Yang PY, Luo CY, Jiang T, Hu P, Xu ZF. Carrier screening and prenatal diagnosis for spinal muscular atrophy in 13,069 Chinese pregnant women., 2020, 22(6): 817–822.
Application of multiplex competitive PCR combined with capillary electrophoresis in carrier screening of spinal muscular atrophy
Yerong Li, Juan Lv, Yuguo Wang, Jianxin Tan, Binbin Shao, Jingjing Zhang
Spinal muscular atrophy (SMA) is an autosomal recessive, fatal neurological disorder in children. The pathogenic gene of SMA is survival motor neuron1 (). There are many methods to detectgene copy number, but few techniques are suitable for large-scale population screening. In order to find a rapid and accurate experimental technique for mass screening of SMA carriers in the population, thegene copy number of 12 SMA patients and their parents was analyzed by multiplex competitive PCR combined with capillary electrophoresis. Meanwhile, the copy number ofgene in 151 healthy pregnant women in Jiangsu was screened with the MLPA technology to confirm their copy number of thegenes. The results showed that the 12 SMA patients had 0 copy ofgene, and all their parents had 1 copy ofgene only. Among 151 healthy subjects, 3 cases (2.0%) had 1 copy ofgene, and hence designated as SMA carriers. One hundred and thirty-four cases (88.7%) had 2 copies of thegene. There were 14 cases (9.3%) with more than 2 copies of thegene. Therefore, multiplex competitive PCR combined with capillary electrophoresis is a rapid, simple and accurate method for the detection of SMA carriers; and potentially applicable to mass screening of SMA carriers in the population.
spinal muscular atrophy; multiplex competitive PCRcombined with capillary electrophoresis;gene; carrier screening
2022-03-29;
2022-05-13;
2022-06-01
国家自然科学基金项目(编号:82001612)资助[Supported by the National Natural Science Foundation of China (No.82001612)]
李烨荣,在读硕士研究生,专业方向:妇产科学,优生学。E-mail: lyr15835364548@163.com
张菁菁,博士,副主任医师,研究方向:妇产科学,优生学。E-mail: 18913384682@163.com
10.16288/j.yczz.22-042
(责任编委: 卢大儒)