植物RNA聚合酶IV调控DNA甲基化和发育的研究进展
2022-07-27许梦萱周明
许梦萱,周明
植物RNA聚合酶IV调控DNA甲基化和发育的研究进展
许梦萱,周明
浙江大学生命科学学院植物生物学研究所,杭州 310058
DNA甲基化是一类稳定可遗传的表观遗传修饰,在调控基因表达、沉默转座子和维持基因组稳定性等方面发挥重要作用。植物中,DNA从头甲基化通过RNA指导的DNA甲基化(RNA-directed DNA methylation, RdDM)途径建立。植物特有的DNA依赖的RNA聚合酶IV (DNA-dependent RNA polymerase IV, Pol IV)是RdDM途径核心蛋白,转录产生非编码RNA,通过RdDM途径引导从头建立DNA甲基化,进而调控植物基因表达和生长发育。Pol IV行使功能受多个蛋白调控:组蛋白阅读器SHH1 (SAWADEE homeodomain homolog 1)识别H3K9甲基化引导Pol IV到基因组特定位点;染色质重塑因子CLSY (CLASSY)蛋白家族协助Pol IV识别靶位点;RNA依赖的RNA聚合酶2 (RNA-dependent RNA polymerase 2, RDR2)将Pol IV转录产生的单链RNA转换成双链RNA。本文总结了Pol IV及其调控蛋白调控植物DNA甲基化和发育的研究进展,以期为DNA甲基化研究和农作物育种提供参考。
DNA甲基化;RdDM途径;RNA聚合酶IV;表观基因组;发育
表观遗传是指核苷酸序列不发生改变的情况下,基因表达发生可遗传改变,导致表型变化的生物学现象。DNA甲基化(DNA methylation)是一类发生在胞嘧啶(cytosine, C) 5号位碳原子上、可稳定遗传的表观遗传修饰,在动、植物基因组中广泛存在[1~3]。DNA甲基化在基因表达调控、转座子沉默和基因组稳定性维持等生命活动中起重要作用[4,5]。动物中,调控DNA甲基化的关键基因突变往往导致胚胎致死[6~8]。植物能够耐受较高程度的DNA甲基化改变,相关基因突变仍可以存活。因此,植物是研究DNA甲基化的理想材料[2,9]。
植物中DNA甲基化包括CG、CHG和CHH(H指A、T或C)3种类型,分别由不同的DNA甲基转移酶建立和维持。以拟南芥()为例,DNA甲基转移酶MET1 (methyltransferase 1)、CMT3 (chromomethylase 3)和CMT2 (chromomethylase 2)分别在DNA复制过程中维持大多数CG、CHG和CHH甲基化[10~13]。DRM2 (domains rearranged methylteransferase 2)主要负责DNA从头甲基化,它可以建立和维持所有类型的DNA甲基化,以CHH甲基化为主[14]。同时,ROS1 (repressor of silencing 1)、DME (demeter)、DML2 (demeter-like protein 2)以及DML3调控DNA去甲基化[15~19]。这些DNA甲基转移酶和去甲基化酶共同维持植物体内DNA甲基化动态平衡。
RNA指导的DNA甲基化途径是植物DNA从头甲基化的主要途径[1,5,20]。该途径由植物特有的DNA依赖的RNA聚合酶IV (Pol IV)起始,经由一系列辅助蛋白参与,产生24个核苷酸小干扰RNAs (24- nucleotide small interfering RNAs, 24 nt-siRNAs)。这些24 nt-siRNAs进一步指引DNA甲基转移酶DRM2从头建立DNA甲基化,调控基因表达、沉默转座子和维持基因组稳定性[5]。
长期以来,植物如何在基因组上找到正确的位置从头建立DNA甲基化?如何建立组织或细胞间特异的甲基化模式?如何控制DNA甲基化信息的跨代传递?这些都是表观遗传调控领域亟待回答的基本问题。近期,植物Pol IV及其调控因子,如组蛋白阅读器SHH1 (又称DNA-binding transcription factor, DTF1)、染色质重塑因子CLSY蛋白家族和RDR2调控DNA甲基化从头建立的研究,为回答这些问题提供了十分有价值的线索。本文归纳并总结了Pol IV及其共纯化因子调控植物DNA甲基化模式和发育的研究进展。
1 Pol IV是RdDM途径的核心蛋白
基因沉默是自然界普遍存在的一种调控基因表达的现象。早在20世纪90年代,研究人员在矮牵牛(L.)的花色转基因实验中发现了基因沉默(当时称为共抑制)现象[21,22]。研究发现,除了3种典型的DNA依赖的RNA聚合酶(Pol I、Pol II和Pol III),植物中还有第四、第五种DNA依赖的RNA聚合酶,即Pol IV和Pol V。它们转录产生非编码RNA,通过RdDM途径引导从头建立DNA甲基化,在基因沉默和维持基因组稳定性等生物学过程中发挥重要作用[23,24]。
1.1 植物通过RdDM途径从头建立DNA甲基化
拟南芥RdDM途径大致可以分为两个主要步骤:Pol IV主导的siRNA合成和Pol V介导的DNA甲基化。首先,Pol IV被SHH1、CLSY家族蛋白招募到基因组特定位点转录出大量30~40个核苷酸长度的单链RNA (single-stranded RNA, ssRNA)[25];同时,RNA依赖的RNA聚合酶2 (RDR2)将这些ssRNA复制成双链RNA (double-stranded RNA, dsRNA)[26~28]。这些dsRNA经过核酸内切酶Dicer样蛋白3 (dicer- like protein 3, DCL3)切割成双链24 nt-siRNAs[29]。随后,HEN1 (HUA enhancer 1)在这些双链24 nt- siRNAs的3'末端的2'OH加上一个甲基基团,提高其稳定性[30]。被甲基化的双链24 nt-siRNAs出核后,其中一条链与AGO4 (argonaute)、AGO6或AGO9结合,形成复合物再次进入细胞核[31,32]。其次,SUVH2 (SU(VAR) homologs 2)与SUVH9的SRA (SET and RING finger-associated)结构域可以结合甲基化的DNA,且可以和DRD1 (defective in RNA-directed DNA methylation 1)、DMS3 (defective in meristem silencing 3)和RDM1 (RNA-directed DNA methylation 1)组成的DDR复合物互作[33]。DMS3和RDM1首先形成DR复合体,然后DR复合体进一步招募DRD1形成DDR蛋白复合体。DRD1是一种染色质重塑因子,它可能招募Pol V到基因组特定位点[34]。然后,Pol V转录出的基因间非编码RNAs (intergenic noncoding RNAs, IGN RNAs),与AGO蛋白内的24 nt-siRNA通过序列互补配对相结合。同时,AGO复合物还能与Pol V相互作用,招募DNA甲基转移酶DRM2,进而在相应位置从头建立DNA甲基化[14,35](图1)。在RdDM途径中,Pol IV起始siRNA前体转录,在RNA指导的DNA从头甲基化途径中发挥核心作用。
1.2 Pol IV是起始RdDM途径的关键蛋白
Pol IV是一个由12个亚基组成的多亚基复合物。Pol IV和Pol II共用约一半的亚基,包括NRPD6/8/ 9/10/11/12[36]。研究认为,Pol IV的非共有亚基,是Pol II对应亚基随着植物的进化,通过独立的重复事件逐步形成[37]。与陆地植物亲缘关系最近的轮藻目Charales中,RNA聚合酶II最大亚基NRPB1复制产生了最早的Pol IV第一大亚基NRPD1,它与Pol II共享第二大亚基。之后,Pol II第二亚基在苔藓、蕨类等陆地植物中复制产生了Pol IV的第二大亚基NRPD2[38]。Pol IV的其他Pol II对应亚基也快速进化,通过复制被保留并且亚功能化[39]。这些亚基经历逐步复制和多样化,形成Pol IV独特的亚基,不同亚基的进化模式与亚基的功能有关。Pol IV亚基按照功能可以分为催化亚基和非催化亚基两类。其中,两个最大亚基NRPD1、NRPD2组成催化中心;其他亚基调控聚合酶的不同功能,如模板选择、起始、延伸和终止等步骤[40]。近期,冷冻电镜进一步揭示了Pol IV的三维结构。Pol IV的结构与Pol II类似,形如一个蟹爪,但在其活动中心裂隙(cleft)中关键基序的构象不同。此外,Pol IV缺乏Pol II起始和延伸因子的锚定表面,说明Pol IV具有不同于Pol II的转录起始机制[28]。Pol IV亚基及结构的独特性,决定了Pol IV在植物中发挥不同于Pol II的功能,即在RdDM途径中转录产生24 nt-siRNAs的前体ssRNA。
目前,Pol IV不同亚基的功能还不完全清楚,已有研究主要集中在Pol IV最大亚基NRPD1。在拟南芥中敲除导致全基因组范围内超过90%的24 nt-siRNAs消失[41]。DNA甲基化组研究发现,失活RdDM途径的不同组分对DNA甲基化的影响程度不同。当失活Pol IV的最大亚基NRPD1,会导致RdDM途径依赖的DNA甲基化几乎完全消失[42]。这些数据表明Pol IV是RdDM途径的核心蛋白,起始RdDM途径。因此,研究Pol IV如何被调控尤为重要。

图1 RNA指导的DNA甲基化途径示意图
Pol IV被招募到靶位点主要依赖SHH1和CLSY家族蛋白。SHH1可以识别组蛋白H3K9甲基化,通过蛋白互作招募Pol IV至特定位点。CLSY家族不同成员具有不同的功能来招募或辅助Pol IV到达靶位点:CLSY1、CLSY2可以和SHH1相互作用共同招募Pol IV;CLSY3、CLSY4主要和CG甲基化相关。Pol V被招募到靶位点需要SUVH2/9蛋白以及DRD1、DMS3和RDM1形成的DDR蛋白复合体。
2 Pol IV调控蛋白
除Pol IV自身12个亚基外,对Pol IV最大亚基NRPD1进行亲和纯化还鉴定出多个共纯化蛋白,例如SHH1、CLSY1-4、RDR2和RDM4等[43]。研究表明,这些蛋白参与引导Pol IV到基因组特定位点,重塑染色质协助Pol IV识别靶位点,将Pol IV转录产生的ssRNA转换成dsRNA等关键过程。
2.1 组蛋白阅读器SHH1引导Pol IV靶向基因组位置
Pol IV如何在基因组上找到其靶位点是植物表观遗传学研究的一个热点。Pol II通常被转录因子招募到特定的基因组位置,起始转录功能。到目前为止,还没有证据表明转录因子能够辅助或招募Pol IV行使转录功能,但Pol IV仍需要被招募到基因组特定位点。利用生化和质谱手段分析发现,拟南芥SHH1与Pol IV直接互作[43]。染色质免疫沉淀测序(chromatin immunoprecipitation sequencing, ChIP-seq)显示在突变体中,Pol IV不能结合到受SHH1调控的位点,说明Pol IV到达这些位点依赖SHH1蛋白。结构和生化数据显示SHH1蛋白包含一个SAWADEE结构域,可通过独特的串联Tudor样折叠(tandem Tudor-like fold)识别未甲基化的H3K4和甲基化的H3K9[44,45]。拟南芥中,组蛋白H3K9甲基化信号往往和依赖RdDM途径的DNA甲基化位点重叠。这样,SHH1蛋白将DNA从头甲基化和组蛋白H3K9甲基化联系起来,从而形成一个自身相互加强的循环。因此,SHH1可以通过识别染色质上H3K9甲基化,招募Pol IV到基因组特定位置行使转录功能。然而,拟南芥基因组中仅有44%Pol IV依赖的24 nt-siRNAs受SHH1调控,说明还存在其他蛋白调控Pol IV的功能,例如CLSY蛋白。
2.2 染色质重塑因子CLSY家族完整调控Pol IV功能
除SHH1蛋白外,还有一类SNF2 (sucrose nonfermenting 2)染色质重塑因子通过共纯化和遗传实验被鉴定出来与Pol IV互作[43,46]。因该蛋白序列中包含一个“CLASSY”的氨基酸基序,将其称为CLASSY蛋白,简称CLSY蛋白[46]。拟南芥基因组共有4个基因,形成CLSY家族,4个成员分别称为CLSY1-4[43]。在进化上,CLSY家族蛋白与DRD1同属于植物特异性的DRD1亚家族[47]。在CLSY蛋白家族内部,CLSY1和CLSY2属于一个分支;CLSY3和CLSY4属于另一个分支。这种进化上的分化,暗示它们可能行使不同的功能[48]。CLSY蛋白包含一个SNF2_N结构域(也称为DEXDc结构域)和一个解旋酶C结构域(helicase C domain)[46]。这两个结构域构成了对染色质重塑活性十分重要的ATPase (ATP synthase)域[49,50]。结构模拟显示CLSY蛋白还存在结合和水解三磷酸腺苷(adenosinetriphosphate, ATP)的motif以及一个KKRKK核定位信号[46]。这些信息表明,CLSY家族蛋白可能在核内通过水解ATP释放的能量来重塑核小体,改变DNA的可及性(accession),从而招募或调控Pol IV行使功能[51,52]。
通过对不同突变体的表观组测序和遗传学研究发现,失活单个基因,会不同程度地降低24 nt-siRNAs积累和减少对应区域的DNA甲基化水平,且不同基因调控的位点绝大部分都不重合,表明单个基因以位点特异性的方式调控DNA甲基化。其中,影响24 nt-siRNAs位点的范围最大。尽管4个基因分别影响了不同基因组区域的DNA甲基化,但是它们影响的24 nt-siRNAs只占突变体的一部分,说明它们存在功能冗余[53]。双突变体组合(和)相对于单突变体影响更多位置的24 nt-siRNAs产生和DNA甲基化,且和的作用范围不重合,与进化分析结果吻合。四突变体()和突变体几乎等同,影响拟南芥几乎所有Pol IV依赖的24 nt-siRNAs的积累和DNA甲基化水平。因此,拟南芥中4个染色质重塑因子CLSY1-4能够完整地调控Pol IV的功能,并且单个CLSY家族成员或双成员组合以位点特异性的方式调控24 nt- siRNAs的产生和DNA甲基化[54]。
利用ChIP-seq进一步发现Pol IV主要富集在24 nt-siRNAs产生位点。四突变体中,Pol IV的富集信号几乎完全消失,表明Pol IV到基因组特定位点行使功能依赖4个CLSY蛋白组合[54]。遗传分析结合小RNA测序发现与高度相似,而与作用位点不重叠,说明SHH1和CLSY1/2一起发挥作用。生化数据证明CLSY1/2影响SHH1和Pol IV的相互作用。一方面SHH1通过识别染色质上的H3K9甲基化,招募Pol IV到基因组特定位置;同时CLSY1/2发挥染色质重塑因子的作用,通过改变染色质构象辅助或招募Pol IV起始ssRNA转录。另一方面,遗传证据显示CLSY3/4调控的位点与CG甲基化关联,但CLSY3/4如何找到其靶位点,目前还不清楚。尽管有证据显示CLSY3可能通过识别特定的DNA motif招募Pol IV行使功能,但还需要证明CLSY3直接结合该motif[55]。
综上所述,CLSY家族单个蛋白可能通过改变染色质构象,以位点特异性的方式辅助或招募Pol IV到基因组特定位点起始ssRNA转录,CLSY家族可以完整调控Pol IV功能(图1)。
2.3 Pol IV-RDR2复合物将ssRNA转换成dsRNA
Pol IV被招募到其靶位点后行使转录功能,产生ssRNA。由于Pol IV转录产生的ssRNA被RDR2复制为dsRNA后,随即被DCL蛋白快速切割,使得Pol IV转录产物很长一段时间都没有被检测出来。在三突变体背景中,由于3个功能冗余的基因失活,Pol IV转录产物得以积累,因此Pol IV转录产物可以通过测序被鉴定出来[27]。研究发现,Pol IV转录区域的两侧富含A/T序列,这一特点与Pol Ⅱ转录区域的序列模式类似,显示出Pol II和Pol IV介导转录的相似性。但Pol IV转录产物具有5'单磷酸,在3'末端缺少poly(A)尾,且不含内含子,这些特征又将它们与Pol II转录产物区分开来。在基因组上,Pol IV转录产物主要来自基因间区域,其中65%与转座子或重复序列重叠,约9%与基因重叠[27]。Pol IV转录产物长度仅为30~40个核苷酸,推测一个Pol IV转录产物加工成一个24 nt-siRNA[25]。此外,Pol IV转录产物的3'末端含有碱基错配,研究发现模板链上的DNA甲基化导致不当碱基的错误整合是Pol IV转录终止的一种机制。在DNA甲基化位点附近优先终止Pol IV转录将促进先前存在的DNA甲基化位点附近的siRNA生成,从而形成一个自我强化的循环[25]。
Pol IV转录产生的ssRNA如何被传递到RDR2?近期,Pol IV-RDR2复合物三维结构的解析,为理解这一过程提供了细节。冷冻电镜结构显示,拟南芥Pol IV-RDR2形成一个全酶(holoenzyme),它们的活性位点是直接连接的,形成一个聚合酶RNA通道。首先,Pol IV合成一定长度的ssRNA后会发生转录暂停并倒退,此时该ssRNA能够直接穿过这个聚合酶RNA通道,到达RDR2活性位点,随后被RDR2作为模板合成dsRNA。完成dsRNA的合成后,又回到转录起点,开始下一轮的合成。这既能保证RDR2底物的特异性,又能确保dsRNA的产生限制在基因组的RdDM位点,同时不被RNA核酸酶降解[28]。
2.4 其他蛋白
在Pol IV的亲和纯化中,除SHH1、4个CLSY蛋白和RDR2外,还鉴定出了RDM4 (RNA-directed DNA methylation 4;也称为defective in meristem silencing 4, DMS4)蛋白[43]。RDM4是一种从酵母到人类都保守的蛋白质,该蛋白质与酵母IWR1(与RNA Pol II相互作用)具有序列相似性。突变除了减少RdDM靶位点的DNA甲基化,还导致拟南芥植株变小和部分不育等发育缺陷[56]。进一步发现,RDM4的突变直接或间接影响Pol IV依赖的siRNAs、Pol V依赖的IGN RNAs合成和许多Pol II驱动基因的表达。这些数据表明RDM4可能是这几种RNA聚合酶的共同调节因子[57]。此外,研究还发现,RDM4部分通过CBF (C-repreat-binding factor)调节拟南芥对冷胁迫的反应,RDM4在冷胁迫反应中的作用与RdDM途径无关[58],也说明RDM4在植物其他途径中发挥作用。
3 Pol IV调控蛋白CLSY家族塑造植物表观基因组模式
3.1 CLSY家族基因塑造组织和细胞特异性表观基因组模式
植物中DNA甲基化模式在不同组织和细胞间存在较大差异。例如,根尖分生组织中不同类型细胞具有独特的DNA甲基化模式。其中,中央根冠细胞的CHH甲基化水平在根尖分生组织中最高,甚至高于生殖细胞[59]。此外,拟南芥胚发育到种子萌发过程中CG和CHG甲基化较稳定,而CHH甲基化的变化程度较大[60,61]。这些研究表明,不同组织间差异化的CHH甲基化主要与DRM2和RdDM途径有关,但具体的调控基因和调控机制还不清楚。最新研究发现,CLSY家族基因具有组织特异性表达,参与调控拟南芥组织间差异化的DNA甲基化模式。
拟南芥中,CLSY家族基因表达具有很强的组织特异性。GUS报告基因系显示:CLSY1主要在叶片、花苞等的维管组织中表达;CLSY3在成熟但未受精的胚珠中富集表达;CLSY2和CLSY4的表达量较低,在叶原基和花原基中有少量表达[55]。共聚焦显微镜观察CLSY3-Venus荧光报告株系,在幼苗和叶片中无法观察到荧光信号,但在心皮和花药中荧光信号富集[62](图2)。玉米()中(required to maintain repression1,拟南芥同源基因)主要在根尖、幼苗的叶片、芽顶端分生组织和未成熟的穗等有丝分裂程度高的组织中表达[63]。CLSY家族成员的组织特异性表达模式,暗示它们可能会协同Pol IV,通过RdDM途径建立和调控组织特异性24 nt-siRNAs和DNA甲基化模式。
拟南芥不同组织间小RNA和DNA甲基化存在明显差异。例如,胚珠中数百个位点24 nt-siRNAs的丰度就占到其所有24 nt-siRNAs的约90%,这些位点被高度甲基化[55]。对突变体进行小分子RNA和DNA甲基化测序分析发现,CLSY家族基因在全基因组范围内调控组织特异性24 nt-siRNAs产生和DNA甲基化模式。CLSY1在叶片中表达最高,相应地叶片中绝大多数24 nt-siRNAs的积累和DNA甲基化模式依赖CLSY1。胚珠中,24 nt-siRNAs和DNA甲基化模式则主要受CLSY3调控[55]。甚至改变一个到两个CLSY基因就可以实现不同组织间DNA甲基化模式转换。例如:花序中,clsy1,2突变体显示出与野生型胚珠相似的24 nt-siRNAs特征;胚珠中,clsy3,4突变体显示出与野生型叶片相似的24 nt-siRNAs特征;而在叶片中,clsy1突变体则显示出与nrpd1突变体相似的24 nt-siRNAs特征[55]。这些数据表明,CLSY家族蛋白是调控拟南芥不同组织间DNA甲基化模式差异的关键因子(图2)。
除了调控DNA从头甲基化,CLSY4还被发现在DNA去甲基化中发挥作用。突变体高甲基化区域的核小体占有率降低,说明CLSY4可能通过核小体重塑促进活性DNA去甲基化[53]。同时突变体中非CG序列的高甲基化取决于RdDM途径。进一步研究表明4个CLSY蛋白除了可以共同调控经典RdDM途径;每个CLSY蛋白还可以介导依赖RdDM途径的DNA甲基化位点的去甲基化。4个CLSY蛋白在调控拟南芥基因组DNA甲基化过程中发挥双重作用,协调从头甲基化途径和主动去甲基化途径来进行基因座特异性DNA甲基化动态调节[53]。
综上所述,CLSY家族成员的表达有很强的组织特异性,它们能够单独或组合在一起通过调控RdDM途径和DNA去甲基化途径共同塑造拟南芥不同组织特异的表观基因组模式。
3.2 CLSY3调控表观遗传信息跨代传递
植物有性生殖过程中往往伴随着表观遗传信息的重编程,在这个过程中,保持转座子沉默和维持基因组的稳定性对遗传信息跨代传递十分重要[64,65]。拟南芥中,RdDM途径介导雄性生殖细胞中特异性DNA甲基化模式的建立,调节生殖细胞基因表达[66]。近期研究表明,CLSY家族蛋白在表观遗传信息跨代传递过程中发挥关键作用。
拟南芥中,特异地在花药绒毡层细胞中高度表达,而不能在减数分裂细胞中表达。小分子RNA和DNA甲基化测序发现,而不是其他基因,调控绒毡层细胞24 nt-siRNAs产生和DNA甲基化。但在突变体中不表达的雄性减数分裂细胞,原来高甲基化转座子的DNA甲基化水平和24 nt-siRNAs丰度却发生了丢失,表现出类似于突变体的特征。这表明绒毡层细胞中依赖的24 nt-siRNAs可能从绒毡层细胞(体细胞)移动到减数分裂细胞(雄性生殖细胞),通过顺式()或反式()作用方式引导建立精细胞的表观基因组模式,沉默转座子和维持基因组的稳定性,使得表观遗传信息通过精细胞在跨代传递中保持稳定(图2)。因此,CLSY家族对雄性生殖细胞特异性DNA甲基化模式的建立十分重要。值得注意的是,绒毡层细胞产生的24 nt-siRNAs在减数分裂细胞中是以不完全匹配(允许3个碱基错配)的方式靶向目标位点,引导建立DNA甲基化[62]。最近的研究表明,RdDM途径的启动并不需要24 nt-siRNAs与其核靶序列之间完全序列配对[67]。这为人们理解RdDM途径如何调控植物表观遗传信息跨代传递提供了重要线索。此外,还被发现在雌性器官胚珠中特异富集表达,调控siren siRNAs (small- interfering RNA in endosperm)的表达和DNA甲基化模式建立[55]。尽管还缺乏雌性器官细胞层面的数据,我们推测CLSY3可能是一个调控雄性和雌性生殖发育细胞DNA甲基化模式的关键蛋白。
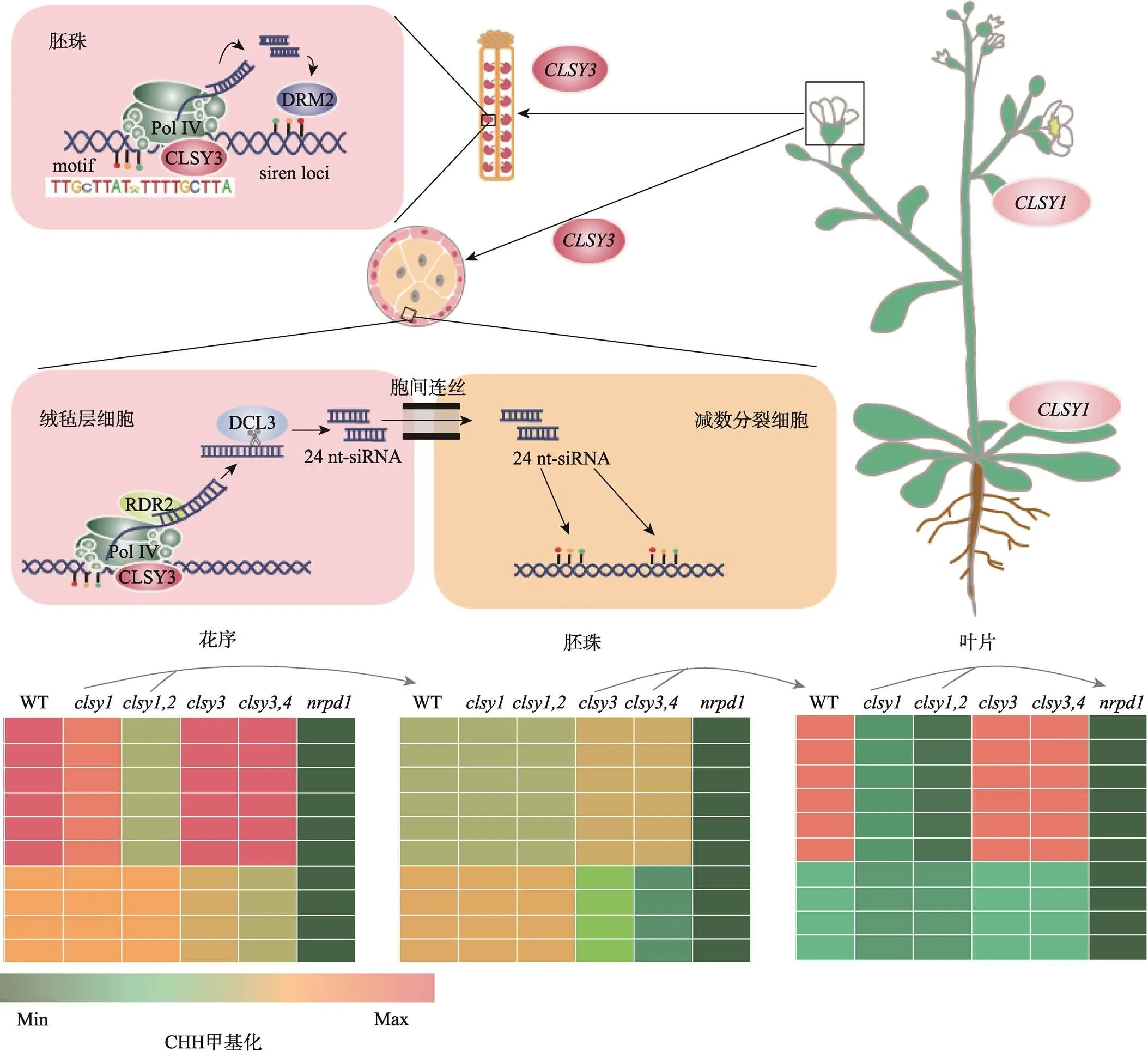
图2 CLSY家族调控拟南芥组织和细胞特异性DNA甲基化模式
以拟南芥为例,主要在成熟的叶片和莲座叶中表达,主要在胚珠和绒毡层细胞中特异性表达,和的表达量较低,图中未显示。胚珠中,CLSY3招募Pol IV与一个特定的motif相关,且可以调控siren loci的甲基化。绒毡层细胞中,CLSY3招募Pol IV产生的24 nt-siRNAs通过胞间连丝进入减数分裂细胞,调控精细胞中24 nt-siRNAs水平和DNA甲基化状态,控制拟南芥跨代遗传信息传递。不同的组织中,CLSY家族基因调控拟南芥组织特异性DNA甲基化模式。CLSY家族基因的DNA甲基化模式示意图根据文献[55]修改绘制。绒毡层中的表达根据文献[62]修改绘制。
4 Pol IV调控植物发育
Pol IV作为RdDM途径的核心蛋白,其功能缺失导致全基因组范围内24 nt-siRNAs丢失和DNA甲基化水平变化。如果这些变化位于基因附近,会影响基因的表达,进而导致植物多种发育缺陷;在水稻()、玉米等富含转座子的农作物中尤为明显。
拟南芥突变体表型较弱,突变体中诱导晚开花表型的(flowering wageningen)基因启动子区域CHH甲基化减少,该基因表达被激活,导致开花时间延迟[68,69]。拟南芥中,转座子和重复序列集中在基因很少的着丝粒周围区域[70]。这可以部分解释为什么拟南芥RdDM途径关键成分突变体没有明显的发育异常。
尽管拟南芥突变体没有明显的发育表型,但在其他双子叶植物中,缺失Pol IV会导致严重的发育缺陷[71]。与拟南芥同为十字花科的荠菜()基因组中含有大量的转座子,Pol IV功能丧失对荠菜的影响比拟南芥更强。在荠菜中,突变体花粉发育停滞在小孢子发育期、种子变小且数量变少,花粉管生长调节子()、()和等基因可能参与这一过程,但详细机制还有待进一步验证[72]。白菜型油菜()突变体种皮颜色变深。是调控种皮颜色的转录因子,在种子发育过程中显著上调,导致种皮颜色变深。此外,突变体种子变的更小、更干瘪[73]。在番茄()中,突变体的花和叶也出现明显的发育异常,且高度不育[74]。
在单子叶植物水稻中,转座子占基因组的35%以上,且部分位于常染色质区域[42]。如果DNA甲基化变化区域位于基因附近,往往会影响相邻基因的表达,进而调控植物的生长发育。在水稻中,敲除或敲低Pol IV最大亚基的两个同源基因和导致水稻株高降低、分蘖数增多、穗变小和育性变差等重要农艺性状改变。研究发现,和通过调控(ideal plant architecture 1,也称为)控制水稻分蘖。Pol IV可以调控启动子和基因下游微型反向重复转座序列(miniature inverted-repeat transposable elements, MITE)的DNA甲基化水平。在水稻突变体中,随着这些MITE位置DNA甲基化丢失,被激活而被抑制,导致水稻分蘖增多[42]。此外,Pol IV还参与水稻对病毒的抗性,水稻草状矮化病毒(rice grassy stunt virus, RGSV)编码的P3蛋白可以诱导一个U-box类型E3泛素连接酶(P3-inducible protein 1, P3IP1)特异性高表达,与OsNRPD1a互作并导致其泛素化和UPS (ubiquitin-proteasome system)依赖性降解,进而导致水稻植株矮化和分蘖增多(图3)。Pol IV的缺失还能进一步促进RGSV的侵染[75]。Pol IV功能丧失对单子叶植物玉米的植物发育也有显著的影响。玉米Pol IV最大亚基NRPD1参与叶片极性、性别决定、开花时间和副突变的调控。(required to maintain repression 6)编码玉米Pol IV最大的亚基,突变体显示多种发育缺陷:开花时间延迟,营养节间较短导致植株矮小,幼年到成年营养生长发育转变的时间变长,同时近轴-远轴极性改变,部分顶端雌蕊不发生败育导致两性花的形成。RMR6通过限制顶端花序()的功能来维持玉米的雌雄同体的性别决定模式[76]。在玉米突变体中,(liguleless 2)表达下降,该基因可以调节营养期到生殖期的转变,最终导致玉米突变体开花显著延迟[77]。()调控花青素色素合成,等位基因纯合会使营养组织,花药颜色变成深紫色,会抑制等位基因的表达。正常情况下,糊粉层中的表达会被抑制,避免色素沉积;玉米缺失不能维持抑制状态导致籽粒颜色变深[78~80]。
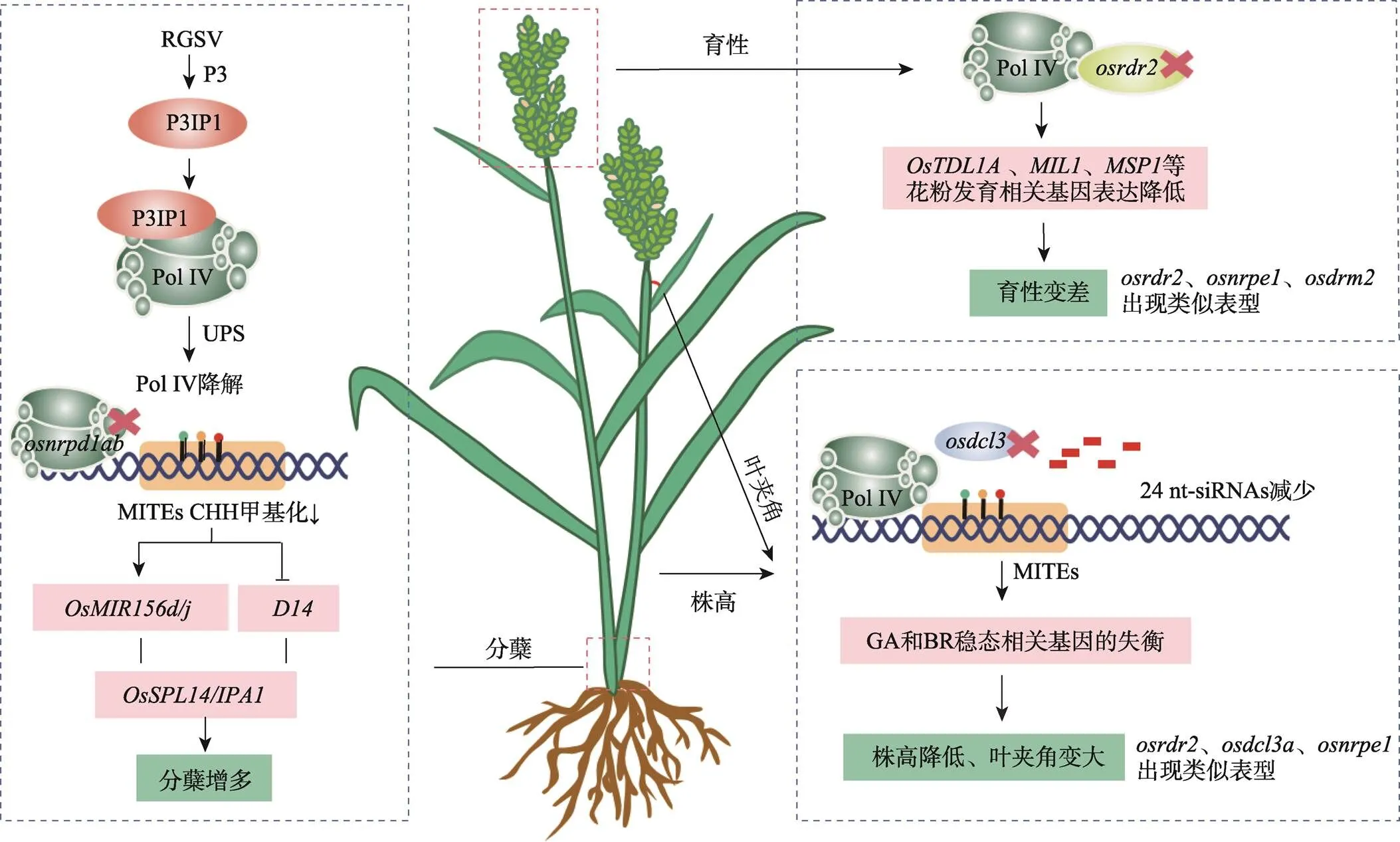
图3 Pol IV调控水稻重要农艺性状
在水稻中,水稻草状矮化病毒(RGSV)P3蛋白可以诱导P3IP1特异性高表达,增强OsNRPD1a的泛素化,通过泛素蛋白酶体系统(UPS)依赖性降解。水稻和表达的降低导致微型反向重复转座元件(MITE)CHH甲基化显著降低,从而影响调控水稻分蘖的关键农艺学重要基因(和)的表达,进而导致水稻分蘖增多。在突变体中,Pol IV转录产生的ssRNA无法复制成dsRNA,进而导致、、等花粉发育相关基因表达降低,水稻育性变差。在突变体中,24 nt-siRNAs数量显著变少,同时GA和BR稳态相关基因的失衡,水稻植株出现株高降低和叶夹角变大的表型。
除了Pol IV之外,RdDM途径中其他关键蛋白的缺失也常出现类似的异常发育表型。以水稻为例,突变体株高降低、穗变小、雌蕊,雄蕊发育异常。研究表明,依赖的DNA甲基化可能会调节参与雄蕊发育、减数分裂和花粉活力的多个关键基因的表达。例如雄蕊发育相关基因、和的表达在突变体中下降,这些基因表达的变化可能最终导致育性异常[81]。的敲低导致植物株高降低,叶夹角变大。在突变体中,赤霉素(gibberellin, GA)和油菜素内酯(brassinosteroid, BR)代谢稳态失衡从而导致形态异常,编码GA失活酶的(elongated uppermost internode)基因在敲低植物中表达上调,导致水稻矮化。外源施加赤霉素可以挽救植物的矮化表型。此外,OsDCL3a的直接靶标(GAST family gene in rice 1)可以促进BR合成,影响水稻的叶夹角的大小[82]。OsAGO4a通过调节位于启动子中的两个串联微型转座子影响水稻对稻瘟病菌的抗性[83]。的敲除同样会导致植物株高的降低和结实率的下降[84]。定向破坏,会损害水稻的营养和生殖发育,导致植株变矮、抽穗延迟或无抽穗、小穗形态异常,并且完全不育[85]。综上所述,RdDM途径关键蛋白的缺失影响植物正常生长发育,说明由Pol IV起始的RdDM途径对植物的生长发育至关重要(图3)。
5 结语与展望
Pol IV是植物特有的DNA依赖的RNA聚合酶,由12个亚基组成。Pol IV的三维结构为理解Pol IV
作用方式提供了更多细节。拟南芥中Pol IV-RDR2复合物组成一个全酶,并在内部形成一个RNA通道,使得Pol IV转录的ssRNA能准确、及时地传递给RDR2,产生dsRNA。Pol IV的共纯化蛋白中,SHH1能够识别H3K9甲基化,进而招募Pol IV到基因组靶位点。4个CLSY蛋白作为染色质重塑因子,完整调控Pol IV的功能,协助Pol IV起始转录,产生24 nt-siRNAs,起始RdDM途径,从头建立DNA甲基化,调控基因表达、沉默转座子和维持基因组稳定性。
目前,尽管围绕Pol IV的研究很多,但还有一些机制不清楚。比如,Pol II的转录过程以及其不同亚基的功能已比较清楚,但Pol IV的亚基是如何组装的,具体细节还有待厘清。组装好的Pol IV如何被招募到CLSY3/4依赖的位点,详细的分子机制还有待进一步阐明。与拟南芥不同,水稻和玉米中存在不止一个NRPD1的同源基因,他们是否存在功能上的分化,也缺乏系统研究。另外,Pol IV和RdDM途径在植物抗病方面发挥重要作用,是否参与调控其他胁迫反应,还有待进一步探索。
Pol IV对植物发育的影响在拟南芥、水稻、玉米等单、双子叶模型中都有研究。敲除Pol IV最大亚基NRPD1在拟南芥中没有明显发育表型,但在水稻、玉米、番茄和荠菜等植物中都出现了严重的发育缺陷。Pol IV调控蛋白对植物发育也有重要影响,如调控水稻株高、分蘖和育性等重要农艺性状。但目前对CLSY家族和SHH1的功能研究还较缺乏,尤其是在农作物中的研究较少。CLSY家族能够完整调控Pol IV的功能,预示着SHH1、CLSY家族的单个基因或多基因组合可能在这些农作物的发育中发挥重要作用。
Pol IV及其调控蛋白在农作物生长发育中发挥重要作用,为通过表观遗传调控改良农作物提供了新途径。近期,基于CRISPR技术的DNA甲基化靶向编辑已在植物中建立。例如,拟南芥中,基于细菌CG特异性DNA甲基转移酶MQ1的变体与失活的Cas9 (nuclease-dead mutants of Cas9, dCas9)融合,开发了基于CRISPR的CG甲基化特异性靶向系统,可以靶向甲基化拟南芥基因组特定CG位点的胞嘧啶[86]。利用带有烟草() DRM甲基转移酶催化结构域的SunTag系统可以有效地靶向基因启动子,使拟南芥开花提前[87]。在水稻中,基于SunTag系统的靶向DNA去甲基化系统可以对蛋白质编码基因和转座子进行靶向DNA去甲基化[88]。这些研究的发展为从DNA甲基化层面改良农作物提供了技术支持。
总之,对Pol IV及其调控蛋白的研究将为植物DNA甲基化研究和从表观遗传调控层面改良农作物提供有益的参考和线索。
[1] Law JA, Jacobsen SE. Establishing, maintaining and modifying DNA methylation patterns in plants and animals., 2010, 11(3): 204–220.
[2] He XJ, Chen TP, Zhu JK. Regulation and function of DNA methylation in plants and animals., 2011, 21(3): 442–465.
[3] Elhamamsy AR. DNA methylation dynamics in plants and mammals: overview of regulation and dysregulation., 2016, 34(5): 289–298.
[4] Wang RX,Xu JH. Genomic DNA methylation and histone methylation., 2014, 36(3): 191–199.
王瑞娴, 徐建红. 基因组DNA甲基化及组蛋白甲基化. 遗传, 2014, 36(3): 191–199.
[5] Zhang HM, Lang ZB, Zhu JK. Dynamics and function of DNA methylation in plants., 2018, 19(8): 489–506.
[6] Li E, Bestor TH, Jaenisch R. Targeted mutation of the DNA methyltransferase gene results in embryonic lethality., 1992, 69(6): 915–926.
[7] Robertson KD. DNA methylation and human disease., 2005, 6(8): 597–610.
[8] Cortellino S, Xu JF, Sannai M, Moore R, Caretti E, Cigliano A, Le Coz M, Devarajan K, Wessels A, Soprano D, Abramowitz LK, Bartolomei MS, Rambow F, Bassi MR, Bruno T, Fanciulli M, Renner C, Klein-Szanto AJ, Matsumoto Y, Kobi D, Davidson I, Alberti C, Larue L, Bellacosa A. Thymine DNA glycosylase is essential for active DNA demethylation by linked deamination-base excision repair., 2011, 146(1): 67–79.
[9] Finnegan EJ, Peacock WJ, Dennis ES. Reduced DNA methylation inresults in abnormal plant development., 1996, 93(16): 8449–8454.
[10] Lindroth AM, Cao XF, Jackson JP, Zilberman D, Mccallum CM, Henikoff S, Jacobsen SE. Requirement of CHROMOMETHYLASE3 for maintenance of CpXpG methylation., 2001, 292(5524): 2077–2080.
[11] Saze H, Scheid OM, Paszkowski J. Maintenance of CpG methylation is essential for epigenetic inheritance during plant gametogenesis., 2003, 34(1): 65–69.
[12] Stroud H, Greenberg MVC, Feng SH, Bernatavichute YV, Jacobsen SE. Comprehensive analysis of silencing mutants reveals complex regulation of themethylome., 2013, 152(1–2): 352–364.
[13] Stroud H, Do T, Du JM, Zhong XH, Feng SH, Johnson L, Patel DJ, Jacobsen SE. Non-CG methylation patterns shape the epigenetic landscape in., 2014, 21(1): 64–72.
[14] Cao XF, Jacobsen SE. Role of theDRM methyltransferases inDNA methylation and gene silencing., 2002, 12(13): 1138–1144.
[15] Gong ZZ, Morales-Ruiz T, Ariza RR, Roldán-Arjona T, David L, Zhu JK. ROS1, a repressor of transcriptional gene silencing in, encodes a DNA glycosylase/lyase., 2002, 111(6): 803–814.
[16] Ortega-Galisteo AP, Morales-Ruiz T, Ariza RR, Roldán- Arjona T.DEMETER-LIKE proteins DML2 and DML3 are required for appropriate distribution of DNA methylation marks., 2008, 67(6): 671–681.
[17] Tang K, Lang ZB, Zhang H, Zhu JK. The DNA demethylase ROS1 targets genomic regions with distinct chromatin modifications., 2016, 2(11): 16169.
[18] Liu R, Lang ZB. The mechanism and function of active DNA demethylation in plants., 2020, 62(1): 148–159.
[19] Kima S, Park J, Lee J, Lee KK, Park O, Choi H, Seo PJ, Cho H, Frost JM, Fischer RL, Choi Y, Ohme-Takagi M. The DME demethylase regulates sporophyte gene expression, cell proliferation, differentiation, and meristem resurrection., 2021, 118(29): e2026806118.
[20] Matzke MA, Mosher RA. RNA-directed DNA methylation: an epigenetic pathway of increasing complexity., 2014, 15(6): 394–408.
[21] Napoli C, Lemieux C, Jorgensen R. Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans., 1990, 2(4): 279–289.
[22] van der Krol AR, Mur LA, Beld M, Mol JN, Stuitje AR. Flavonoid genes in petunia: addition of a limited number of gene copies may lead to a suppression of gene expression., 1990, 2(4): 291–299.
[23] Herr AJ, Jensen MB, Dalmay T, Baulcombe DC. RNA polymerase IV directs silencing of endogenous DNA., 2005, 308(5718): 118–120.
[24] Wierzbicki AT, Haag JR, Pikaard CS. Noncoding transcription by RNA polymerase Pol IVb/Pol V mediates transcriptional silencing of overlapping and adjacent genes., 2008, 135(4): 635–648.
[25] Zhai JX, Bischof S, Wang HF, Feng SH, Lee T, Teng C, Chen XY, Park SY, Liu LS, Gallego-Bartolome J, Liu WL, Henderson IR, Meyers BC, Ausin I, Jacobsen SE. A one precursor one siRNA model for Pol IV-dependent siRNA biogenesis., 2015, 163(2): 445–455.
[26] Blevins T, Podicheti R, Mishra V, Marasco M, Wang J, Rusch D, Tang H, Pikaard CS. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guidingDNA methylation in., 2015, 4: e09591.
[27] Li SF, Vandivier LE, Tu B, Gao L, Won SY, Li SB, Zheng BL, Gregory BD, Chen XM. Detection of Pol IV/RDR2-dependent transcripts at the genomic scale inreveals features and regulation of siRNA biogenesis., 2015, 25(2): 235–245.
[28] Huang K, Wu XX, Fang CL, Xu ZG, Zhang HW, Gao J, Zhou CM, You LL, Gu ZX, Mu WH, Feng Y, Wang JW, Zhang Y. Pol IV and RDR2: a two-RNA-polymerase machine that produces double-stranded RNA., 2021, 374(6575): 1579–1586.
[29] Singh J, Mishra V, Wang F, Huang H, Pikaard CS. Reaction mechanisms of Pol IV, RDR2, and DCL3 drive RNA channeling in the siRNA-directed DNA methylation pathway., 2019, 75(3): 576–589.
[30] Yang ZY, Ebright YW, Yu B, Chen XM. HEN1 recognizes 21-24 nt small RNA duplexes and deposits a methyl group onto the 2' OH of the 3' terminal nucleotide., 2006, 34(2): 667–675.
[31] Havecker ER, Wallbridge LM, Hardcastle TJ, Bush MS, Kelly KA, Dunn RM, Schwach F, Doonan JH, Baulcombe DC. TheRNA-directed DNA methylation argonautes functionally diverge based on their expression and interaction with target loci., 2010, 22(2): 321–334.
[32] Duan CG, Zhang HM, Tang K, Zhu XH, Qian WQ, Hou Y, Wang BS, Lang ZB, Zhao Y, Wang XG, Wang PC, Zhou JP, Liang GM, Liu N, Wang CG, Zhu JK. Specific but interdependent functions forAGO4 and AGO6 in RNA-directed DNA methylation., 2015, 34(5): 581–592.
[33] Johnson LM, Du JM, Hale CJ, Bischof S, Feng SH, Chodavarapu RK, Zhong XH, Marson G, Pellegrini M, Segal DJ, Patel DJ, Jacobsen SE. SRA- and SET-domain- containing proteins link RNA polymerase V occupancy to DNA methylation., 2014, 507(7490): 124–128.
[34] Wongpalee SP, Liu SH, Gallego-Bartolomé J, Leitner A, Aebersold R, Liu WL, Yen L, Nohales MA, Kuo PH, Vashisht AA, Wohlschlegel JA, Feng SH, Kay SA, Zhou ZH, Jacobsen SE. CryoEM structures ofDDR complexes involved in RNA-directed DNA methylation., 2019, 10(1): 3916.
[35] El-Shami M, Pontier D, Lahmy S, Braun L, Picart C, Vega D, Hakimi MA, Jacobsen SE, Cooke R, Lagrange T. Reiterated WG/GW motifs form functionally and evolutionarily conserved ARGONAUTE-binding platforms in RNAi-related components., 2007, 21(20): 2539–2544.
[36] Zhou M, Law JA. RNA Pol IV and V in gene silencing: Rebel polymerases evolving away from Pol II's rules., 2015, 27: 154–164.
[37] Ream TS, Haag JR, Wierzbicki AT, Nicora CD, Norbeck AD, Zhu JK, Hagen G, Guilfoyle TJ, Pasa-Tolić L, Pikaard CS. Subunit compositions of the RNA-silencing enzymes Pol IV and Pol V reveal their origins as specialized forms of RNA polymerase II., 2009, 33(2): 192–203.
[38] Luo J, Hall BD. A multistep process gave rise to RNA polymerase IV of land plants., 2007, 64(1): 101–112.
[39] Huang Y, Kendall T, Forsythe ES, Dorantes-Acosta A, Li SF, Caballero-Pérez J, Chen XM, Arteaga-Vázquez M, Beilstein MA, Mosher RA. Ancient origin and recent innovations of RNA polymerase IV and V., 2015, 32(7): 1788–1799.
[40] Wang YQ, Ma H. Step-wise and lineage-specific diversification of plant RNA polymerase genes and origin of the largest plant-specific subunits., 2015, 207(4): 1198–1212.
[41] Zhang XY, Henderson IR, Lu C, Green PJ, Jacobsen SE. Role of RNA polymerase IV in plant small RNA metabolism., 2007, 104(11): 4536–4541.
[42] Xu L, Yuan K, Yuan M, Meng XB, Chen M, Wu JG, Li JY, Qi YJ. Regulation of rice tillering by RNA-directed DNA methylation at miniature inverted-repeat transposable elements., 2020, 13(6): 851–863.
[43] Law JA, Vashisht AA, Wohlschlegel JA, Jacobsen SE. SHH1, a homeodomain protein required for DNA methylation, as well as RDR2, RDM4, and chromatin remodeling factors, associate with RNA Polymerase IV., 2011, 7(7): e1002195.
[44] Law JA, Du JM, Hale CJ, Feng SH, Krajewski K, Palanca AMS, Strahl BD, Patel DJ, Jacobsen SE. Polymerase IV occupancy at RNA-directed DNA methylation sites requires SHH1., 2013, 498(7454): 385–389.
[45] Zhang H, Ma ZY, Zeng L, Tanaka K, Zhang CJ, Ma J, Bai G, Wang PC, Zhang SW, Liu ZW, Cai T, Tang K, Liu RY, Shi XB, He XJ, Zhu JK. DTF1 is a core component of RNA-directed DNA methylation and may assist in the recruitment of Pol IV., 2013, 110(20): 8290–8295.
[46] Smith LM, Pontes O, Searle I, Yelina N, Yousafzai FK, Herr AJ, Pikaard CS, Baulcombe DC. An SNF2 protein associated with nuclear RNA silencing and the spread of a silencing signal between cells in., 2007, 19(5): 1507–1521.
[47] Kanno T, Mette MF, Kreil DP, Aufsatz W, Matzke M, Matzke AJM. Involvement of putative SNF2 chromatin remodeling protein DRD1 in RNA-directed DNA methylation., 2004, 14(9): 801–805.
[48] Hu YF, Zhu N, Wang XM, Yi QQ, Zhu DY, Lai Y, Zhao Y. Analysis of rice Snf2 family proteins and their potential roles in epigenetic regulation., 2013, 70: 33–42.
[49] Lusser A, Kadonaga JT. Chromatin remodeling by ATP- dependent molecular machines., 2003, 25(12): 1192–1200.
[50] Mohrmann L, Verrijzer CP. Composition and functional specificity of SWI2/SNF2 class chromatin remodeling complexes., 2005, 1681(2–3): 59–73.
[51] Clapier CR, Cairns BR. The biology of chromatin remodeling complexes., 2009, 78: 273–304.
[52] Hohmann AF, Vakoc CR. A rationale to target the SWI/SNF complex for cancer therapy., 2014, 30(8): 356–363.
[53] Yang DL, Zhang GP, Wang LL, Li JW, Xu DC, Di CR, Tang K, Yang L, Zeng L, Miki D, Duan CG, Zhang HM, Zhu JK. Four putative SWI2/SNF2 chromatin remodelers have dual roles in regulating DNA methylation in., 2018, 4: 55.
[54] Zhou M, Palanca AMS, Law JA. Locus-specific control of theDNA methylation pathway inby the CLASSY family., 2018, 50(6): 865–873.
[55] Zhou M, Coruh C, Xu GH, Martins LM, Bourbousse C, Lambolez A, Law JA. The CLASSY family controls tissue-specific DNA methylation patterns in., 2022, 13(1): 244.
[56] He XJ, Hsu YF, Zhu SH, Liu HL, Pontes O, Zhu JH, Cui XP, Wang CS, Zhu JK. A conserved transcriptional regulator is required for RNA-directed DNA methylation and plant development., 2009, 23(23): 2717–2722.
[57] Kanno T, Bucher E, Daxinger L, Huettel B, Kreil DP, Breinig F, Lind M, Schmitt MJ, Simon SA, Gurazada SG, Meyers BC, Lorkovic ZJ, Matzke AJM, Matzke M. RNA-directed DNA methylation and plant development require an IWR1-type transcription factor., 2010, 11(1): 65–71.
[58] Chan ZL, Wang YP, Cao MJ, Gong YH, Mu ZX, Wang HQ, Hu YL, Deng X, He XJ, Zhu JK. RDM4 modulates cold stress resistance inpartially through the CBF-mediated pathway., 2016, 209(4): 1527–1539.
[59] Kawakatsu T, Stuart T, Valdes M, Breakfield N, Schmitz RJ, Nery JR, Urich MA, Han XW, Lister R, Benfey PN, Ecker JR. Unique cell-type-specific patterns of DNA methylation in the root meristem., 2016, 2(5): 16058.
[60] Kawakatsu T, Nery JR, Castanon R, Ecker JR. Dynamic DNA methylation reconfiguration during seed development and germination., 2017, 18(1): 171.
[61] Narsai R, Gouil Q, Secco D, Srivastava A, Karpievitch YV, Liew LC, Lister R, Lewsey MG, Whelan J. Extensive transcriptomic and epigenomic remodelling occurs duringgermination., 2017, 18(1): 172.
[62] Long JC, Walker J, She WJ, Aldridge B, Gao HB, Deans S, Vickers M, Feng XQ. Nurse cell-derived small RNAs define paternal epigenetic inheritance in., 2021, 373(6550): eabh0556.
[63] Hale CJ, Stonaker JL, Gross SM, Hollick JB. A novel snf2 protein maintains trans-generational regulatory states established by paramutation in maize., 2007, 5(10): 2156–2165.
[64] Feng Sh, Jacobsen SE, Reik W. Epigenetic reprogramming in plant and animal development., 2010, 330(6004): 622–627.
[65] Kawashima T, Berger F. Epigenetic reprogramming in plant sexual reproduction., 2014, 15(9): 613–624.
[66] Walker J, Gao HB, Zhang JY, Aldridge B, Vickers M, Higgins JD, Feng XQ. Sexual-lineage-specific DNA methylation regulates meiosis in., 2018, 50(1): 130–137.
[67] Fei Y, NyikóT, Molnar A. Non-perfectly matching small RNAs can induce stable and heritable epigenetic modifications and can be used as molecular markers to trace the origin and fate of silencing RNAs., 2021, 49(4): 1900–1913.
[68] Pikaard CS, Haag JR, Ream T, Wierzbicki AT. Roles of RNA polymerase IV in gene silencing., 2008, 13(7): 390–397.
[69] Pontier D, Yahubyan G, vega D, Bulski A, Saez-vasquez J, Hakimi MA, Lerbs-Mache S, Colot V, Lagrange T. Reinforcement of silencing at transposons and highly repeated sequences requires the concerted action of two distinct RNA polymerases IV in., 2005, 19(17): 2030–2040.
[70] Initiative AG. Analysis of the genome sequence of the flowering plant., 2000, 408(6814): 796–815.
[71] Zhang S, Wu XQ, Xie HT, Zhao SS, Wu JG. Multifaceted roles of RNA polymerase IV in plant growth and development., 2020, 71(19): 5725–5732.
[72] Wang ZX, Butel N, Santos-González J, Borges F, Yi J, Martienssen RA, Martinez G, Köhler C. Polymerase IV plays a crucial role in pollen development in., 2020, 32(4): 950–966.
[73] Grover JW, Kendall T, Baten A, Burgess D, Freeling M, King GJ, Mosher RA. Maternal components of RNA-directed DNA methylation are required for seed development in., 2018, 94(4): 575–582.
[74] Gouil Q, Baulcombe DC. DNA Methylation signatures of the plant chromomethyl transferases., 2016, 12(12): e1006526.
[75] Zhang C, Wei Y, Xu L, Wu KC, Yang L, Shi CN, Yang GY, Chen D, Yu FF, Xie Q, Ding SW, Wu JG. A Bunyavirus-inducible ubiquitin ligase targets RNA polymerase IV for degradation during viral pathogenesis in rice., 2020, 13(6): 836–850.
[76] Parkinson SE, Gross SM, Hollick JB. Maize sex determination and abaxial leaf fates are canalized by a factor that maintains repressed epigenetic states., 2007, 308(2): 462–473.
[77] Forestan C, Farinati S, Aiese Cigliano R, Lunardon A, Sanseverino W, Varotto S. Maize RNA Pol IV affects the expression of genes with nearby TE insertions and has a genome-wide repressive impact on transcription., 2017, 17(1): 161.
[78] Hollick JB, Kermicle JL, Parkinson SE. Rmr6 maintains meiotic inheritance of paramutant states in., 2005, 171(2): 725–740.
[79] Erhard KF Jr, Stonaker JL, Parkinson SE, Lim JP, Hale CJ, Hollick JB. RNA polymerase IV functions in paramutation in., 2009, 323(5918): 1201–1205.
[80] Hollick JB. Paramutation and related phenomena in diverse species., 2017, 18(1): 5–23.
[81] Wang LL, Zheng KZ, Zeng LJ, Xu DC, Zhu TX, Yin YM, Zhan HD, Wu YF, Yang DL. Reinforcement of CHH methylation through RNA-directed DNA methylation ensures sexual reproduction in rice., 2022, 188(2): 1189–1209.
[82] Wei LY, Gu LF, Song XW, Cui XK, Lu ZK, Zhou M, Wang LL, Hu FY, Zhai JX, Meyers BC, Cao XF. Dicer-like 3 produces transposable element-associated 24-nt siRNAs that control agricultural traits in rice., 2014, 111(10): 3877–3882.
[83] Deng YW, Zhai KR, Xie Z, Yang DY, Zhu XD, Liu JZ, Wang X, Qin P, Yang YZ, Zhang GM, Li Q, Zhang JF, Wu SQ, Milazzo J, Mao B, Wang E, Xie H, Tharreau D, He ZH. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance., 2017, 355(6328): 962–965.
[84] Zheng KZ, Wang LL, Zeng LJ, Xu DC, Guo ZX, Gao XQ, Yang DL. The effect of RNA polymerase V on 24-nt siRNA accumulation depends on DNA methylation contexts and histone modifications in rice., 2021, 118(30):e2100709118.
[85] Moritoh S, Eun C, Ono A, Asao H, Okano Y, Yamaguchi K, Shimatani Z, Koizumi A, Terada R. Targeted disruption of an orthologue of DOMAINS REARRANGED METHYLASE 2, OsDRM2, impairs the growth of rice plants by abnormal DNA methylation., 2012, 71(1): 85–98.
[86] Ghoshal B, Picard CL, Vong B, Feng SH, Jacobsen SE. CRISPR-based targeting of DNA methylation inby a bacterial CG-specific DNA methyltransferase., 2021, 118(23): e2125016118.
[87] Papikian A, Liu WL, Gallego-Bartolomé J, Jacobsen SE. Site-specific manipulation ofloci using CRISPR-Cas9 SunTag systems., 2019, 10(1): 729.
[88] Tang SJ, Yang C, Wang D, Deng X, Cao XF, Song XW. Targeted DNA demethylation produces heritable epialleles in rice., 2022, 65(4): 753–756.
Advances of RNA polymerase IV in controlling DNA methylation and development in plants
Mengxuan Xu, Ming Zhou
DNA methylation is a type of stable epigenetic modifications that plays crucial roles in regulating gene expression, silencing transposons and maintaining genome stability. In plants, theDNA methylation is established via a pathway termed as RNA-directed DNA methylation (RdDM). The plant-specific DNA-dependent RNA polymerase IV (Pol IV) as the core protein in RdDM pathway produces non-coding RNAs that direct the establishment of DNA methylation, regulates gene expression and controls plant development.Pol IV function is regulated by several proteins including SHH1, which recognizes H3K9 methylation and guides Pol IV to genome specific sites, the chromatin remodeling factor CLSY family that is involved in assisting Pol IV chromatin association and RDR2 that converts Pol IV produced single-stranded RNA into double-stranded RNA. In this review, we summarize the latest progress on Pol IV and its co-regulators, and focus on their functions in shaping epigenome and development in plants, which might provide implications for studying of DNA methylation and crop breeding.
DNA methylation; RdDM pathway; Pol IV; epigenome; development
2022-03-08;
2022-06-07;
2022-06-21
中央高校111计划(编号:B14027)和浙江大学“百人计划”资助[Supported by 111 Project (No. B14027) and the Hundred-Talent Program of Zhejiang University]
许梦萱,在读硕士研究生,专业方向:植物学。E-mail: 21907017@zju.edu.cn
周明,博士,研究员,研究方向:温度调控植物发育的遗传和表观遗传基础。E-mail: mingzhou@zju.edu.cn
10.16288/j.yczz.22-063
(责任编委: 陆发隆)