分子伴侣作用下的蛋白质稳定与进化
2022-07-15唐宇琦叶松涛刘嘉张鑫
唐宇琦,叶松涛,刘嘉,张鑫
(1西湖大学理学院化学系,浙江 杭州 310030;2浙江西湖高等研究院,理学研究所,浙江 杭州 310024)
蛋白质在生物体内起到十分重要的作用,从基础的能量储存到复杂的生物代谢都离不开蛋白质。蛋白质在生物体内通常会被折叠成多级结构,蛋白质折叠的复杂程度决定了其结构和功能的多样性。大多数天然蛋白质的折叠仅具有边缘稳定性(marginal stability),这种边缘稳定性在保证蛋白质功能正常的情况下,赋予了蛋白质在结构上偏离最稳定状态去行使功能的能力。边缘稳定性的弊端是蛋白质容易发生错误折叠,导致蛋白质丧失正常功能。因此,生物体内有一套复杂的质量控制机制用来帮助蛋白质正确折叠、修复或降解错误折叠的蛋白质。但是作为生物体内重要的工具,蛋白质在维持稳定的同时,也同样在进化。在实验室里,科学家们通过定向进化(directed evolution)的方法来产生并优化蛋白质的新功能,用于开发新型催化剂、新型药物抗体、生产可再生燃料等。蛋白质的定向进化需要具有新功能的蛋白质突变体,这就要求蛋白质折叠过程能够适应翻译错误的新突变体,并将它们折叠成具有新功能的复杂多级结构。蛋白质的稳定性(stability)是生物体内生理活动正常进行的基础,而蛋白质的可进化性(evolability)则是生物体适应自然条件选择的关键因素。围绕这一关键问题,本文首先简述了蛋白质个体的稳定性(protein stability)以及蛋白质稳态(protein homeostasis)的异同,并详尽介绍了以热休克蛋白(heat shock protein,Hsp)为主的分子伴侣(molecular chaperone)对于维持蛋白质个体稳定性的重要作用,以及通过多种手段维持内环境蛋白质稳态的研究。随后本文总结了蛋白质稳定性与可进化性关系的研究进展,并通过分子伴侣辅助定向进化、分子伴侣自身定向进化两方面实例,阐述了蛋白质稳定性对于可进化性的重要意义。
1 蛋白质的稳定性
1.1 蛋白质的天然结构仅具有边缘稳定性
多肽链经核糖体合成后通常需要折叠成一个在体内具有生理功能的多级结构,也称为天然结构(native structure)。诺贝尔奖得主Christian Anfinsen的早期实验提出蛋白质的天然结构是其正常折叠时的最低能量态。尽管如此,人们通过对天然蛋白质的氨基酸序列进行突变发现,天然蛋白质序列并非保持生理功能前提下的最稳定序列,多数蛋白质可以在保持乃至提高功能的情况下得到热力学更稳定的突变体。其中蛋白酶作为天然蛋白质的代表,因催化活性检测方法比较简单、直观,并且研究体系也不复杂,成为被广泛研究的体系。例如,Giver 等早在1998 年通过将野生型(wild-type)对硝基苄基酯酶(pNBE)进行体外定向进化,得到了既能保持低温活性又具有更好热稳定性的突变体[1];同一年,Van Den Burg通过定点突变增强嗜热菌蛋白酶样蛋白酶(TLP-ste)的刚性结构,得到能够在极端环境中保持其活性的突变体[2];Miyazaki等人在2000年也仅通过7个氨基酸残基的突变就得到稳定性大幅增强的嗜冷酶突变体,并发现该突变体稳定性的增加来源于与Ca2+结合能力的增强[3]。虽然这些研究都只是针对天然蛋白质氨基酸序列及其特定催化功能的探究,但以实践表明天然蛋白质并非处于最稳定状态。除此以外,蛋白质错误折叠时形成的无规蛋白质沉淀(amorphous protein aggregates)和淀粉样蛋白质纤维(amyloid fibrils)均被证实比天然结构更稳定。
蛋白质的稳定性不仅仅决定于氨基酸序列,还决定于其他与蛋白质相互作用的生物大分子以及周围的物理化学环境。例如,研究发现生物素(biotin)与链霉亲和素(streptavidin)的紧密结合能够显著提高后者在高温下的稳定性[4];许多DNA/RNA 结合蛋白与DNA/RNA 的多价相互作用(multivalent interactions)也能够帮助提高蛋白质的稳定性,如TDP-43、FUS、TAF15 等[5-7];甚至是细胞中常见离子浓度的变化就能够在一定程度上影响蛋白质的折叠[8-10]。人们不仅通过突变得到热力学更稳定的突变体,还运用计算发现多数天然蛋白质的折叠自由能(∆Gf)仅在-10 kcal/mol左右,即轻微的扰动就很有可能影响蛋白质的正确折叠,揭示出天然蛋白质具有边缘稳定性(marginal stability),为进一步探究蛋白质构效关系提供更深层次的理解[1,11-12]
1.2 蛋白质组的边缘稳定性
除了单一蛋白质以外,蛋白质组也被证实具有边缘稳定性。Ghosh 等人在2010 年通过建立量化模型对多种生物体的蛋白质组进行分析,提出蛋白质组的稳定性并不取决于组内蛋白质折叠自由能的平均值,而取决于其折叠自由能分布函数中靠近尾端的蛋白质,即蛋白质组中折叠稳定性相对较差的蛋白质[13]。例如,在大肠杆菌(Escherichia coli)里的4300 多种蛋白质中,约有15%的蛋白质折叠自由能(∆Gf)高于-4 kcal/mol。对于这部分蛋白质,少许突变或轻微的环境扰动就可能导致蛋白质不能够正常折叠,进而影响其功能。为了验证这一点,他们将环境温度从37°C升高到41°C,发现尽管4°C 的温度变化对蛋白质组的平均折叠自由能影响不大(∆Gf从-7 kcal/mol提高到-5.6 kcal/mol),却会直接导致分布函数中靠近尾端的蛋白质变得不稳定。这些不稳定蛋白质的错误折叠不仅会影响蛋白质组中其他蛋白质的折叠和运作,还需要占用大量分子伴侣和泛素-蛋白酶体系统(ubiquitin-proteasome system)来修复或降解。当细胞中的质量控制系统不足以抵挡错误折叠蛋白质带来的负面影响,蛋白质组整体稳定性被破坏,即打破了蛋白质稳态。总体来说,这些实验结果不仅强调了蛋白质个体的边缘稳定性,也阐述了少量蛋白质的不稳定折叠会直接破坏整体蛋白质组的稳态,即蛋白质组也存在边缘稳定性。蛋白质稳态是细胞开展正常生理活动的保障,而蛋白质稳态的破坏会引发细胞、组织甚至器官的生理功能异常,并会直接导致含阿尔兹海默病(Alzheimer’s disease,AD)、亨廷顿病(Huntington’s disease,HD)以及帕金森病(Parkinson’s disease,PD)在内的多种神经退行性疾病(neurodegenerative diseases)。
2 分子伴侣在蛋白质折叠中的重要作用
2.1 分子伴侣家族简介
由于天然蛋白质具有边缘稳定性,蛋白质自身的突变或周围环境的细微变化都可能引发蛋白质的错误折叠。蛋白质的错误折叠不仅会使得蛋白质丧失正常功能;错误折叠蛋白质的积累还会破坏细胞内蛋白质稳态网络并导致疾病[14-17]。因此,蛋白质折叠过程的质量控制对蛋白质组的平衡甚至是细胞和有机体的健康至关重要。正因如此,生物进化出了一系列质量控制网络,从介导蛋白质正确折叠的分子伴侣,到降解错误折叠或聚集蛋白质的泛素-蛋白酶体系统,共同维持细胞内环境蛋白质稳态[18-20]。分子伴侣通常可以被定义为帮助其他生物大分子(如蛋白质)折叠或组装成相应功能活性构象,但不参与最终结构组成的任何蛋白质[21]。最具代表性的分子伴侣是热休克蛋白。热休克蛋白在细胞受环境温度、压力或化学物质的刺激时被上调,从参与新生肽的折叠、错误折叠蛋白质的展开以及重新折叠,到参与寡聚物的组装、异常蛋白质的降解和功能蛋白质的运输,在维持细胞内环境稳定的同时协助细胞完成特定生理功能[22-25]。
近几十年来,分子生物学家主要在原核生物中探究分子伴侣的作用机制,这是因为相比于真核生物,原核生物体系更为简单。真核生物蛋白质通常较大,并具有复杂结构域,蛋白质构效关系也更为多元化。不仅如此,为了有效配合细胞正常执行功能,真核生物中分子伴侣分布更为广泛,例如细胞质、细胞核以及线粒体[26]。原核生物体系,尤其是以大肠杆菌为代表的细菌常被作为宿主大量表达用于生物研究的重组蛋白,在细胞生物学中被用于探究生化反应机理,或在生物工程中被用于探究蛋白质的定向进化。分子伴侣被证实能够有效提升外源蛋白质在大肠杆菌内的折叠效率,在提升活性外源蛋白质的产率上起到重要作用[27]。了解原核生物体系分子伴侣的作用机理,对于理解更为复杂的真核生物体系,如哺乳动物细胞中分子伴侣的构效关系是十分必要的。因此,本章节主要以大肠杆菌为体系阐述部分重要的分子伴侣的结构及其作用机制。
首先,根据分子伴侣分子量的大小可以把它们进行简单分类,如Hsp90家族(约90 kDa)、Hsp70家族(约70 kDa)、Hsp60家族(约60 kDa)以及一些小分子热休克蛋白(sHsp,12~43 kDa)[23]。不同的分子伴侣侧重的功能也不尽相同。以大肠杆菌分子伴侣为例,主要可以把它们分为:保持酶(holdase)、折叠酶(foldases)和解聚酶(disaggregases)。保持酶主要负责稳定新生肽链或部分折叠的蛋白质,其中包括触发因子(trigger factor, TF)和IbpA/IbpB(大肠杆菌内小分子热休克蛋白同源物)[28-29]。折叠酶主要负责在ATP 的驱动下帮助新生肽链折叠,展开错误折叠的蛋白质并将其进行重新折叠,其中包括DnaK(大肠杆菌内Hsp70 同源物)、GroEL(大肠杆菌内Hsp60 同源物)和HptG(大肠杆菌内Hsp90 同源物)[28,30]。其余不可逆转的蛋白质沉淀则被解聚酶ClpB(大肠杆菌内Hsp100 同源物)溶解(图1)[31]。
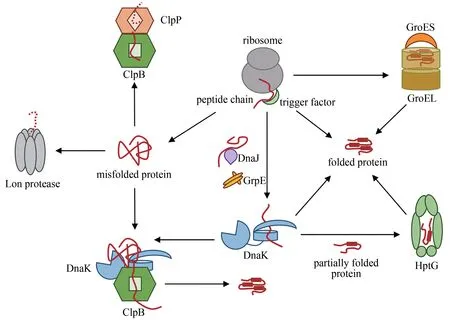
图1 大肠杆菌中由分子伴侣和蛋白酶调节的蛋白质稳态网络Fig.1 Proteostasis network modulated by molecular chaperones and proteases in E.coli
2.2 保持酶
TF 是大肠杆菌内作用于新生肽链的首要分子伴侣,主要负责稳定新生肽链,防止其错误折叠[32-33]。它不依赖于ATP,而是通过与核糖体相互作用形成动态反应循环[24]。首先,与核糖体的结合将TF 保持在开放的、活化的构象中。当新生肽链从核糖体出口处离开核糖体时,TF 会随即与该段肽链结合,并迅速脱离核糖体(约0.10 秒)。相较于其他分子伴侣,TF 不具有特定底物的结合位点,而是将整个蛋白质内腔用于与底物的结合。TF 内腔表面混合分布着疏水性和亲水性氨基酸残基,并同时通过疏水作用和亲水作用最大效率帮助TF 与新生肽链结合。随着肽链的不断延长,多个TF 会相继进入核糖体对接位点,持续保护新生成的肽链。在核糖体完成翻译后,新生肽链可以直接被转移到下游的分子伴侣如DnaK 和GroEL,并在它们的帮助下完成折叠[28,34-35]。TF 在细胞中处于单体和二聚体之间的动态转化,前者主要存在于同核糖体结合时的功能状态,而后者主要负责协助细胞质中蛋白质的折叠以及其他细胞活动[36]。
IbpA 和IbpB 是大肠杆菌里两种小分子热休克蛋白,与大肠杆菌中包涵体(inclusion body)的形成有关[37]。IbpA/IbpB 主要作为保持酶,与错误折叠的蛋白质结合,并将其传递给DnaK/DnaJ、ClpB 分子伴侣系统进行重新折叠[38]。这两种分子伴侣有48%的序列相似性,并且在大肠杆菌内以异源二聚体的形式执行其功能[39]。然而,IbpA 和IbpB 在功能上存在显著差异:前者更倾向于与错误折叠的蛋白质共沉积,甚至形成纤维状聚集体;而IbpB 能够激活Lon 蛋白酶对IbpA 的降解[40]。尽管目前对IbpA/IbpB 的作用机理缺乏更为详细的阐述,但是可以确定的是IbpA 与IbpB 间相互作用对错误折叠蛋白质的解聚有重要影响。
2.3 折叠酶
Hsp70/Hsp40、Hsp60/Hsp10 以 及Hsp90 是 帮助蛋白质折叠的代表性分子伴侣。这些分子伴侣通过与新生多肽或是错误折叠蛋白质暴露出来的疏水性氨基酸结合来阻止其进一步的聚集,并在ATP和其他辅助因子的共同作用下进行功能构型的动态循环,实现对底物的结合与释放,从而广泛参与了蛋白质从头折叠以及重新折叠[22,41]。虽然大肠杆菌内大多数新合成的蛋白质能够自行折叠成其特定三维结构,但是仍有约20%~30%的蛋白质被证明需要分子伴侣帮助其折叠成相应的功能构型[22-23,42]。
Hsp70/Hsp40 分子伴侣系统在帮助蛋白质折叠以及维护蛋白质稳态方面起到重要作用[23]。Hsp70主要在共分子伴侣Hsp40 以及核苷酸交换因子(nucleotide exchange factor, NEF)的辅助下完成对底物蛋白质的折叠[43]。Hsp40 首先和底物结合,并将底物带到Hsp70附近[44]。Hsp70从真核生物到原核生物都高度同源,因此,在许多研究中均以大肠杆菌内Hsp70 的同源物——DnaK 为模型来探究其结构和功能。Hsp40在大肠杆菌里的同源物为DnaJ,与Hsp70 不同的是,Hsp40 进化出种类更多的同源物。Hsp40 的多元化决定了不同Hsp40 家族成员具有不同底物结合位点,从而允许对底物的特异性选择[45]。DnaK 是一个二聚体,通过其氨基末端ATP 酶结构域(ATPase domain)与羧基末端肽链结合结构域(peptide-binding domain)的变构偶联实现对底物的结合和释放[46-47]。其中肽链结合结构域主要由控制底物进出的α-螺旋盖(α-helical lid)和β-夹心(β-sandwich)组成,后者负责与DnaJ 带来的舒展且富含疏水性氨基酸的残基片段结合(图2)[48-50]。当DnaK 的氨基末端与ATP 结合时,α-螺旋盖开放,此时DnaK 对肽链的亲和力较低[51]。DnaJ 和DnaK 之间的相互作用加速了ATP的水解,并促使DnaK 构型发生变化,即α-螺旋盖闭合形成密闭结构,同时羧基末端与底物的相互作用增强(图2)[44,52-55]。当DnaJ 从DnaK 解离后,核苷酸交换因子GrpE 的结合促进了ADP 的解离。而在下一个ATP 与DnaK 结合并开始新一轮循环时,α-螺旋盖再次开放,同时释放出底物[42,56]。其中一部分底物已经完成折叠或仍处于未折叠状态,还有一部分底物可能需要被进一步输送到下游分子伴侣如Hsp90,完成后期加工才能被正常使用[57]。DnaK/DnaJ 在与底物结合的过程中提供了保护机制,防止蛋白质自身或在外界物质的影响下错误折叠,Hsp70的过表达也被证明能够有效阻止蛋白质的聚集。不仅如此,一些DnaJ 还能够与已经折叠的蛋白质结合,而在这种情况下,DnaK发挥展开作用[54];Hsp70 还可以与Hsp100 ATP 酶合作,分解不可逆大聚集体[58];Hsp70 的构象循环还可以作为传输工具,将蛋白质递送给转运酶,随后转运酶帮助蛋白质穿过细胞器膜[59-60]。总的来说,Hsp70的作用不仅仅局限于帮助底物折叠,还能够引导底物的展开与解聚,以及充当运输工具。
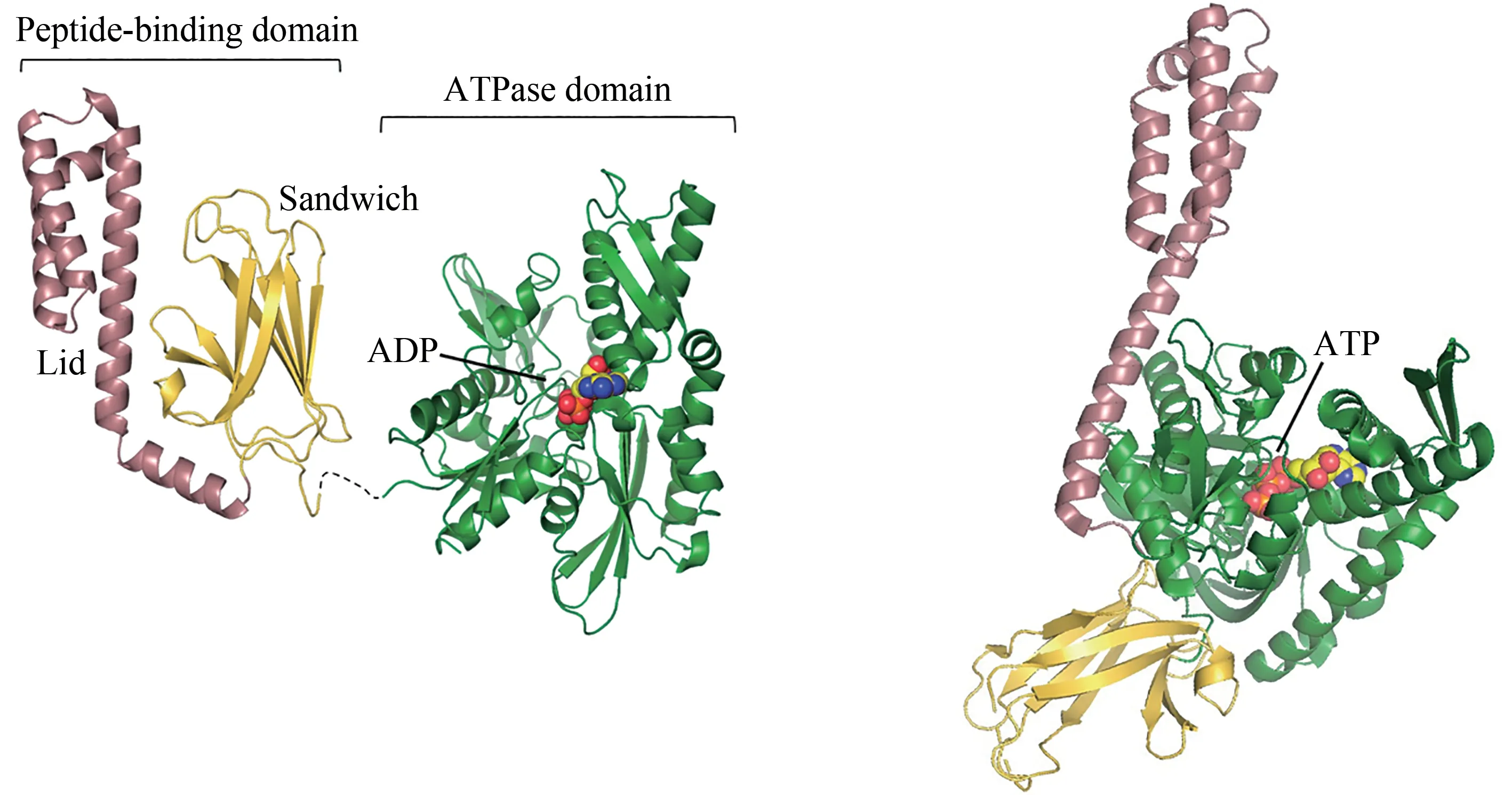
图2 ADP结合(左)和ATP结合(右,PDB代码:4B9Q)[55]的Hsp70构象[在ADP结合状态下,肽链结合结构域(PDB代码:1DKZ)[50]和ATP酶结构域(PDB代码:3HSC)[51]通过灵活的氨基酸链连接]Fig.2 ADP-bound(left)and ATP-bound(right,PDB code:4B9Q)[55]conformations of Hsp70[In the ADP-bound state,the peptide-binding domain(PDB code:1DKZ)[50]and ATPase domain(PDB code:3HSC)[51]are connected by a flexible linker]
作为Hsp60/Hsp10 的同源物,GroEL/GroES 是大肠杆菌中被研究得最多的分子伴侣[61]。GroEL由14 个相同亚基组成,它们以对称的方式排列成两个背对背的七元环,从而堆叠成一个高约15 nm的圆柱体折叠笼(图3)[62-65]。折叠笼内部是一个直径约为5 nm 的开口空腔,内壁形成光滑的疏水表面,以此选择性地捕获因未折叠或错误折叠而暴露出的疏水性肽链。作为GroEL 的共分子伴侣,GroES 是一个由7 个相同的β 桶组成的七元环,通过与GroEL 的顶端结构域相互作用形成折叠笼的盖子[66-67]。当与ATP 结合后,GroEL 迅 速招 募GroES覆盖折叠笼的空腔,同时进行结构重组,将疏水性内腔转化为亲水性、带负电的封闭室[68-69]。受到封闭的环境限制,蛋白质一旦进入亲水腔内部,或被迫保持展开状态,或遵循其氨基酸序列决定的三维结构进行折叠。当ATP 水解完成后,折叠笼被重新打开,并释放出蛋白质。GroEL的两个七元环可以不间断交替进行ATP 结合和ATP 水解的循环,所以当其中一个环释放出的蛋白质仍处于展开状态时可以立即被另外一个环捕获,并再次进行折叠尝试(图3)[70]。Hsp60 在细胞中不只是作为折叠机器,越来越多的证据表明Hsp60还能够协助很多细胞活动,例如物质运输、信号传导和细胞增殖,甚至还在免疫系统中发挥重要作用[71]。
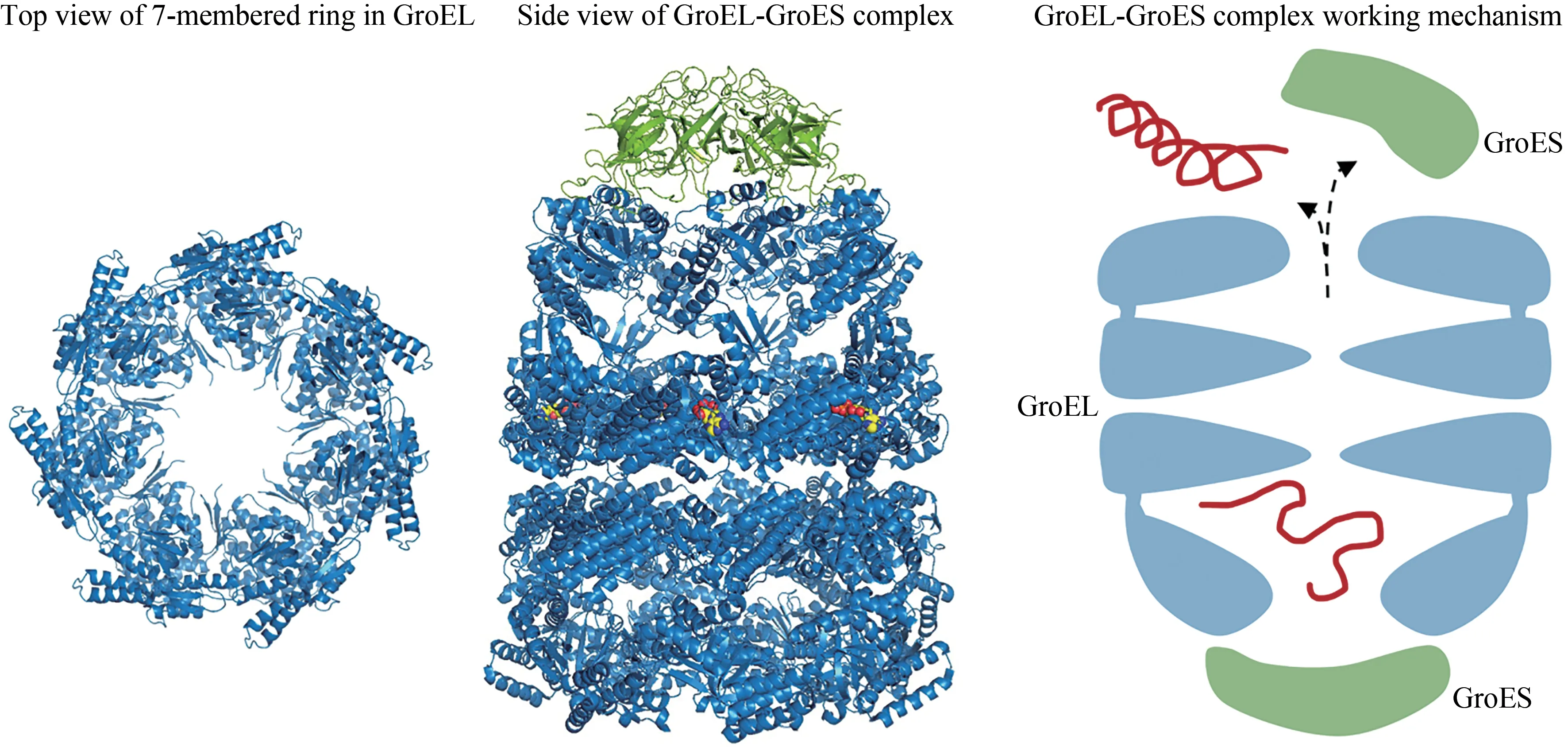
图3 Hsp60七元环(左,PDB代码:1IOK)[65]和Hsp60/Hsp10复合物(中,PDB代码:1AON)[67]的结构,以及Hsp60/Hsp10作用机理(右)Fig.3 Structures of the 7-membered ring in GroEL(left,PDB code:1IOK)[65]and the Hsp60/Hsp10(middle,PDB code:1AON)[67],as well as the working mechanism of Hsp60/Hsp10(right)
Hsp90 是一种高度保守且依赖ATP 的分子伴侣。与其他热休克蛋白一样,Hsp90能够帮助蛋白质折叠并预防蛋白质聚集[72-73]。与其他分子伴侣不同的是,Hsp90功能更加专一,主要负责细胞信号传导和发育相关蛋白质的后期折叠,如类固醇激素受体、激酶和肿瘤抑制因子p53 等[74]。对大肠杆菌内Hsp90 的同源物HptG 的研究表明,这类分子伴侣呈现出更为动态的构象循环,以类似剪刀的展开闭合形式快速切换。HptG 二聚体通过羧基端连接形成一个打开的“V”字形,并在内部形成疏水结构(图4)[75-76]。ATP 与HptG 两氨基端结合引起二聚体明显的构象变化,进而氨基端相连接,形成扭曲的“分子钳”(图4);ATP 的水解和ADP从氨基端的解离使得HptG 回到初始的“V”字状态[77-79]。在这个循环中还有多个辅助因子的调节,如AHA1 刺激ATP 的水解,而CDC37 负责抑制ATP 酶的活性[80-81]。这些辅助因子共同帮助HptG进行构象转变,从而实现对靶蛋白的捕获与释放。除了单独作用外,Hsp90还能和其他分子伴侣,例如Hsp70 相互作用,共同优化关键调控蛋白的折叠[22]。在这个过程中,Hsp70 负责底物初期的折叠,通过共分子伴侣将释放出的底物直接传递给下游的Hsp90完成最终的折叠[57]。
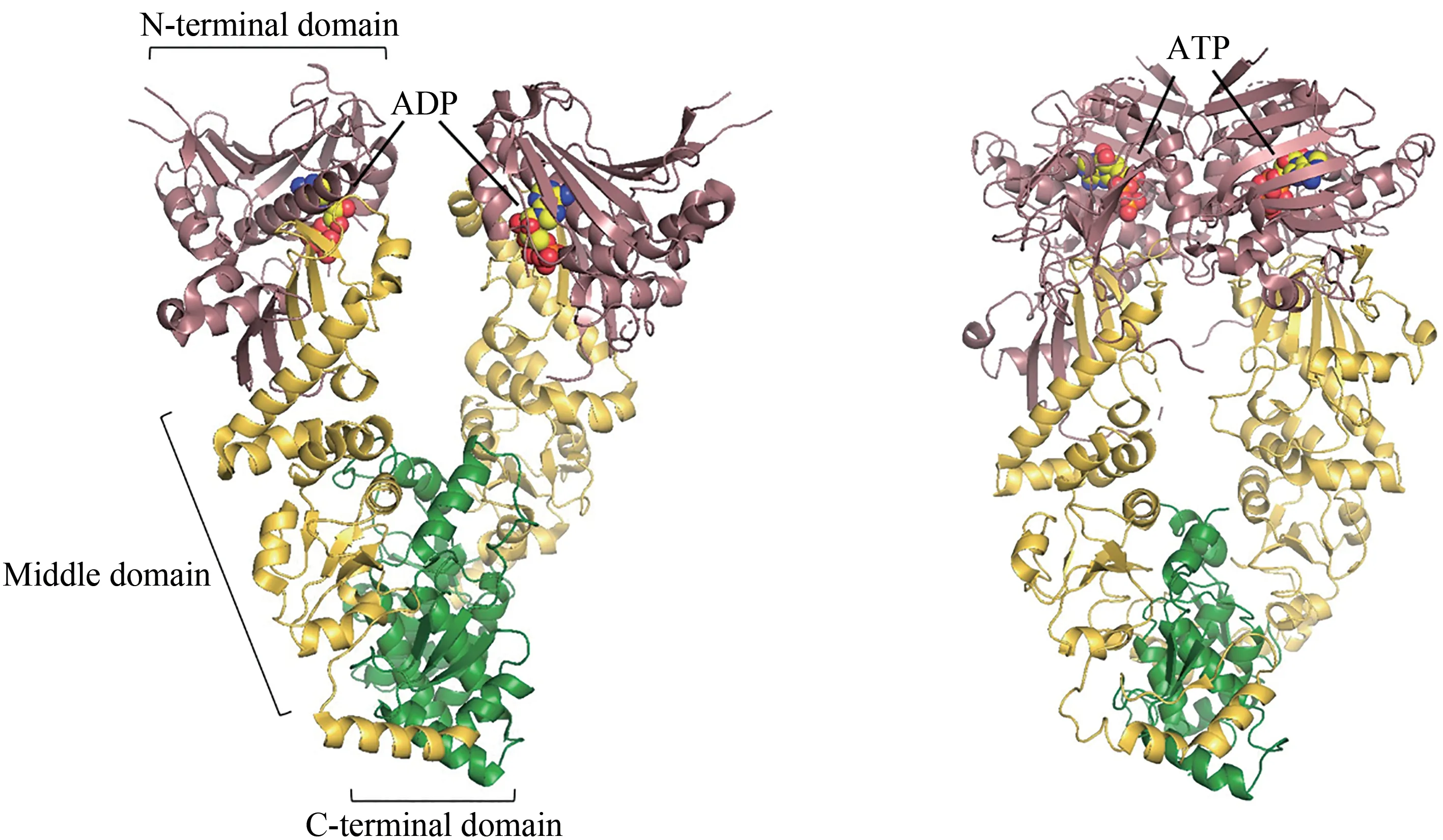
图4 ADP结合(左,PDB代码:2O1V)[79]和ATP结合(右,PDB代码:2CG9)[76]的Hsp90构象Fig.4 ADP-bound(left,PDB code:2O1V)[79]and ATP-bound(right,PDB code:2CG9)[76]conformations of Hsp90
除了大肠杆菌,上述所有分子伴侣在其他原核生物以及真核生物中都有许多对应的同源物,执行着相似或不同的功能。例如,人类Hsp70同源物HspA 主要负责协调细胞应激反应。其中,HspA1A 被发现可以促进肝癌细胞增殖;HspA1B也在细胞增殖以及肿瘤生长上起到重要作用[82-83]。Hsp40在人类细胞中也存在相应同源物,主要可以分为DnaJA、DnaJB与DnaJC[84]。其中DnaJA与大肠杆菌中DnaJ 最为相似,而有些DnaJA 和DnaJB分子伴侣能够独立展开工作[85]。Hsp60 在人类线粒体中的同源物HspD 主要负责线粒体中蛋白质的折叠,但是与细菌中GroEL 不同的是,HspD 活性结构可以在单环和双环间交替,并且两个环可以同时和HspE(Hsp10在人类线粒体中的同源物)结合,以此提升折叠效率[86]。在细胞质中,Hsp60的同源物为CCT,由8个不同的亚基组成,主要负责细胞质中蛋白质的折叠[87]。Hsp90在人类细胞质中的同源物为Hsp90α 和Hsp90β,同时,Hsp90 还存在于线粒体(TRAP-1)以及内质网(GRP94),它们在细胞中的广泛分布强调了Hsp90在细胞稳态中的重要作用[88]。
2.4 解聚酶
错误折叠蛋白质的聚集被视为细胞衰老以及疾病的标志。为了维持正常的细胞环境,无法被重新折叠或者重新折叠失败的蛋白质会被进一步水解。蛋白水解系统识别错误折叠的蛋白质,并使用AAA+(ATPases associated with diverse cellular activities)相关的蛋白酶如ClpP,以及ATP 酶如ClpA/X,直接参与维持蛋白质稳态。ClpP 是一个高度保守的丝氨酸蛋白酶,由两个堆叠的七元环形成空腔,并允许蛋白质在空腔内部发生降解。每个ClpP 亚基中N 端环可以控制轴向开口;核心结构域有负责水解底物的丝氨酸-组氨酸-天冬氨酸催化三联体;相邻亚基在ClpP顶端形成疏水裂缝,作为ATP 酶的结合位点。ClpA/X 是Hsp100 分子伴侣家族在大肠杆菌中的同源物,分别通过与ClpP形成复合物控制ClpP 的水解功能。这些ATP 酶由6 个相同的亚基形成六元环。每个亚基都有不止一个AAA+结构域,该结构域负责ATP 的结合和水解。通过ATP 的结合和水解,ClpA/X 构型发生变化,并运用积累的能量展开底物,随后将底物推入ClpP 空腔内部进行进一步水解。除了大肠杆菌中的ClpA/ClpX,维管植物中也存在多种Hsp100同源物,如拟南芥的叶绿体中就有ClpC1、ClpC2、ClpD 和ClpB3。Hsp100 不仅能够通过与蛋白酶相互作用促进底物的水解,还能够与其他分子伴侣形成双分子伴侣体系。例如,酵母中发现的Hsp104 以及大肠杆菌中的ClpB 均为Hsp100 的同源物,它们均能够与相应体系Hsp70的同源物协同作用,展开异常蛋白质,并帮助其重新折叠。研究表明,该双分子伴侣体系对底物有一定的选择性,即ClpB 与DnaK 的亲和力根据DnaK 结合的底物种类有很大差异。例如,当DnaK 与大分子量蛋白质聚集体结合时,ClpB对DnaK表现出更高的亲和力。不仅如此,ClpB-DnaK 的激活需要两个分子伴侣的耦合活性,才能够有效将底物从DnaK 转移至ClpB。
除了ClpP,Lon蛋白酶也能帮助降解并清除受损或错误折叠的蛋白质,从而调节蛋白质稳态。Lon 蛋白酶是一种高度保守的蛋白酶,存在于细菌、真核生物线粒体以及其他细胞器中。同许多分子伴侣一样,Lon 蛋白酶也是一种压力应激蛋白,同时也属于AAA+蛋白家族。Lon 蛋白酶是第一个被发现的ATP 依赖性蛋白酶,由相同亚基组成同源寡聚体。与ClpP 不同的是,Lon 蛋白酶每个亚基同时携带ATP 酶和蛋白酶结构域。其N 端结构域负责与底物结合,中间ATP 酶结构域负责调节ATP的结合和水解,C端的丝氨酸和赖氨酸形成催化二联体,在底物水解中起重要作用。大肠杆菌中,Lon蛋白酶还能降解许多具有调节功能的蛋白质,而这些蛋白质并非处于错误折叠状态。除此之外,Lon蛋白酶同样也在正常生理功能中起着重要调节作用。例如,在哺乳动物中,Lon蛋白酶展现出类似于分子伴侣的特征,能够在细胞内质网应激和缺氧期间被上调,为细胞提供保护功能。
3 提高蛋白质(组)稳定性手段的研究
3.1 过量表达分子伴侣
既然分子伴侣能够帮助蛋白质正确折叠,那么针对容易聚集的蛋白质,是否可以通过过量表达分子伴侣来降低蛋白质聚集的风险?早在1996 年Thomas 和Baneyx 就已经在大肠杆菌内验证了这种方法的可行性[27]。他们选取的蛋白模型是一个三组分融合蛋白(preS2-S'-β-galactosidase),该蛋白质两结构域之间被插入一段含有30 个氨基酸的短疏水序列,使其高度易聚集[89]。在明确了过度表达分子伴侣对融合蛋白的合成速率没有显著影响后,他们直接过量表达质粒编码的DnaK/DnaJ 来探究分子伴侣的过度表达对融合蛋白酶活性的影响。研究发现DnaK/DnaJ 的过度表达不仅能够显著增强该蛋白质在高温下(42°C)的酶活性,还能够在低温(30°C)和正常温度(37°C)下进一步提高活性酶的回收率。
关于过度表达分子伴侣对酶活性的提升,主要可以从两种机理解释:一是分子伴侣能够通过与蛋白质相互作用促进其正确折叠。在没有过量表达分子伴侣时,小部分的蛋白质消耗掉了环境中可用的分子伴侣,剩下大部分没有结合分子伴侣的蛋白质更易错误折叠从而聚集;随着分子伴侣浓度增加,更多的蛋白质能够在其帮助下正确折叠,从而降低错误折叠蛋白质的比例。二是分子伴侣能够解聚已经聚集的蛋白质。即通过解聚已经存在于包涵体内的蛋白质聚集体,从而帮助恢复蛋白质正确折叠。进一步的免疫共沉淀实验结果指向前一种机理,即该融合蛋白的短疏水序列具有与DnaK 特异性结合的条件,并能够与DnaK 形成稳定复合物。因此,DnaK 通过直接与新生多肽相互作用,促进新合成蛋白质的正确折叠以及组装,从而产生更多具有活性的融合蛋白。
3.2 过量表达热休克转录因子
尽管已经有很多研究证明过量表达分子伴侣能够帮助更多目标蛋白质正确折叠,但这并非调节蛋白质组稳定性的最佳解决办法。首先,分子伴侣对底物具有一定的特异性,蛋白质组的折叠需要多种分子伴侣协同作用。这往往需要大量的实验来确定能够作用于特定蛋白质的分子伴侣组合;并且分子伴侣与目标蛋白同时过表达会因细胞内代谢环境限制而相互影响,例如二者对于有限的RNA 聚合酶的竞争,或是目标蛋白转录产物的大量积累,可能会抑制分子伴侣的表达。因此,该方法需要筛选出最佳条件来优化分子伴侣效应。其次,除了前面提到的直接过量表达质粒编码的分子伴侣,还可以通过增加细胞内环境压力,如升温或化学诱导来间接实现分子伴侣的过量表达。但这种方法也很容易导致细胞内蛋白质稳态被破坏,从而造成细胞损伤甚至是凋亡。因此,人们想到了在维持细胞内环境稳定的同时,通过过量表达负反馈缺陷型热休克转录因子σ32来提高大肠杆菌内重组蛋白的产量[90]。σ32能够在细胞承受压力时与核心RNA 聚合酶结合,从而诱导许多分子伴侣以及辅助因子以自然进化出特定的化学计量进行表达[91-92]。相比于单一的某个或多个分子伴侣的过表达,这种整体水平提升的方式不需要特别明确途径中哪些代表性分子伴侣对目标蛋白质有显著的作用,使得同时增加多种正确折叠的蛋白质成为可能。尽管已经有工作尝试过表达σ32来提升重组蛋白的产量,但是σ32会受到分子伴侣介导的负反馈途径的影响,如细胞内GroEL/GroES 水平的升高会降低σ32的活性,所以野生型σ32的作用只是短暂的。然而,Yura 等发现仅仅单位点的突变就可以在几乎不改变σ32与RNA 聚合酶结合能力的同时突破负反馈调节的影响,从而实现σ32突变体的长效作用[93]。Zhang 等人将其中一个σ32突变体I54N 运用于优化大肠杆菌重组目标蛋白的表达,即通过整体重塑或加强蛋白质稳态网络来最大限度地提高重组目标蛋白的数量和质量[90]。σ32-I54N 的过表达使得细胞内形成类似热休克响应情况下的蛋白质网络,并且除了热休克响应相关的基因,绝大多数大肠杆菌蛋白质组都没有受到影响。两种重组蛋白——逆醛缩酶(retro-aldolase,RA)和内切木聚糖酶(endoxylanase,XynA),作为模型蛋白被用来研究σ32-I54N 表达的情况下增强的蛋白质稳态网络对重组蛋白的影响。在正常情况下,这两种模型蛋白主要在大肠杆菌内形成包涵体;然而在增强的蛋白质稳态网络中,两种模型蛋白的溶解度分别提高了2 倍和3 倍。不仅如此,具有正常功能的模型蛋白数量也在增强的蛋白质稳态网络中得到了不同程度的提升。这些结果表明σ32-I54N 表达不仅可以提升正确折叠蛋白质的产量,还能够增加具有正常功能的蛋白质数量。后面针对转甲状腺素蛋白(transthyretin,TTR)A25T 突变体的实验也进一步证实σ32-I54N 在重组蛋白质过表达时的积极作用。这种方法有望被广泛运用到不同体系实现不同重组蛋白过表达的优化,尤其是具有复杂结构的大分子量蛋白质,为体外探究特定生化反应机理提供技术支持。
3.3 化学手段重塑胞内蛋白质折叠环境
除了过量表达,对分子伴侣的抑制有时也可以作为针对蛋白质稳态被破坏的潜在治疗手段,如抑制Hsp90。Hsp90 是一种在细胞中极为丰富的分子伴侣,在正常情况下约占细胞总蛋白质数量的1%~2%,但在细胞应激时被过度表达,总占比提升至4%~6%。不仅如此,人们发现Hsp90 在帮助实现或维持关键致癌蛋白的活性构象方面起到重要作用,这些蛋白质在细胞中功能十分广泛,从简单的信号传导、蛋白质运输,到染色质重塑和细胞增殖[94-95]。目前,处于临床评估的绝大多数Hsp90 抑制剂通过阻断ATP 与Hsp90 N 端结构域的结合起作用,从而诱导许多Hsp90依赖性致癌蛋白的降解[96-98]。与之类似,研究发现对HSF1(heat shock factor 1)的调控也能在一定程度上影响内环境的稳态。除了正常的细胞生存和增殖进化,HSF1 还可以帮助细胞适应蛋白质稳态失衡导致的病理生理紊乱,从而引发恶性疾病[99]。Dai 等通过基因敲除证明HSF1 不仅促进小鼠体内肿瘤的初步形成,还可能帮助具有潜在恶性缺陷的人类癌细胞的生长和存活[99]。因此,针对HSF1 相关转录网络的抑制可以作为广泛且有效治疗癌症的策略。截至目前,已经有很多相关的抑制剂被接连报道,例如化合物KRIBB11 被发现可以通过与HSF1 直接作用或通过影响HSF1 下游靶蛋白的表达来抑制肿瘤的生长[100];还有一种RNA 适体(RNA aptamer)被证实能够阻止HSF1 与其靶基因结合,从而诱导细胞凋亡并避免了癌细胞的聚集[101]。Vilaboa 等报道了一例能够直接靶向HSF1的小分子化合物IHSF115,该化合物能够与分离的HSF1 DNA 结 合 域片段 结 合[102]。IHSF115 不 影响热休克条件下HSF1 的寡聚化、细胞核定位以及与特定DNA 的结合。然而,免疫沉淀物的蛋白质印迹分析显示IHSF115 能够干扰HSF1 与激活转录因子1(activating transcription factor 1, ATF1)之间的相互作用,从而影响HSF1 靶基因的热诱导转录。实验发现,绝大多数情况下IHSF115 的存在抑制了受HSF1 控制的相关基因的表达,其中主要包括控制蛋白质折叠的基因,如包涵体组装的负调控、蛋白质的重新折叠;还有一部分包括控制生物正常功能的基因,如对激素刺激的反应、骨骼肌分化、细胞生长的负调节等。不仅如此,实验还发现IHSF115 能够有效激活热休克响应时HSF1 抑制的特定基因,如核小体的组装、以DNA 为模板的转录调控等。总体而言,IHSF115 能够通过与HSF1 的相互作用影响其调控的靶基因,从而帮助重塑细胞内蛋白质折叠环境。
4 分子伴侣与定向进化
4.1 蛋白质稳定性是影响蛋白质进化的重要因素
生物体内绝大多数生理活动都离不开蛋白质,例如:免疫蛋白参与机体防御机制[103-104],多肽类激素参与调节细胞代谢[105-106],以及酶参与底物的特异性催化[107-108]。蛋白质稳定性决定生物体内生化反应是否能够正常、持续进行,然而生物多样性的形成离不开蛋白质的进化。蛋白质进化往往与基因突变和环境选择有关。其中,基因突变与蛋白质稳定性也有着密不可分的联系,因此,人们猜测蛋白质的稳定性影响蛋白质的进化。围绕这一猜想的关键问题是蛋白质的稳定性和可进化性如何相关,蛋白质稳定性的增强究竟是提升了可进化性还是降低了可进化性。
2005 年Drummond 等在使用酿酒酵母(Saccharomyces cerevisiae)探究高表达蛋白质进化缓慢的机理时,提出蛋白质稳定性的增强会延缓蛋白质进化的假设[109]。他们认为高表达蛋白质产生错误翻译的可能性提高,导致其更容易发生错误折叠。而这些错误折叠的蛋白质会在细胞内聚集,丧失其功能并产生细胞毒性。为降低下游错误折叠蛋白质产生的负面作用,细胞产生了相应机制,使得蛋白质即使翻译错误也能维持正常折叠,也就是提升对翻译错误的容忍性,被称为“翻译稳健性”(translational robustness)。适度增加蛋白质热力学稳定性能够增加对蛋白质错误折叠的包容度。因此,在表达水平增加时,细胞内压力使得蛋白质稳定性增强,从而增强对蛋白质突变或翻译错误引起错误折叠的包容度,并减缓蛋白质的进化。Bloom 等在2004 年利用一个简单模型蛋白质也得到类似的结论[110]。该模型蛋白质仅有18 个氨基酸,可以自行折叠成二维晶体结构,并能与特定配体结合。在五个不同温度参数下对模型蛋白质进行500代进化,结果发现在更高温度参数下,即对蛋白质的稳定性强选择的情况下,蛋白质突变体与原配体结合能力降低。因此,他们认为具有低稳定性的蛋白质才能更有效地进化配体结合功能。
然而就在两年后,Bloom 等修正了之前的理论,并认为具有较高稳定性的蛋白质才能增加有益变异的可能性[111]。同之前一样,他们利用类似的模型蛋白质来探究蛋白质稳定性和可进化性的关系。但与之前不同的是,本次实验引入了新的筛选机制,即增加了4种新配体。首先,他们发现折叠更稳定的蛋白质能容纳更多的突变体。与天然蛋白质相似,原始亲本蛋白仅具有边缘稳定性(∆Gf= -0.5 kcal/mol)。通过两轮随机诱变筛选出的突变体——稳定亲本蛋白对同种配体有相同的结合力度,却更加稳定(∆Gf= -1.5 kcal/mol)[图5(a)]。其中,两种模型蛋白质折叠自由能的差值可被视为稳定亲本蛋白具有的额外稳定性。同原始亲本蛋白相比,稳定亲本蛋白能够产生更多数量的正常折叠的突变体,这证实了更稳定的蛋白质对突变的包容度更大。同时他们还发现,提高蛋白质的稳定性可以增加有益变异的可能性。例如,尽管绝大多数稳定亲本蛋白的改良突变体稳定性都有所下降,但多个改良突变体与4种新配体的结合力度增强[图5(a)、(b)],初步证实了稳定性更好的蛋白质可进化性也更高。为了进一步验证这一结论,他们对细胞色素P450 BM3 血红素结构域(heme domain)的两种突变体21B3 和5H6进行随机突变,并筛选出它们在抗炎药萘普生(naproxen)、3-苯氧基甲苯、苯氧乙酸、β-肾上腺素受体阻滞剂普萘洛尔(propranolol)和2-甲基苯并呋喃5种底物上功能改进的突变体。21B3和5H6这两种突变体均可催化12-对硝基苯氧基羧酸的羟基化反应,且对5 种底物的催化活性也基本一致。但相比于21B3,5H6 热稳定性更强,稳定性的增强提升了5H6 产生的功能改进的突变体数量(从4个到13个),进一步证实了稳定的蛋白质具有更大的进化潜力[图5(c)、(d)]。
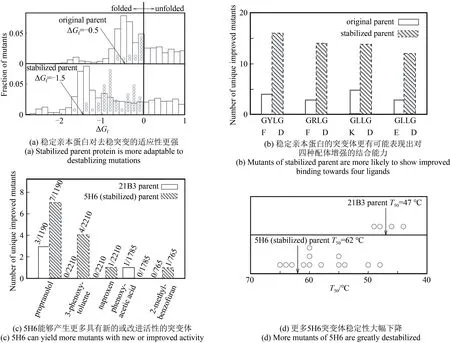
图5 稳定性的增加增强了模型晶格蛋白和P450 BM3血红素结构域的可进化性[111]Fig.5 Increased stability enhances evolvability of model lattice protein and P450 BM3 heme domain[111]
Bloom 等前后两次实验得到完全相反的结论,主要原因在于实验方法的不同。首先,前一次实验主要通过强选择得到稳定性相同的蛋白质突变体;后一次实验主要通过对两种具有不同稳定性的模型蛋白质进行随机突变。更为重要的是,前一次的实验仅关注于突变体与原配体的结合力度,限制了研究对象;而后一次实验注重于突变体与5 种新配体的结合力度,更能够体现出突变体改进的功能,即进化。因此,从实验方案的合理性上看,后者得到的结论更为可靠,即更稳定的蛋白质对突变更稳健,从而能够积累一定程度的突变,增加进化的可能性。
Zheng 等用黄色荧光蛋白(yellow fluorescent protein,YFP)的例子同样证明了增强蛋白质的稳定性有利于蛋白质的进化[112]。在实验的第一阶段,他们首先对具有黄色荧光表型的祖先种群进行不同强度的选择,即强选择(种群S)、弱选择(种群W)或不选择(种群N)。结果显示强选择进化出的种群S相比于其他两种选择显示出更好的热力学稳定性。为了了解强进化种群对突变的包容程度,他们接着对前面不同选择强度下进化出的种群进行随机诱变。对比实验结果发现种群S产生了更多保持黄色荧光的突变体,说明稳定性的提高增强了原始黄色荧光表型的突变稳健性。在实验的第二阶段,他们在相同的选择压力下将种群向具有绿色荧光的新表型进行定向进化。结果显示种群S最快产生适应性进化,并且进一步的研究发现种群S氨基酸变化数量和遗传多样性方面的增加均大于种群W 和N。因此,该实验说明了强选择条件下稳定性的增强提高了突变稳健性,从而有助于增加遗传多样性。与此同时,这个结论也证实了达尔文的进化论,即在通过强选择增加种群进化潜力的情况下,面对气候变化等环境挑战时种群的进化拯救可能会更容易。
区别于天然蛋白质仅有单一折叠状态(即能量最低态)的传统观点,还有其他观点认为许多天然蛋白质存在多个折叠形态,是一种多构象的集合(native state ensemble)[113]。蛋白质的功能与这个集合中的折叠形态一一对应。由于这些折叠状态能量相同或者近似,蛋白质可以在不同折叠状态间切换。Tawfik等认为这些折叠状态的多样性是蛋白质进化的重要基础,并探究了关键位置突变对于蛋白质稳定性和进化性的影响[114]。他们发现蛋白质关键位置的突变会打破多构象的相近折叠能分布,产生一个或几个热力学更加稳定的优势构象,这些新构象为新功能的产生提供了结构基础,并在自然选择下发生进化。Kamerlin等的工作也通过计算的手段详述了蛋白质复杂构象动力学(conformational dynamics)对于其可进化性的重要意义[115]。Sun等于近期发表的工作运用实验与计算相结合的手段,创新性开发了锌依赖性乙醇脱氢酶(zinc-dependent alcohol dehydrogenase fromThermoanaerobacter brockii,TbSADH)定向进化的方法[116]。TbSADH 存在着一段结构灵活的底物结合位点区域,野生型的TbSADH 依靠这一区域产生复杂的构象动力学行为。该区域由9个氨基酸组成,其与底物相互作用关键的残基是第84 位的脯氨酸(P84)。作者通过对P84进行大规模的突变筛选,改变了原有酶活性位点的大小与底物手性识别能力,并进化出具有可容纳新底物、对底物具有手性选择性的新突变。传统的蛋白质定向进化领域侧重于在蛋白质不改变天然结构的情况下进行探讨,而这些工作提出的新观点,即蛋白质通过突变定向进化出具有新功能的新构象,极大地丰富了蛋白质定向进化领域的认知。
无论是Drummond 等认为的“翻译稳健性”,还是Bloom、Zheng 等提出的“蛋白质增强的稳定性更加包容突变”,都强调了蛋白质稳定性对蛋白质进化的重要影响,却得到完全不同的结论。其中的主要原因在于实验条件的选择。达尔文进化论肯定了基因突变和自然选择在物种进化上的重要影响,蛋白质进化同样也离不开这两个重要因素。Drummond 等并没有考虑“条件选择”这一因素;不仅如此,他们也没有意识到蛋白质高表达时细胞面对压力产生的质量调控机制,将实验条件过分简化,从而得到相反的结论。天然蛋白质的边缘稳定性在一定程度上限制了进化,但是“边缘稳定”并不代表“不稳定”;恰恰相反,“边缘稳定”维护了生物系统的整体稳定性。例如,绝大多数蛋白质的突变体都会比“边缘稳定”的野生型更加不稳定,进而很快就会被淘汰,这在一定程度上维持了蛋白质性质和功能的稳定性;蛋白质的降解也变得更加容易,因为细胞不需要花费过多能量去强制展开过度稳定的蛋白质,从而有利于维持内环境稳态。辩证性地看待蛋白质稳定性与进化的关系能够更好地帮助我们理解大自然的进化规律,同时深化对疾病或癌症的机理性研究,促进药物开发以及做到提前预防。
4.2 分子伴侣协助蛋白质定向进化
除了帮助蛋白质折叠,分子伴侣还可以作为不稳定突变的缓冲系统,促进遗传的多样性和加快适应速度。Tokuriki 等在2009 年发现Hsp60/Hsp40分子伴侣系统可以协助不稳定酶突变体的折叠,从而促进酶进化[117]。其中,他们主要探究大肠杆菌内GroEL/GroES 对于携带改变或改进酶功能的突变体的作用。在对4种GroEL/GroES依赖性不同的酶进行随机突变漂移(random mutational drifts)后,他们发现GroEL/GroES 过表达的情况下,依赖于GroEL/GroES 的酶产生了更多正常折叠且具有催化活性的突变体。在对这些突变体序列进行比对后发现,依赖于GroEL/GroES 的酶能够承受更多在核心区域的残基替代。相比于蛋白质表面,在蛋白质内部核心区域发生的残基替代更有可能导致蛋白质错误折叠,这也解释了依赖于GroEL/GroES 的酶突变体平均稳定性更低的原因。因此,GroEL/GroES可能在与蛋白质相互作用过程中缓冲了突变带来的不稳定性。不仅如此,他们还发现在GroEL/GroES 过表达情况下,适应突变体改进的特异性和活性是没有过表达情况下的至少10 倍,说明分子伴侣的存在更有利于有益变异的产生。总的来说,GroEL/GroES的过表达可以显著增加具有改进功能或新功能的酶突变体的数量和倍数,是分子伴侣协助蛋白质定向进化的有力证据。
2018 年Phillips 等发现分子伴侣能够促进流感核 蛋 白(nucleoprotein, NP) 产 生 适 应 性 突变[118]。他们采用化学诱导HSF1 抑制剂的方法降低细胞溶质分子伴侣水平,并结合深度突变扫描探究单个氨基酸的取代在宿主细胞缺失分子伴侣情况下的适应性。结果发现在细胞处于类似发热温度(39°C)时,绝大多数NP突变体在没有分子伴侣的条件下适应性降低,只有几个特定位点氨基酸的取代显著升高了NP 的适应性。其中,位点283 的脯氨酸对于人类抗病毒限制因子MxA(myxovirus resistance protein A)免疫系统逃逸有很大贡献,而其他氨基酸如甲硫氨酸和丙氨酸在该位点的取代使得NP 变体在没有分子伴侣的情况下相比于野生型更稳定。进一步研究发现脯氨酸在这个位点的取代降低了282~284 位点α-螺旋含量,导致Pro283 NP 变体结构更加不稳定,且更容易聚集。这种不稳定性在环境温度升高且没有分子伴侣的情况下更为明显,说明分子伴侣可以帮助调节流感中免疫逃逸突变体的适应性。除了流感核蛋白,他们还研究了宿主细胞内质网分子伴侣对分泌途径蛋白突变的影响。同样的,他们发现内质网分子伴侣能够影响流感血凝素(hemaggluttinin,HA)对突变的耐受程度[119]。当环境温度升高时,许多HA 变体变得不稳定,从而数量急剧降低。但是在XBP1s 或是ATF6f/XBP1s存在的情况下,内质网分子伴侣水平上升,并帮助这些不稳定HA 变体正确折叠。这些工作指出了分子伴侣在协助蛋白质定向进化中的重要作用,为抗病毒药物的研发提供了理论依据。
除了前面提到的蛋白质类分子伴侣外,人们发现化学伴侣(chemical chaperone)也可以帮助体内蛋白质进化。化学伴侣是一类能够影响蛋白质稳定性的小分子,主要被分为渗透液、疏水化合物和药理伴侣。Bandyopadhyay 等在研究渗透液对蛋白质突变的缓冲能力时,发现细胞的化学环境可能改变细胞内特定蛋白质突变体折叠的途径[120]。首先,他们采用模型渗透液三甲胺正氧化物(trimethylamineN-oxide,TMAO)来研究渗透液是否能通过帮助蛋白质折叠来缓冲遗传变异。TMAO 是一种胆碱、甜菜碱和肉碱通过肠道微生物代谢产生的小分子[121]。在研究TMAO 对野生型麦芽糖结合蛋白(maltose binding protein, MBP)及其突变体折叠速率的影响时发现,MPB 仅单位点氨基酸的取代就可大幅提高蛋白质折叠对TMAO 的依赖程度,进而提升蛋白质在渗透液中的折叠速率。这些MBP 突变体与野生型具有类似的天然构型,说明TMAO 的辅助功能可能主要由蛋白质的折叠途径决定。为了验证这一假设,他们通过引入甘氨酸增加蛋白质链的灵活性,并从实验中发现TMAO 能够特异地缓冲导致蛋白质折叠途径中柔性驱动陷阱(flexibility-driven traps)的突变,而非通过产生其他分子伴侣来作用。不仅如此,在通过对5000 个单位点突变体的筛选后发现,不同的渗透液对不同突变的缓冲能力也不尽相同,主要体现在不同的渗透液能够独特地缓冲不同的突变区间。例如,TMAO 允许在蛋白质高度保守且容易导致聚集的区域进行替换;而脯氨酸允许在蛋白质表面且不易导致聚集的区域进行替换。这表明不同的渗透液可能以不同的方式驱动蛋白质进化,主要基于化学伴侣在蛋白质折叠途径上的不同作用机制。因此,研究化学伴侣对细胞内蛋白质折叠以及特殊变异的缓冲,不仅为探究蛋白质稳定性与可进化性关系提供多维线索,还能够从化学小分子层面探测影响蛋白质构效关系的决定性因素。
4.3 分子伴侣自身的定向进化
通常来讲,分子伴侣的普遍性使得分子伴侣在自然进化过程中会高度保守,因为一旦发生变异就很有可能丧失掉相应的功能,从而不被保留下来。其中,Hsp70就是这样一个进化保守的分子伴侣。不管在原核生物还是真核生物细胞内,Hsp70家族有超过一半的相似氨基酸序列。然而并非所有分子伴侣都是这样。比如Hsp40家族,它们仅仅只有一个结构域J 是相似的,而且就是通过这个结构域刺激ATP 酶活性帮助Hsp70 折叠蛋白质[85]。J 结构域包含70 个氨基酸残基,由四个螺旋以及螺旋Ⅱ和Ⅲ之间的环区组成[122]。人们发现,在生物体内,Hsp40 的种类数量远多于Hsp70。例如,在大肠杆菌中发现了6 种DnaJ 的同系物[123];在恶性疟原虫(Plasmodium falciparum)中,至少发现了10 种DnaJ 相关的蛋白质[124];而在人类蛋白质组中,也有140 种蛋白质与DnaJ 相关。不仅如此,不同的DnaJ 蛋白也进化出了不一样的功能。例如,在酵母内质网中发现了3种DnaJ的同系物,每一种都专门用于调节酵母内Hsp70的同系物——Kar2的不同功能。其中Sec63是一种重要的跨膜蛋白,可帮助Kar2 将新生蛋白转运至内质网,Scj1 可以帮助Kar2 在内质网腔中折叠和组装蛋白质,而Jem1 主要通过与Kar2p 相互作用,在核膜融合中发挥重要作用[125-127]。由此可见,在不同条件下,底物蛋白质的进化也能够刺激分子伴侣的进化,以提升其对底物蛋白质的特异性,为物种多样性奠定了基础。
认识到分子伴侣在蛋白质折叠以及进化过程中起到的重要作用后,人们想到可以通过定向进化已知分子伴侣来优化其功能。Wang 等早在2002年就发现可以通过定向进化GroEL/GroES 优化其对特定底物的折叠效率[128]。首先,他们使用多轮体内筛选和体外DNA 改组,筛选出能够显著提高绿色荧光蛋白(green fluorescent protein, GFP)折叠能力的GroEL/GroES 突变体。在对突变体序列进行分析后发现,突变主要集中在负责与ATP酶结合的区域。ATP的结合与水解为GroEL/GroES折叠循环提供动力,同时也与GroEL 功能构象变化有关。实验分析表明,ATP结合区域的突变显著提高ATP 酶的催化活性。不仅如此,他们还发现GroES突变体带电氨基酸残基增多。作为折叠腔的盖子,GroES静电环境的改变降低了折叠腔内部的疏水性。因此,他们推测GroEL/GroES 突变体这两个方面的改变显著提升了GFP 的折叠效率,即GroEL/GroES在底物范围方面具有可塑性。然而有趣的是,在GroEL/GroES 变体提升对GFP 的折叠效率时,对其他种类蛋白质如HrcA 的折叠效率却降低,揭示出分子伴侣特异性和普遍性可能的内在冲突。这种冲突说明了分子伴侣能够根据特定底物的性质产生进化,从而为探究分子伴侣与底物相互作用机制、底物对分子伴侣进化的影响提供理论支持。
分子伴侣的定向进化也为治疗由蛋白质错误折叠引起的几种常见神经退行性疾病提供了新思路。受到酵母(yeast)中Hsp104 对朊病毒严格调控的启发,Jackrel 等在2015 年报道了Hsp104 的增强突变体,该突变体可被用于有效解聚与肌萎缩侧索硬化(amyotrophic lateral sclerosis,ALS)和帕金森病相关的错误折叠蛋白质[129]。野生型Hsp104 对人类神经退行性疾病相关蛋白质没有明显的解聚效果。然而,通过对Hsp104 不同结构域的随机变异,他们发现中部的螺形线圈结构域的突变能够明显增强Hsp104 对TDP-43、FUS 以及αsynuclein 在酵母中有毒聚集体形成的抑制。值得注意的是,只需要将Hsp104 第503 位的丙氨酸突变成任意氨基酸(脯氨酸除外),该突变体就能够降低上述蛋白质在酵母中的毒性。除此之外,Hsp104 突变体增强的抑制作用并不来自于对蛋白质质量控制体系的激活,如自噬或泛素-蛋白酶体系统;而是作为遗传抑制因子根除蛋白质聚集体,同时加速底物的易位。进一步对Hsp104 突变体作用机制进行探究后,发现与野生型不同的是,这些功能增强的突变体并不需要Hsp70/Hsp40 的协助;而对Hsp70/Hsp40 的依赖只会妨碍其抑制作用。Hsp104 增强突变体的独立性有望被运用到不同生物体系,帮助优化不稳定蛋白质的过量表达,并进一步开发专门针对特定一种或多种蛋白质的增强变体,以最大限度减少脱靶效应,从而开发出高效率治疗剂。
5 总结与展望
生物体的正常运行离不开蛋白质的稳定性,生物的多样性离不开蛋白质的可进化性。天然蛋白质正常折叠时并非处于其最稳定状态,因为多数天然蛋白质能够在保持甚至提高功能的同时得到稳定性增强的突变体。蛋白质的折叠自由能为-10 kcal/mol 左右,仅具有边缘稳定性。蛋白质组的稳定性取决于蛋白质组中折叠不稳定的蛋白质,因而也具有边缘稳定性。蛋白质的进化往往离不开一级结构上的关键氨基酸突变。绝大多数突变,甚至是可能导致新功能的突变都会在蛋白质边缘稳定的基础上进一步降低其稳定性。这些不稳定的蛋白质突变体很容易被细胞质量调控网络清除,从而不再具有进化的可能性。因此,蛋白质的边缘稳定性限制了蛋白质的进化。为了突破这个限制,科学家们想到了从源头上解决问题——调控蛋白质的稳定性。蛋白质稳定性的增加提升了其对突变的包容度,即能够在产生更多突变时仍正确折叠,从而在功能不损伤的情况下积累一定的有益突变。其实,积累有益突变的过程就是在进化。从许多实验室定向进化得到的具有增强或更新功能的突变体来看,往往少数的突变,甚至是一个位点的突变,就能够部分或完全改变蛋白质的功能。分子伴侣除了可以帮助蛋白质折叠、展开错误折叠的蛋白质,还作为缓冲系统帮助蛋白质积累一定的突变,从而帮助蛋白质进化。同样作为蛋白质的分子伴侣也能够进化。在实验室中,分子伴侣能够通过定向进化来优化其折叠功能,或用作疾病相关易聚集蛋白质的解聚剂。自然界中的分子伴侣在不同生物体内,甚至是同种生物不同细胞器内也进化出功能相似或完全不同的同源物,为物种多样性提供物质基础。因此,分子伴侣和蛋白质的进化是相辅相成的:分子伴侣可以帮助蛋白质进化,而蛋白质的进化也需要分子伴侣产生相应改变来适应进化的蛋白质。希望在不久的将来,我们能够利用更为丰富的蛋白质结构数据库以及更精密的计算模型系统地探索分子伴侣与蛋白质进化的相互作用关系,从而为模拟自然界生物进化提供更为坚实的理论基础。作为影响蛋白质稳定和进化的重要分子机器,分子伴侣不仅在基础生物学研究领域,还在生物工程和生物医药等领域都彰显出不可小觑的实力。其中,定向进化分子伴侣抑制细胞内蛋白质的聚集为治疗相关神经退行性疾病提供了新方向。相比于以往的小分子药物,分子伴侣更易生产、可操控性高且生物相容性好,有望作为预防和治疗蛋白质错误折叠相关疾病的重要手段。
