基于高通量测序技术分析青藏高原风干牦牛肉中细菌多样性
2022-07-08弋凯鸽吴寒冰李帅康池福敏辜雪冬
弋凯鸽,吴寒冰,李帅康,王 卫,耿 放,杨 林,池福敏,辜雪冬,
(1.西藏农牧学院食品科学学院,西藏林芝 860000;2.西藏特色农牧资源研发省部共建协同创新中心,西藏林芝 860000;3.成都大学肉类加工四川省重点实验室,四川成都 610106)
牦牛被誉为“高原之舟”,是我国高原地区所特有的珍稀牛种[1],其肉中富含高蛋白和氨基酸,且脂肪和热量都较低,因而深受消费者喜爱。风干牦牛肉大多是藏区人民家庭式生产用来过节招待客人或者自己食用的一种传统生食肉制品。它的制作工艺是将牦牛肉进行分割切条后,充分利用高原地带冬季低温干燥低气压的特殊自然气候条件风干而成[2]。风干牦牛肉也有小作坊生产和小型企业的批量生产,受藏族人民传统的饮食习俗和不规范的制作工艺等方面的影响,其成品可能存在安全卫生及污染等方面的问题[3]。在风干牦牛肉的制作过程中,空气和土壤中含有大量微生物,其中可能存在一定数量对形成风干牦牛肉品质有积极作用或有潜在益生特性的菌株,也可能存在一些致病菌及条件致病菌[4]。
高通量测序技术可以对一个物种的基因组进行全面分析,且具备处理大规模样品的优势[5]。因此,利用高通量测序技术测定不同环境中样品的微生物,已经成为很多学者使用的一种手段。对比传统微生物研究方法,如平板分离纯化法、定量PCR 等,具有高灵敏度、高效性、准确性、无偏向性等优点,主要应用于微生物学、分子生物学、遗传学等领域。目前关于风干牦牛肉中微生物研究多侧重在加工以及品质形成方面[6−7],而在微生物多样性方面的研究多侧重在对细菌数量的变化[8]。因此,为探究西藏不同地区风干牦牛肉中细菌群落组成对其品质和安全性的影响,本文利用MiSeq 技术对西藏风干牦牛肉中的细菌群落结构以及多样性进行分析,为后续风干牦牛肉的加工与安全控制提供一定的参考。
1 材料与方法
1.1 材料与仪器
风干牦牛肉 于2021 年4 月采自西藏不同地区的当地农户家庭,分别是那曲班戈县、昌都丁青县、日喀则谢通门县、拉萨达孜县、山南扎囊县。将不同的地区分成5 组,编号为那曲(NQ)、昌都(CD)、日喀则(RKZ)、拉萨(LS)和山南(SN),每个地区分别采集三份来自于不同农户家庭的样品;DNeasy Powe Water DNA 提取试剂盒 德国Qiagen公司;AxyPrep DNA Gel Extraction DNA 凝胶回收试剂盒 美国Axygen 公司;DNA Marker DL2000宝生物工程(大连)有限公司;琼脂糖凝胶电泳试剂美国Invitrogen 有限公司。
P7589 荧光分光光度计 美国Invitrogen 有限公司;BG-gdsAUTO(130)凝胶成像设备 北京百晶生物技术有限公司;DYY-6C 电泳仪 北京六一生物科技有限公司;Flx800 酶标仪 美国伯腾有限公司;2720 PCR 扩增仪 美国ABI 公司。
1.2 实验方法
1.2.1 样品前处理 在采集点将样品放置于无菌样品采集袋,在采集袋上进行样品编号,储存于装有冰袋的泡沫箱中尽快送回实验室,4 ℃冰箱中保存备用。
1.2.2 DNA 提取 采用 Mo Bio/QIAGEN 公司的DNeasy PowerWater Kit 进行抽提,之后在260 和280 nm处用荧光分光光度计分别测定抽提DNA 的吸光值,得到DNA 浓度,并使用1%琼脂糖凝胶电泳检测DNA 的质量。调整DNA 溶液浓度,于4 ℃下保存DNA 工作液,−20 ℃下保存储存液。
1.2.3 PCR 扩增 首先针对样本DNA 特定16S rRNA V3V4 区进行PCR 扩增338F(5'-ACTCCTACGGGG AGGAGCA-3')和806R(5'-GGACTACHVGGGTWT CTAAT-3'),对目标条带大批量PCR 扩增后进行割胶回收,得到纯化的样本。用酶标仪对各个样品定量,最后采用标准的Illumina TruSeq DNA 文库制备实验流程构建所需的上机文库,在上海派森诺公司的Illumina Novseq PE250 平台进行测序。
1.2.4 序列分析 首先调用QIIME2 进行修改,使用demux 插件解复用,用cutadapt 对序列的引物片段进行切除,删去未匹配引物的序列,然后通过dada2对序列进行质量过滤、去噪、合并、去除嵌合体[9]。使用RDP FrameBot 软件,基于RDP 网站下载的相应功能基因的seeds 蛋白序列,对核酸序列中的插入和缺失错误进行纠正,对样本中所包含的高质量序列的长度分布进行统计[10]。
1.3 数据处理
使用R 语言stat 包的uclust 函数,默认采用UPGMA 算法对beta 多样性距离矩阵进行聚类分析,采用QIIME2 的classify-sklearn 算法与SILVA132的Greengenes 数据库进行物种注释分析,得到在每个分类水平上的分类学组成信息。通过Qiime 软件计算Chao1、Simpson、Shannon 等指数,使用R 软件根据多样性指数绘制稀释曲线。基于Qiime 软件Jaccard 和Bray-Curtis 等两种距离矩阵,对这些距离矩阵做PCoA 分析,并通过R 软件进行PCoA 分析[11]。
2 结果与分析
2.1 测序数据统计与稀释曲线分析
测序获得不同风干牦牛肉样品的原始序列数据后,进行筛选,除去不合格的序列,得到的有效序列共386693 条,主要集中分布在404~433 bp 范围内。稀释曲线的走势表明样品测序深度对多样性的影响,当曲线走势趋于平缓,说明该结果充分覆盖了目前样本中包含的所有物种[12]。随机从各样本的原始数据中抽取一定数量的序列,获得样本在一定的条件下所可能包含的物种总数及其中每个物种的相对丰度,得到稀释曲线(图1)。结果表明,随着数据量的增加,曲线逐渐平缓,当前样本已足够反应了所包含的多样性,具有科学性。
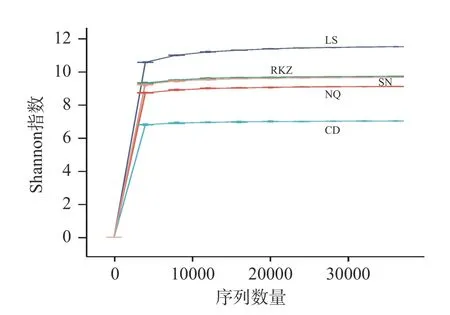
图1 样品稀疏曲线图Fig.1 Sample sparseness curve
2.2 Alpha 多样性分析
为探究风干牦牛肉样品中细菌群落的丰富度和多样性,采用多样性指标的Observed_species、Chao1、Shannon、Simpson 及Coverage 指数进行评估[13]。Coverage 指所测序列对样本的覆盖率,值越高,则表明测出的样本序列概率越高,可以准确地反映出样品中微生物多样性的真实情况。Simpson 与Shannon均被用来估算微生物多样性,微生物多样性主要包括丰富度和均匀度。Simpson 侧重相对丰度,而Shannon 更侧重物种丰富度。当二者指数值越大,说明群落多样性越高。Chao1 指数用于计算群落丰度,样品的群落丰度与值的大小呈正比[14]。如表1 所示,5 组样品的Coverage 指数都达到了0.98 以上,表明测序结果真实可靠。LS 组的风干牦牛肉样品中Chao1、Shannon 及Simpson 值最高,表明该地区的物种丰度最高,其菌群结构也更复杂。这可能与拉萨地区气候相对温和湿润有利于部分细菌生长有关。CD 组的风干牦牛肉样品的Chao1、Shannon、Simpson 指数均为最小,这说明了该组样品中菌群群落丰度及多样性最低。原因可能是由于昌都地区的人们在制作风干牦牛肉时,采用整条后腿肉直接风干后再切割食用的方式而导致部分细菌分布不均。其余组的丰度指数和多样性指数也有明显的差别,那曲、日喀则、山南地区均采用传统切割分条风干的方式,其中,那曲气候最为寒冷干燥,而导致部分细菌不易生长。这说明不同的风干环境和农户制作方式导致细菌群落的多样性存在差异。
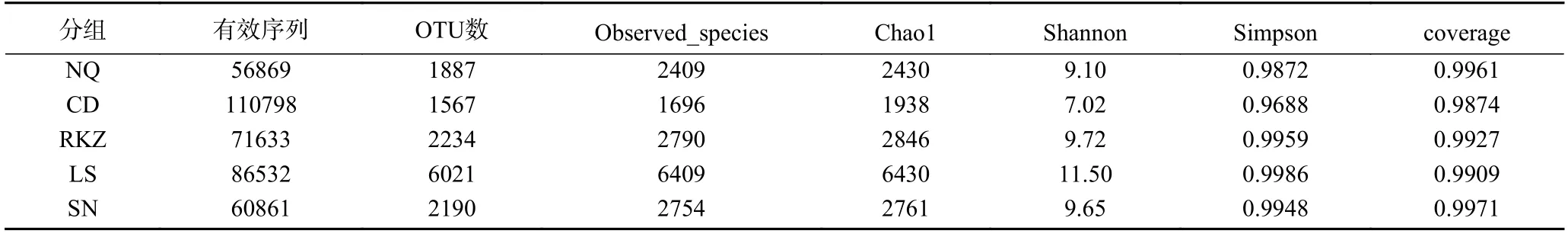
表1 α-多样性指数Table 1 α-diversity index
2.3 OTU 的花瓣图分析
基于不同组的样本中所含的OTU 总数绘制韦恩图,利用该图对不同样本的群落进行分析,可以直观表达各组样本共有以及特有的OTU 数[15]。如图2所示,LS 组样品OTU 数有6021 个,RKZ 组样品OTU数有2234 个,SN 组样品OTU 数有2190 个,NQ 组样品OTU 数有1887 个,CD 组样品OTU 数有1567个,而各样本共有的OTU 数为27 个,表明各样本之间的菌群结构具有明显的差异。
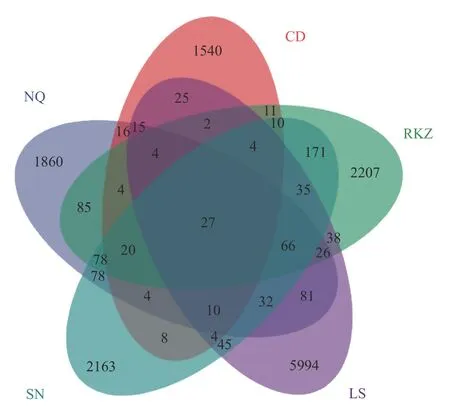
图2 风干牦牛肉中细菌的OTU 分析图Fig.2 OTU analysis of bacteria in air dried yak meat
2.4 不同地区风干牦牛肉样品细菌群落结构分析
2.4.1 基于门水平的细菌群落结构分析 在门水平上,5 组风干牦牛肉样品的群落组成如图3 所示。5 组样品中微生物群落构成基本相同,主要差别在微生物的相对丰度上。微生物群落主要包括放线菌(Actinobacteria)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、蓝细菌门(Cyanobacteria)、酸杆菌门(Acidobacteria)和芽单胞菌门(Gemmatimonadetes)等。NQ 组的风干牦牛肉样品细菌组成以厚壁菌门和放线菌门为主,相对丰度分别为38.91%和25.22%,CD 组菌群结构以变形菌门(65.62%)和厚壁菌门(22.01%)为主,RKZ 组以放线菌门(48.82%)和厚壁菌门(21.40%)为主,LS 组以放线菌门(35.86%)和厚壁菌门(29.21%)为主,SN 组以放线菌门(39.80%)和厚壁菌门(24.49%)为主。结果发现,NQ、RKZ、LS、SN 组的优势菌群是厚壁菌门和放线菌,CD 组的优势菌群为变形菌门和厚壁菌门。这可能与风干牦牛肉制作时间都处于天气寒冷干燥时期有关,此环境条件下有利于其生存。其中,厚壁菌门中的细菌可以抵抗脱水和极端环境。芽孢杆菌纲内的物种由于大多可形成抗逆性极强的芽孢,而具有极强的环境适应性[16],因此,在各类生态系统中发挥着重要的生物学作用。放线菌门中大多数细菌是一种呈菌丝状生长和以孢子繁殖的原核生物,且孢子具有耐干燥的能力[17]。变形菌门中大多数细菌营兼性或者专性厌氧及异养生活[18]。
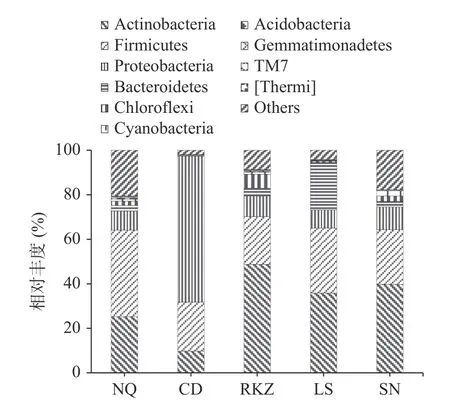
图3 基于门水平上风干牦牛肉的细菌群落分布图Fig.3 Distribution map of bacterial community based on air dried yak meat at door level
2.4.2 基于科水平的细菌群落结构分析 不同组样品中在科水平上的群落分布,如图4 所示。5 组样品中细菌群落大体上一致,主要差别在群落的相对丰度上。5 组样品中微生物群落主要包括微球菌科(Micrococcaceae)、肠杆菌科(Enterobacteriaceae)、假单胞菌科(Pseudomonadaceae)、链球菌科(Streptococcaceae)、蓝球菌科(Planococcaceae)、间孢囊菌科(Intrasporangiaceae)、棒状杆菌科(Corynebacteriaceae)、瘤胃球菌科(Ruminococcaceae)、诺卡菌科(Nocardioidaceae)和乳杆菌科(Lactobacillaceae)等。NQ 组的风干牦牛肉样品菌群组成以乳酸杆菌科(13.56%)为主。乳酸杆菌科主要在厌氧环境下繁殖,广泛存在于发酵食物中,可提高食品中的风味物质[19]。肠杆菌科(35.62%)和假单胞菌科(27.67%)为CD 组主要群落,为食物中的优势腐败菌群,具有分解蛋白质和脂肪的能力[20]。RKZ 组以间孢囊菌科(10.39%)为主,该菌的菌丝体可能含有间囊泡,但不含孢子[21],可能来源于自然环境。LS 组以微球菌科(14.71%)为主,部分菌属于非致病菌,营呼吸与发酵代谢,可能来源于原料。棒状杆菌科(8.63%)为SN组主要群落,为肠道菌群中正常存在的微生物,具有一定的耐受能力[22]。
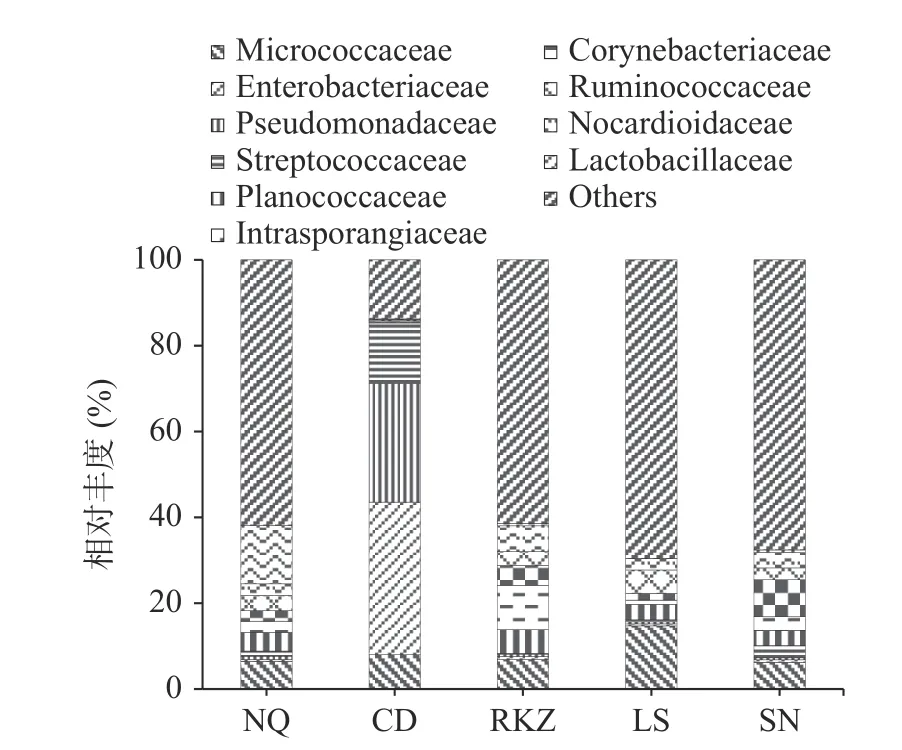
图4 基于科水平上风干牦牛肉的细菌群落分布图Fig.4 Bacterial community distribution map of air dried yak meat based on family level
2.4.3 基于属水平的细菌群落结构分析 基于属分类水平的5 组风干牦牛肉样品的细菌群落结构如图5 所示。5 组样品中细菌群落除未分类的菌株,在构成和含量上有明显的差别。细菌群落主要包括假单胞菌属(Pseudomonas)、节杆菌属(Arthrobacter)、棒状杆菌属(Corynebacterium)、乳酸杆菌属(Lactobacillus)、链球菌属(Streptococcus)、普雷沃菌属(Prevotella)、动物杆菌属(Planomicrobium)、梭菌属(Clostridium)、小双孢菌属(Microbispora)和乳球菌属(Lactococcus)等。由图可以看出,NQ 组的优势菌群为乳酸杆菌属(12.75%),CD 组为假单胞菌属(27.27%),RKZ 组为棒状杆菌属(4.59%),LS 组为节杆菌属(10.99%)和普雷沃菌属(10.20%),SN 组为棒状杆菌属(8.63%)。这与田建军等[23]的研究结果基本一致。其中,乳酸菌作为发酵食物中重要的菌株,可以利用原料肉中的蛋白质和碳水化合物进行异质发酵产酸,赋予风干牦牛肉独特的风味。假单胞菌、节杆菌属和棒状杆菌属属于优势腐败菌,可以分解食物中的蛋白质和碳水化合物[24−26]。普雷沃菌属是人体健康肠道菌群中重要的菌群的一种,适量存在对人体健康有益[27]。
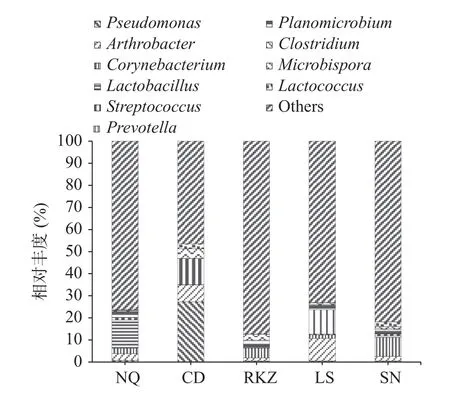
图5 基于属水平上风干牦牛肉的细菌群落分布图Fig.5 Bacterial community distribution map of air dried yak meat based on genus level
2.4.4 物种丰度聚类热图 为更直观展现各地区风干牦牛肉优势细菌的丰度变化以及相似性,对相对丰度排名在前20 的属绘制热图。由图6 聚类分析结果可得,那曲与拉萨地区样品间结构相近,山南与日喀则地区样品间结构相近,可能是由于这四个地区在地理位置上比较相邻,农户家庭的加工方式也大致相同。昌都样品采集地区的位置距离其他四个地区较远,且加工方式也有较大差异。各地区优势细菌的相似性也有较大的差异,昌都与拉萨地区的优势细菌树枝距离最近,相似性较高。日喀则与山南地区优势细菌之间的树枝距离最近,相似度较高。这两个地区的优势菌与那曲地区的相近,相似度较高。
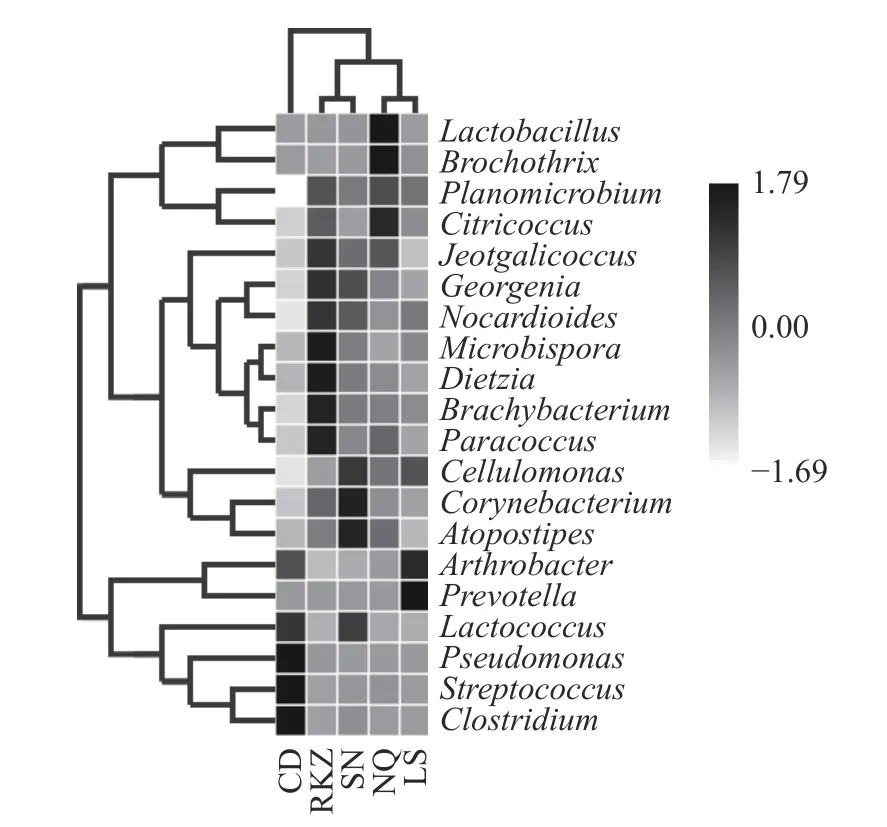
图6 属分类水平风干牦牛肉的细菌丰度热图Fig.6 Heat map of bacterial abundance of air dried yak meat at genus classification level
3 讨论
本文以西藏地区的风干牦牛肉作为研究对象,通过对5 个地区样品进行细菌多样性分析,发现各地区间细菌种类以及丰度相差较大,这些细菌主要来源于风干牦牛肉的原料肉以及生产环境中。在门水平分类上,优势菌群为厚壁菌门、变形菌门以及放线菌门。这与研究发酵肉制品中的结果基本一致[26,28]。相关研究表明,厚壁菌门中清酒乳杆菌在发酵肉制品中产酸,导致pH 降低,抑制杂菌生长,加速亚硝酸盐分解,致使亚硝胺减少[29]。厚壁菌门也是存在于肉制品中的一种优势腐败菌[28],它存在于肠道中通过吸收食物中的热量而导致肥胖[30]。变形菌门与放线菌门中多为病原菌,例如:变形菌门中的沙门氏菌会引起食物中毒[31]。放线菌门中多为腐生菌,可以使食物腐败变质[32]。因此,为避免出现食物中毒的情况,采取在原料肉前处理时进行无菌处理,改进加工方法以及食用前进行加热等方式,尽可能降低有害菌导致的危害。在科分类水平上,各地区风干牦牛肉的菌群种类以及丰度差别较大,可能是与各地区的加工方式以及环境不同有关。其中,乳酸杆菌科为发酵食物中的有益菌株,利用营养物质产生风味物质而形成独特的风味,同时抑制一些食源性病原菌的生长,保证了食品的品质与风味[33]。假单胞菌科、肠杆菌科和棒状杆菌科多为优势腐败菌,尹德风等[34]对生鲜肉类产品中的腐败细菌进行研究,认为腐败微生物可能会出现在屠宰加工环节。因此,控制原料肉的处理极为重要。在属水平以及丰度热图上,发现了各地区的细菌菌落组成与丰度之间的差异与相互关系,从而说明加工环境以及方式对风干牦牛肉的品质有重要的影响。马静等[35]发现青海不同海拔牦牛乳的微生物组成和丰度有差异,认为环境对微生物多样性有重要影响。综合分析发现,不同地区的风干牦牛肉在门、科和属水平上存在种类及含量的差异。这可能与每个地区的环境有一定的关系,其次在加工过程中,不同原料肉的前处理方式以及农户的食用习惯,也有可能导致群落差异的产生。
4 结论
本研究通过对西藏风干牦牛肉进行高通量测序分析,研究发现在门分类上那曲、日喀则、拉萨以及山南组的优势菌为厚壁菌门和放线菌门,昌都组为变形菌门和厚壁菌门。在科分类上,那曲组优势菌为乳酸杆菌科,昌都组为肠杆菌科和假单胞菌科,日喀则组为间孢囊菌科,拉萨组为微球菌科,山南组为棒状杆菌科。在属分类上,那曲组为乳酸杆菌属,昌都组为假单胞菌属,日喀则和山南组均为棒状杆菌属,拉萨组为节杆菌属和普雷沃菌属,表明不同风干牦牛肉在菌落组成和丰度上有所不同。通过对西藏风干牦牛肉进行测序,了解藏区风干牦牛肉细菌群落情况,发现风干牦牛肉中既存在对品质形成以及产生风味的有益菌,也可能存在优势腐败菌以及致病菌,或与藏区农户对原料肉的前处理以及制作环境有很大关系。因此,提高藏区农户的食品安全意识,关注致病菌以及腐败菌污染的隐患,改善风干牦牛肉的加工方式十分重要。
