鸟类TLR 基因进化分析
2022-07-07梁春鸿李丹蕾
梁春鸿,李丹蕾,朱 英,何 珂
(1. 浙江农林大学动物科技学院、动物医学院,浙江 杭州 311302;2. 海南浙江大学研究院,海南三亚 572000;3. 西南民族大学青藏高原研究院,四川成都 610041)
Toll 样受体是一类参与非特异性免疫的重要蛋白,参与宿主对病原微生物感染的免疫反应,是宿主防御的重要介质,被称为宿主防御系统的警报受体.它是一种模式识别受体(pattern recognition receptor,PRRs),是连接天然免疫和适应性免疫的桥梁,在病原体或者宿主自身刺激下被激活.TLR基因与病原体相关分子模式(pathogen-associated molecular patterns,PAMPs)结合介导调控细胞内信号转导,分泌相关炎症因子抵御病原微生物侵染[1-2].
目前已发现TLR基因在脊椎动物、无脊椎动物和植物中广泛存在. 在脊椎动物中,大量的TLR基因已被识别,并且在结构和功能上均呈现出较高的相似性.但在不同物种中,TLR基因的种类和数量却存在较大的差异.在脊椎动物中发现的23 个TLR基因,鸟类中仅含有其中部分(10 种),分别是TLR1A、TLR1B、TLR2A、TLR2B、TLR3、TLR4、TLR5、TLR7、TLR15 和TLR21[3].其中,TLR15 和TLR21 是鸟类特有的TLR基因,而哺乳动物中存在的TLR8 和TLR9 基因已经在鸟类中缺失[4-5]. 鸟类中甚至还存在TLR1 和TLR2基因的重复事件,形成了TLR1A、TLR1B和TLR2A、TLR2B[6-7].
根据配体来源的不同,可将TLR基因分为外源性配体和内源性配体. 因识别的配体不同,又可将其分为病毒型(ViralTLRs)和非病毒型(Non-ViralTLRs).TLR3、TLR7 等为 ViralTLRs,只被表达在如内质网和溶酶体的胞内膜中,识别外源性配体,如肽聚糖、微生物核酸等.TLR1,TLR2,TLR4,TLR5 等为Non-Viral TLRs,能被表达在细胞表面,识别内源性配体,如脂质、脂蛋白等微生物膜组分[8].在禽类的研究中,鸡的TLR3、TLR7 和TLR21 基因均有抗病毒功能,其中TLR7 基因在不同类型的细胞上的细胞定位不同,且随时间的变化而移动,即先定位于内质网,移动至核内体、溶酶体等细胞器进行相关功能发挥[9]. 鸡的TLR21 基因具有识别细菌基因组DNA、合成寡聚脱氧核苷酸的核苷酸受体的功能[10].
研究结果表明,在哺乳类和鸟类中,ViralTLRs和Non-ViralTLRs基因会表现出不同的进化模式[11],但关于TLR 基因进化的研究多在脊椎动物和两栖动物中展开,对鸟类的TLR基因进化研究相对较少. 在两栖动物的研究中已发现ViralTLRs比Non-ViralTLRs受到更多的选择压力,并且两栖动物的TLR 基因家族组成介于鱼类和羊膜动物之间[12].
不同类型的鸟类面临不同环境的病原体选择压力,如具有长距离迁徙行为的雁形目物种是多种病原生物的重要载体和病毒的天然储存库. 因此,研究鸟类TLR基因家族的进化机制将有助于我们深入理解不同类群鸟类的适应性进化.本研究利用基因组学结合生物信息学研究手段,从基因组数据中挖掘鸟类TLR基因,分析TLR基因家族在鸟类中的进化模式,旨在明确鸟类TLR基因物种间拷贝数量差异和基因进化选择,进一步了解鸟类免疫系统适应性进化的机制.
1 材料和方法
1.1 实验数据
本研究所采用的基因组数据来源于国家生物技术中心信息检索数据库(National Center for Biotechnology Information Search database,NCBI),由于三代测序技术能够获得更长的测序结果,更有利于基因家族位点预测及序列获取[13],因此主要采用其获得的基因组数据进行分析.
鸟类被划分为古颚下纲(古颚总目)和新颚下纲,新颚下纲包括鸡雁小纲(鸡形目和雁形目)和新鸟小纲(包括雀形目等95%的物种). 所采用的数据由两部分组成,分别是三代测序得到的基因组数据,共有41 种,属16 个目;及二代测序得到的基因组数据(由于古鹗总目物种缺少三代测序数据,但其在鸟类进化上不可或缺,因此我们采用二代数据进行补充;雁形目也用二代数据进行补充分析),共32 种,属7 个目(见表1).

表1 本项目所用基因组数据Table.1 Genome data used in this project

images/BZ_29_207_362_2274_408.pngSylvia atricapilla 黑顶林莺 GCA_009819655.1 Taeniopygia guttata 斑胸草雀 GCA_008822105.2佛法僧目Coraciiformes Merops nubicus 洋红蜂虎 GCA_009819595.1鸽形目Psittaciformes Streptopelia turtur 欧斑鸠 GCA_901699155.1鹳形目Pteroclidiformes Pterocles gutturalis 黄喉沙鸡 GCA_009769525.1 Balearica regulorum 灰冠鹤 GCA_011004875.1 Cariama cristata 红腿叫鹤 GCA_009819825.1 Grus nigricollis 黑颈鹤 GCA_004360235.1鸻形目Charadriiformes Alca torda 刀嘴海雀 GCA_008658365.1 Pluvialis apricaria 欧金鸻 GCA_017639485.1 Sterna hirundo 普通燕鸥 GCA_009819605.1火烈鸟目Phoenicopteriformes Phoenicopterus ruber 大火烈鸟 GCA_009819775.1鸡形目Galliformes Centrocercus minimus 甘迅尼松鸡 GCA_005890655.1 Gallus gallus 原鸡 GCF_000002315.6 Pavo cristatus 蓝孔雀 GCA_005519975.1鹃形目Cuculiformes Cuculus canorus 大杜鹃 GCA_017976375.1 Tauraco erythrolophus 红冠蕉鹃 GCA_009769465.1隼形目Accipitriformes Aquila chrysaetos 金雕 GCA_900496995.3 Falco naumanni 黄爪隼 GCA_017639645.1 Falco rusticolus 海东青 GCA_015220075.1犀鸟目Bucerotiformes Bucorvus abyssinicus 地犀鸟 GCA_009769605.1夜鹰目Caprimulgiformes Nyctibius grandis 大林鸱 GCA_013368605.1 Pogoniulus pusillus 红额67 补images/BZ_29_1356_1194_1382_1221.pngGCA_015220805.1鹦形目Psittaciformes Melopsittacus undulatus 虎皮鹦鹉 GCA_012275295.1 Strigops habroptila 鸮鹦鹉 GCA_004027225.2雨燕目Apodiformes Calypte anna 红喉蜂鸟 GCA_003957575.1雁形目Anseriformes Anas platyrhynchos 绿头鸭 GCA_015476345.1 and GCA_900411745.1 Aythya fuligula 凤头潜鸭 GCA_009819795.1 Cygnus atratus 黑天鹅 GCF_013377495.1 Cygnus olor 疣鼻天鹅 GCA_009769625.1以下为2 代测序数据雁形目Anseriformes Anas zonorhyncha 斑嘴鸭 GCA_002224875.1 Anser brachyrhynchus 粉脚雁 GCA_002592135.1 Anser indicus 斑头雁 GCA_006229135.1 Anseranas semipalmata 鹊雁 GCA_013399115.1 Asarcornis scutulata 白翅栖鸭 GCA_013398475.1 Branta canadensis 加拿大黑雁 GCA_006130075.1 Cairina moschata 疣鼻栖鸭 GCA_009194515.1 Chauna torquata 冠叫鸭 GCA_013399475.1 Cygnus cygnus 大天鹅 GCA_014362685.1 Heteronetta atricapilla 黑头鸭 GCA_011075105.1 Nettapus auritus 厚嘴棉凫 GCA_011076525.1 Oxyura jamaicensis 棕硬尾鸭 GCF_011077185.1 Stictonetta naevosa 澳洲斑鸭 GCA_011074415.1无翼鸟目Apterygiformes Apteryx haastii 大斑几维鸟 GCA_003342985.1 Apteryx owenii 小斑奇威鸟 GCA_003342965.1 Apteryx mantelli 北岛褐几维鸟 GCA_001039765.1 Apteryxrowi 奥卡里托几维鸟 GCF_003343035.1鸵鸟目Struthionformes Struthio camelus 非洲鸵鸟 GCA_000698965.1鹤鸵目Casuariiformes Dromaius novaehollandiae 鸸鹋 GCA_016128335.1 Casuarius casuarius 鹤鸵 GCA_003342895.1美洲鸵鸟Rheiformes Rhea americana 大美洲鸵 GCA_003343005.1 Rhea pennata 美洲鸵鸟 GCA_003342835.1images/BZ_29_265_2292_291_2319.png形目Tinamiformes Tinamus guttatus 白喉images/BZ_29_1284_2292_1310_2319.pngGCA_000705375.2 Crypturellus cinnamomeus 棕穴images/BZ_29_1284_2328_1310_2354.pngGCA_003342915.1 Crypturellus undulatus 波斑穴images/BZ_29_1313_2363_1339_2390.pngGCA_013389825.1 Nothoprocta ornata 丽色斑images/BZ_29_1313_2399_1339_2425.pngGCA_013398335.1 Crypturellus soui 小穴images/BZ_29_1284_2434_1310_2461.pngGCA_013389845.1 Eudromia elegans 凤头images/BZ_29_1284_2470_1310_2496.pngGCA_003342815.1 Nothoprocta pentlandii 安第斯斑images/BZ_29_1342_2505_1369_2532.pngGCA_013398315.1 Nothoprocta perdicaria 智利斑images/BZ_29_1313_2540_1339_2567.pngGCF_003342845.1 Nothocercus julius 茶胸林images/BZ_29_1313_2576_1339_2603.pngGCA_013398735.1 Nothocercus nigrocapillus 斗篷林images/BZ_29_1313_2611_1339_2638.pngGCA_013398345.1
1.2 实验方法
1.2.1 鸟类TLR基因筛选
利用BLAST 程序[14]对鸟类基因组进行目的序列的查找,参数为e value ≤10-5.检索序列来源于原鸡(Gallus gallus)和斑胸草雀(Zebra finch)(见表2)[15].利用原鸡相关序列集进行非雀形目鸟类TLR基因搜索;斑胸草雀相关序列集进行雀形目相关分析.

表2 TLR 基因BLAST 检索的相关序列Table.2 ID of TLR query genes in avian
由于预测到的结果长短不一,在原始数据上,通过以下标准筛选出有效的TLR基因:(1)基因的长度应满足目标序列原长的60%;(2)比对相似度百分比应大于70%;(3)bit score 值要尽可能大且比对期望值要小于10-5. 在判定过程中,只有同时满足上述三者的才被认定是真基因,作为后续分析.
1.2.2 不同物种的TLR基因拷贝位点分析
利用Phylogeny subsets(http:/ /birdtree. org/subsets/)建立本研究中采用的鸟类系统进化关系,并在iTOL(https:/ /itol. embl. de/itol. cgi)中进行可视化.同时,TLR基因拷贝数目用R 软件中的Pheatmap 包和ggplot2 包进行可视化.
1.2.3 选择压力分析
对筛选后得到的TLR基因位点进行选择压力分析.首先,利用 samtools(https:/ /www. htslib. org. )对下载的73 种鸟类基因组进行目的序列提取;其次,利用MEGA-X(10.2.5)软件对序列进行比对;采用非同义突变速率和同义突变速率比值(ω =dN/dS)进行选择压力分析.其中ω >1 表示基因存在正向选择作用,ω =1 为中性选择,ω <1 为净化选择.
1.2.4TLR相关基因的系统树构建
由于TLR3、TLR7 和TLR21 在选择压力中出现选择差异,因此利用这三个基因进行系统进化关系分析.利用MEGA-X(10.2.5)软件对所有有效的核酸序列进行比对,选择建树模型为Tamura-Nei(TN),建树中采用Bootstrap 重复取样次数为1 000 次,接着采用最大似然法(Maximum likelihood)进行系统树的构建.系统树利用人、黑猩猩等相关序列作为外类群.
2 结果
2.1 TLR 基因数目分析
本实验中73 个物种均能有效预测到TLR基因,说明采用的BLAST 方法可靠、可行. 在鸟类基因组中,预测到的TLR基因0 ~4 个不等,我们筛选了具有代表性(TLR基因存在或者重复)的共33 个物种进行作图(图1).

图1 BLAST 预测的部分鸟类TLR 位点数目进化树中红色分支表示古颚总目下的物种,黄色表示鸡形目,绿色表示雁形目,蓝色表示雀形目.图中不同的色块表示真基因不同的数目.Fig.1 BLAST predicts the number of TLR sites for some birdsThe red branch in the evolutionary tree represents the species under the order Palaeognathus,yellow represents Galliformes,green represents Anseriformes,and blue represents passerines.The different color blocks in the heatmap represent the different numbers of true genes.
预测到的鸟类10 个TLR基因共可以分为以下三类:(1)TLR1A、TLR2A、TLR2B、TLR3、TLR4 和TLR5 中均出现少量基因多拷贝现象,其中TLR2A和TLR2B仅在非雀形目中出现多拷贝现象,TLR3 和TLR5 多拷贝现象仅出现在雀形目中. (2)TLR1B、TLR15 和TLR21 没有出现基因多拷贝现象,其中TLR21 基因在34 个物种中没有成功预测(有24 个物种未检测到TLR21 基因,其余则是不满足真基因的筛选标准).(3)有17 个物种出现TLR7 多拷贝现象(占所有物种的23.28%),雀形目的多拷贝现象(92.31%)和拷贝数目多于非雀形目(8.33%),并且雀形目内拷贝数目存在物种间差异.
2.2 选择压力分析
根据鸟类进化过程中的主要类群进行分类群选择压力分析,判断是否在不同的进化簇中存在不同的进化选择压力.由于雀形目是特别快速辐射的类群,因此我们又将其从新鸟小纲中分离出来.
结果表明,其中TLR1A、TLR5 和TLR15 在所有类群中均体现出净化选择;其他TLR基因在不同类群中存在差异的选择作用(见表3). 如,TLR1B在古鹗总目和雁形目中为正向选择,其余类群中为净化选择或中性选择;TLR2A仅在雀形目存在微弱的正向选择,其他类群均为净化选择;TLR2B仅在鸡形目中为正向选择;TLR3 在除古颚总目的其他类群中均为正向选择;TLR4 仅在古颚总目中为正向选择;TLR7 的正向选择发现在古颚总目(作用强烈)和鸡形目中(作用微弱);TLR21 在新鸟小纲中均体现出正向选择作用.

表3 不同类群中TLR 基因进化选择分析Table3 Selection of TLR gene in different evolutionary groups
2.3 序列进化系统树分析
由于TLR3、TLR7 和TLR21 在不同类群选择压力中出现差异,因此选择这三个基因进行系统进化关系分析(图2).总体来说,三个基因的系统进化关系基本上符合物种进化关系,物种均按照“目”进行聚类,相同目之间聚类的置信值基本均超过75%. TLR3 在多数物种中没有出现基因重复事件,并且系统进化关系符合鸟类各目的进化聚类(图2a),其出现的正向选择作用可能和其介导的病原体呈递选择有关.TLR7尽管在多数物种中存在基因重复事件,但在系统树中未出现明显的不同基因位点分枝(图2b),推测该基因经历了严重的纯化选择,同个物种的重复基因座之间的相似度很高.在TLR21 基因的进化树中,古颚总目形成了独特的一显著分枝,而其他鸟类形成另一显著分枝,两者之间距离较远(图2c).
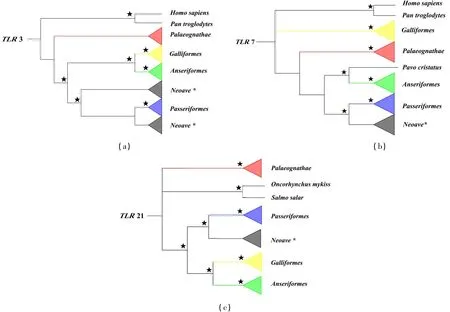
图2 TLR3(a)、TLR7(b)和TLR21(c)基因的系统进化关系分析★代表置信值≥75%.红色表示Palaeognathe(古颚总目);黄色表示Galliformes(鸡形目);绿色表示Anseriformes(雁形目);蓝色表示Passeriformes(雀形目);黑色表示Neoave*(新鸟小纲(除雀形目)).系统树中所采用的外类群为:TLR3(人XM_009 448543.3 和黑猩猩 NM_003265.3),TLR7(人 NM_016562.4 和黑猩猩 NM_001130133.2),TLR21(虹鳟 XM_021591169.2 和大西洋鲑鱼XM_014201342.2).Fig.2 Phylogenetic trees of TLR3(a),TLR7(b) and TLR 21(c) genes★represents the confidence value ≥ 75%.Red for Palaeognathe;yellow for Galliformes;green for Anseriformes;blue for Passeriformes (except passerine).The outgroups used in the phylogenetic tree were:TLR3 (Homo sapiens XM_009448543.3 and Pan troglodytes NM_003265.3),TLR7 (Homo sapiens NM_016562.4 and Pantroglodytes NM_001130133.2),TLR21 (Oncorhynchus mykiss XM_021591169.2 and Salmo salar XM_014201342.2).
3 讨论
TLR基因是一种在动植物中广泛存在的古老的免疫基因,具有保守的进化模式. 以往大多数的研究发现,TLR基因在哺乳动物进化上往往表现出净化选择的作用[16-17],这和我们的结果一致. 在本研究结果中,我们发现鸟类类群均存在不同程度的进化选择.
鸟类广泛分布在不同的环境中,如突胸总目、鸡形目等陆禽主要以陆地为生境,雁形目等水禽生活史中大量涉及水环境,雀形目大多数为林鸟. 外部环境和食物种类对病原微生物的多样性和组成起着关键作用[18].在长期的进化过程中,不同环境下的鸟类为了更好地适应其所在环境,免疫功能也随之进化,TLR基因可能存在不同的进化方向.
根据研究结果我们发现,古颚总目的TLR3、TLR4和TLR7 基因的选择作用和其他鸟类存在差异. 相较于其他鸟类翼长且有飞行的能力,古鹗总目的鸟类均翼短不能飞翔,仅能在地面行走,这使得古颚总目需要面对更加复杂多样化的环境和更多的病原微生物.根据研究表明,TLR3 基因主要识别双链RNA 病毒,TLR4 基因主要识别脂多糖类,TLR7 基因主要识别单链RNA 病毒[7].因此我们推测环境中差异的病原微生物丰度可能是古颚总目TLR基因的选择作用与其他鸟类存在差异的原因之一.雀形目为中、小型飞鸟,体型小于其他鸟类,多栖息于树木生长的环境中,食物多为杂食性,以昆虫、浆果等为食.研究结果发现雀形目的TLR2A基因选择作用与其他鸟类存在差异,TLR7 基因有明显的基因重复事件,TLR21 基因相较于其他鸟类存在更少的基因丢失情况.我们推测雀形目的这些TLR基因差异可能与其需要发挥特异的免疫作用有关.
在基因重复上,在TLR1A、TLR2A、TLR2B、TLR3、TLR4、TLR5 和TLR7 基因发现了基因拷贝现象. 其中TLR7 重复在雀类中较为常见,我们推测该基因重复事件发生在雀形目快速辐射之前,但在系统树中未体现出位点相关的进化枝,而TLR3 和TLR21 在系统树中未存在类群相关特征,推测可能为独立事件.此外,我们发现新鸟小纲中除了雀形目,其他大多数鸟类的TLR21 基因未能被预测到,我们猜测可能是因为基因组装配上出现了问题,从而导致未能成功预测到TLR21 基因位点.古颚总目的TLR21 基因在进化中形成了一个独特的进化分枝,而TLR21 基因在古颚总目中的缺失可能是因为其在进化中丢失了.
4 结论
本研究通过BLAST 在鸟类基因组中成功筛选出TLR基因家族,发现TLR基因的多拷贝现象在物种间存在差距,TLR7 基因是出现基因多拷贝现象最多的基因,且雀形目基因多拷贝数量明显多于非雀形目.在鸟类不同类群中,TLR基因的选择存在差异,尤其是古颚总目的基因进化和其他鸟类不同.在大多鸟类物种中,TLR3 基因和TLR21 基因表现出正向选择,形成适应性变化.推测鸟类TLR基因存在不同的进化选择机制,本研究结果为之后鸟类的TLR基因研究提供了一定基础.
