多粘类芽孢杆菌LY1 的全基因组测序及分析
2022-06-22聂俊辉张立新郭建军
聂俊辉,王 平,张立新,王 通,郭建军,曾 静,袁 林
(1.江西省科学院微生物研究所,江西 南昌 330096;2.上饶市农林水科学研究中心,江西 上饶 334000)
多粘类芽孢杆菌(Paenibacillus polymyxa)属芽孢杆菌科(Bacillaceae)类芽孢杆菌属(Paenibacillus),是一种产芽孢的革兰氏阳性菌[1]。多粘类芽孢杆菌在自然界中分布广泛,在玉米、高粱、小麦等多种植物体内外以及根际土壤中均有被发现,是一类重要的根际有益菌[2]。多粘类芽孢杆菌对植物生长发育的有益作用主要表现在促进植物产生激素、促进植物吸收养分、促进植物光合作用、增强植物固氮作用等方面[3];同时,多粘类芽孢杆菌还能通过分泌抗菌杀虫物质如抗菌肽、细菌素、脂肽等增强植物抵御病虫害的能力,是一种不可多得的生物防治原料,目前已广泛应用于农业生产领域,具有较高的经济价值[4-5]。
近年来,高通量测序技术快速发展,使得细菌基因组的高效解析成为现实,为细菌的功能及其作用机制研究提供了重要手段[6-7]。赵兴丽等[8]通过高通量测序技术发现土壤中有益微生物的含量在一定程度上可间接反映茶树的抗病性,有益微生物菌群所占比率与发病程度呈负相关。陈德纯等[9]对一株牦牛源产细菌素植物乳杆菌的基因组进行测序,发现其基因组中包括多个细菌素基因以及细菌素转运系统的功能序列,为该菌株的应用研究提供了重要的参考数据。高通量测序技术的普及有效地促进了微生物的研究。
目前,有关多粘类芽孢杆菌的研究多集中在其生物特性及其对植物与生物防治的作用机理上,而对其基因序列的分析研究报道较少[10-12]。笔者从土壤中分离出一株多粘类芽孢杆菌LY1,利用基因组测序技术全面解析LY1 菌株的基因组序列,再通过生物信息学的方法比较不同菌株基因间的差异性和相似性,探索其基因结构特征,明确其基因功能,从全基因组水平了解其系统发育进化关系,以期为全面了解多粘类芽孢杆菌的遗传背景,实现多粘类芽孢杆菌的遗传改造提供基本线索。
1 材料与方法
1.1 供试菌株
多粘类芽孢杆菌LY1 从土壤中分离,由江西省科学院微生物研究所分离保存。
1.2 试验方法
1.2.1 测序及数据质控取菌体委托上海生工生物工程股份有限公司使用Illumina Hiseq platform 进行基因组测序。Illumina Hiseq™得到的原始图像数据文件经CASAVA 碱基识别 (Base Calling)分析转化为原始测序序列(Sequenced Reads),对原始数据质量值等信息进行统计,并使用FastQC 对样本的测序数据质量进行可视化评估。测序得到的原始数据,里面含有带接头的、低质量的序列。为了保证信息分析质量,必须对原始数据进行过滤,得到Clean 数据。随机从Clean 数据中抽取10 000 条序列与NCBI NT 数据库进行blastn 比对,取evalue ≤1E-10 并且相似度>90%、coverage >80%的比对结果,计算其物种分布,同时进行污染检测。
1.2.2 数据拼装使用SPAdes 对二代测序数据进行拼装。SPAdes 首先会对原始序列进行序列错误校正,然后通过多Kmer 值进行组装,最终综合各Kmer 值组装结果获得最佳结果。再采用GapFiller 对拼接得到的contig 进行Gap 修补,最后采用PrInSeS-G 进行序列矫正,修正拼接过程中的剪辑错误以及小片段的插入缺失。
1.2.3 基因预测与注释采用Prokka 对组装结果进行基因元件预测。Prokka 是一系列基因元件预测工具的集合,调用Prodigal 预测编码基因,Aragorn 预测tRNA,RNAmmer 预测rRNA,Infernal 预测miscRNA,预测出的各类基因元件汇总并完成初步注释。采用Repeat Modeler 对组装结果进行重复序列Denovo 预测,再利用RepeatMasker 寻找基因组区段上各类型重复序列出现的位置和频率。NCBI Blast+用于CDD、KOG、COG、NR、NT、PFAM、Swissprot、TrEMBL注释。基于Swissprot 和TrEMBL 的蛋白注释结果根据Uniprot 的注释信息得到GO 注释。使用KAAS,KEGG Automatic Annotation Server 进行KEGG 注释。
1.2.4 系统发育树构建将基因预测得到的16S rRNA序列与NCBI 的16S 数据库进行Blast 比对,设置参数identify >95。然后选取identify 最高的前30 条16S rRNA序列,利用muscle软件进行序列多重比对后,采用FastTree 软件构建系统发育树。
2 结果与分析
2.1 测序数据质控
二代测序和三代测序原始数据经过去除带接头的、低质量的序列,过滤后得到Clean 数据。Illumina Hiseq 原始测序数据过滤处理后,总共得到6 495 188个Clean Reads,总碱基数为951 905 276 bp,平均Read长度为146.56 bp;其中,Q10、Q20、Q30 的比例分别为100.00%(碱基数951 903 300 bp)、98.18%(碱基数934 538 164 bp)、93.79%(碱基数892 808 488 bp);GC含量为45.63%(碱基数434 383 207 bp)。PacBio RSII原始测序数据过滤处理后,总共得到333 829 个Clean Reads,总碱基计数为925 726 342 bp,平均Reads 长度2 773.06 bp;其中,Reads ≥1 000 bp 、≥5 000 bp和≥10 000 bp 的分别占比47.34%、11.45%和5.30%,而Bases in Reads ≥1 000 bp、≥5 000 bp 和≥10 000 bp 的分别占比90.25%、61.52%和46.25%;GC 含量为44.40%。综上所述,2 组数据质量较高,可用于下一步基因组组装。
2.2 基因预测与基因组基本特征
2.2.1 基因组的组成与信息2 组测序数据经过序列矫正、Gap 修补、剪辑错误修复、拼装等程序后获得菌株的基因组信息,如图1 所示,多粘类芽孢杆菌LY1 的基因组为一条环状闭合DNA,大小 5 765 474 bp,共预测到6 086 个蛋白质编码基因、162 个 tRNA基因、42 个rRNA 基因、74 个small RNA ;最短基因长度为70 bp,最长基因长度为42 252 bp,基因平均长度为889.05 bp,长度≥500 bp 的基因有4 107 个,长度≥1 000 bp 的基因有1 916 个;重复区域计数为216 个,重复比例为1.45%;Low_complexity 为25 个,Simple_repeat 为117 个。基因长度和GC 含量的分布如图2 所示。
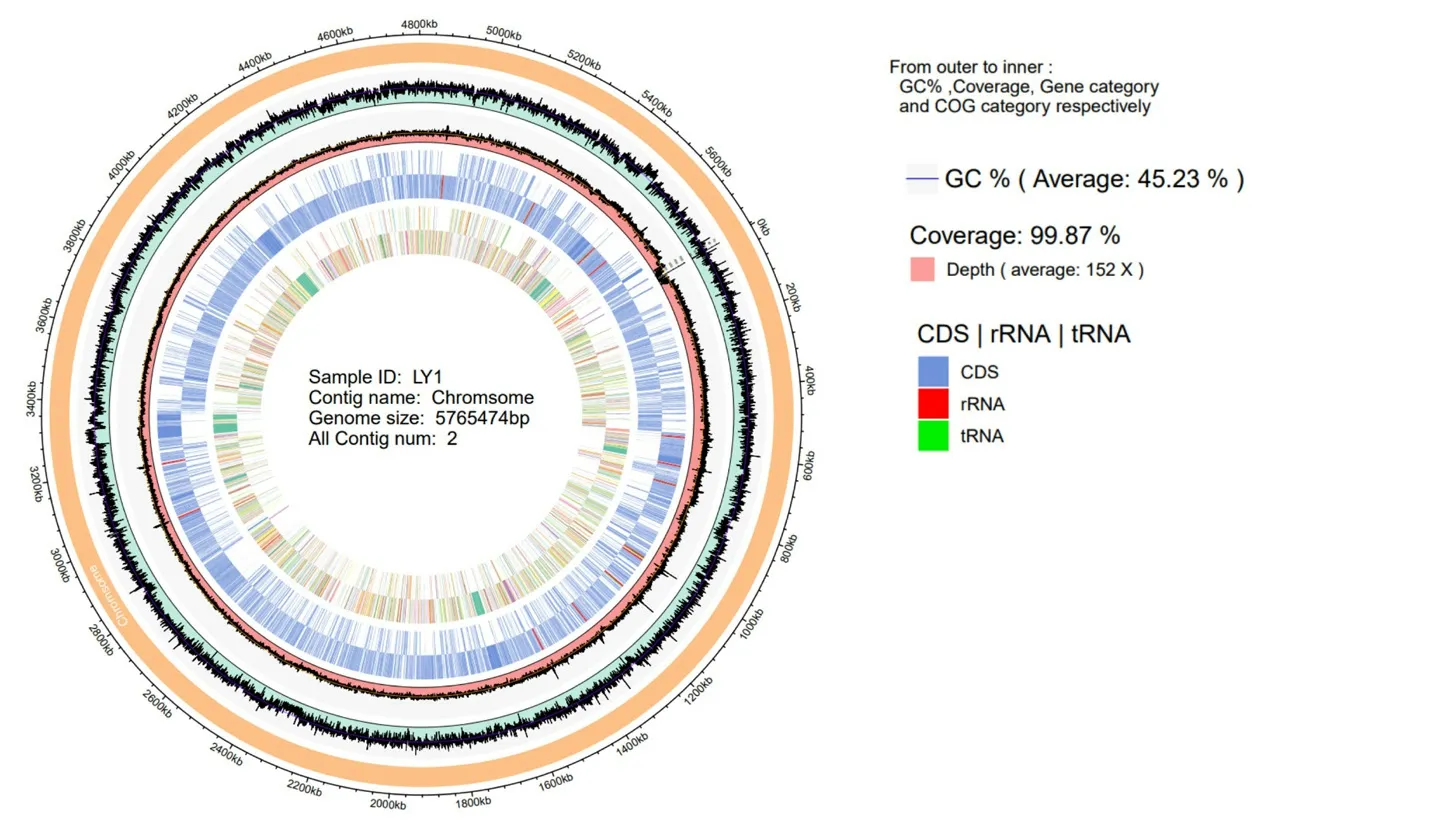
图1 多粘类芽孢杆菌LY1 的基因组图
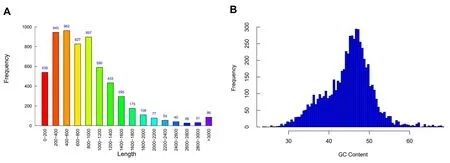
图2 多粘类芽孢杆菌LY1 的基因长度(A)和GC 含量分布(B)
2.2.2 基因组注释将预测的编码基因与多个数据库进行比对,注释到CDD(Conserved Domain Database)、KOG(euKaryotic Ortholog Groups)、NR(NCBI nonredundant protein sequences)、PFAM(Protein family)、Swissprot(A manually annotated and reviewed protein sequence database)、TrEMBL、GO(Gene Ontology)、KEGG(Kyoto Encyclopedia of Genes and Genomes)的基因比例分别为72.92%、64.27%、98.14%、69.57%、66.32%、97.52%、66.66%和37.92%,unigenes 总共5 813 个,同时注释到NR、KEGG、Swissprot、COG的基因有2 133 个,注释到所有数据库的基因共有2 066 个,占unigenes 的35.54%。
2.2.3 NR 数据库注释由上面的数据可知,注释到NR 数据库的基因最为丰富,共有5 705 个,占比98.14%。NR 数据库是一个非冗余的蛋白质数据库,内容全面,通过与NR 数据库的对比,可以查看物种转录本序列与相近物种的近似情况以及同源序列的功能信息[13]。由图3 可知,鉴定到LY1 菌株有3 792 个基因与多粘类芽孢杆菌同源,与Paenibacillus polymyxaM1 菌株的相似性相对较高,在M1 的编码基因中注释到318 个基因,而在Paenibacillus polymyxaSQR-21、Paenibacillus polymyxaSC2 编 码 基因中则分别注释到76 和29 个基因。
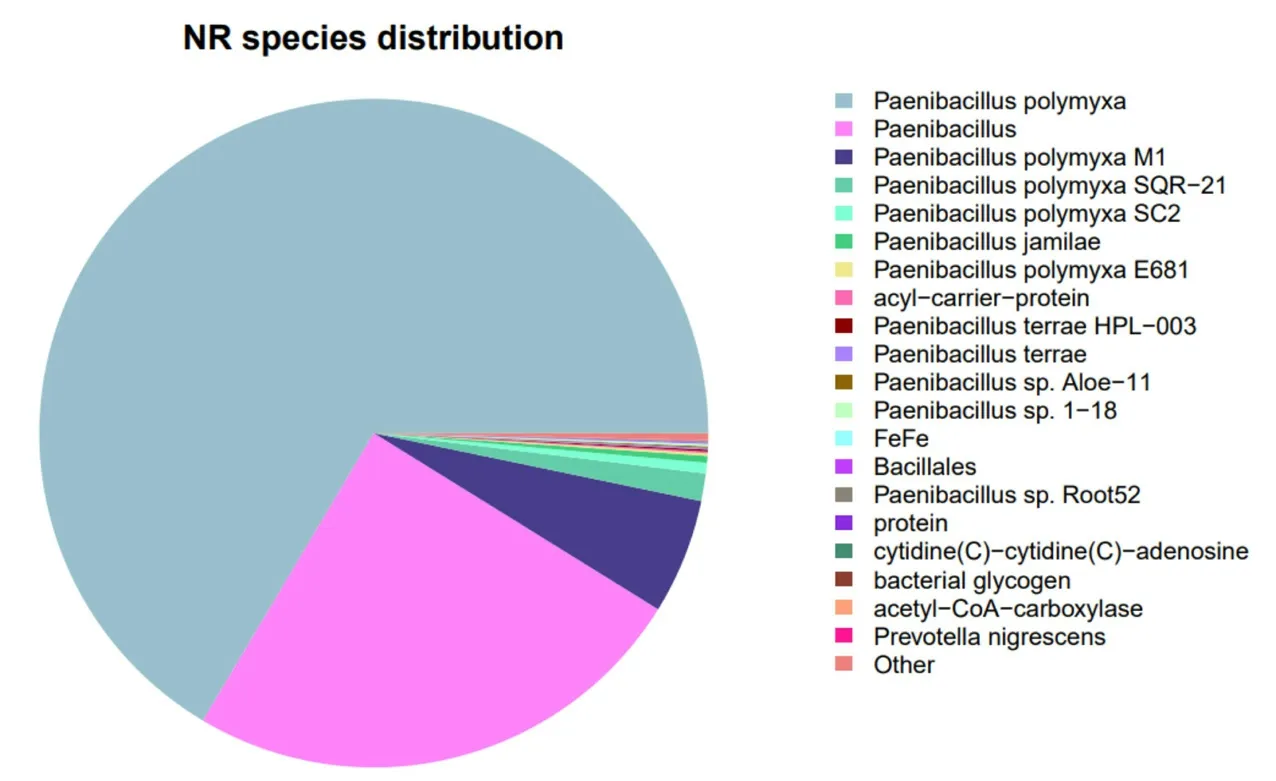
图3 多粘类芽孢杆菌LY1 基因的NR 数据库比对
2.3 基因功能注释
2.3.1 GO 功能注释GO (Gene Ontology) 是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表(controlled vocabulary)来全面描述生物体中基因和基因产物的属性[14]。GO 总共有3 个ontology,分别描述基因的分子功能(MF,molecular function)、细胞组分(CC,cellular component)、参与的生物过程(BP,biological process)[15]。通过将基因进行GO 注释和分类,可判断基因参与的主要功能。多粘类芽孢杆菌LY1 基因的3 大分类统计结果如图4 所示。在生物过程中,分类到代谢过程(metabolic process)和细胞过程(cellular process)中的基因占比最多,分别为2 370(40.77%)和2 266 个(38.98%);在细胞组分分类中,分类到细胞(cell)和细胞组成(cell part)中的基因占比最多,均为2 045 个(35.18%);在分子功能分类中,分类到催化活性(catalytic activity)和结合(binding)中的基因占比最多,分别为2 345(40.34%)和2 080 个(35.78%)。
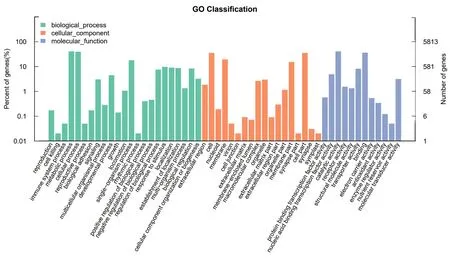
图4 多粘类芽孢杆菌LY1 的GO 功能注释
2.3.2 KEGG 注释KEGG(Kyoto Encyclopedia of Genes and Genomes,京都基因与基因组百科全书)是基因组破译方面的数据库,在给出染色体中一套完整基因的情况下,它可以对蛋白质交互网络在各种细胞活动起的作用作出预测,方便地寻找与行使某一类功能相关的所有注释上的基因[16-17]。由图5 可知,LY1 菌株的基因通过KEGG 分类主要分为5 大类,分别为细胞过程(Cellular Processes)、环境信息处理(Environmental Information Processing)、遗传信息处理(Genetic Information Processing)、代谢(Metabolism)和有机系统(Organismal Systems),其中注释到代谢的相关基因最多,达2 255 个,注释到细胞过程、环境信息处理、遗传信息处理和有机系统的基因数分别为99、613、334 和31 个。
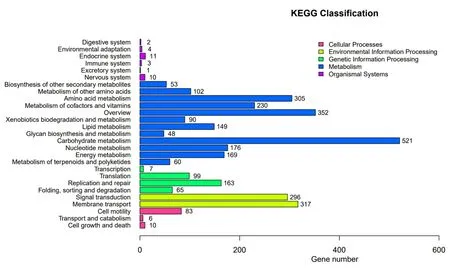
图5 多粘类芽孢杆菌LY1 的KEGG 功能注释
2.3.3 COG 注释蛋白质直系同源簇数据库(COG,Cluster of Orthologous Groups of Prot-eins)是用于同源蛋白注释的数据库,是将细菌、藻类和真核生物等完整基因组的编码蛋白根据系统进化关系构建而成,蛋白编码序列与之比对后可以预测蛋白的功能[18-19]。多粘类芽孢杆菌LY1 的COG 功能注释结果如图6 所示,注释到碳水化合物转运及代谢(Carbohydrate transport and metabolism)和转录(Transcription)分类的基因最多,分别为433(11.59%)和398 个(10.65%)。
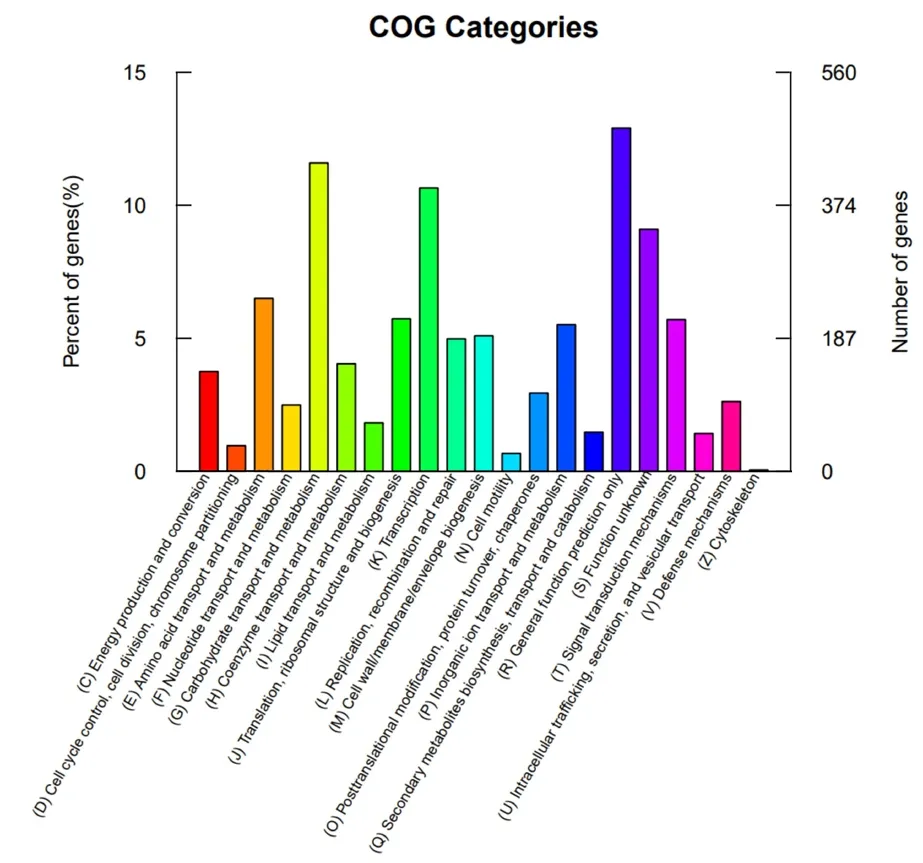
图6 多粘类芽孢杆菌LY1 的COG 功能注释
2.4 毒力因子分析
把菌株LY1 的基因蛋白序列在VFDB 致病因子数据库(Virulence Factors of Pathogenic Bacteria)中进行比对,将基因与其对应的毒力因子(Virulence Factors,VF)功能注释信息相结合,发现注释到SetB 组(full dataset,共30 053 个与毒力因子相关的基因)的蛋白序列共326 个,占比5.61%;注释到SetA 组(core dataset,共2 585 个与毒力因子相关的基因)的蛋白序列共301 个,占比5.18%(表1)。

表1 多粘类芽孢杆菌LY1 的基因组基本特征
2.5 系统发育进化分析
16S rRNA 基因是细菌上编码rRNA 相对应的DNA 序列,存在于所有细菌的基因组中,具有高度的保守性和特异性,是病原菌检测和鉴定的一种有效参照[20-21]。如图7 所示,LY1 菌株与Paenibacillus polymyxaM1、Paenibacillus polymyxaOSY-DF、Paenibacillus polymyxaSC2、Paenibacillus peoriae、Paenibacillus jamilae、Paenibacillus polymyxaE681 等菌株的距离较近。
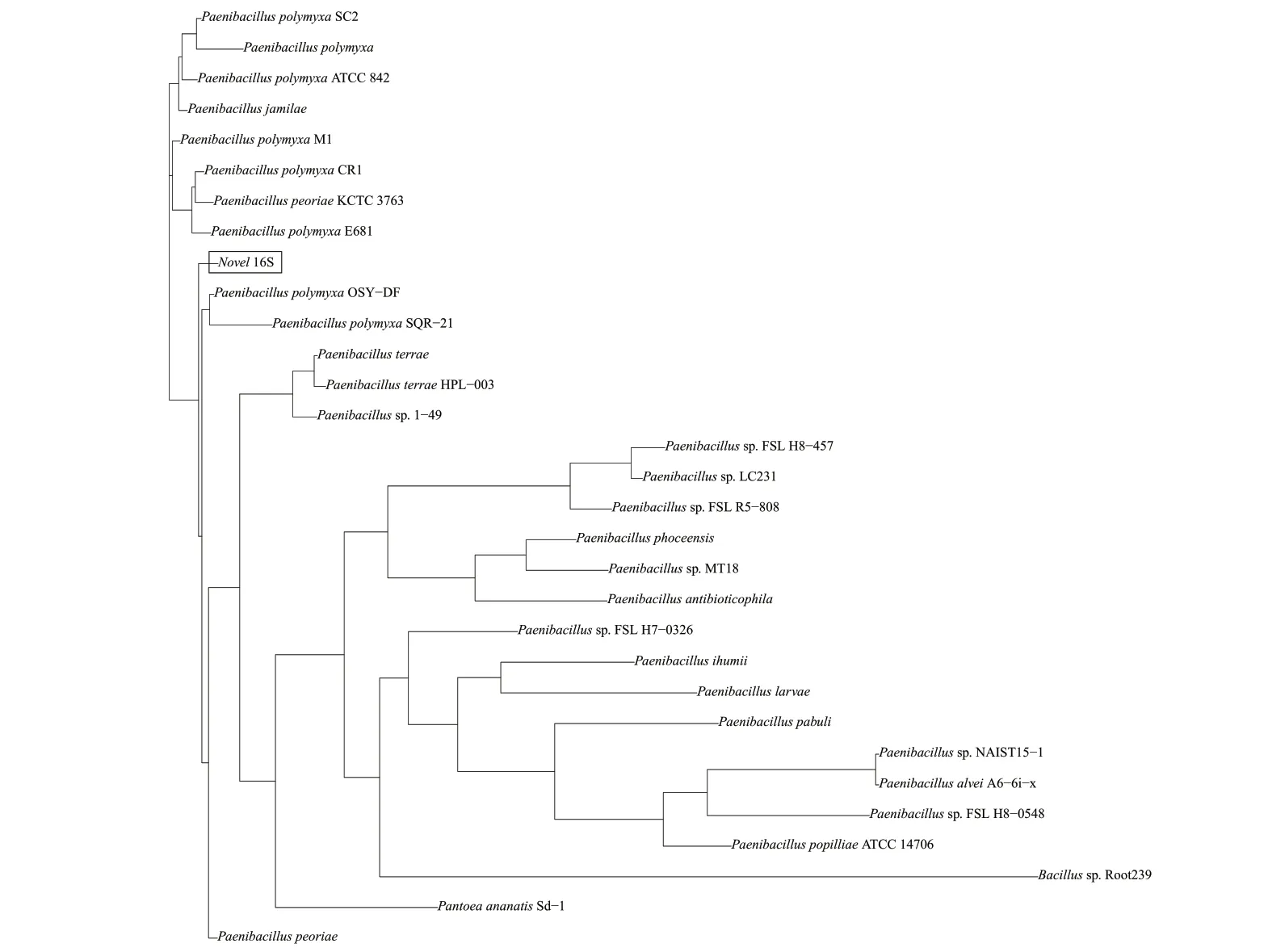
图7 菌株LY1 基于16S rRNA 的系统发育树
3 小 结
目前,人们对多粘类芽孢杆菌的功能有了较为全面地了解,但其分子作用机制以及遗传工程改造等方面的研究报道还不多[3]。该研究通过全基因组测序分析了多粘类芽孢杆菌LY1 的基因组序列,获得其基因组基本特征、基因功能注释及分类、系统发育进化关系等关键信息,为其后续的功能挖掘、遗传操作系统建立、基因工程改造等提供了研究基础与前提条件。结果表明,多粘类芽孢杆菌LY1 的基因组长度为5 765 474 bp,共有6 086 个蛋白质编码基因、162 个 tRNA 基因、42 个rRNA 基因,GC 含量约为45.23%;在GO 功能注释结果中,在生物过程分类下,代谢过程和细胞过程中的基因占比最多,分别占比40.77%和38.98%;在细胞组分分类下,分类到细胞和细胞组成中的基因占比最多,为35.18%;在分子功能分类下,分类到催化活性和结合中的基因占比最多,分别为40.34%和35.78%;系统发育进化分析显示,菌株LY1 与Paenibacillus polymyxaM1、Paenibacillus polymyxaOSY-DF、Paenibacillus polymyxaSC2、Paenibacillus polymyxaE681 等菌株进化距离较近。同时,根据多粘类芽孢杆菌LY1 的基因组信息及发育进化信息,笔者认为有望将其开发成新型生物防治制剂和农业肥料添加剂等在农业生产领域广泛应用。
