茶树CLH基因家族的鉴定与转录调控研究及其在白化茶树中的表达分析
2022-06-21王涛王艺清漆思雨周喆陈志丹孙威江
王涛,王艺清,漆思雨,周喆,陈志丹,孙威江*
茶树CLH基因家族的鉴定与转录调控研究及其在白化茶树中的表达分析
王涛1,3,4,5,王艺清1,3,4,5,漆思雨1,3,4,5,周喆1,3,4,5,陈志丹2,3,4,5*,孙威江1,3,4,5*
1. 福建农林大学园艺学院,福建 福州 350002;2. 福建农林大学安溪茶学院,福建 泉州 362400;3. 福建省茶产业工程技术研究中心,福建 福州 350002;4. 海峡两岸特色作物安全生产省部共建协同创新中心,福建 福州 350002;5. 福建省茶产业技术开发基地,福建 福州 350002
叶绿素酶(Chlorophyllase,CLH)是叶绿素降解过程中的关键酶,将叶绿素a脱去植醇,形成脱植基叶绿素a。以白化茶树白鸡冠新梢叶片为材料,克隆获得3条基因cDNA全长序列,并进行生物信息学分析。结果表明,3条基因分布于2个亚家族,其蛋白质编码区(Coding sequence,CDs)长度为894~975 bp,编码氨基酸个数为297~324,蛋白质分子量为31.99~34.91 kDa,等电点为4.89~7.61,不稳定系数为38.94~48.24,其中CsCLH1.1和CsCLH1.2为不稳定蛋白,CsCLH2为稳定蛋白。Cell-PLoc亚细胞定位预测结果表明,3个CsCLHs蛋白均定位于叶绿体;而WolfPsort亚细胞定位预测结果显示,CsCLH1.1和CsCLH1.2定位于细胞质,CsCLH2定位于叶绿体。遮阴和恢复光照处理下的qRT-PCR结果显示,遮阴抑制白鸡冠叶片的表达,光照诱导白鸡冠叶片的表达。不同品种中表达模式分析表明,在白化叶中高表达。另外,酵母单杂交结果表明,可以与和启动子结合。综上所述,CsCLHs在白化茶树叶片中可能参与叶绿素降解,在叶片白化过程中发挥重要作用,结果可为进一步探究茶树CLH基因家族的功能及茶树叶片白化机理提供参考。
茶树;叶绿素酶;基因家族;表达差异
叶片白化是绿色植物叶绿素缺陷出现的表型[1-3],植物白化通常会造成减产和品质下降[4-5],但在白化茶树的叶片中有氨基酸含量高,儿茶素、咖啡碱含量低的特点[6-7]。茶树白化类型可划分为光照敏感型和温度敏感型,光照敏感型白化茶树在强光下芽叶呈现黄白色,在遮阴处理后芽叶转绿,同时叶绿素含量显著上升[8-10],但恢复光照后,叶片叶绿素含量显著下降[11],且显著低于绿叶茶树品种[12-13]。研究表明,茶树叶绿素合成途径关键基因的低表达与叶绿素降解途径关键基因的高表达是造成茶树芽叶白化的主要原因[11,14-15]。
叶绿素酶(Chlorophyllase,CLH)作为叶绿素降解途径中的限速酶,参与叶绿素降解途径的第一步,将叶绿素a脱去植醇(Phytol)[16]。研究表明,在花色叶片中,非绿色部分的叶绿素酶活性高于绿色部分[17],在叶片花色由浅至深的转化过程中,叶绿素酶的活性也逐渐减小,表明叶绿素的含量与叶绿素酶活性紧密关联[18]。CLH基因在拟南芥()[19]、草地早熟禾()[20]等植物中被相继克隆,但前人对植物CLH的研究多集中于植物衰老和贮藏方面,目前关于CLH在白化茶树中的作用机制尚无深入研究。
白化茶树是优异、特异种质资源,研究其叶绿素降解过程中的分子机制,对于揭示白化茶树体内的叶色调控及叶片发育调控机理等有重要的理论价值,同时在茶树良种选育方面有重要的应用价值。因此,本研究以光照敏感型白化茶树白鸡冠为材料,克隆了3条的cDNA全长序列,并对其进行生物信息学分析和表达分析,初步研究遮阴与恢复光照处理下的表达差异,以及在不同白化茶树品种间的表达模式,为深入研究茶树白化的分子机制提供试验基础。
1 材料与方法
1.1 材料处理
以武夷星种质资源圃内的白鸡冠为试验材料,选取白鸡冠茶树白化表型最显著的第二叶,用锡箔纸进行遮阴处理,处理于2018年9月进行。遮阴方法如下:取处理前未遮阴样品为对照(NS),分别于遮阴处理1 d和3 d时取样,依次标记为S1和S3;遮阴3 d取样后去除锡箔纸开始恢复光照,分别在恢复光照1 d和3 d时取样,标记为RL1和RL3。取样标准为叶片生长发育状况和形状大小相似,所取样品用液氮速冻后保存于–80℃冰箱,用于提取RNA。所有样品均设置3个重复,由北京百迈客生物科技有限公司进行转录组测序。
以白鸡冠、黄化品系、黄金芽、金光、千年雪、御金香、中黄1号和金牡丹为试验材料,分别取其一芽二叶新梢,于液氮中速冻,用于不同白化品种中的表达量分析。并以铁观音为试验材料,分别取其嫩叶、成熟叶、老叶、嫩茎、老茎、花苞、果实和根等组织器官,于液氮中速冻,用于的组织表达模式分析。
1.2 茶树CsCLHs基因家族成员鉴定
在拟南芥数据库TAIR(www.arabidopsis.org/index.jsp)下载拟南芥AtCLH1、AtCLH2的氨基酸序列,从TPIA数据库下载茶树基因组数据。用已鉴定的拟南芥AtCLH1、AtCLH2氨基酸序列作为种子序列,在TPIA上进行BLASTP对茶树基因组进行序列比对搜索(E-value≤10-5),得到茶树CsCLHs成员氨基酸序列,同时通过在线数据库Pfam(pfam.xfam.org)下载Chlorophyllase结构域文件,通过SPDE软件[21]HMMER Search功能进行比对,将比对出的序列通过NCBI CDD(www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)验证候选蛋白结构域,以确定其含有Chlorophyllase(PF07224)完整保守结构域。
1.3 茶树CsCLHs基因的克隆及测序
采用RNAprep Pure多糖多酚植物总RNA提取试剂盒[天根生化科技(北京)有限公司]提取白鸡冠叶片总RNA,采用Nano Drop 2000c超微量分光光度计(美国Thermo Fisher公司)测定RNA浓度,2%琼脂糖凝胶电泳检测所得RNA的完整性。根据Promega goscriptTMreverse transcription system试剂盒说明书步骤进行cDNA反转录。利用Primer Premier 5软件设计各个基因CDs区全长特异性引物(表1)。使用GeneStar公司的2×SuperStar Plus PCR Mix对各基因的CDs区全长进行扩增,对目的片段进行纯化并连接至pEASY-Blunt Cloning载体(北京全式金生物技术有限公司),经菌落PCR验证后送至生工生物工程(上海)股份有限公司测序。
1.4 茶树CsCLHs蛋白质结构预测
通过ProtParam网站(web.expasy.org/protparam)对CsCLHs蛋白分子量大小、理论等电点等理化特征进行分析预测;通过Cell-PLoc 2.0(www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2)和Wolfpsort(wolfpsort.hgc.jp)对CsCLHs编码的蛋白进行亚细胞定位预测;使用在线工具SignalP 5.0(www.cbs.dtu.dk/services/SignalP)对CsCLHs各蛋白的信号肽进行预测。通过TMHMM Server v.2.0(services.healthtech.dtu.dk/service.php?TMHMM-2.0)工具对CsCLHs蛋白的跨膜结构域进行预测;通过SOPMA(https://npsaprabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在线预测CsCLHs亚家族蛋白的二级结构,使用Phyre2(www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)工具预测各蛋白的三级结构模型,并通过Ramachandran plot(http://mordred.bioc.cam.ac.uk/~rapper/rampage.php)对所得蛋白的三级结构模型进行评估。
1.5 茶树CsCLHs基因家族进化树构建
利用NCBI BLASTP(www.ncbi.nlm.nih.gov)下载已经报道的各植物CLHs蛋白序列,并通过MEGA 7.0软件与茶树CsCLHs氨基酸序列进行Clustal W多序列比对,多序列比对参数为默认值。随后使用近邻相接法(Neighbor-Joining)构建系统发生树,其参数均为默认值。
1.6 茶树CsCLHs基因家族保守基序与结构预测
利用Pfam在线数据库(pfam.xfam.org)和NCBI CDD对茶树CsCLHs家族蛋白结构进行预测。使用在线工具MEME(meme-suite.org/tools/meme)分别对茶树CsCLHs氨基酸序列保守基序进行预测分析,基序数量设置为10个,其余参数均为默认值。从TPIA数据库下载茶树基因结构注释文件gff3,通过TBtools软件[22]对CsCLHs系统进化树、基序分布以及内含子、外显子分布进行可视化。
1.7 茶树CsCLHs基因家族顺式作用元件预测
在TPIA数据库中下载茶树基因组,通过TBtools软件提取家族成员起始密码子ATG上游2 000 bp序列,利用启动子在线预测软件PlantCARE(bioinformatics.psb.ugent.be/webtools/plantcare/html)进行顺式作用元件预测,利用TBtools进行顺式作用元件可视化处理。
1.8 茶树CsCLHs上游调控转录因子预测及表达量测定
利用PlantTFDB数据库(http://plantregmap.gao-lab.org/index.php)中Regulation prediction功能,输入序列为ATG上游2 000 bp启动子区域,以拟南芥为参考,预测上游调控转录因子,并通过TPIA数据库BLASTP获得拟南芥同源基因的茶树基因序列号,基于转录组数据中FPKM值为参考,分析可调控转录因子在遮阴及恢复光照条件下的表达模式。
基于基因组和转录组数据,选取CSS0007067(BPC6)和CSS002729(CDF5)进行酵母单杂交验证,根据和启动子序列与BPC6和CDF5编码区序列设计引物进行PCR扩增(表2)。对pAbAi载体和pGADT7载体分别进行双酶切,采用ClonExpress Ⅱ One Step Cloning Kit(南京诺唯赞生物科技股份有限公司)构建pAbAi-Pro、pAbAi-Pro、pGADT7-BPC6和pGADT7-CDF5重组载体。将重组载体通过PGE/LiAc法转化至酵母Y1H感受态中。为筛选出具有抑制Y1H(Pro)和Y1H(Pro)生长的AbA(金担子素A)浓度,以pAbAi-P53为阳性对照,空载Y1H为阴性对照,分别在含有AbA(25、50、150、300、500 ng·mL-1)的SD/-URA培养基上培养3~5 d,观察是否有菌落出现。确定AbA抑制浓度后,将pGADT7-BPC6和pGADT7-CDF5分别转入Y1H(Pro)和Y1H(Pro)感受态中,得到酵母重组菌株Y1H(Pro+BPC6)、Y1H(Pro+CDF5)、Y1H(Pro+BPC6)和Y1H(Pro+CDF5),在SD/-Leu和SD/-Leu/AbA150培养基上培养3~5 d,以Y1H(Pro)和Y1H(Pro)菌株为阴性对照,观察是否有菌落出现。
1.9 茶树CsCLHs基因家族表达分析

表2 酵母单杂交试验所用引物
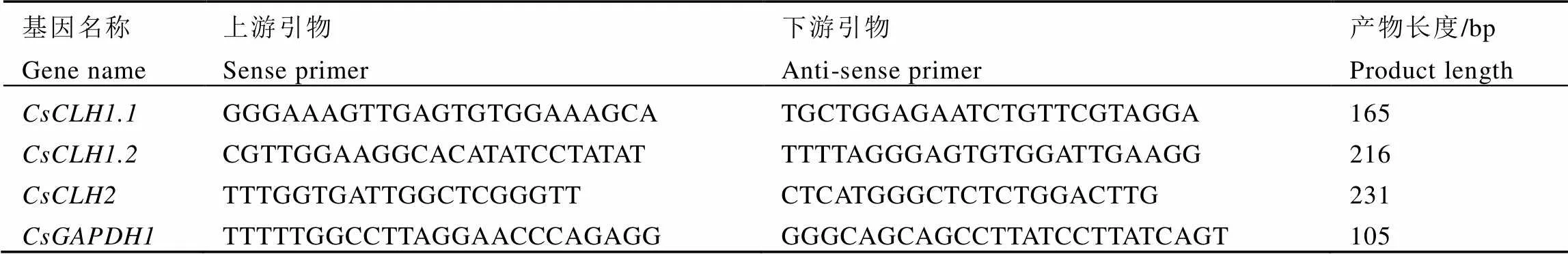
表3 实时荧光定量PCR引物序列信息
2 结果与分析
2.1 茶树CsCLHs基因家族成员的鉴定与克隆
根据拟南芥AtCLH1和AtCLH2的蛋白序列,在茶树基因组数据库中得到5条茶树基因,通过NCBI CDD蛋白质结构域在线预测网站对所获得的蛋白质序列进行分析,只有3条蛋白序列包含完整的叶绿素酶结构域。将茶树CLHs与葡萄、拟南芥等植物CLHs的蛋白质序列使用Clustal W进行多重比对,构建系统进化树。结果显示(图1),茶树与葡萄、拟南芥等植物CLHs聚类后可分为3组,其中CSS004684和CSS0033432与葡萄等CLH1聚为一类,CSS0028283与葡萄、拟南芥等CLH2聚为一类。根据进化树聚类分析与在茶树染色体的定位(图2),将CSS004684、CSS0033432和CSS0028283分别命名为、和。以上述基因的基因组序列为参考,以白鸡冠新梢叶片的cDNA为模板,通过RT-PCR的方式克隆得到CsCLHs家族的3条基因(图3)。测序结果表明,茶树中3条的CDs区长度为894~975 bp。
2.2 茶树CsCLHs家族蛋白质特征分析
CsCLHs家族的氨基酸长度差异较小,在297~324个氨基酸(表4),蛋白质分子量为31.99~34.91 kDa,等电点为4.89~7.61,不稳定系数为38.94~48.24,其中CsCLH1.1和CsCLH1.2为不稳定蛋白,CsCLH2为稳定蛋白,CsCLHs蛋白脂肪族氨基酸含量较高,GRAVY值在–0.001~0.200,其中CsCLH1.1和CsCLH1.2为疏水性蛋白,CsCLH2为亲水性蛋白。信号肽预测表明,CsCLHs均不含有信号肽。TMHMM跨膜结构分析表明,CsCLHs均不含有跨膜结构(表5)。
2.3 茶树CsCLHs基因结构、染色体定位与保守基序分析
通过MEME在线工具对CsCLHs蛋白的10个保守基序进行预测分析,在CsCLHs各蛋白中基序3、基序2、基序4、基序1和基序5的排列顺序较一致,CsCLH2缺少基序8(图4-A),因此推测CsCLH1s和CsCLH2在叶绿体中所发挥的作用存在一定差异。通过NCBI CDD在线数据库分析发现,茶树CsCLHs蛋白都含有Chlorophyllase结构域(PLN00021),该结构域属于cl21492/水解酶超家族(图4-B)。CsCLH1.1、CsCLH1.2和CsCLH2蛋白成员的Chlorophyllase结构域分别分布在第1~300个氨基酸、第1~297个氨基酸和第21~324个氨基酸,各蛋白的保守结构域相对保守。外显子-内含子结构分析表明,家族成员的外显子和内含子数量存在差异,外显子数量2~6个,无内含子(图4-C)。
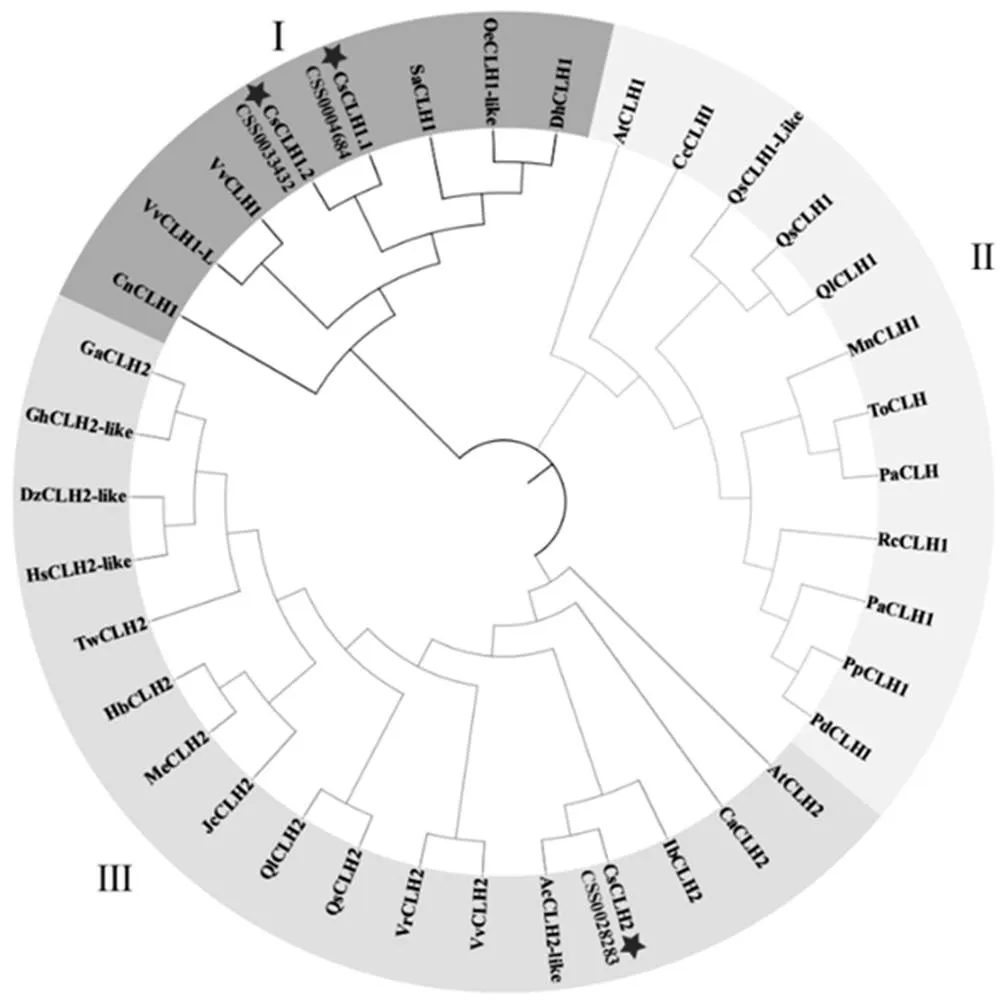
注:茶树,CsCLH1.1,CsCLH1.2,CsCLH2;拟南芥,AtCLH1(NP_564094.1),AtCLH2(NP_199199.1);山谷栎,QlCLH1(XP_030946150.1),QlCLH2(XP_030924067.1);东方苔草,ToCLH(PON83887.1);欧洲栓皮栎,QsCLH1(XP_023917005.1),QsCLH1-like(XP_023873288.1),QsCLH2(XP_023900495.1);桃,PpCLH1(XP_007200272.1);扁桃,PdCLH1(XP_034228050.1);欧洲甜樱桃,PaCLH1(XP_021821960.1);山黄麻,PaCLH(PON76085.1);月季,RcCLH1(XP_024162290.1);大麻,CsCLH(XP_030509823.1);桑,MnCLH(XP_010096444.1);海枣,PdCLH1(XP_008787534.2);旋蒴苣苔,DhCLH1(KZV50452.1);独角金,SaCLH1(GER41106.1);木豆,CcCLH1(XP_020203352.1);椰子,CnCLH1(KAG1361310.1);葡萄,VvCLH1(RVX20904.1),VvCLH2(RVX22292.1);河岸葡萄,VrCLH1-like(XP_034690185.1),VrCLH2(XP_034684712.1);木樨榄,OeCLH1-like(XP_022879035.1);猕猴桃AcCLH2-like(PSS01417.1);橡胶,HbCLH2(XP_021691950.1);木薯,MeCLH2(XP_021608126.1);麻风树,JcCLH2(XP_012069695.1);雷公藤,TwCLH2(XP_038705126.1);陆地棉,GhCLH2-like(XP_016699405.2);澳洲棉,GaCLH2(XP_016699405.2);甘薯,IbCLH2(AWK91225.1);小果咖啡,CaCLH2(XP_027116339.1)
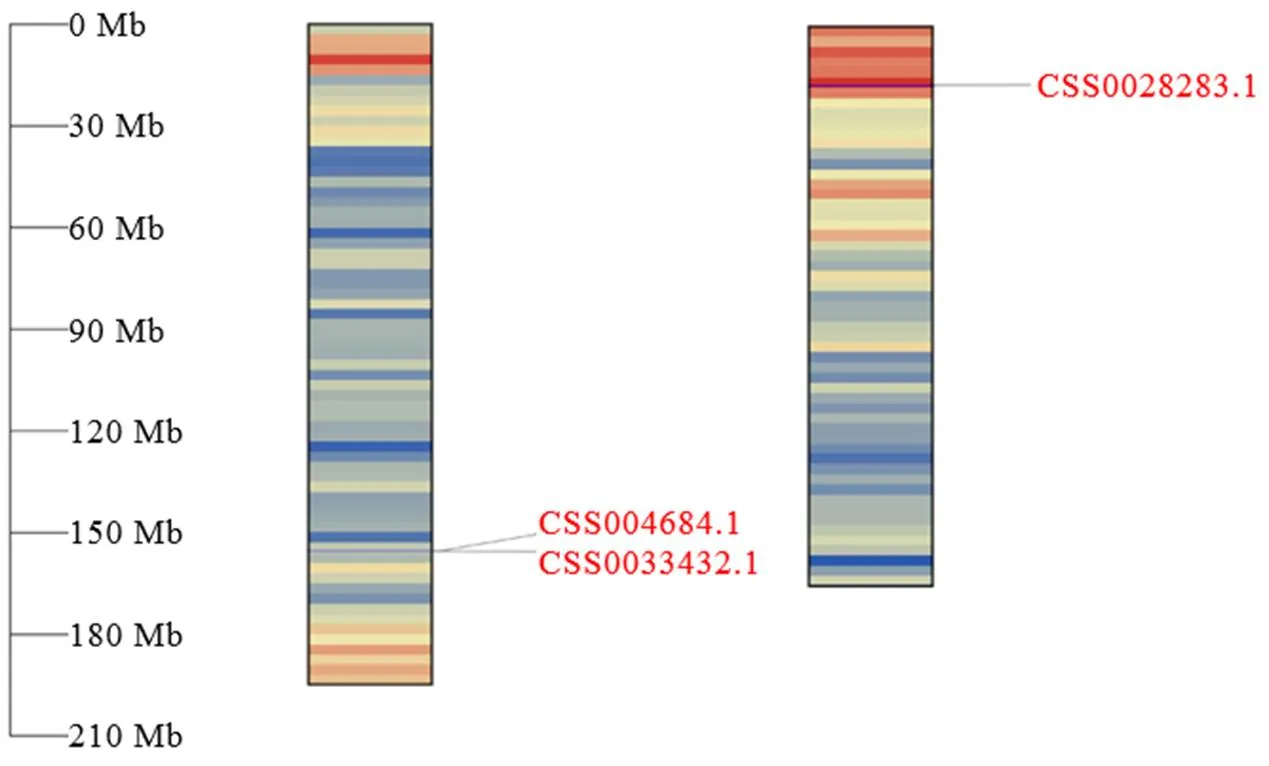
图2 CsCLHs家族基因在茶树染色体分布图

注:M:2 000 bp DNA marker

表4 CsCLHs鉴定及亚细胞定位
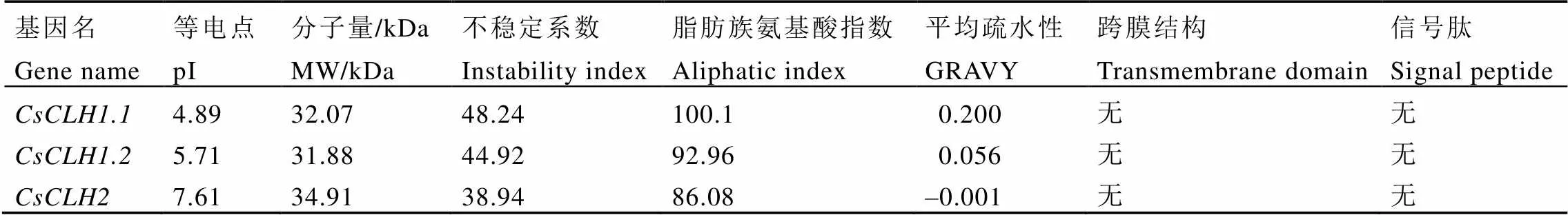
表5 CsCLHs理化性质
2.4 茶树CsCLHs蛋白二级、三级结构分析
CsCLHs蛋白的二级结构预测结果表明(表6),3个蛋白均含有螺旋、转角、无规则卷曲和延伸链。其中无规则卷曲是CsCLHs蛋白中的主要结构,占比在48.51%~50.93%;其次为螺旋和延伸链,分别占比24.38%~29.63%和16.50%~19.33%;转角在CsCLHs蛋白的二级结构组成中占比最低。使用Phyre2在线工具的Intensive模式对CsCLHs蛋白的三级结构进行预测(图5),预测所得到的CsCLHs蛋白结构与模板的覆盖度均达到96%以上。通过Ramachandran在线评估蛋白质结构,结果显示位于允许区域范围内的氨基酸位点为93.2%~97.6%,表明所构建的CsCLHs蛋白的三级结构模型准确可信。
2.5 茶树CsCLHs启动子与顺势作用元件分析
从TPIA上下载上游2 000 bp序列,利用PlantCARE在线预测网站分析其顺式作用元件。结果表明(图6),除了具有典型核心启动子CAAT-box、TATA-box外,还包含大量的光响应元件、激素响应元件、逆境响应元件及较少的组织特异性元件和MYB识别位点。激素类响应元件主要包括脱落酸、水杨酸、赤霉素、乙烯、茉莉酸甲酯和生长素,其中均含有脱落酸响应元件,生长素响应元件、茉莉酸甲酯响应元件(as-1、TGACG-motif和CGTCA-motif)和赤霉素响应元件(TATC-box)只分布于和,茉莉酸响应元件(TCA元件)和乙烯响应元件(ERE元件)只分布于和;非生物胁迫响应元件中,干旱诱导响应元件(DRE core)、缺氧诱导响应元件(GC-motif)、压力响应元件(TC-rich repeats和STRE元件)只分布于,损伤诱导响应元件(WUN-motif)只分布于,低温诱导响应元件(LTR元件)只分布于。在家族中,中响应激素和胁迫的顺式作用元件数量最多,推测其可能在激素和胁迫响应中的作用更为显著。
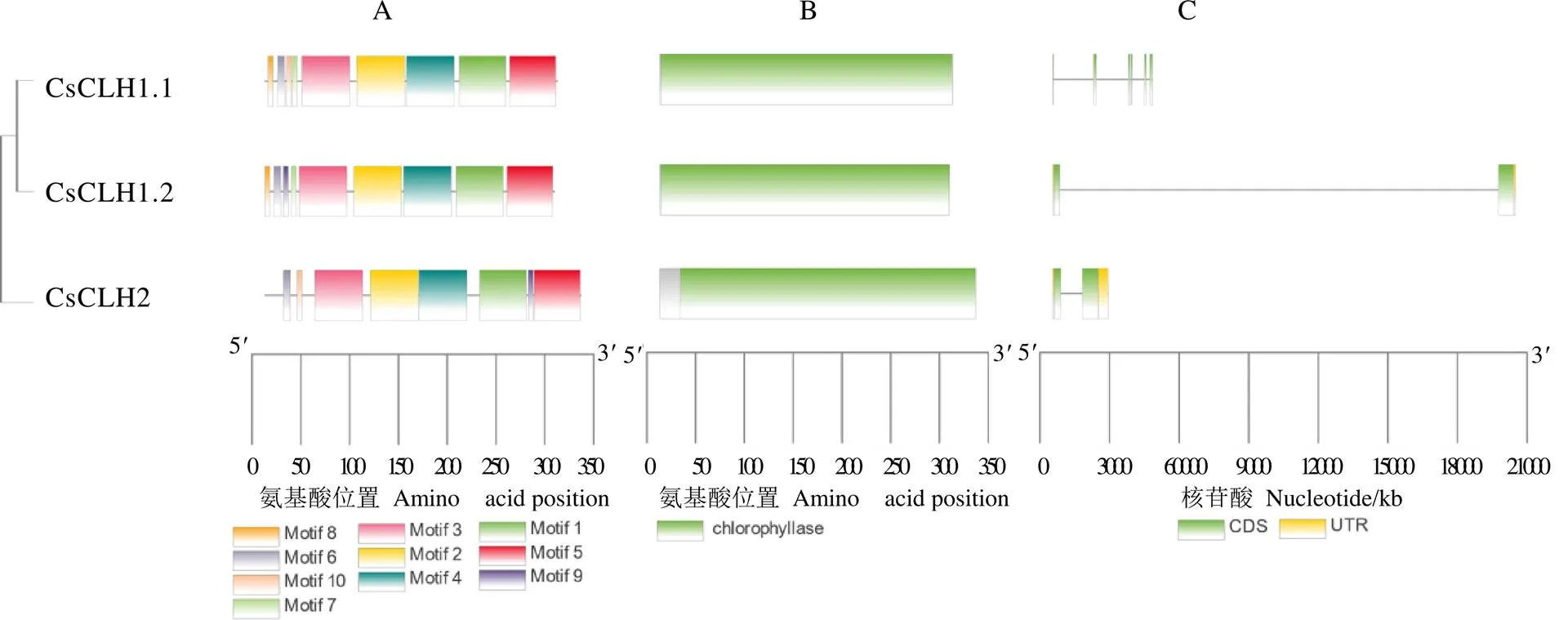
注:A:CsCLHs保守基序分布;B:蛋白质保守结构域;C:CsCLHs内含子-外显子结构分布图

表6 茶树CsCLHs家族蛋白二级结构预测
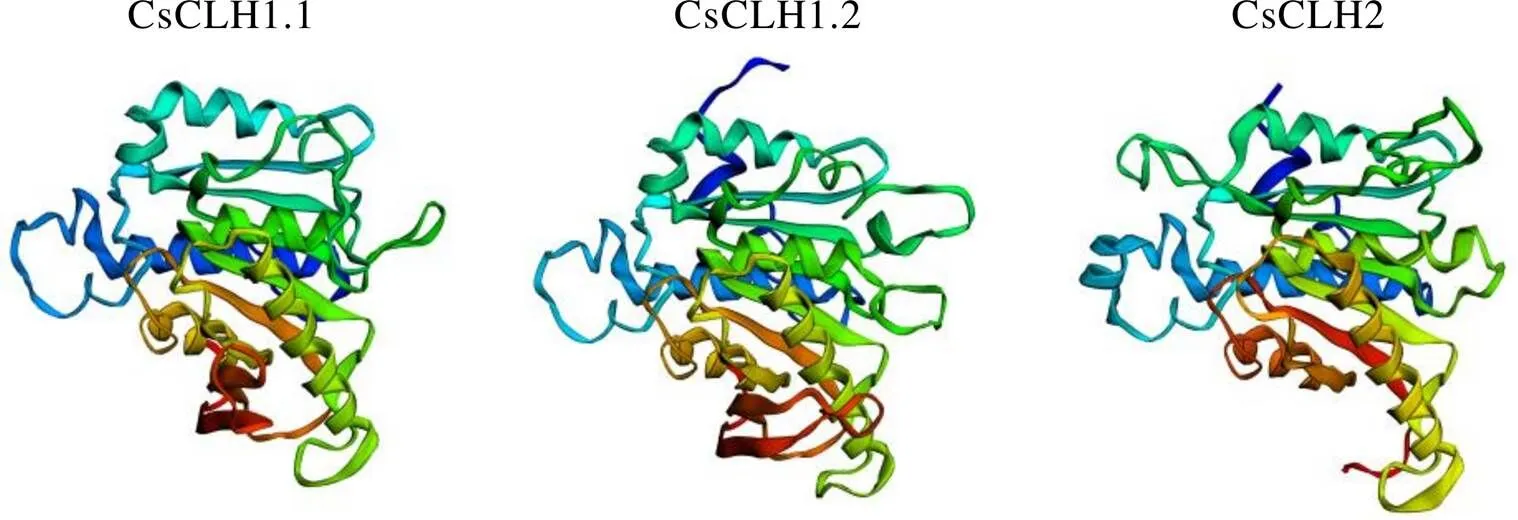
图5 茶树CsCLHs蛋白三级结构预测
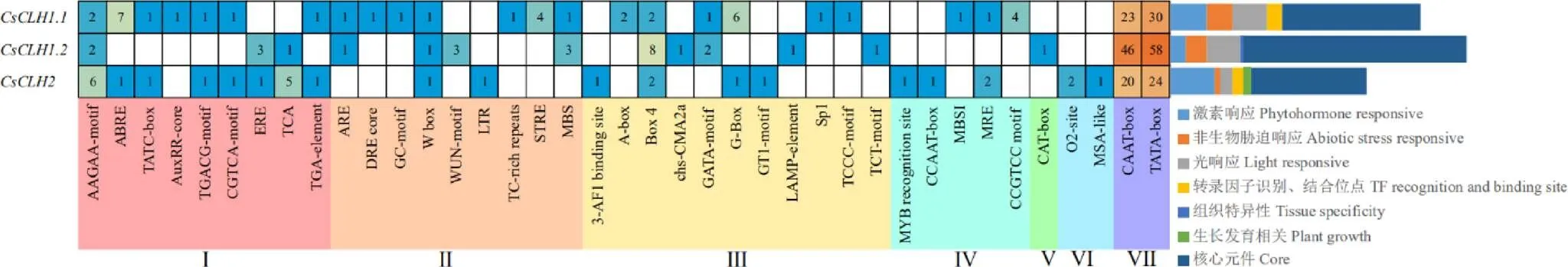
注:Ⅰ:激素响应;Ⅱ:非生物胁迫响应;Ⅲ:光响应;Ⅳ:转录因子识别、结合位点;Ⅴ:组织特异性元件;Ⅵ:生长发育相关元件;Ⅶ:核心元件
2.6 茶树CsCLHs上游调控转录因子预测及表达量分析
以模式植物拟南芥为参考,通过PlantTFDB在线数据库预测与上游启动子结合的转录因子并进行同源序列比对,共鉴定出22个转录因子,其中有12个转录因子同时与3个启动子结合(表7)。通过分析遮阴和恢复光照处理下白鸡冠第二叶转录组数据发现(图7),22个转录因子中有6个差异表达基因,其中有1个(CSS0047505)表达量呈现出遮阴后下调,恢复光照后上调的表达趋势,有2个(CSS0007067和CSS0027296)表达量呈现出遮阴后上调,恢复光照后下调的表达变化。
由酵母单杂交结果可知,pAbAi+Pro和pAbAi+Pro单转Y1H后培养在SD/-URA培养基中,通过AbA筛选抑制酵母生长的浓度,发现在150 ng·mL-1AbA中酵母不生长,因此Pro和Pro启动子的自激活质量浓度为150 ng·mL-1。以Y1H-pAbAi(Pro)和Y1H-pAbAi(Pro)为阴性对照,将pGADT7-BPC6和pGADT7-CDF5分别转入Y1H-pAbAi(Pro)和Y1H-pAbAi(Pro)后在SD/-Leu和SD/-Leu/AbA 150 ng·mL-1平板培养。由图8可知,Y1H(Pro+BPC6)、Y1H(Pro+CDF5)、Y1H(Pro+BPC6)和Y1H(Pro+CDF5)在SD/-Leu平板上正常生长,未转入pGADT7-BPC6和pGADT7-CDF5的Y1H(Pro)和Y1H(Pro)无法生长,表明pGADT7-BPC6和pGADT7-CDF5成功转入,而在SD/-Leu/AbA 150 ng·mL-1的平板上,Y1H(Pro+BPC6)和Y1H(Pro+BPC6)无法生长,Y1H(Pro+CDF5)和Y1H(Pro+CDF5)正常生长,表明CDF5能结合和的启动子。
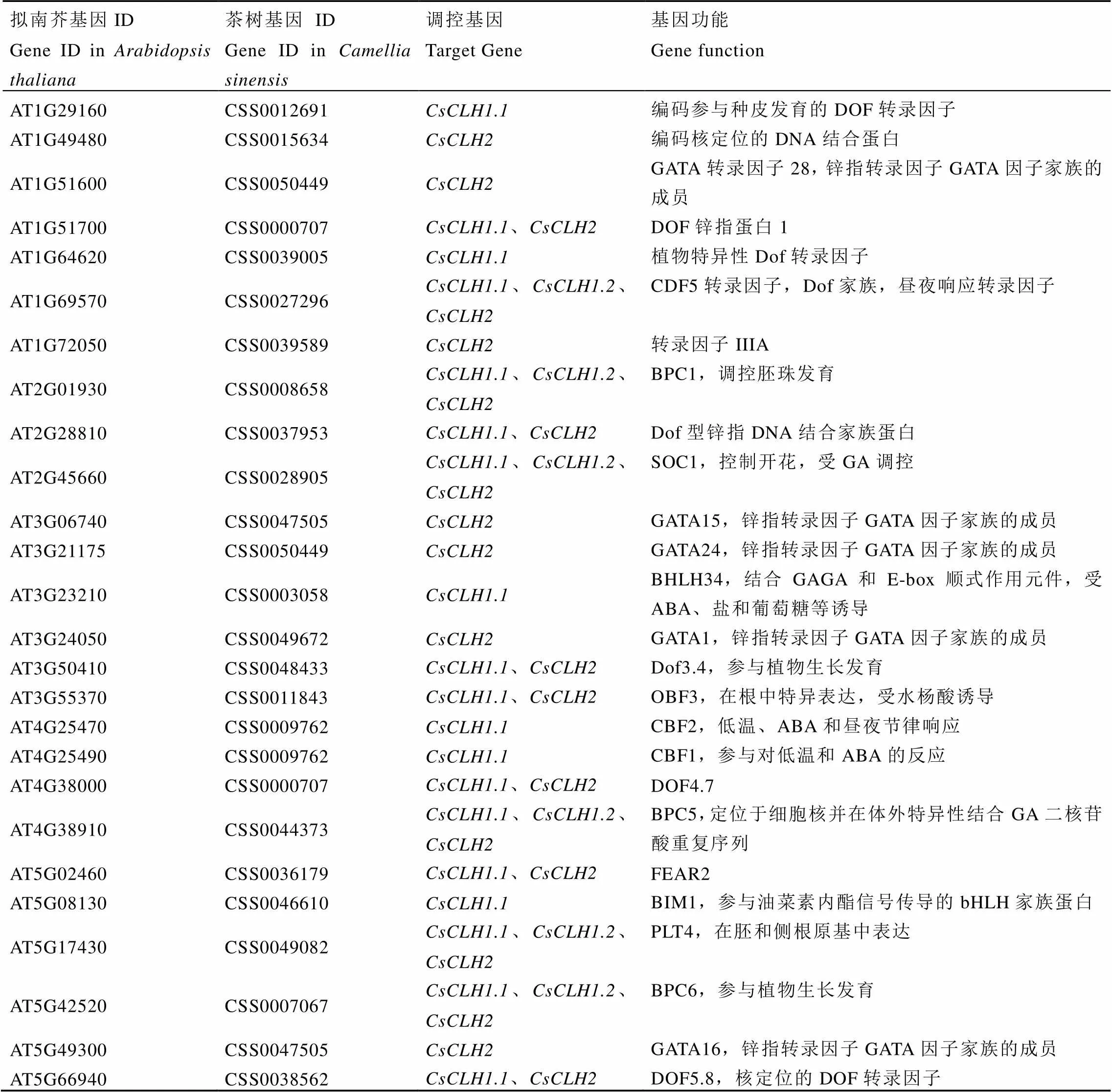
表7 茶树CsCLHs上游调控转录因子基因组信息
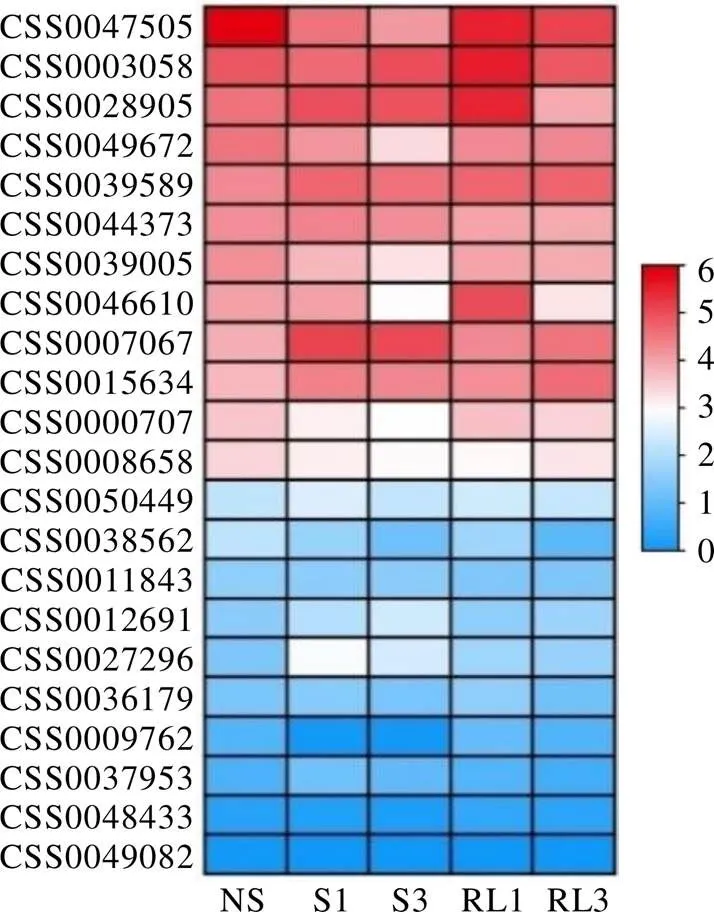
图7 预测调控CsCLHs的转录因子表达量

注:A:SD/-Ura/AbA培养基;B:SD/-Leu/AbA培养基
2.7 茶树CsCLHs基因在白化叶片中的表达分析
通过实时荧光定量PCR分析茶树在遮阴和恢复光照处理下的表达情况,结果表明(图9),在遮阴处理1 d和3 d,均呈现下调表达;恢复光照1 d,的表达量下调,相对表达稳定,而表达量极显著上调;恢复光照2 d,的表达量均呈现极显著上调趋势。
为了进一步探究在调控茶树叶色白化过程中的作用,以正常绿叶品种金牡丹为对照,对不同茶树品种中的表达量进行检测。结果表明(图10),在白化茶树中的表达量均显著高于金牡丹;的表达模式显示,除黄金芽和中黄1号外,其余品种的表达量均显著高于金牡丹;的表达图谱显示,仅白鸡冠和黄金芽的表达量高于金牡丹。
2.8 茶树CsCLHs在不同组织中的表达分析
为了解家族基因在茶树不同组织的表达模式,利用荧光定量PCR对的表达模式进行分析。结果表明(图11),在嫩叶、成熟叶和老茎中的表达量较高,在根中表达量最低;在嫩叶和果实中的表达量较高,在其余各部位的表达量均处于较低水平;而在各个组织部位中均有较高的表达量,其中在成熟叶中表达量最高。

注:以CsCLH1.1在不遮阴叶片中的表达量为对照;颜色刻度表示差异倍数log2转换均一化的值,红色为高水平表达,蓝色为低水平表达。NS,不遮阴;S1,遮阴1 d;S3,遮阴3 d;RL1,恢复光照1 d;RL2,恢复光照2 d
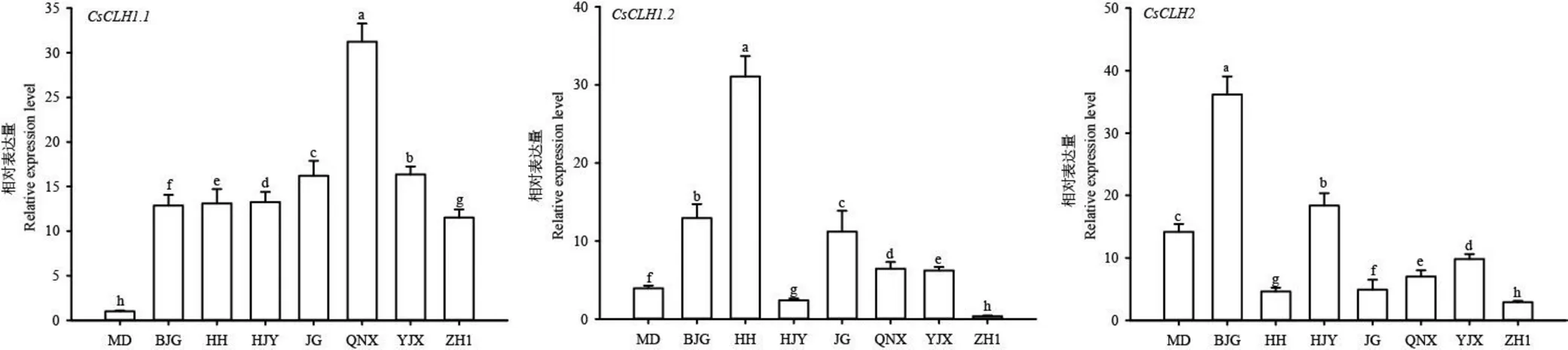
注:MD:金牡丹;BJG:白鸡冠;HH:黄化品系;HJY:黄金芽;JG:金光;QNX:千年雪;YJX:御金香;ZH1:中黄1号;不同小写字母表示品种间具有显著差异,P<0.05
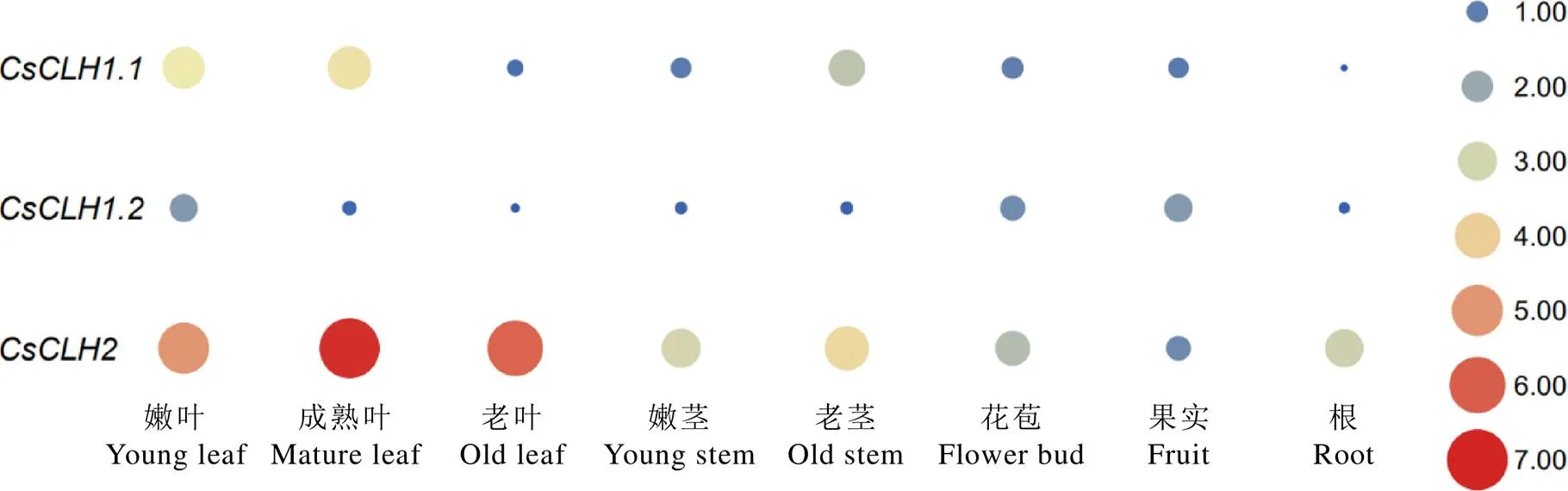
注:以CsCLH1.1在花苞中的表达量为对照;颜色刻度表示差异倍数log2转换均一化的值,红色为高水平表达,蓝色为低水平表达
3 讨论
植物叶绿素降解存在两条途径,分别是以叶绿素为底物的叶绿素酶降解途径和以脱镁叶绿素为底物的脱镁叶绿素酶降解途径[23],其中叶绿素酶被认为是叶绿素代谢途径中的第一个酶[24],在叶绿素分解代谢中作为限速酶进行翻译后调节[25]。在叶绿素酶理化研究方面,已有对波罗蜜[26]、鸡爪槭[27]、小麦[28]等植物叶片和采后贮藏过程中的叶绿素酶活性变化的研究报道,而在茶树中主要针对叶绿素酶的分离和纯化,加工过程叶绿素酶活性变化,以及不同茶树品种和不同叶位叶绿素酶活性的差异等进行研究。研究表明,在不同季节中春茶新梢叶绿素酶活性最高,其次为夏季、秋季,在茶树不同叶位中,叶绿素酶活性随着叶片衰老而降低[29]。与叶绿素酶理化性质研究相比,关于叶绿素酶分子水平上的研究尤其是基因克隆和转录表达水平的研究甚少。本研究在茶树中共克隆出3条,根据前人的报道,CLH基因家族在不同植物中包含的基因数量不同,如拟南芥[16]、青花菜[30]、发财树[31]等只有两条CLH基因。系统进化树分析结果显示,茶树CsCLH1s与葡萄等CLH1s聚在同一类亚群,茶树CsCLH2和葡萄、拟南芥等植物的CLH2聚在一个亚群。亚细胞定位预测发现,Cell-PLoc和WolfPsort预测结果存在差异,Cell-PLoc预测CsCLH1s定位于叶绿体,而WolfPsort预测CsCLH1s定位于细胞质中,此结果和前人报道一致,在银杏叶[32]、柑橘[33]等植物中,定位于嫩叶的叶绿体中,而在成熟叶或衰老叶中定位于细胞质、液泡膜和内质网中[34-36],表明叶绿素酶在植物不同组织间的功能存在差异。
CDF(Cycling dof factors)转录因子是植物中特有的转录因子家族,参与植物生长发育、光照和激素等非生物胁迫响应、碳氮代谢平衡等功能[37]。通过预测启动子的上游调控因子,结合基因组和白鸡冠茶树第二叶遮阴和恢复光照处理的转录组数据,鉴定出两个可能参与调控的转录因子(CSS0007067)和(CSS0027296),酵母单杂交结果表明可以与和的启动子结合。Henriques等[38]研究发现,CDF5的表达受到光调控,在黑暗条件下高表达,在光照条件下低表达。本研究转录组数据中在遮阴后表达量上调,恢复光照后表达量下调,与呈现相反的表达模式,表明在白鸡冠茶树白化叶片中可能负调控和的表达。
叶绿素酶是叶绿素降解过程中的重要组成部分,顺式作用元件分析结果表明,茶树基因启动子区包含了大量光响应有关的顺式作用元件,因此本研究分析了白鸡冠第二叶在遮阴和恢复光照处理下的表达模式。家族基因在遮阴及恢复光照处理后表达水平发生变化,即在遮阴下低表达,在恢复光照后高表达,这与周喆等[11]研究白鸡冠茶树第二叶在遮阴及恢复光照后的叶绿素含量和表达模式变化规律一致。在光照敏感型黄化品种黄金芽中也有类似的结果[39],表明在遮阴条件下,叶绿素降解途径中叶绿素和脱镁叶绿素的降解过程受阻,叶绿素和脱镁叶绿素在叶片中积累,叶色复绿,而在光照条件下叶绿素酶和脱镁叶绿素酶高表达,影响白鸡冠茶树叶片叶色变化。
研究表明,叶绿素酶在芽和嫩叶中有较高的活性[29],本研究中在嫩叶中表达量最高,在各部位中均有表达,其中在成熟叶、老叶和嫩叶中表达量较高,与前人研究结果一致。为了探究基因家族成员在调控茶树叶色白化过程中的作用,本研究对金牡丹和白鸡冠、黄金芽等8个品种表达量进行检测,其中在白化品种中具有较高的表达量,李远华等[29]研究表明茶树叶绿素酶活性与叶绿素含量呈负相关,推测基因参与白化茶树叶片白化过程。
本研究在茶树全基因组范围内鉴定获得3条家族基因,并分析了其结构和功能,研究结果可为CsCLHs参与茶树叶绿素代谢途径并调控芽叶白化的机制研究提供参考。
[1] Zhong X M, Sun S F, Li F H, et al. Photosynthesis of a yellow-green mutant line in maize [J]. Photosynthetica, 2015, 53(4): 499-505.
[2] Li W X, Yang S B, Lu Z G, et al. Cytological, physiological, and transcriptomic analyses of golden leaf coloration inL [J]. Horticulture Research, 2018, 5: 12. doi: 10.1038/s41438-018-0015-4.
[3] Gang H X, Liu G F, Chen S, et al. Physiological and transcriptome analysis of a yellow-green leaf mutant in birch (×) [J]. Forests, 2019, 10(2): 120. doi: 10.3390/f10020120.
[4] Slattery R A, VanLoocke A, Bernacchi C J, et al. Photosynthesis, light use efficiency, and yield of reduced-chlorophyll soybean mutants in field conditions [J]. Frontiers in Plant Science, 2017, 8: 549. doi: 10.3389/fpls.2017.00549.
[5] 元世昌, 黄亚伟, 王若兰, 等. 优质稻黄变期间营养组分的变化规律研究[J]. 食品科技, 2019, 44(6): 156-161.
Yuan S C, Huang Y W, Wang R L, et al. Changes of nutrient components during high quality rice yellowing [J]. Food Science and Technology, 2019, 44(6): 156-161.
[6] Ma L L, Liu Y L, Cao D, et al. Quality constituents of high amino acid content tea cultivars with various leaf colors [J]. Turkish Journal of Agriculture and Forestry, 2018, 42(6): 383-392.
[7] Shin Y H, Yang R, Shi Y L, et al. Light-sensitive albino tea plants and their characterization [J]. Hortscience, 2018, 53(2): 144-147.
[8] 范延艮, 赵秀秀, 王翰悦, 等. 黄金芽不同色泽叶片生理特性研究[J]. 茶叶科学, 2019, 39(5): 530-536.
Fan Y G, Zhao X X, Wang H Y, et al. Study on physiological charateristics of leaves with different colors of ‘Huangjinya’ [J]. Jounal of Tea Science, 2019, 39(5): 530-536.
[9] 吴全金. ‘白鸡冠’茶树响应光调控的基因差异及理化特征分析[D]. 福州: 福建农林大学, 2015.
Wu Q J. Gene differential analysis and physicochemical characteristics ofcv.in response to light [D]. Fuzhou: Fujian Agriculture and Forestry University, 2015.
[10] Li N, Yang Y P, Ye J H, et al. Effects of sunlight on gene expression and chemical composition of light-sensitive albino tea plant [J]. Plant Growth Regulation, 2016, 78(2): 253-262.
[11] 周喆, 陈志丹, 吴全金, 等. 白鸡冠茶树基因全长cDNA克隆与表达分析[J]. 茶叶科学, 2020, 40(1): 43-54.
Zhou Z, Chen Z D, Wu Q J, et al. Cloning and expression analysis ofgene in tea plant () [J]. Jounal of Tea Science, 2020, 40(1): 43-54.
[12] Dong F, Zeng L T, Yu Z M, et al. Differential accumulation of aroma compounds in normal green and albino-induced yellow tea () leaves [J]. Molecules, 2018, 23(10): 2677. doi: 10.3390/molecules23102677.
[13] Wang L, Yue C, Cao H L, et al. Biochemical and transcriptome analyses of a novel chlorophyll-deficient chlorina tea plant cultivar [J]. BMC Plant Biology, 2014(14): 352. doi: 10.1186/s12870-014-0352-x.
[14] Wu Q J, Chen Z, Sun W J, et al.sequencing of the leaf transcriptome reveals complex light-responsive regulatory networks incv.[J]. Frontiers in Plant Science, 2016, 7: 332. doi:10.3389/fpls.2016.00332.
[15] 李旭敏. 光敏型白化茶转录组分析及叶绿素代谢途径相关基因研究[D]. 杭州: 浙江大学, 2019.
Li X M. Study on transcriptome and gene expression with regard to chlorophylls metabolism pathway in photosensitive albino tea plant [D]. Hangzhou: Zhejiang University, 2019.
[16] Tsuchiya T, Ohta H, Okawa K, et al. Cloning of chlorophyllase, the key enzyme in chlorophyll degradation: finding of a lipase motif and the induction by methyl jasmonate [J]. PNAs, 1999, 96(26): 15362-15367.
[17] Chen C M M, Chao P Y, Huang M Y, et al. Chlorophyllase activity in green and non-green tissues of variegated plants [J]. South African Journal of Botany, 2012, 81(4): 44-49.
[18] 梁俊林, 顾国军, 唐实玉, 等. 银杏叶变色期的生理特征[J]. 四川农业大学学报, 2020, 38(1): 65-70.
Liang J L, Gu G J, Tang S Y, et al. Physiological characteristics of leaf color change period ofL. [J]. Journal of Sichuan Agricultural University, 2020, 38(1): 65-70.
[19] Tian Y N, Zhong R H, Wei J B, et al.CHLOROPHYLLASE 1 protects young leaves from long-term photodamage by facilitating FtsH-mediated D1 degradation in photosystem Ⅱ repair [J]. Molecular Plant, 2021, 14(7): 1149-1167.
[20] 张兰, 滕珂, 尹淑霞. 草地早熟禾叶绿素酶1基因的克隆和表达分析[J]. 中国草地学报, 2016, 38(4): 1-7.
Zhang L, Teng K, Yin S X. Cloning and expression analysis of chlorophyllase 1 genefromL. [J]. Chinese Journal of Grassland, 2016, 38(4): 1-7.
[21] Xu D, Lu Z C, Jin K M, et al. SPDE: a multi-functional software for sequence processing and data extraction [J]. Bioinformatics, 2021, 37(20): 3686-3687.
[22] Chen C J, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data [J]. Molecular Plant, 2020, 13(8): 1194-1202.
[23] Schelbert S, Aubry S, Burla B, et al. Pheophytin pheophorbide hydrolase (pheophytinase) is involved in chlorophyll breakdown during feaf senescence in[J]. Plant Cell, 2009, 21(3): 767-785.
[24] Takamiya K I, Tsuchiya T, Ohta H. Degradation pathway(s) of chlorophyll: what has gene cloning revealed? [J]. Trends in Plant Science2000, 5(10): 426-431.
[25] Harpaz-Saad S, Azoulay T, Arazi T, et al. Chlorohyllase is a rate-limiting enzyme in chlorophyll catabolism and is posttranslationally regulated [J]. The Plant Cell, 2007, 19(3): 1007-1022.
[26] 周丹, 罗灿, 于旭东, 等. 波罗蜜叶片突变体叶绿素含量测定和超微结构观察[J]. 热带作物学报, 2021, 42(10): 2935-2941.
Zhou D, Luo C, Yu X D, et al. Determination of chlorophyll content and observation of ultrastructure in leaves of mutants of[J]. Chinese Journal of Tropical Crops, 2021, 42(10): 2935-2941.
[27] 梁俊林, 李俭, 代鑫, 等. 施加Fe2+和Cu2+对鸡爪槭叶色变化的生理影响[J]. 应用与环境生物学报, 2021, 27(3): 549-554.
Liang J L, Li J, Dai X, et al. Physiological effects of Fe2+and Cu2+application on leaf color changes in[J]. Chinese Journal of Applied and Environmental, 2021, 27(3): 549-554.
[28] 王绘艳. 小麦叶绿素和脱镁叶绿素酶基因的作用及表达分析[D]. 太原: 山西农业大学, 2015.
Wang H Y. The function and gene expression analysis of chlorophyllase and pheophttinase in[D]. Taiyuan: Shanxi Agricultural University, 2015.
[29] 李远华, 顾玮, 倪德江, 等. 茶树叶绿素酶活性的变化研究[J]. 茶叶科学, 2011, 31(1): 27-32.
Li Y H, Gu W, Ni D J, et al. Study on variation of chlorophyllase activity in tea plant () [J]. Journal of Tea Science, 2011, 31(1): 27-32.
[30] 樊艳燕, 刘玉梅, 李占省, 等. 青花菜衰老过程中叶绿素降解相关基因的表达分析[J]. 园艺学报, 2015, 42(7): 1338-1346.
Fan Y Y, Liu Y M, Li Z X, et al. Analysis of the expression of chlorophyll degrading genes during senescence of[J]. Acta Horticulturae Sinica, 2015, 42(7): 1338-1346.
[31] Chen M C M, Yang J H, Liu C H, et al. Molecular, structural, and phylogenetic characterization of two chlorophyllase isoforms in[J]. Plant Systematics and Evolution. 2014, 300: 633-643.
[32] Okazawa A, Tang L, Itoh Y, et al. Characterization and subcellular localization of chlorophyllase from[J]. Zeitschrift für Naturforschung C, 2006, 61(1/2): 111-117.
[33] Shemer T A, Harpaz-Saad S, Belausov E, et al.chlorophyllase dynamics at ethylene-induced fruit color-break: a study of chlorophyllase expression, posttranslational processing kinetics, and in situ intracellular localization [J]. Plant Physiology, 2008, 148(1): 108-118.
[34] Hrtensteiner S, Krutler B. Chlorophyll breakdown in higher plants [J]. Biochimica et Biophysica Acta, 2011, 1807(8): 977-988.
[35] Nicole S A, Silivia S B, Marion K C, et al. The chlorophyllases AtCLH1 and AtCLH2 are not essential for senescence-related chlorophyll breakdown in[J]. FEBS Letters, 2007, 581(28): 5517-5525.
[36] Hu X, Makita S, Schelbert S, et al. Reexamination of chlorophyllase function implies its involvement in defense against chewing herbivores [J]. FEBS Letters, 2015, 167(3): 660-670.
[37] Begoa R M, Laura C, Jose D F, et al. CDF transcription factors: plant regulators to deal with extreme environmental conditions [J]. Journal of Experimental Botany, 2020, 71(13): 3803-3815.
[38] Henriques R, Wang H, Liu J, et al. The antiphasic regulatory module comprising CDF5 and its antisense RNA FLORE links the circadian clock to photoperiodic flowering [J]. The New phytologist, 2017, 216(3): 854-867.
[39] Fan Y G, Zhao X X, Wang H Y, et al. Effects of light intensity on metabolism of light-harvesting pigment and photosynthetic system inL. cultivar 'Huangjinya' [J]. Environmental and Experimental Botany, 2019, 166: 103796. doi: 10.1016/j.envexpbot.2019.06.009.
Identification and Transcriptional Regulation of CLH Gene Family and Expression Analysis in Albino Tea Plants ()
WANG Tao1,3,4,5, WANG Yiqing1,3,4,5, QI Siyu1,3,4,5, ZHOU Zhe1,3,4,5, CHEN Zhidan2,3,4,5*, SUN Weijiang1,3,4,5*
1. College of Horticulture, Fujian Agriculture and Forestry University, Fuzhou 350002, China; 2. Anxi College of Tea Science, Fujian Agriculture and Forestry University, Quanzhou 362400, China; 3. Engineering Technology and Research Center of Fujian Tea Industry, Fuzhou 350002, China; 4. Ministerial and Provincial Joint Innovation Centre for Safety Production of Cross-Strait Crops, Fujian Agriculture and Forestry University, Fuzhou 350002, China; 5. Tea Industry Technology Development Base of Fujian Province, Fuzhou 350002, China
Chlorophyllase (CLH) is the key enzyme in the degradation of chlorophyll, stripping its phytol to form dephytolithochlorophyll a. The full-length cDNA sequences of threegenes were obtained from the second leaves of albino tea cultivar ‘Baijiguan’, and bioinformatics analysis was performed. The results show that the threegenes could be divided into two subfamilies. The full length ofwas 894-975 bp, encoding 297-324 amino acids. The protein molecular weights were 31.99-34.91 kDa. The isoelectric points were 4.89-7.61, and the instability coefficients were 38.94-48.24. CsCLH1.1 and CsCLH1.2 were unstable proteins, while CsCLH2 was a stable protein. The subcellular localization prediction results of Cell Ploc show that three CsCLH proteins were located in chloroplast, while the results of Wolf Psort show that CsCLH1.1 and CsCLH1.2 were located in cytoplasm and CsCLH2 was located in chloroplast. The qRT-PCR results on the ‘Baijiguan’ leaves indicated that expressions ofwere inhibited by shading treatment and light induced’ expressions. Expression pattern analysis ofshows thatwere highly expressed in the albino cultivars. In addition, it was identified thatcould bind to theandpromoters according to the yeast one hybrid system. In conclusion, CsCLHs in albino tea leaves might be involved in chlorophyll degradation and play an important role in the process of albino leaf, which provided a reference for further investigation in the function of the CLH gene family and the albinism of leaves in tea plants.
, chlorophyllase, gene family, sequence analysis, differential expression
S571.1
A
1000-369X(2022)03-331-16
2021-12-14
2022-01-24
福建省高校产学合作项目(2019N5007)、国家自然科学基金(31770732)、国家重点研发计划(2019YFD1001601)、福建农林大学“双一流”建设科技创新能力提升培育计划(KSYLC005)、福建张天福茶叶发展基金会科技创新基金(FJZTF01)
王涛,男,硕士研究生,主要从事茶树栽培育种与生物技术方面研究。*通信作者:asbulletdan@163.com;swj8103@126.com
(责任编辑:黄晨)
