瘤胃微生物群对奶牛亚急性瘤胃酸中毒易感性的影响
2022-06-17牟瑞营吐尼牙孜买买提赵才军张乃生胡晓宇付云贺
李 爽,牟瑞营,吐尼牙孜·买买提,赵才军,张乃生,胡晓宇*,付云贺*
(1 吉林大学 动物医学学院,吉林 长春130062;2 临朐县畜牧局,山东 临朐 261000)
在奶牛的泌乳初期或高峰期,往往需要对奶牛饲喂大量的精饲料以满足产奶的营养需求。然而,这些易发酵碳水化合物饲料的长期饲喂往往会导致瘤胃pH值的降低,进而导致亚急性瘤胃酸中毒(SARA)的发生[1-3]。SARA在给乳业带来了巨大挑战的同时,也给农业经济带来了巨大的损失,并且给动物福利也带来了负面影响。研究表明,SARA与腹泻、蹄叶炎、真胃变位、肝脓肿、全身炎症、饲料利用率低以及乳脂降低密切相关[4-5]。
当奶牛饲喂高精饲料时,瘤胃中的非纤维性碳水化合物会促进淀粉酶的发酵和有机酸的产生,尤其是乳酸[6-9]。在早期阶段,大量乳酸可以被乳酸利用菌快速代谢,转化为短链脂肪酸(SCFAs),主要是乙酸、丙酸和丁酸[10]。然而,一旦乳酸的产生超过乳酸利用菌的代谢能力,大量乳酸开始在瘤胃中积累,导致瘤胃液pH值迅速下降,诱导SARA的发生[11-12]。临床实践表明,即使采用相似的饲养和管理方式,奶牛对SARA的敏感性也存在很大差异[13]。造成这种变化的原因尚不清楚,但可能是生理和行为的综合影响,包括瘤胃微生物群组成。一项重要的研究表明,瘤胃微生物群的差异是影响高谷物饲粮耐受或适应的重要因素。此外,也有研究报道,与饲喂小麦粉碎物的绵羊相比,瘤胃内接种小麦耐受绵羊的瘤胃液后,这些接种瘤胃液的绵羊不会出现进食过度而引发的消化不良[14]。这些证据表明,瘤胃微生物群与SARA易感性密切相关。
越来越多的研究证明,SARA奶牛瘤胃微生物群发生显著的改变[15-16],并且调节瘤胃微生物群可以有效改善高精饲料诱发的SARA[17-18]。然而,瘤胃细菌群落与SARA的易感性之间的关系尚未见报道。因此,在本研究中,我们评估了SARA耐受奶牛和SARA易感奶牛的瘤胃液中细菌和真菌群落的特点,旨在找到某些能够使奶牛适应瘤胃中高谷物饲料的菌种,以此为依据筛查SARA耐受奶牛并为SARA的预防提供一个新的方向。
1 材料与方法
1.1 实验动物与方法试验选用16头来自山东省青州市某牛场的荷斯坦奶牛(2~3岁,泌乳天数和体质量相似)。试验前,所有动物都饲喂低精饲料(精饲料和粗饲料比值为4∶6),自由饮水,适应环境15 d后,所有的奶牛都饲喂高精饲料(精饲料和粗饲料比值为7∶3),每天饲喂2次(5:00和18:00),自由采食和饮水。6周后,基于瘤胃液的pH值(12 h内 pH<5.8的时长超过3 h可判定为SARA),将奶牛分为SARA耐受组(高精饲料喂养6周后SARA阴性的动物)和SARA易感组(高精饲料喂养6周后SARA阳性的动物)。方案由吉林大学伦理委员会批准(图1)。
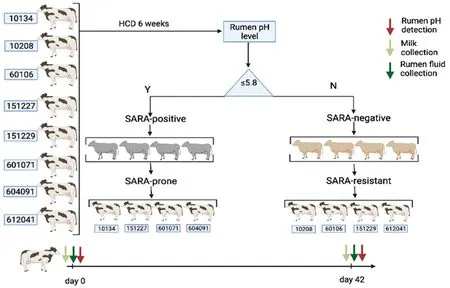
图1 技术路线
1.2 样品采集和分析使用2.0 mm×120 mm针头和50 mL注射器[19-20]通过瘤胃穿刺收集每个样本的瘤胃液。分别在喂食高精饲料开始前和6周后采取瘤胃液,将采集样本在液氮中冷冻并在-80℃保存。进行16S rRNA和ITS1-1F检测分析。
1.3 DNA提取和Illumina MiSeq测序以及生物信息学分析使用CTAB/SDS方法提取瘤胃液中的总基因组DNA样本,并使用无菌水稀释至1 mg/L。使用特异性引物(16S V4:515F-806R或ITSI-1F-F-ITSI-1F-R)扩增不同区域的16S rRNA基因或ITSI-1F。所有的PCR反应都使用Phusion®高保真PCR Master Mix(New England Biolabs)进行。PCR产物以相等的比例混合,并使用Qiagen凝胶提取试剂盒(Qiagen,Germany)进行纯化。使用TruSeq®DNA PCR-Free Sample Preparation Kit(Ilumina,USA)生成测序文库,并在Qubit@ 2.0 Fluorometer(Thermo Scientific)和Agilent Bioanalyzer 2100系统上评估文库质量。最后在IluminaHiSeq2500平台上对文库进行测序,得到250 bp的配对末端读数。
测序深度由稀疏曲线监测,其最小一致性截止值为97%,最小对齐长度截止值为100 bp。采用alpha多样性指数(包括Ace和Shannon指数)评价各样本的物种丰富度和多样性。所有指标用QIIME(1.7.0版)计算,用R软件(2.15.3版)显示。利用生物信息学和进化基因组学原理建立的Venn图分析不同类群样品中核心属瘤胃液的数量。采用线性判别分析结合效应大小(LEfSe)分析,鉴定了不同组样本瘤胃液细菌群落中差异表达的细菌类群。
2 结果
2.1 瘤胃液pH值高精饲料饲养6周后,根据瘤胃液pH值将奶牛分为2组,有8头奶牛为SARA阳性组(瘤胃液pH值为5.77±0.10),其余8头牛为SARA阴性组(瘤胃液pH值为6.32±0.09)(表1)。

表1 瘤胃液pH值
2.2 SARA耐受和SARA易感组奶牛瘤胃内细菌微生物群落组成的比较在通过质量控制测试的热测序读数中,共有1 405 280个读数,瘤胃液中每个样品平均有87 830个读数。通过观察检测样本物种积累箱型图可以得到本次测序量和测序深度符合样本分析要求(图2)。
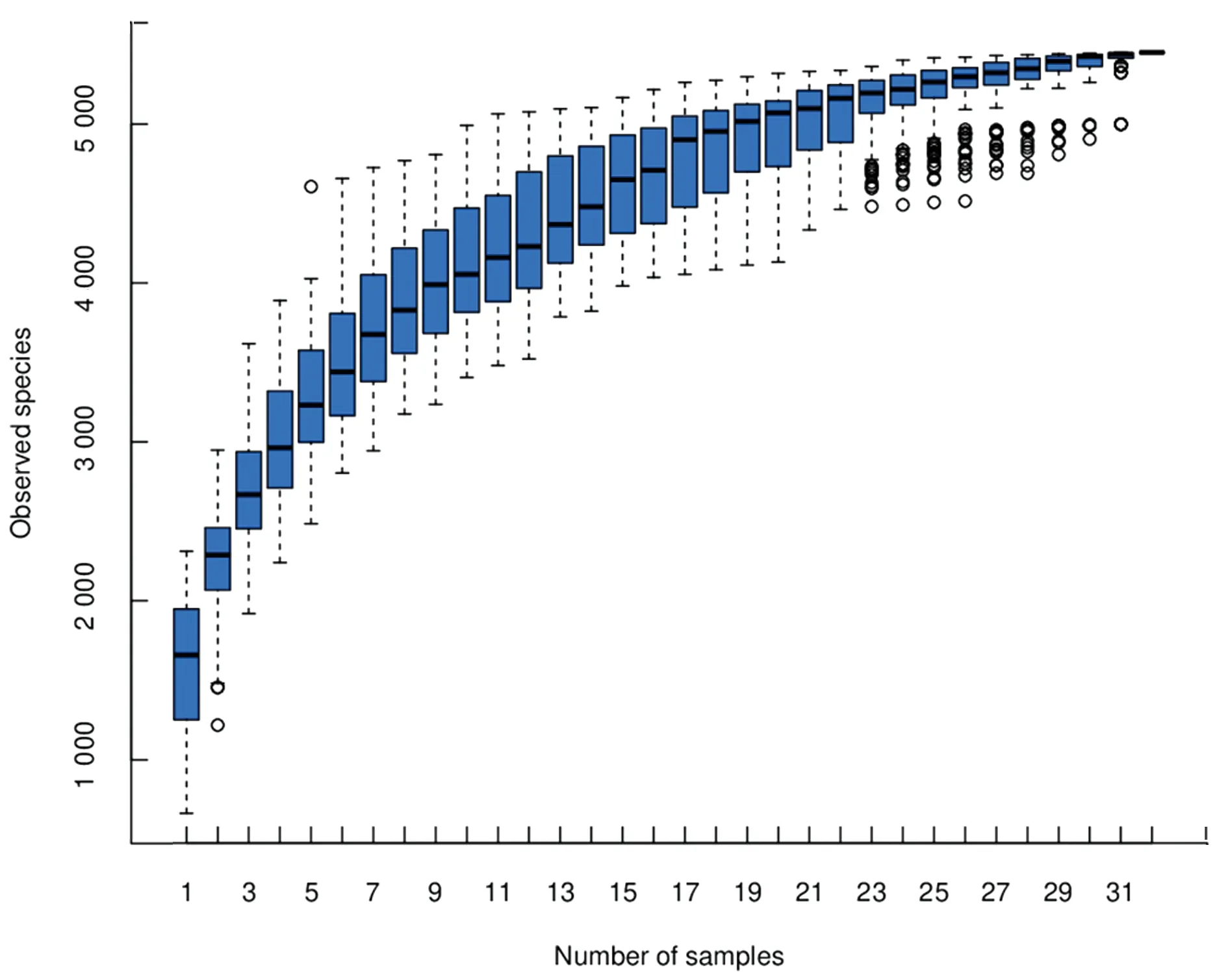
图2 检测样本物种累计箱型图
为了探索SARA的敏感性是否与奶牛的瘤胃微生物群有关,我们分析了SARA耐受和SARA易感奶牛瘤胃液中的细菌群落。Ace(P= 0.81)和Shannon指数(P= 0.37)分析表明,SARA易感奶牛瘤胃液的多样性高于SARA耐受奶牛,但差异不明显(图3A,B)。此外,未加权UniFrac指标的PCoA显示,SARA耐受和SARA易感奶牛瘤胃液的细菌群落结构发生变化(图3C)。
2.3 SARA耐受和SARA易感奶牛瘤胃内细菌群落在门类和属种层面的比较在门水平上,结果表明变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、软壁菌门(Tenericutes)和螺旋菌门(Spirochaetes)在SARA耐受奶牛和SARA易感奶牛瘤胃液中最为丰富(图4A)。在属水平上,寡养单胞菌(Stenotrophomonas),未鉴定的瘤胃球菌(unidentified_Ruminococcaceae),支原体(Mycoplasma),未鉴定的普雷沃菌(unidentified_Prevotellaceae),未鉴定的理研菌(unidentified_Rikenellacea),未鉴定的拟杆菌(unidentified_Bacteroidales),未鉴定的毛螺菌(unidentified_Lachnospiraceae),未鉴定的蓝藻细菌(unidentified_Cyanobacteria),琥珀酸菌(Succiniclasticum),乳酸杆菌(Lactobacillus)在SARA易感奶牛和SARA耐受奶牛瘤胃液中丰度较高(图4B)。
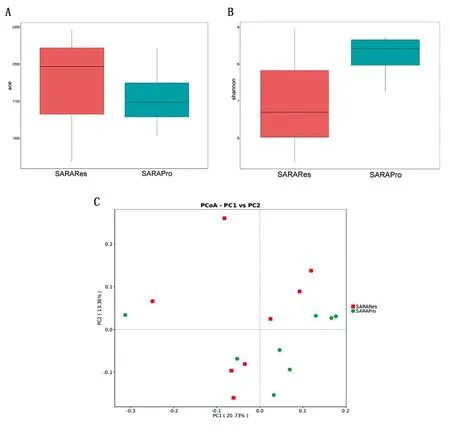
A.Ace细菌微生物区系丰富度;B.Shannon微生物多样性;C.SARA耐受奶牛和SARA易感奶牛瘤胃液细菌群落组成的PCoA显示未加权UniFrac度量差异;SARARes.SARA耐受奶牛;SARAPro.SARA易感奶牛
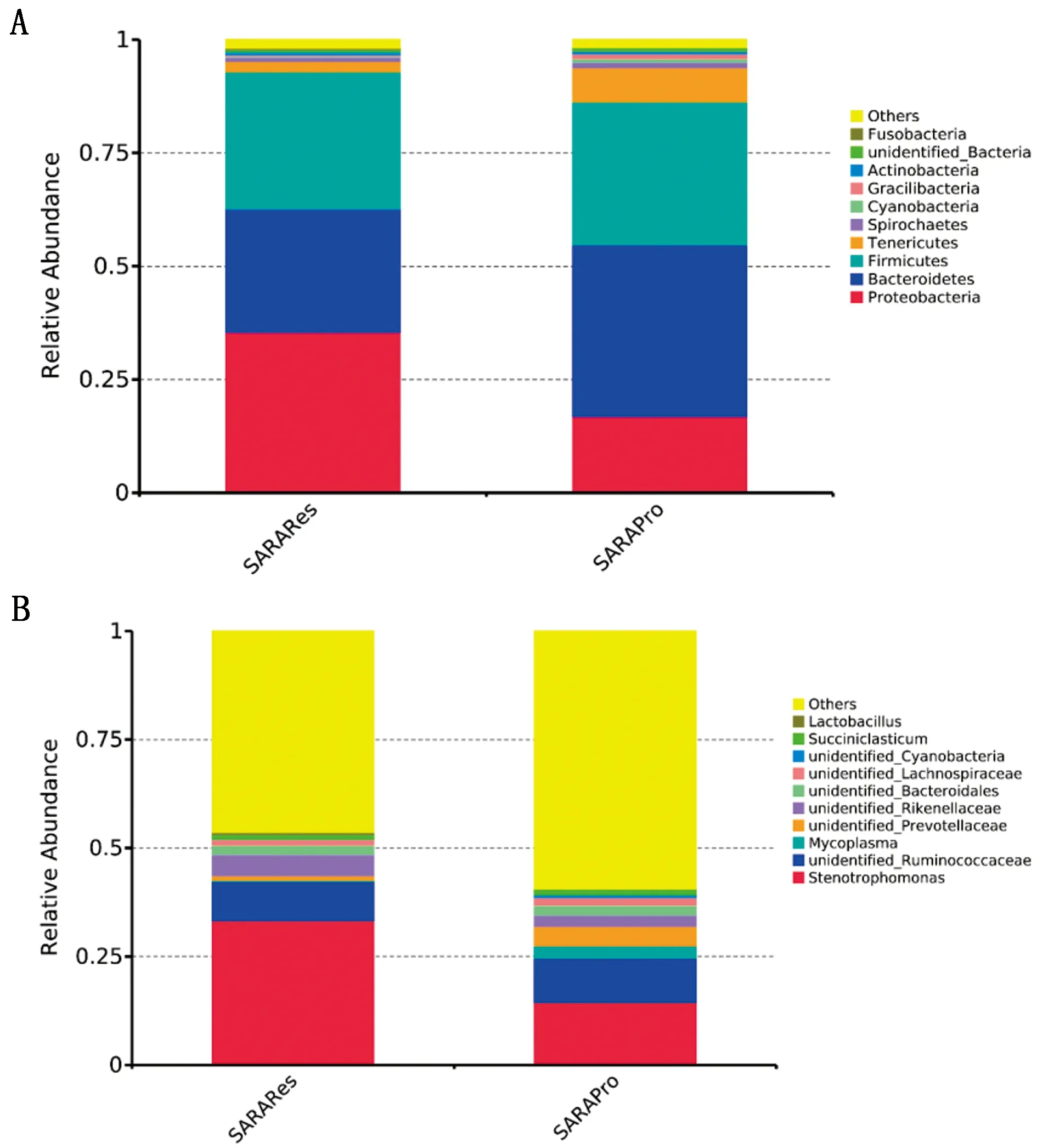
A.SARA耐受和SARA易感奶牛的瘤胃液中前10个门类的相对丰度;B.SARA耐受和SARA易感奶牛的瘤胃液中前10个属的相对丰度
Venn图用于评估细菌群落的独特性是否与SARA的发生有关。如图5A所示,大多数属在SARA耐受组和SARA易感组奶牛之间共享。核心菌属在SARA耐受组中占所有瘤胃细菌的66.92%,在SARA易感组奶牛中占75.48%。此外,t检验分析SARA耐受组和SARA易感组奶牛瘤胃液中细菌属的差异显示,SARA易感组奶牛的unidentified_Spirochaetaceae和Anaeroplasma明显高于SARA耐受组的奶牛(图5B)。此外,用LEfSe来分析属水平的生物标志物,线性判别分析(LDA=3.4)也检测到unidentified_Spirochaetaceae在SARA易感组奶牛中富集(图5C,D)。此外,瘤胃普雷沃菌(Prevotella_ruminicola)属的丰度在SARA易感奶牛中也富集,而泰泽雷拉菌属(Tyzzerella_sp_Marseille_P3062)的丰度在SARA耐受奶牛中富集。这些结果表明,细菌群落概况,特别是瘤胃液中unidentified_Spirochaetaceae、Anaeroplasma和Prevotella_ruminicola属的丰度增加,可能与奶牛对SARA的易感性有关。
2.4 SARA耐受和SARA易感奶牛瘤胃内真菌群落的比较通过高通量测序(HTS), 16个瘤胃样品的ITS扩增片段共获得1 097 391条,平均每个瘤胃样品的ITS扩增片段数为68 586条。通过观察检测样本物种积累箱型图可以得到本次测序量和测序深度符合样本分析要求(图6)。
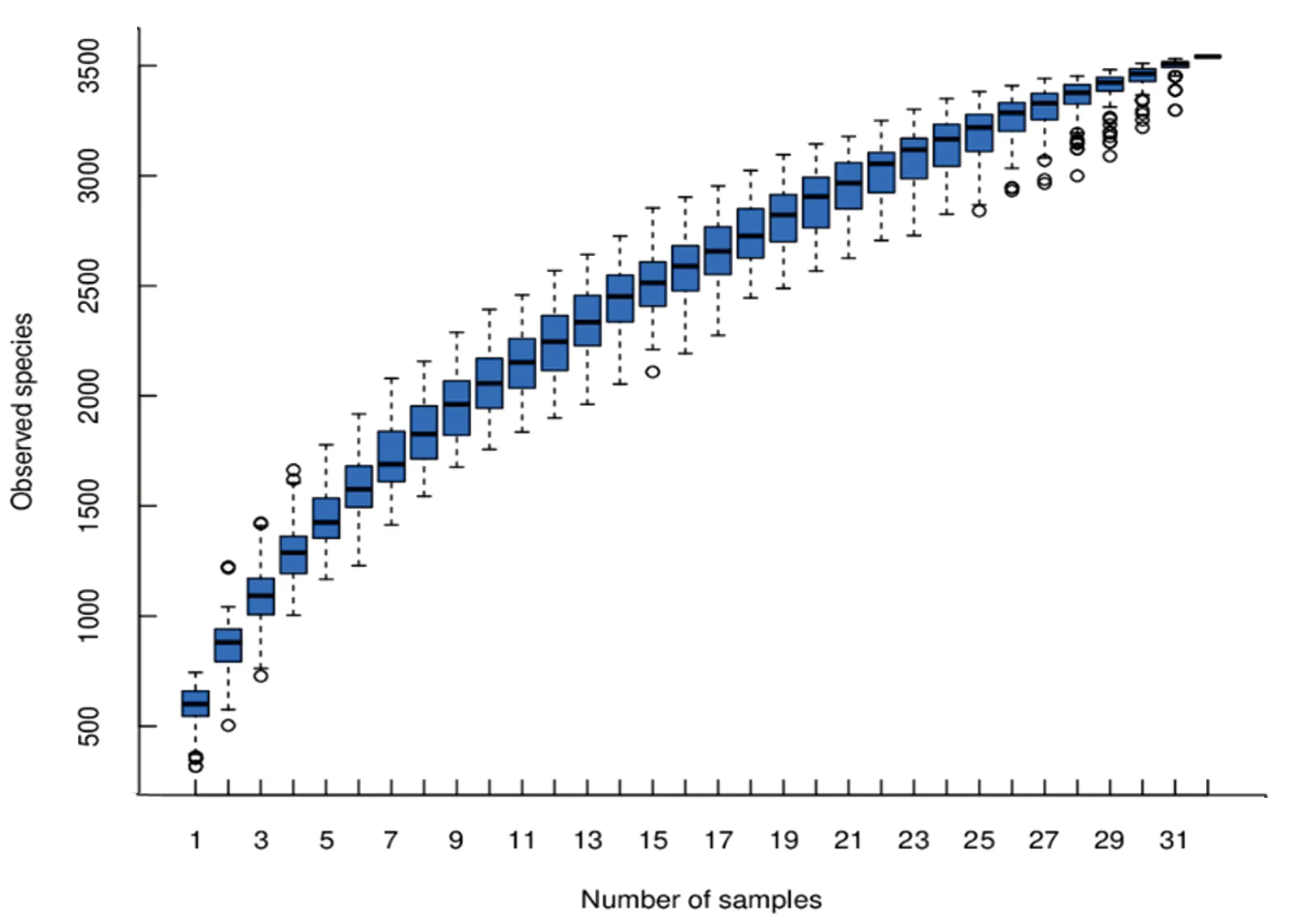
图6 检测样本物种累计箱型图
对SARA耐受奶牛和SARA易感奶牛瘤胃真菌进行α-多样性分析,Ace(P=0.94)和Shannon(P=0.13)指数结果表明,瘤胃真菌丰富度没有显著差异,而瘤胃真菌多样性SARA耐受奶牛高于SARA易感奶牛,虽然差异不显著(图7A,B)。此外,未加权UniFrac度量的PCoA显示,SARA耐受奶牛和SARA易感奶牛之间的瘤胃液真菌群落特征没有明显差异(图7C)。
2.5 SARA耐受和SARA易感奶牛瘤胃内真菌群落在门类和属种层面的比较进一步分析表明,在SARA耐受组和SARA易感组奶牛中,瘤胃真菌种群以子囊菌门(Ascomycota)、薪假丝酵母菌门(Neocallimastigomycota)、担子菌门(Basidiomycota)和莫蒂埃雷利菌门(Mortierellimycota)为主(图8A)。此外,在SARA耐受和SARA易感组奶牛瘤胃液中相对丰度居前10位的菌属有节担菌属(Wallemia)、镰刀菌属(Fusarium)、枝顶孢属(Acremonium)、癣囊腔菌(Plectosphaerella)、假丝酵母属(Candida)、枝孢霉属(Cladosporium)、钉孢属(Passalora)、曲霉菌(Aspergillus)、茎点霉属(Phoma)、乳头状瘤菌(Papiliotrema)(图8B)。
此外,Venn图显示,大多数真菌属在SARA耐受和SARA易感奶牛之间是共享的(图9A)。在这些OTU中,在SARA耐受组奶牛中核心真菌微生物群大约占69.57%,在SARA易感组奶牛中核心真菌微生物群大约占60.72%。T检验显示,SARA易感奶牛中的Sarocladium丰度较高,而Aspergillus和Papiliotrema的丰度则低于SARA耐受组奶牛(图9B)。此外,LEfSe还用于鉴定SARA耐受和SARA易感奶牛瘤胃液中真菌类群的丰度。可知在SARA易感组奶牛中富含Fusarium_oxysporum,SARA耐受组奶牛中Aspergillusgenus和Papiliotrema_laurentii较多(图9C,D)。这些结果表明,瘤胃中Sarocladium、Aspergillus、Papiliotrema_laurentii和Fusarium_oxysporum丰度的变化可能与SARA的易感性有关。

A.Ace真菌微生物区系丰富度;B.Shannon微生物多样性;C.SARA耐受奶牛和SARA易感奶牛瘤胃液真菌群落组成的PCoA显示未加权UniFrac度量差异;SARARes.SARA耐受奶牛;SARAPro.SARA易感奶牛

A.SARA耐受和SARA易感奶牛的瘤胃液中前10个门类的相对丰度;B.SARA耐受和SARA易感奶牛的瘤胃液中前10个属的相对丰度
3 讨论
近年来,世界范围内奶牛的产奶量大幅提高。同时,牧场经常通过增加饲料中精料的比例来满足奶牛泌乳的营养需求。由此,大量有机酸在瘤胃中积累,通常会诱发SARA的发生[21-22]。SARA的发生通常伴随着瘤胃微生物群和生理学的变化,诱发奶牛系统性代谢紊乱[23],最终导致奶牛业的重大经济损失[5,24]。近年来SARA奶牛的瘤胃内微生物群,包括细菌、古细菌、真菌和原生动物受到了广泛的关注。随着高通量测序技术的发展,健康奶牛和SARA奶牛的瘤胃微生物群落的差异已经被发现[25-26]。然而,奶牛本身的瘤胃细菌或真菌微生物群的结构是否与SARA的易感性有关还没有报道。事实上,了解SARA不同易感性背后的机制是非常重要的,特别是探索瘤胃微生物群在SARA发展中的作用,这将有助于开发奶牛SARA潜在有效的预防策略。因此,我们评估了SARA耐受和SARA易感奶牛瘤胃内细菌和真菌微生物群的差异。结果表明,瘤胃内细菌和真菌群落的差异,特别是unidentified_Spirochaetaceae、Anaeroplasma和Prevotella_ruminicola、Sarocladium、Aspergillus、Papiliotrema_laurentii和Fusarium_oxysporum丰度的变化可能与奶牛SARA的易感性有关。

A.Venn图显示了SARA耐受和SARA易感的奶牛之间瘤胃真菌群落中共享和独特属的数量; B.通过T检验分析检测了SARA耐受奶牛SARA易感奶牛在属水平上的真菌群落差异;C.LEfSe分析的线性判别分析(LDA)评分,显示了SARA耐受和SARA易感奶牛瘤胃真菌中的生物标志物分类群(LDA>3.5);D.LEfSe分析产生的支系图,表明分类群之间的关系属于门、类、目、科和属
瘤胃被认为是影响奶牛各种疾病发生和发展的最重要器官之一,瘤胃中的微生物群发挥着多种重要的生理功能。研究表明,接种健康奶牛的瘤胃液可以加速患瘤胃酸中毒的羊的瘤胃细菌群落平衡和瘤胃细菌功能的恢复[27]。因此,我们假设SARA易感性的不同可能与奶牛本身的瘤胃微生物群落的结构不同有关。α-多样性分析表明,SARA耐受和SARA易感奶牛之间的微生物群丰富度和真菌多样性存在差异。此外,与SARA耐受奶牛相比,SARA易感奶牛的瘤胃细菌中unidentified_Spirochaetaceae、Anaeroplasma和Prevotella_ruminicola的丰度,以及瘤胃真菌Sarocladium和Fusarium_oxysporum的丰度较高,而Aspergillus和、Papiliotrema_laurentii的丰度则较低。研究表明,螺旋体科与瘤胃中的纤维降解能力有关[28]。基于与瘤胃相关的微生物进行生物增殖以降解木质纤维素的研究表明,螺菌科未培养的成员在厌氧消化器中生产挥发性脂肪酸方面具有重要作用[29]。Anaeroplasma是由ROBINSOR等[29-30]首次从牛瘤胃液中分离出来的。它是一种革兰阴性细菌,属于支原体目,拥有催化酶,能够部分消化被杀死的革兰阴性细菌或酪蛋白。瘤胃Anaeroplasma需要脂多糖、胆固醇和可溶性淀粉,并且有能力产生乙醇和乳酸[30]。LAU等[31]提出,Anaeroplasma可以产生淀粉溶解酶,有助于淀粉酶的分解。WANG等[32]也表明,Anaeroplasma可以将糖类发酵成乙酸盐和甲酸盐,瘤胃液中Anaeroplasma的相对丰度与CH4的产生呈正相关。Prevotella_ruminicola是一种专性厌氧细菌,属于拟杆菌门,在瘤胃中非常突出,在蛋白质和肽的代谢中起着重要作用,并促进瘤胃中半纤维素的利用[33-35]。如上所示,SARA耐受和SARA易感奶牛瘤胃中的大多数有差异的细菌都与产酸有关。此外,Fusarium_oxysporum是镰刀菌属中的丝状真菌种类,是植物、动物和人类病原体之间的大型物种复合体,以宿主特异性方式攻击不同的物种[36-37]。Papiliotrema_laurentii是一种通常在土壤和植物中抑制的酵母,有能力水解聚酯涂层和涂层成分[38]。研究还表明,Papiliotrema_laurentii可以从菊粉水解物中积累细胞内脂质[39]。我们也知道,曲霉菌的一些种类有能力产生黄曲霉的次级代谢物[40]。BAN等[41]建议将酵母菌和曲霉菌直接作为反刍动物的饲料中的添加剂。Sarocladium是禾本科植物(Poaaceae)的内生菌,是本地草原的主要科。有证据表明,瘤胃中Sarocladium的丰度越高,牛的体质量就越高[42]。然而,unidentified_Spirochaetaceae、Anaeroplasma、Prevotella_ruminicola、Sarocladium、Aspergillus、Papiliotrema_laurentii、Fusarium_oxysporum在SARA易感性中是如何发挥作用的还有待进一步研究。
总之,本研究表明,SARA易感性可能与瘤胃微生物群的结构差异有关。瘤胃中大量的unidentified_Spirochaetaceae、Anaeroplasma、Prevotella_ruminicola、Sarocladium、Aspergillus、Papiliotrema_laurentii、和Fusarium_oxysporum的变化可能是导致SARA易感性增加的原因。然而,这需要对这些细菌和真菌进行分离和培养,并深入验证它们在奶牛SARA易感性中的作用。
