基于Mask RCNN和U-Net结合的三阶段肾脏与肿瘤分割方法
2022-06-15周少飞秦品乐武志芳
周少飞, 柴 锐, 秦品乐, 武志芳
(1. 中北大学 大数据学院, 山西 太原 030051;2. 生物医学成像与影像大数据山西省重点实验室, 山西 太原 030051;3. 山西医科大学第一医院 核医学科, 山西 太原 030001)
0 引 言
肾脏肿瘤是影响人体泌尿生殖系统健康的恶性肿瘤之一[1]. 临床治疗中, 根治性肾切除手术是最常用的方式, 但存在术后因肾功能缺失而造成生活不便的问题, 所以, 保留肾单位的手术被广泛应用于肾脏肿瘤治疗中[2], 这就要求进行手术之前, 得到肾脏与肿瘤的准确分割结果[3]. 但是, 在腹部扫描图像的成像中存在的组织细节对比度较低、 肾脏与肿瘤形状不规则都会对其精确分割造成困难[4].
在传统分割方法中, 更关注肾脏和肿瘤作为整体的分割, 且大多数都是利用基于水平集、 上下文信息等的方法来定位肾脏位置, 再通过设定阈值、 随机森林等做进一步分割[5-6]. 这些方法不仅速度慢而且精度低, 不适合在临床中使用, 所以对更高效精准的自动分割方法的需求非常迫切. 随着深度学习的发展, 用于图像分割的方法层出不穷. 在自然图像分割领域, He等[7]提出的Mask RCNN表现出了极优的性能, 现已逐步将其应用在医学图像的检测和分割中, 但对医学图像这种数据量少的数据集来说, 其分割效果并不理想. 对于增强CT中肾脏与肿瘤的分割, 基于深度学习方法的应用也日益广泛, Cruz等[8]先对切片进行分类, 然后再利用2D U-Net网络分割肾脏, 该方法没有将肿瘤单独进行分割; Tsai等[9]提出了一种两阶段的分割方法, 先将肾脏与肿瘤作为整体进行分割, 得到感兴趣的区域后再对其进行细分; Guo等[10]先利用阈值获取肾脏中感兴趣的区域, 再使用加入残差和注意力的U-Net进行肾脏与肿瘤的分割, 最后利用条件随机场对分割结果进行校正, 合适的后处理方式对分割精度的提升有一定作用. 这些分割方法都基于多阶段的方式, 提取感兴趣的区域后再对肾脏及肿瘤进行分割, 有效地提高了分割精度. Zhao等[11]提出基于3D U-Net的自动分割方法, 在训练的过程中加入多尺度监督来提高学习效率, 但是该方法是在3D U-Net架构能提取足够的特征的假设下提出的. 3D U-Net等3D网络虽然能充分利用图像信息, 但因为医学图像较自然图像难采集, 数据量较少, 加上硬件设备性能的限制, 直接使用3D数据易造成过拟合的问题[12].
本文提出基于Mask RCNN和U-Net结合的三阶段方法来实现增强CT中肾脏与肿瘤的分割, 第一阶段, 汇总包含肾脏的轴向断层切片序列, 即识别含有肾脏的断层切片, 以缩小肾脏范围, 该阶段并不涉及肾脏和肿瘤的具体分割; 第二阶段, 对上一阶段选定的断层序列进行训练, 实现肾脏与肿瘤的分割; 第三阶段, 对肿瘤进行分割, 再将第二、 第三阶段的结果融合. 第二、 第三阶段训练数据的输入形式为2.5D, 向网络传递沿通道堆叠在一起的相邻轴向断层切片, 既可以提供每张断层切片内部的上下文信息, 又可以提供几张断层切片堆叠组成的体积信息. 通过不断缩小目标断层切片范围使得分割更关注于肾脏及肿瘤的特征信息, 同时在非肾脏区域减少假阳性. 按照多阶段的方式进行分割来提高最终的分割精度.
1 本文方法
本文方法总体流程如图 1 所示, 包括: 1) 数据预处理. 对图像进行窗宽窗位调整及归一化操作, 以提高组织的对比度. 2) 汇总包含肾脏的断层切片. 利用Mask RCNN网络从输入图像中识别肾脏, 汇总包含肾脏的断层切片序列. 3) 肾脏与肿瘤分割. 利用基于U-Net的网络对选定序列进行肾脏与肿瘤的分割, 然后汇总包含肿瘤的断层序列. 4) 肿瘤分割, 结果优化. 对选定序列进行肿瘤分割, 对结果进行优化, 然后与上一阶段肾脏分割的结果融合. 5) 结果后处理. 利用基于三维连通域的方法对分割结果进一步优化.
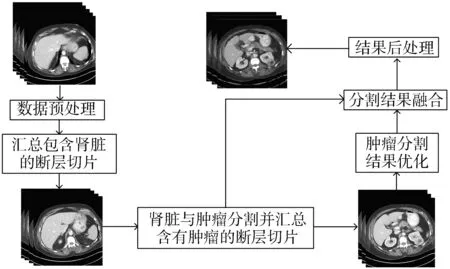
图 1 本文方法流程图
1.1 预处理
为了使图像中的目标组织细节得到增强, 首先对其窗宽窗位进行设置, 具体为: 将CT值大于500 HU(Hounsfield Unit)的值设置为500 HU, CT值小于-200 HU的值设置为-200 HU, 接着归一化至区间[0,1], 可表示为
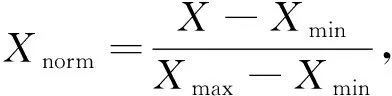
(1)
式中:Xmin为CT值中的最小值;Xmax为CT值中的最大值. 数据预处理效果如图 2 所示, 图像中组织的对比度得到提高.
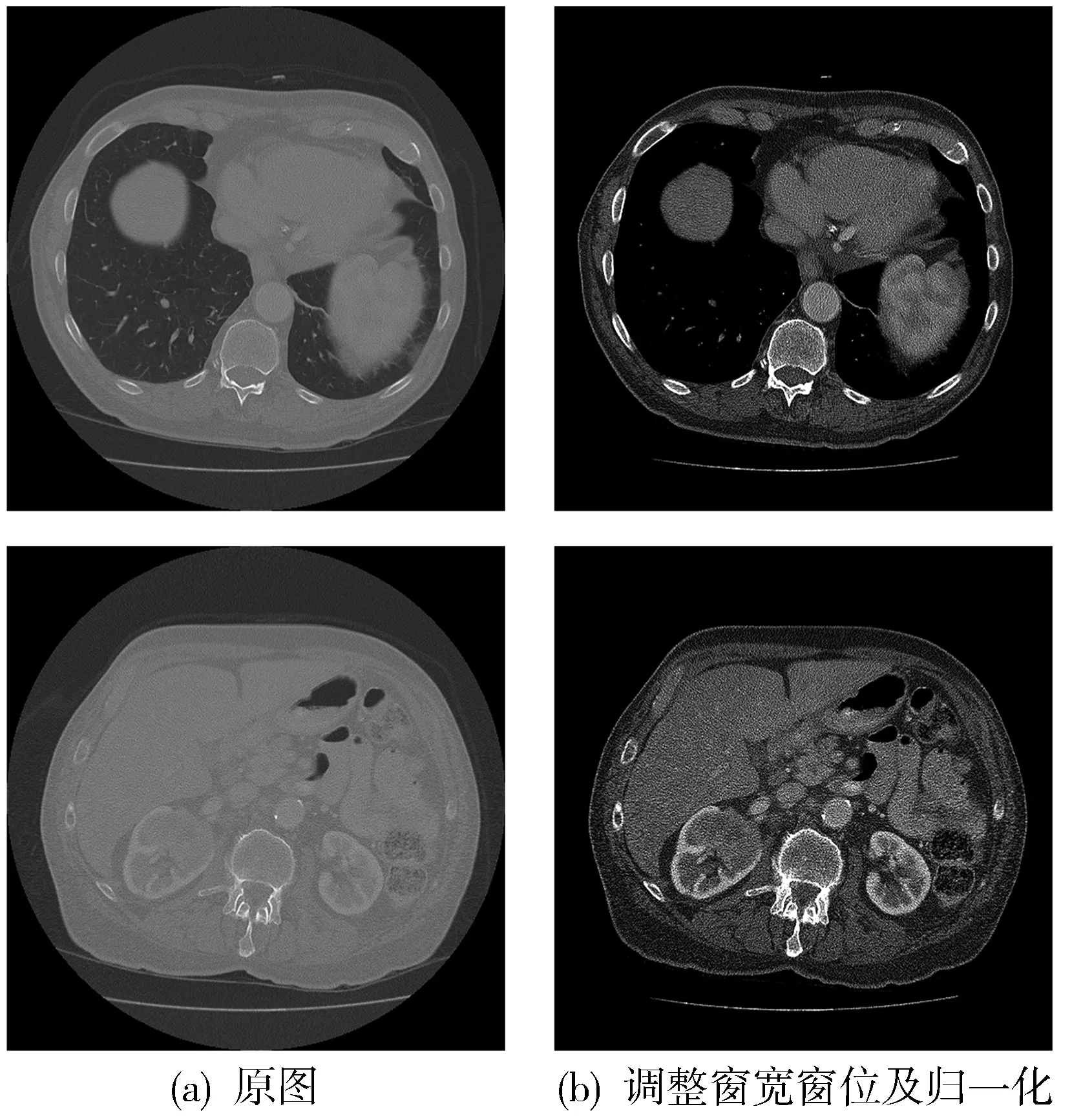
图 2 数据预处理效果对比
1.2 第一阶段: 汇总肾脏断层切片序列
该阶段实现含有肾脏断层切片序列的汇总, 只需要识别断层切片中是否含有肾脏. 在CT图像中, 肾脏只存在于某些连续断层里, 通过将汇总得到的包含肾脏的序列输入下一阶段进行分割, 减少非肾脏断层内的假阳性对结果的影响, 利于最终的分割. 该阶段利用Mask RCNN网络[7]对图像中的肾脏进行识别, 网络结构如图 3 所示, 其中采用预训练的resnet50-fpn[13]作为主干网络, 通过主干网络提取特征得到特征图, 根据特征图生成候选感兴趣区域(Region of Interest, ROI), 然后将ROI送入区域生成网络(Region Proposal Network, RPN)进行二值分类, 过滤掉一部分不合适的ROI, 接着对剩余的ROI进行ROI Align操作, 最后对其进行分类(背景、 肾脏)、 边界框回归和mask生成. 具体是将图像标签作为mask, 每张图像中mask的最小外包矩形作为边界框, 最终在图像上可视化输出概率最大的边界框. 在该过程中将肾脏与肿瘤作为整体进行识别, 第一次出现边界框的断层被认为是肾脏开始的断层, 边界框结束的断层被认为是肾脏结束的断层, 得到这两个断层的层数即得到包含肾脏的断层序列. 在腹部CT图像中, 肾脏是连续的, 所以对每一个患者的CT图像, 只要找到肾脏开始的断层数和肾脏结束的断层数, 就认为两者之间的所有断层切片都含有肾脏, 记录这两个数据, 将得到的结果作为下一阶段输入网络训练的序列.
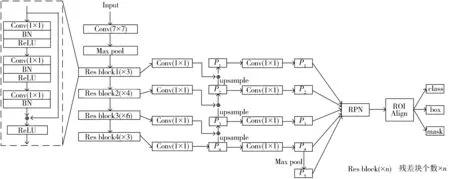
图 3 Mask RCNN网络结构
1.3 第二阶段: 肾脏与肿瘤分割
该阶段对肾脏与肿瘤进行分割, 为了得到更好的分割结果, 希望在训练的过程中可以将提取到的特征信息更完整地传递下去, 该阶段的输入数据为前一阶段汇总的断层序列, 且为整幅图像. 借鉴文献[9] 的网络结构, 本文使用增加密集模块(Dense block)的U-Net网络, 同时在上采样时使用双三次插值. 密集模块如图 4(a) 所示, 其特点是在同一模块中, 每层都与之前所有层连接, 同时也与之后所有层建立连接, 特征在每个连接中得到传递, 它们在通道维度上进行叠加, 可表示为
xl=Ml([x0,x1,…,xl-1]),
(2)
式中:xl表示第l层的输出;M表示非线性变换; [x0,x1,…,xl-1]表示将0到l-1层的特征叠加.
该阶段总体网络结构如图 4 所示, 下采样层采用预训练的densenet161[14]实现特征提取. 在每个密集模块之后采用过渡层(Transtion layer), 其结构如图 4(b) 所示, 包含卷积和平均池化, 可以使特征在不同的密集模块之间传递, 从而减小特征图的尺寸. 在上采样的过程中, 得到的特征图通过跳跃连接与下采样得到的特征图结合, 逐渐增大图像尺寸, 上采样层中使用双三次插值, 使得特征图在该过程中保留更多的特征信息, 利于最终的分割.
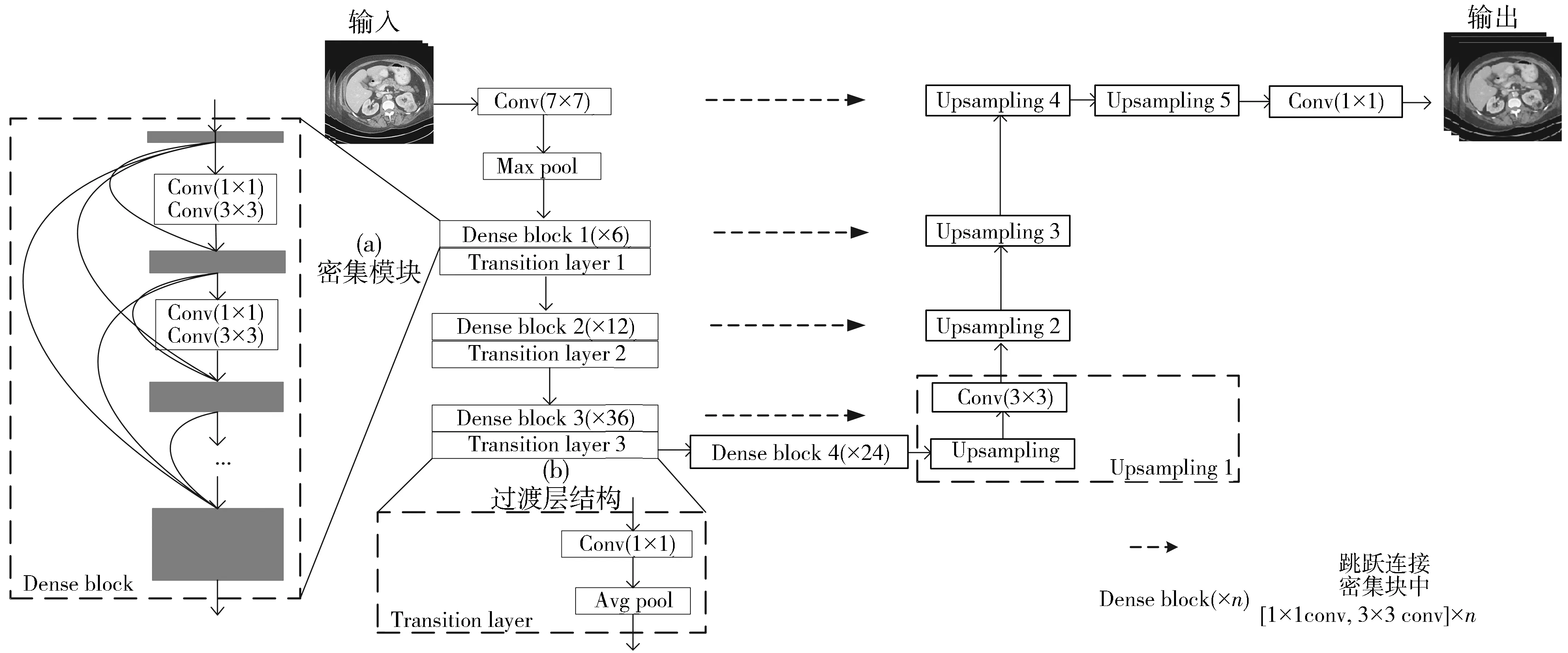
图 4 网络结构
第一、 第二阶段输入的断层切片序列说明如图 5 所示.
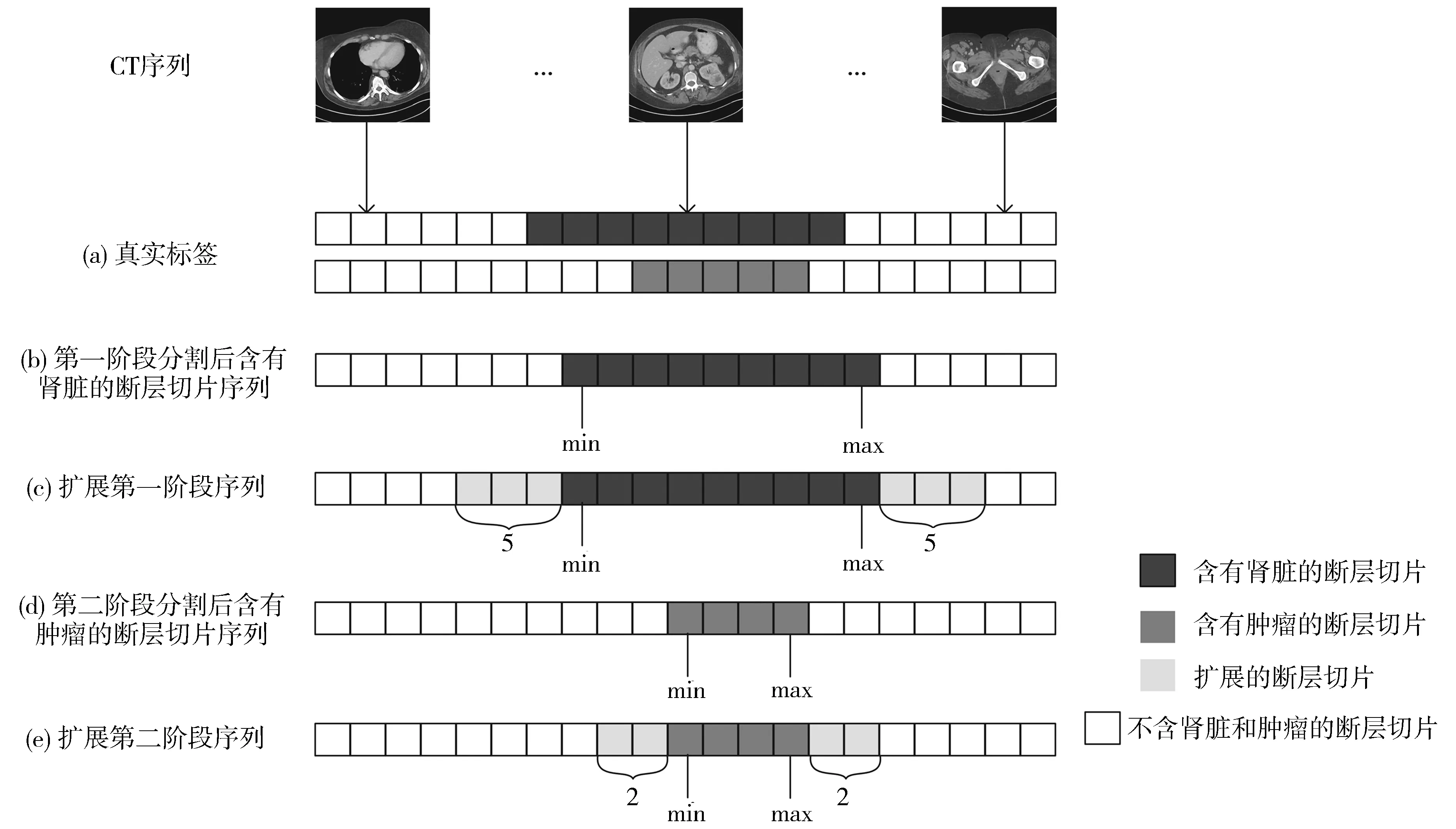
图 5 第一、 第二阶段相关断层切片说明
关于图 5 具体说明如下:
1) (a)表示CT序列中真实包含肾脏和包含肿瘤断层切片的情况.
2) (b)表示第一阶段结束后, 分割得到的包含肾脏的断层切片序列情况. 其中, min表示肾脏开始出现时的断层数, max表示肾脏结束时的断层数.
3) (c)表示对第一阶段得到的分割结果进行扩展后的断层切片序列, 即第二阶段进行肾脏与肿瘤分割时输入的序列. 为减少因为肾脏两端未识别造成的误差, 这里对两端断层切片数量进行扩展, 对比第一阶段汇总的断层序列和真实含有肾脏的断层序列, 当扩展的断层切片数设置为5时, 可以弥补因未识别造成的误差, 所以下一阶段输入训练的断层序列为((min-5),(max+5)).
4) (d)表示第二阶段结束后, 分割得到的包含肿瘤的断层切片序列, 其中min表示肿瘤开始出现时的断层数, max表示肿瘤结束时的断层数.
5) (e)表示对第二阶段得到的序列进行扩展后的结果, 即第三阶段进行肿瘤分割时输入的断层切片序列. 同样为了减少因肿瘤两端未识别造成的误差, 通过与实际包含肿瘤的断层序列对比后, 扩展两端断层的数量, 得到下一阶段输入训练的断层序列((min-2),(max+2)).
1.4 第三阶段: 肿瘤分割
该阶段对肿瘤进行分割, 将肾脏视为背景, 具体将上一阶段得到的包含肿瘤的断层序列作为输入, 且为整幅图像, 单独对肿瘤进行分割, 减少非肿瘤区域对分割的影响, 提高肿瘤的分割精度. 该阶段需要对肿瘤的特征信息进行充分提取, 并使特征尽可能得到保留, 所以该阶段选择使用与第二阶段一致的网络, 最终输出肿瘤的分割结果. 先对结果进行后处理操作, 然后再将第二阶段肾脏、 肿瘤分割结果与第三阶段肿瘤分割结果融合, 作为最终的结果.
1.5 后处理
得到第三阶段肿瘤分割的结果和第二、 第三阶段肾脏肿瘤融合的结果之后, 采用基于三维连通域分析的方法去除假阳性区域. 在本文的分割背景下, 连通域是指CT图像中肾脏或肿瘤区域, 连通域分析是指找到肾脏或肿瘤区域并按要求进行标记. 具体标记连通区域并分析时, 利用腹部CT序列图像是三维的特点, 对检测到的每个标记对象组成的区域体积进行计算. 在去除肿瘤分割结果中的假阳性区域时, 通过对肿瘤体积进行分析, 将区域体积小于最大区域体积0.9倍的部分移除能得到更好的结果; 去除第二、 第三阶段融合后的结果中假阳性区域时, 选择大于阈值的连通区域作为最终分割结果, 在本文中, 阈值设置为50 000.
1.6 损失函数
第一阶段汇总含有肾脏的切片序列, 损失函数计算式为
L=Lcls+Lbox+Lmask,
(3)
Lcls表示分类损失, 计算公式为
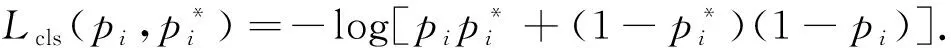
(4)
Lbox表示边界框回归损失, 计算公式为
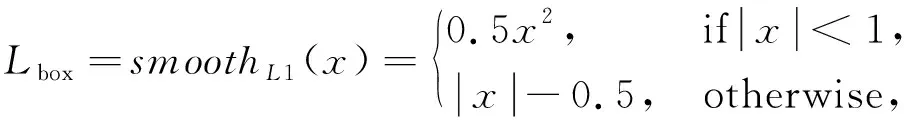
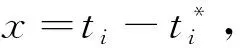
(5)
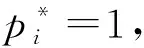
Lmask表示掩码像素分割损失, 通过对像素使用Sigmoid函数, 然后取感兴趣区域中的平均交叉熵得到.
第二阶段实现肾脏与肿瘤的分割, 使用一般Dice损失(Generalized Dice Loss, GDL). GDL的计算公式为
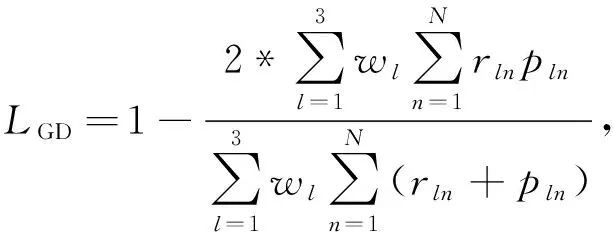
(6)
式中:rln为类别l在第n个像素的真实值;pln为相应的预测结果的概率值;N表示每张图片中的所有像素的数量;wl为每个类别的权重.
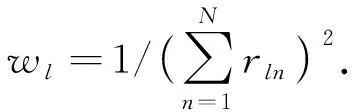
(7)
第三阶段实现肿瘤分割, 使用Dice损失, 计算公式为
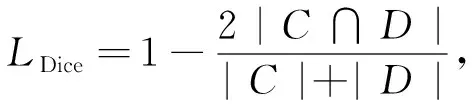
(8)
式中:C为数据集给出的分割结果中肿瘤的像素计数;D为模型预测结果中肿瘤的像素计数. 分母中有重复计算的元素, 所以分子中系数为2.
2 实验结果与分析
2.1 数据集
本文使用的数据为开源数据集(KiTS19[15]), 包含2010年至2018年间接受根治性肾切除或部分肾切除手术的患者腹部的增强CT图像. 在本文中, 主要使用210例(45 965张)患者CT序列图像, 每个序列都由二维扫描图像组成, 且都有完整的肾脏及肿瘤标注信息, 将其按照一定的比例划分为训练集(147例, 31 983张)、 验证集(33例, 8 071张)、 测试集(30例, 5 911张). 最后将训练好的模型在从医院采集到的613张CT图像上进行分割及可视化.
2.2 实验设备及参数设置
使用NVIDIA Tesla V100 32GB图形处理器(Graphic Processing Unit, GPU)进行模型训练, 同时使用中央处理器(Central Processing Unit, CPU)进行测试. 基于服务器的配置情况, 在保证能够正常训练的前提下, 第一阶段初始学习率设置为0.005, 动量因子设置为0.9; 第二、 第三阶段初始学习率设置为0.000 1, 每阶段都设置51个epoch, 每3个epoch进行一次验证, 网络结构由Pytorch实现.
2.3 实验指标与结果分析
第一阶段使用以下定量指标来评价公开数据集中结果的准确度: 召回率(Recall)、 精准率(Precision).
召回率的计算公式为
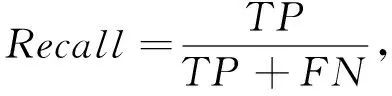
(9)
式中:TP表示正确预测为肾脏部分;FN表示被错误预测为背景的部分.
精准率的计算公式为
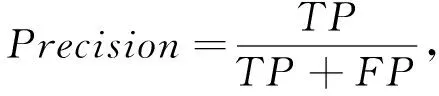
(10)
式中:FP表示将背景错误预测为肾脏的部分.
第二阶段肾脏与肿瘤分割及第三阶段肿瘤分割, 采用以下定量指标来评价分割结果的准确度及精度: Dice系数(Dice coefficient)、 杰卡德系数(Jaccard)、 召回率(Recall)、 精准率(Precision).
Dice系数的计算公式为
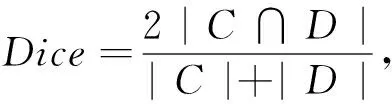
(11)
式中:C为数据集给出的分割结果中肾脏或肿瘤的像素计数;D为模型预测结果中肾脏或肿瘤的像素计数; Dice系数值介于0和1之间, 其值越大, 表明分割与真实值越接近.
杰卡德系数的计算公式为
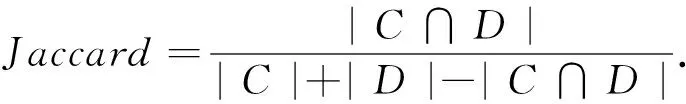
(12)
使用本文提出的方法进行实验, 选取147例训练, 33例验证, 30例测试; 第一阶段对边界框回归的实验结果如表 1 所示; 第二、 第三阶段实验结果如表 2 和表 3 所示. 表 2 所示数据为30例测试集上分割的全局结果, 表 3 所示数据为30例测试集上分割的平均结果, 可以看出, 在第三阶段中肿瘤分割的结果有明显提高.

表 1 第一阶段在测试集上的实验结果

表 2 第二、 第三阶段在测试集上所得实验结果的全局值
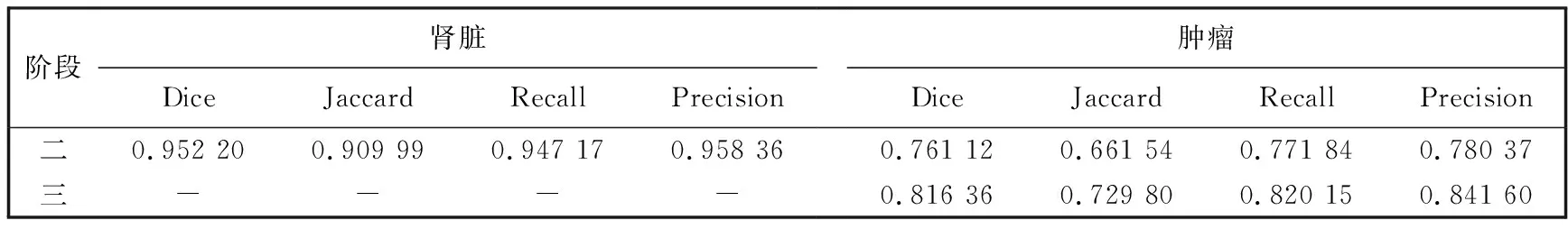
表 3 第二、 第三阶段在测试集上所得实验结果的平均值
对本文方法与其他不同网络结构及方法在数据集上的实验结果进行比较, 结果如表 4 所示. 实验中使用的模型均采用2.5D的输入方式进行, 分割结果为测试集上的平均值. 由表 4 可以看出, 相比于第二阶段使用U-Net网络, 将卷积层替换为残差连接的ResU-Net网络[9]及文献[9]中的两阶段分割方法, 本文方法在定量指标分析上均取得较好结果, 同时增加后处理可以提高分割精度. 每种方法在处理同一序列图像上的耗时分别约为: 135.6 s, 150.2 s, 181.5 s, 192.3 s, 因为本文方法相较其他方法而言模型较为复杂, 在耗时上稍有增加, 但在分割精度上有较大提升, 所以, 本文方法在未增加太多时间消耗的情况下有效提高了肾脏和肿瘤的分割精度.
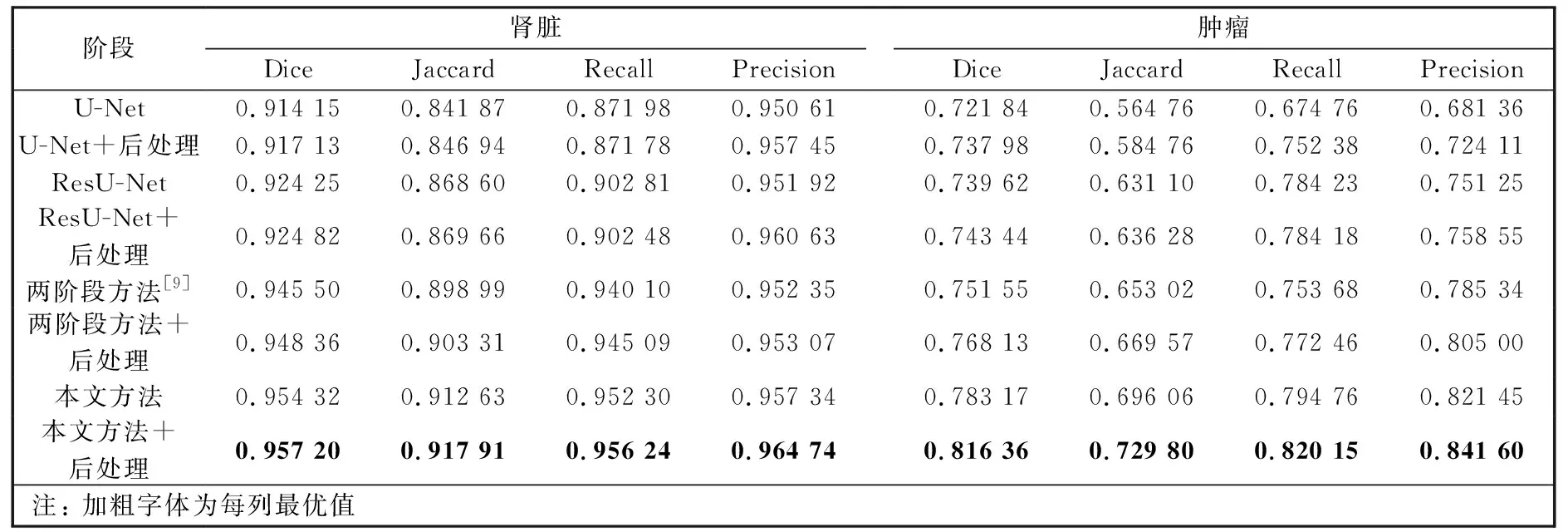
表 4 不同方法所得结果平均值的对比
图 6 为每一阶段分割结果的可视化展示, 浅色表示肾脏分割结果, 深色表示肿瘤分割结果.
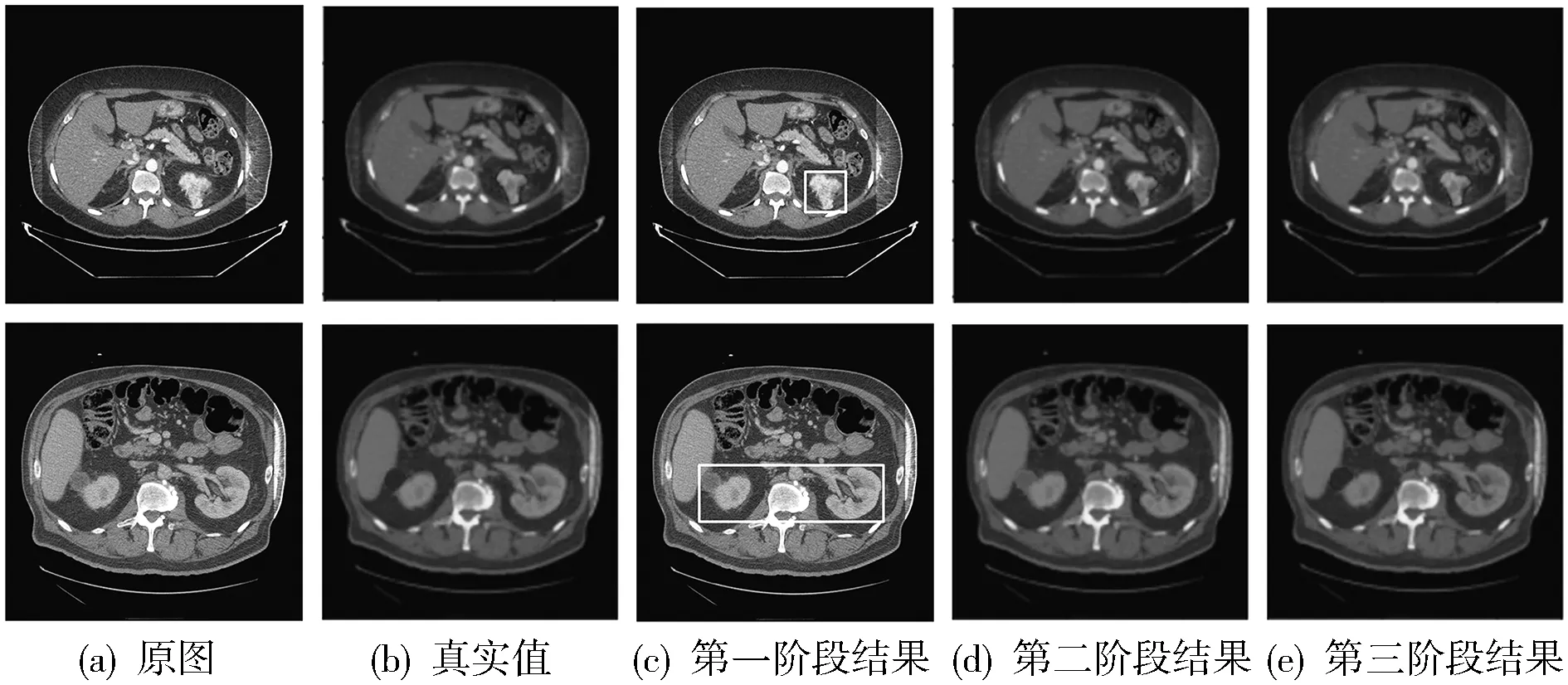
图 6 每阶段结果展示
第一阶段识别断层切片中是否含有肾脏, 通过边界框的有无判断是否含有肾脏; 第二阶段对肾脏和肿瘤进行分割; 第三阶段分割肿瘤. 可以看出, 第三阶段结果中肿瘤分割比第二阶段肿瘤分割结果更为准确, 所以最后将二、 三阶段结果融合后可以获得更好的分割结果.
图 7 为不同方法在公开数据集中对肾脏与肿瘤分割的可视化对比. 可以看出, 其他方法中肾脏和肿瘤的边界不准确, 且存在错误分割的情况, 而本文方法可以得到更准确的肾脏及肿瘤边界, 获得更符合真实标签的分割结果, 说明本文方法能有效进行肾脏和肿瘤分割.
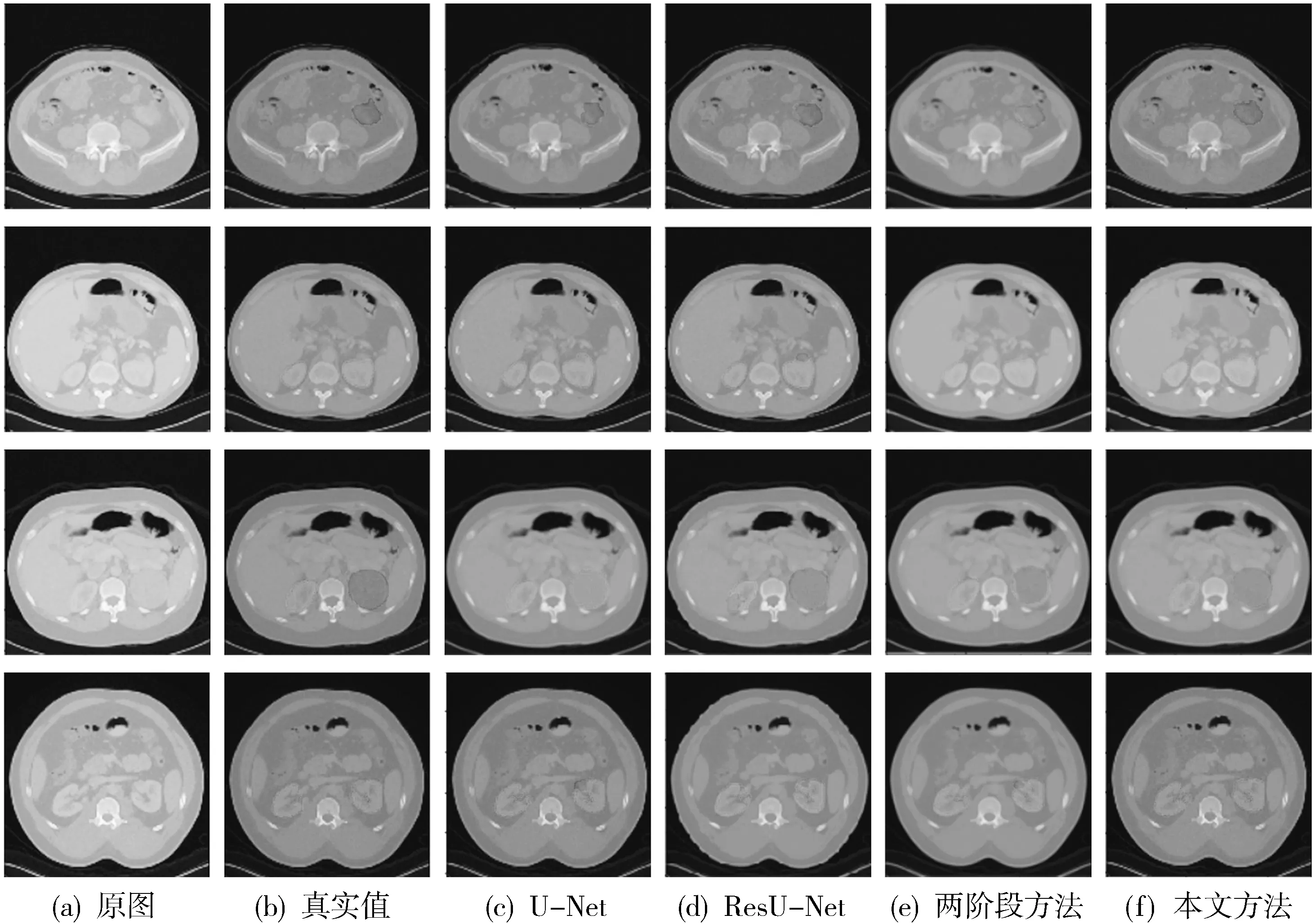
图 7 各方法对公开数据集中测试数据分割的结果
利用本文方法对医院采集的CT图像进行分割及可视化测试, 先对数据进行预处理, 包括调整窗宽窗位和像素值归一化, CT值具体设置为[-200, 500]HU, 归一化区间为[0, 1]. 分割可视化结果如图 8 所示, 可以看出, 本文方法分割结果更好, 得到的肾脏与肿瘤的平均Dice分别为 0.947 96, 0.796 35, 说明本文方法具有一定的泛化性能.

图 8 各方法对医院采集测试数据分割的结果
3 结 论
本文提出基于Mask RCNN和U-Net结合的三阶段分割方法, 用于分割增强CT图像中的肾脏与肿瘤. 主要采用多阶段的方式实现, 通过不断缩小CT图像中目标断层切片的范围来获得更利于最终分割的特征信息. 在训练网络时, 使用2.5D的数据输入方式, 能够在充分利用断层切片的片内特征信息和体积信息的同时缓解硬件设备的压力. 本文还针对肿瘤分割结果和融合结果进行了合适的后处理操作, 以得到更优的分割结果. 但还存在一定的局限性, 如: 需要训练3个模型, 不能够实现端到端训练. 今后的研究会对网络进行适应性调整, 实现更优的深度学习分割算法, 以满足临床应用的需要.
