基于生物信息学的胃部“炎癌转化”过程中关键基因的筛选及其作用**
2022-06-04袁蕴馨付佳音莫非1何芸
袁蕴馨, 付佳音, 莫非1,***, 何芸
(1.贵州医科大学附属医院 临床检验中心, 贵州 贵阳 550004; 2.贵州医科大学 医学检验学院, 贵州 贵阳 550004)
胃癌是全球常见的消化道恶性肿瘤之一,起源于胃黏膜上皮[1-2]。流行病学调查显示,胃癌发病率位居全球肿瘤发病率的第5位,每年全球有超过100万人诊断为胃癌,且病死率居全球第6,严重威胁人类的健康安全[3-4]。研究表明,慢性溃疡型结肠炎、结肠癌、幽门螺杆菌相关的慢性胃炎及胃癌等疾病与长期的非可控性慢性炎症相关[5]。在慢性胃炎向胃癌进展的过程中,存在Correa经典级联反应模式,即慢性非萎缩性胃炎→慢性萎缩性胃炎→肠上皮化生→异型增生或上皮内瘤变→肠型胃癌的进展趋势[6]。由于异型增生或上皮内瘤变有较高的癌变风险,因此称该阶段为癌前病变[7]。临床上常用的早期胃癌筛查手段,如血清标志物、胃蛋白酶等特异性较低,检出率不高,且因胃癌的恶性程度高、侵袭性强,大多数病例诊断时已为晚期[8-9]。尽管各项治疗手段已经取得了进展,但胃癌的预后仍然较差, 在全球的5年生存率低于30%[10]。随着高通量测序技术的高速发展,兴起了以基因芯片表达数据谱为依据、对不同疾病的各种层面开展研究的热潮[6]。目前,基因芯片测序技术广泛用于疾病的检测,Li 等[10]通过对基因芯片表达数据谱的分析,推测了胃癌的发生和发展在表观遗传学中的调节机制,明确枢纽(Hub)基因可作为胃癌诊断的生物标志物,并且具有一定的预后价值;Zhang等[11]通过综合分析5个与胃癌相关的数据表达谱,筛选出与胃癌相关的Hub基因。研究结果显示这些Hub基因在胃癌组织中高度表达,且其过表达与胃癌患者生存率低相关,但目前尚未有研究报道关键Hub基因在胃部“炎癌转化”过程中的作用机制。因此,本研究旨在利用生物信息学分析,对“炎癌转化”发展过程进行系统的分子生物学研究,尤其是对驱动癌变各个阶段进展的分子事件进行分析,以挖掘非侵入性的潜在分子诊断标志物,为延缓、阻断“炎癌转化”进程,提高胃癌早期诊断率提供新思路,并为探索“炎癌转化”进展的分子机制提供依据。
1 材料与方法
1.1 实验材料
1.1.1数据来源 从美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)下的GEO数据库中,以胃炎、胃癌前病变和胃癌为关键词筛选出GSE130823、GSE55696两个微阵列数据集作为挖掘对象。GSE130823是由Zhang 等[11]利用Agilent公司的8×60 K芯片检测的数据,其实验平台为GPL17077,其中包括47例胃炎对照组、17例胃低级别上皮内瘤变、14例胃高级别上皮内瘤变、16例胃肠癌;GSE55696是Xu等[12]利用Agilent公司的4×44 K G4112F芯片检测的数据,其实验平台为GPL6480,其中包括19例慢性胃炎对照组、19例低级别上皮内瘤变、20例高级别上皮内瘤变及19例早期胃癌。
1.1.2标本来源 选取2020年2月—2022年2月有明确病理诊断的胃炎和胃癌患者各15例,要求符合:(1)所有患者皆签署知情同意书;(2)胃镜活检取材的样本直径>2 mm;(3)经病理学分析证实为胃炎及胃癌,且为初次诊断;(4)既往无癌症史;(5)患者未接受过任何治疗(如激素治疗、靶向药物治疗或放疗等辅助治疗)。排除标准:(1)行放疗化疗或全身治疗等辅助治疗的患者;(2)有源于其他肿瘤转移或者合并其他肿瘤可能的患者;(3)有严重感染、凝血障碍及严重肺、肝、肾功能不全的患者等。取胃炎患者胃炎部位的胃组织、胃癌患者的胃癌组织及癌旁组织作为研究标本。
1.1.3主要试剂及仪器 蛋白裂解液(RIPA lysis buffer,RIPA)裂解液、聚丙烯酰胺凝胶电泳(polyacrylamide gelelectrophoresis,SDS-PAGE)凝胶制备试剂盒(上海爱必信生物),辣根过氧化物酶(horseradish peroxidase, HRP)标记羊抗兔免疫球蛋白G(immunoglobulin G,IgG)抗体、RNA提取液、Servicebio®RT First Strand cDNA Synthesis Kit(武汉赛维尔生物),兔抗人H,K-ATPase-α(ATP4A)抗体(美国LSBio),HyPureTMMolecular Biology Grade Water(美国HyClone),低温离心机(美国Beckman),兔抗人紧密连接蛋白3(claudin 3,CLDN3)抗体、蛋白定量(bicinchoninic acid assay,BCA)检测试剂盒(沈阳万类生物),荧光定量聚合酶链式反应(polymerase chain reaction,PCR)仪(美国Bio-rad)。
1.2 实验方法
1.2.1差异表达基因(differentially expressed genes,DEGs)的获取及分析 使用差异分析工具(gene expression omnibus 2R,GEO2R)在线分析工具对GSE130823、GSE55696进行差异表达分析。设置P值和差异倍数(fold change,FC)筛选DEGs。当P<0.05,|Log2FC|>1时差异有统计学意义;利用Draw Venn Diagram(http://bioinformatics.psb.ugent.be/webtools/Venn/)在线韦恩图绘制工具,取2个数据集中重叠的DEGs。
1.2.2功能注释分析 为了从阐明DEGs在“炎癌转化”过程当中所发挥的作用,使用注释、可视化综合发现数据库(the database for annotation, visualization and integrated discovery,DAVID; https://David.Ncifcrf.gov/) 在线工具对交集DEGs从生物过程(biological process,BP)、细胞组成(cellular component,CC)及分子功能(molecular function,MF)3个角度进行基因本体论(gene ontology,GO)分析及京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)富集分析,设置参数P<0.05有统计学意义。
1.2.3蛋白互作( protein protein interaction,PPI) 网络的构建 将重叠DEGs导入交互式基因检索工具(search tool for recurring instances of neighbouring genes,STRING)中进行蛋白质相互作用分析,采用Cytoscape(https://cytoscape.org/)软件将 PPI 网络可视化后,使用cytoHubba插件,以degree值为条件,从 PPI 网络中筛选出排名前10的模块,并将该模块中的基因作为后续研究对象。
1.2.4Hub基因的生存分析 通过Kaplan Meier Plotter(http://kmplot.com/analysis/) 在线软件对排名前5的Hub基因进行生存分析,评价其在胃癌患者中预后判断的价值。
1.2.5Hub基因的验证 (1)采用逆转录-实时荧光定量聚合酶链式反应(quantitative and reverse transcription polymerase chain reaction,qRT-PCR)验证degree值排名靠前的2个Hub基因在胃炎、胃癌组织及癌旁组织中的表达。TRIzol法提取胃炎组织、胃癌组织及癌旁组织中总RNA,并使用Nanodrop 2000检测RNA浓度及纯度;使用Servicebio®RT First Strand cDNA Synthesis Kit将总RNA逆转录为互补脱氧核糖核酸(complementary DNA, cDNA)后用于qRT-PCR,引物序列见表1;(2)Western blot验证degree值排名靠前的2个Hub基因编码的蛋白在癌旁组织、胃炎组织和胃癌组织中的表达:使用RIPA裂解癌旁组织、胃炎组织及胃癌组织,并提取总蛋白,采用BCA法对其进行蛋白定量并测定蛋白浓度;进行SDS-PAGE凝胶电泳,200 mA 恒定电流转模1.5 h,5%脱脂牛奶封闭2 h,按照1 ∶1 000的比例配制ATP4A一抗稀释液,1 ∶500的比例配制CLDN3一抗稀释液,加入抗体孵育盒中,于4 ℃孵育过夜,使用杂交膜清洗液(TBS with tween-20,TBST)漂洗后放于封闭液中,室温震荡封闭2 h;TBST洗膜3次,二抗于室温下孵育1 h;TBST洗膜3次,曝光。

表1 引物序列Tab.1 Primer sequences
1.3 统计学分析
采用SPSS 24. 0软件进行统计分析,组间比较采用t检验,用Kaplan-Meier Plotter 数据库分析Hub基因与胃癌患者预后的关系,P<0.05表示差异有统计学意义。
2 结果
2.1 DEGs筛选
使用Sangbox软件对GSE130823、GSE55696进行标准化处理(图1)。利用GEO2R在线分析工具结果表明,GSE130823筛选出差异表达基因312个,GSE55696筛选出差异表达基因2 493个,二者交集基因有113个,其中73个上调,40个下调(图2)。
注:A、B分别为GSE130823标准化前、后数据,C、D分别为GSE55696标准化前、后数据;蓝色表示标准化前的数据分布,红色表示标准化后的数据分布。图1 GSE130823及GSE55696微阵列数据集的标准化处理Fig.1 Normalization of GSE130823 and GSE55696 microarray datasets
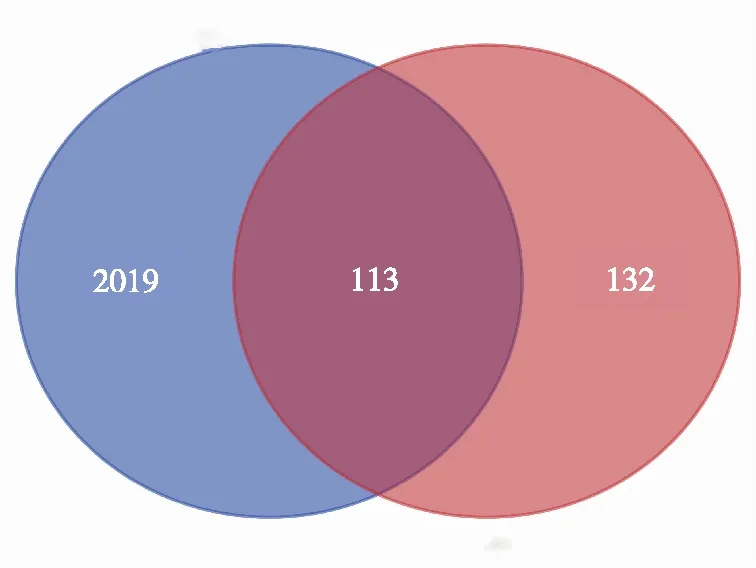
注:蓝色表示GSE55696数据集,红色表示GSE130823数据集,红蓝色交接处表示重叠DEGs。图2 重叠DEGs筛选结果Fig.2 Overlapping DEGs screening results
2.2 重叠DEGs功能注释分析
BP变化的DEGs主要富集在G蛋白偶联(G-protein coupled receptor signaling pathway)、蛋白质分解(proteolysis)、细胞间连接(cell adhesion)等方面。CC变化的DEGs显著定位在细胞膜的有机组成部分(extracellular space)、细胞基质(plasma membrane)、细胞外空间(integral component of membrane)等区域;MF变化的差异基因富集在序列特异性DNA结合(sequence-specific double-stranded DNA binding)、受体结合(receptor binding)、分子结构活性(structural molecule activity)等功能上(图3)。KEGG通路富集分析显示DEGs主要富集在:(1)胃酸分泌(gastric acid secretion);(2)精氨酸和脯氨酸代谢(arginine and proline metabolism);(3)蛋白质的消化与吸收(protein digestion and absorption);(4)细胞粘附(cell adhesion)等通路上(图4)。
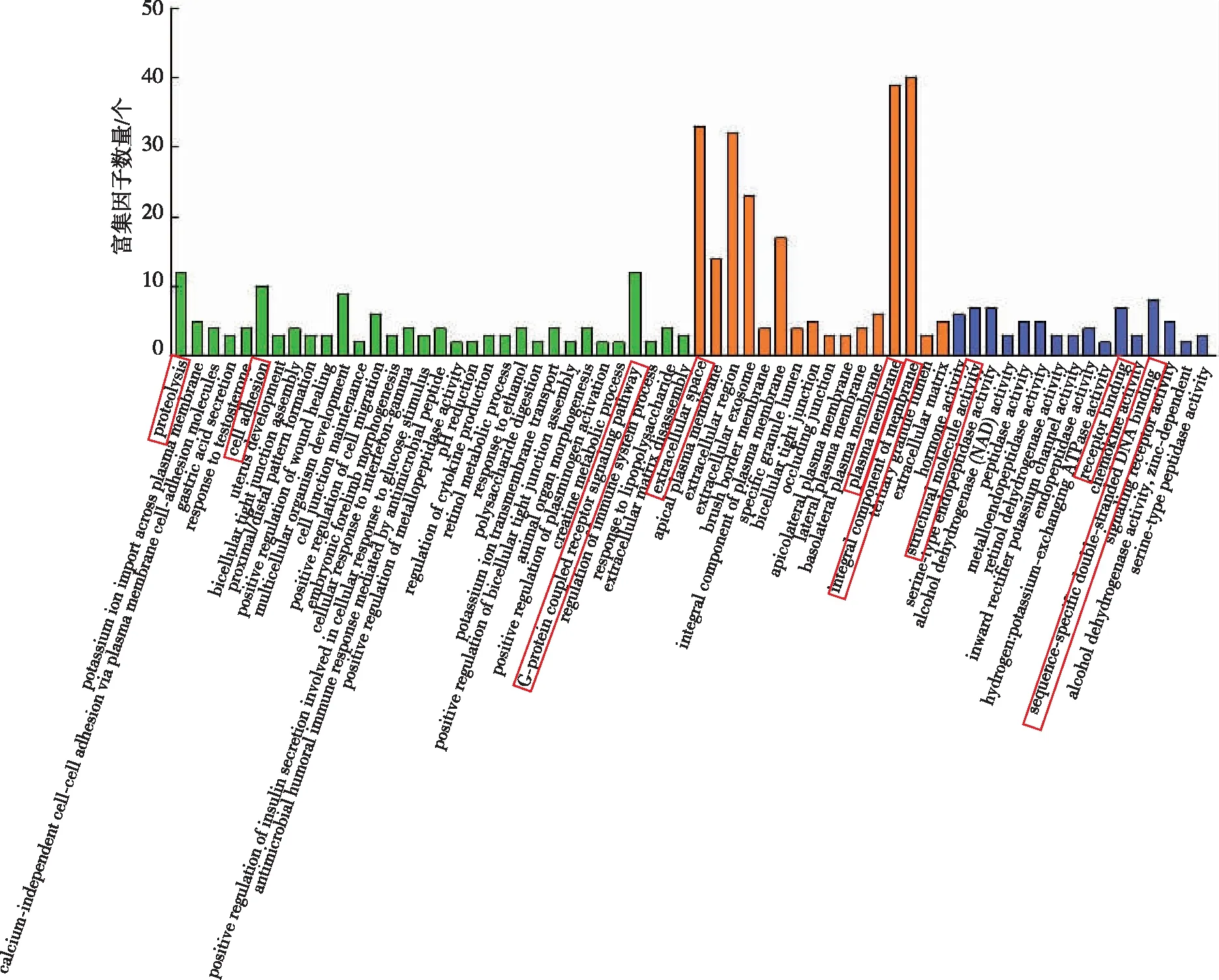
注:绿色表示BP,橙色表示CC,蓝色表示MF;红色框线表示文中已说明的内容。图3 重叠DEGs的GO富集结果Fig.3 GO enrichment results of overlapping DEGs
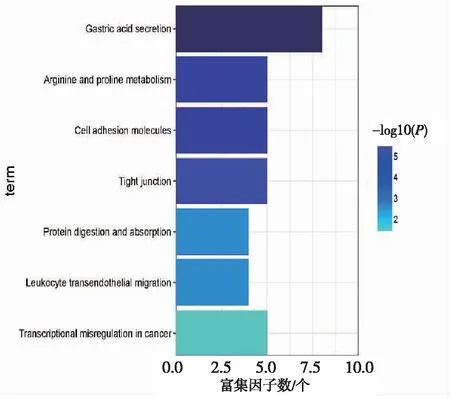
注:颜色越深表示差异越显著。图4 重叠DEGs的KEGG富集结果Fig.4 KEGG enrichment pathway map of overlapping DEGs
2.3 PPI 网络的构建及Hub基因筛选
构建113个重叠DEGs的PPI网络,共包含48个节点和76条边(图5);使用cytoHubba插件,按degree值筛选出排名前10的DEGs,为Hub基因,包括ATPase-H+/K+exchanging-alpha polypeptide(ATP4A)、claudin 3(CLDN3)、claudin 4(CLDN4)、claudin 7(CLDN7)、Chemokine C-X-C motif ligand 1(CXCL1)、Defensin-alpha 6-Paneth cell-specific(DEFA6)、Glucagon(GCG)、Sucrase-isomaltase(SI)、Cadherin 3(CDH3)、claudin 1(CLDN1),用于后续分析(图6)。
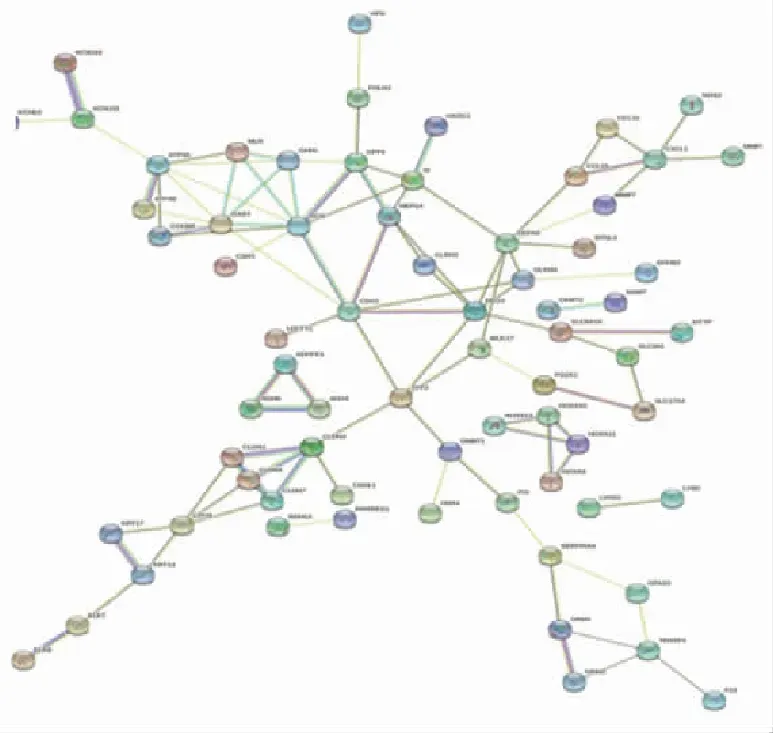
图5 重叠DEGs的PPI网络Fig.5 PPI network with overlapping DEGs
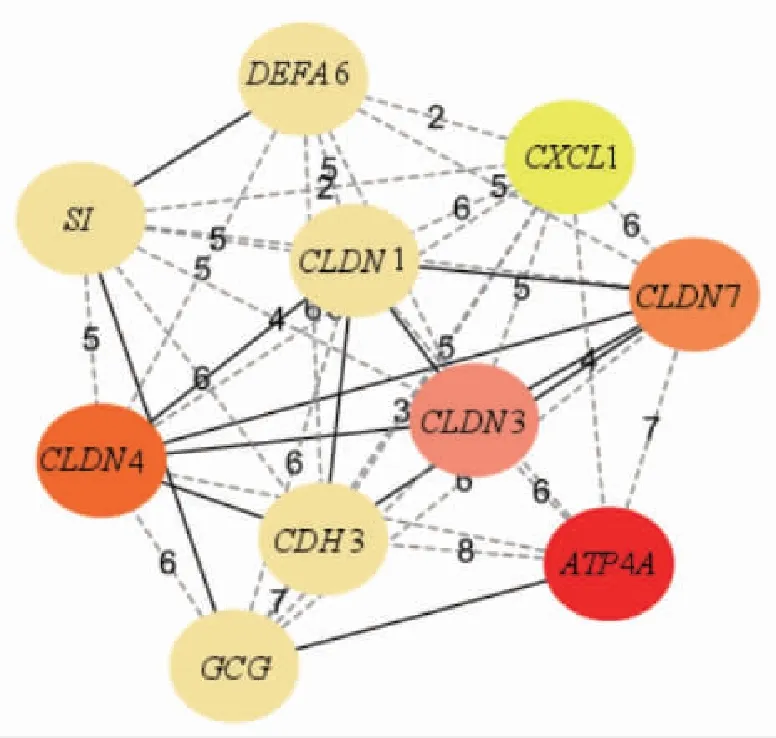
注:红色、粉色、橙色、浅橙色、黄色及浅米色分别表示degree值排名第1、2、3、4、5及6的DEGs;颜色越深排名越靠前。图6 重叠DEGs中Hub基因的筛选Fig.6 Screening of Hub genes in overlapping DEGs
2.4 Hub基因生存分析
Kaplan-Meier Plotter数据库对Hub基因中degree值排名前5的基因进行生存分析,结果表明ATP4A、CLDN3、CLDN4、CLDN7、CXCL1均与胃癌患者的总体生存期(overall survival,OS) 相关(P<0.05,图7)。
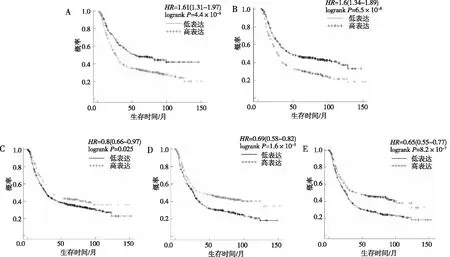
注:A为ATP4A,B为CLDN3,C为CLDN4,D为CLDN7,E为CXCL1。图7 Hub基因生存分析Fig.7 Hub genes survival analysis
2.5 Hub基因的验证
对Hub基因中degree值排名前2位的ATP4A与CLDN3进行临床样本验证,Western blot和qRT-PCR结果显示,与癌旁组织相比,ATP4A在胃炎组织中表达下调,CLDN3在胃炎组织中表达上调;与胃炎组织相比,ATP4A在胃癌组织中表达下调,CLDN3在胃癌组织中表达上调(P<0.05,图8)。
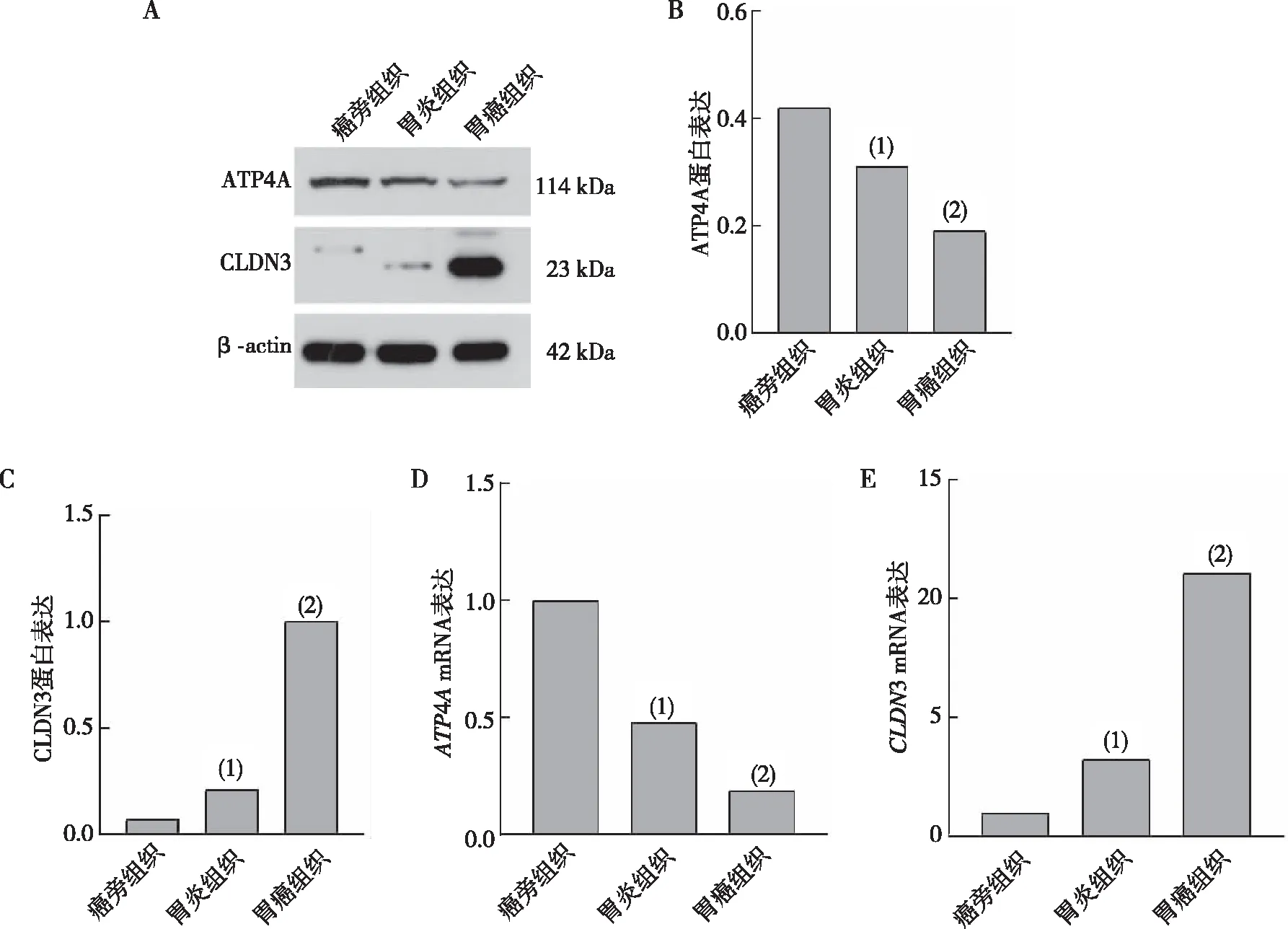
注:A为ATP4A、CLDN3的Western blot检测结果,B、C为ATP4A、CLDN3蛋白定量的结果;D、E为qRT-PCR检测ATP4A、CLDN3 mRNA水平的qRT-PCR检测结果;(1)与癌旁组织比较,P < 0. 05;(2)与胃炎组织比较,P<0. 05。图8 ATP4A、CLDN3蛋白及mRNA的表达Fig.8 Expression of ATP4A and CLDN3 at protein level and mRNA level
3 讨论
1863年,Rudolf等首先提出癌症起源于炎症部位[13]。19世纪,Virchow首次提出“炎癌转化”的构想,并指出“炎癌转化”是炎症疾病从“炎症-癌前病变-癌症” 整个动态过程的总称[14]。有研究指出,非可控性炎症可诱导肿瘤的发生和发展,作为非可控炎症向癌症恶性转化的经典模式,胃部“炎癌转化” 已成为近几年的研究热点[15]。
近年来,生物信息学在各种疾病的研究中得到了广泛的应用,特别是利用GEO数据库筛选潜在的靶基因,这些芯片数据集的充分利用,为生命科学研究提供了宽广的发展前景[16]。本研究通过生物信息学及高通量测序技术,筛选了2个高质量的表达谱数据集,初步鉴定了113个DEGs;通过DAVID数据库对2个芯片数据集中重叠的DEGs 进行 GO 与KEGG 富集分析,富集分析表明DEGs富集的GO条目主要有G蛋白偶联受体信号通路、蛋白水解、细胞粘附等方面,这些基因还介导控制细胞分化、增殖和凋亡等过程;KEGG通路富集分析显示DEGs主要参与胃酸分泌、精氨酸和脯氨酸代谢、蛋白质的消化与吸收等通路,提示重叠DEGs可能通过这些生物途径参与胃炎-胃癌“炎癌转化”的过程。此外,为挖掘DEGs相互之间的关联,本研究使用Cytoscape软件对重叠DEGs进行进一步的分析,按照degree值进行筛选,得到了前10个最有可能参与“炎癌转化”进程的Hub基因,并通过qRT-PCR和Western blot对排名前2位的Hub基因进行验证,结果提示它们的表达趋势与在GEO数据库中一致。
在慢性胃炎向胃癌发展的过程中,胃酸分泌在炎症反应和肿瘤间发挥作用[17]。泌酸功能失调会影响“炎癌转化”的进程,胃酸分泌减少会直接导致幽门螺杆菌的过度生长和微生态的失衡[18]。ATP4A存在于胃粘膜顶细胞中,编码H+/K+-ATP酶,并在胃中介导胃酸分泌,从而维持胃内的酸性环境[19]。Lahner等[20]研究表明,ATP4A与萎缩性胃炎有高度相关性,且其表达水平的降低与胃癌的发生相关。Cao等[19]、Judd等[21]研究显示,ATP4A-/-小鼠的胃部重量和厚度增加了1倍,会出现胃酸过多、高胃酸血症及不完全性肠化生等,并表现为胃黏膜顶细胞膜的变化和胃黏膜变性,引起干细胞标志物CD44表达上调。因此,ATP4A在维持胃部正常功能中起重要作用,其表达异常可能与“炎癌转化”进程相关。
紧密连接蛋白(tight junction ,TJs)是正常上皮细胞与细胞黏附的重要功能组成部分,其异常表达会导致炎症、肿瘤等的发生,在“炎癌转化”过程中,由于细胞极性丧失,细胞间的连接频繁中断,TJs在这一过程中起重要作用[22-23]。Huang等[24]研究显示,敲低CLDN1能够抑制胃癌细胞的生长、迁移与侵袭,CLDN1的下调有助于延缓肿瘤的进展。 Wang等[25]研究结果表明,CLDN1通过β-连环蛋白-T细胞因子(β-catenin-T cell factor,β-catenin-TCF)等信号通路参与肿瘤生成及发展,且CLDN4在70.6%的胃癌组织中高表达,与胃肿瘤的淋巴转移明显相关;Okugawa等[26]发现,CLDN3在胃癌及胃黏膜肠上皮化生细胞中的表达明显高于正常胃粘膜细胞。此外,Wu等[27]发现,CLDN7在胃癌中过表达,能促进胃癌细胞的增殖、侵袭。因此,TJs的表达在胃癌细胞中存在异常,有可能为早期胃癌的诊断、预后等提供新的分子生物学标志物,具有一定的临床研究价值。
趋化因子是一种由肿瘤细胞和基质细胞产生的小分子分泌蛋白,和其相配对的同源受体结合可通过直接或间接的方式调控肿瘤的生长,包括激活信号通路,直接调控肿瘤细胞的增殖与转移。其中,趋化因子CXC亚家族在胃癌细胞的增殖、侵袭及转移过程中具有促进作用;CXCL1在巨噬细胞和中性粒细胞中均有表达,可参与炎症、创伤愈合等过程,与胃癌的不良预后呈正相关[28]。此外,CXCL1可通过激活非受体型蛋白酪氨酸激2-信号传导蛋白和转录激活物3(just another kinase 2- signal transducer and activator of transcription 3,JAK2-STAT3 )通路,增加血管内皮生长因子表达和微血管密度,促进肿瘤的血管生成,介导肿瘤的血行转移节肿瘤细胞的转移及侵袭[29]。趋化因子不仅参与机体组织的分化、发育以及炎症反应,对胃癌的发生、发展也能产生重要影响.因此,调节趋化因子及其受体的表达可能成为未来胃癌特异性治疗的一种策略,还可通过调节趋化因子水平减轻胃粘膜局部炎症反应,为减缓“炎癌转化”的发生提供新的治疗途径。
综上所述,胃部“炎癌转化”是一个多因素、多环节、多步骤的复杂过程。本研究通过GEO数据库,筛选出参与“炎癌转化”过程的关键基因及通路,分析它们在“炎癌转化”进程中发挥的作用,从而为今后研发延缓、阻断“炎癌转化”的药物提供新的治疗靶点及途径,并可为早期胃癌诊断和预防提供潜在的分子生物标记物。
