肾透明细胞癌中具有预后预测价值免疫相关基因的筛选
2022-06-01徐明薛波新阳东荣朱进
徐明 薛波新 阳东荣 朱进
肾细胞癌约占成人恶性肿瘤的3%,其最常见组织学亚型为肾透明细胞癌(clear cell renal cell carcinoma, ccRCC),治疗上以手术为主,但仍有一定比例患者死于肿瘤特异性复发和转移[1-2]。肿瘤微环境是由免疫细胞、间充质细胞、内皮细胞、炎症细胞和细胞外基质分子组成[3-4]。免疫细胞和基质细胞是肿瘤微环境中两种主要的非肿瘤成分,在肿瘤诊断和患者预后评估中具有重要价值。
ccRCC是最早通过免疫疗法治疗有效的恶性肿瘤之一,也是一种在各种临床和基因组研究中发现的高度免疫侵袭性肿瘤[5-7]。Janiszewska等[8]总结相关研究发现,ccRCC患者中约1%的转移自发消退是由大量免疫反应介导的。免疫微环境中基因表达的具体机制在癌症的治疗中起着重要作用。然而,关于ccRCC的高免疫浸润、自发缓解和免疫治疗反应机制的报道仍缺乏可靠和详细的解释。因此,挖掘在ccRCC发生、发展中起关键作用的免疫微环境相关基因具有实际指导意义。
Yoshihara等[6]设计了ESTIMATE算法,该算法可利用癌症基因组图谱(The Cancer Genome Atlas, TCGA)数据库中的基因表达数据估计恶性肿瘤中的基质细胞和免疫细胞。在该算法中,作者通过分析免疫细胞和基质细胞的特异性基因表达特征来计算免疫评分和基质评分,以预测非肿瘤细胞的浸润。随后有学者将ESTIMATE算法应用于胶质母细胞瘤进行免疫/基质预后相关基因筛选,获得并证实了预测该肿瘤不良预后的基因[9],证明了基于大数据的算法是有效且可靠的。在本研究中,我们采用ESTIMATE算法评估ccRCC基因表达数据的免疫评分和基质评分,提取并验证差异表达的肿瘤微环境相关基因,以期为ccRCC的治疗和预后判断提供新的依据。
对象与方法
一、基因表达数据集
从遗传病控制(Genetic Disease Control, GDC)网站(https://www.cancer.gov/about-nci/organization/ccg/research/structuralgenomics/tcga/)下载534例ccRCC患者的基因表达数据,以及对应的患者样本临床信息,临床特征包括性别、N分期、M分期、AJCC分期、总生存(overall survival, OS)信息和无病生存(disease free survival, DFS)信息。在ESTIMATE 算法网站(https://bioinfor-matics.mdanderson.org/estimate/)上查看并下载所有 TCGA所对应ccRCC样本的基质评分和免疫评分。整合上述两类数据,得到同时包含OS记录、ESTIMATE评分和表达谱信息的样本共533例,用于后续验证分析。其中435例ccRCC患者具有DFS信息。为了进一步验证,从NCBI(http://www.ncbi.nlm.nih.gov/geo)下载GEO额外数据集(GSE29609),将该数据集中基因表达谱数据及临床随访数据合并,得到包含OS的表达谱数据样本共39例,用于后续验证分析。
二、数据处理和统计分析
1.免疫评分和基质评分与ccRCC患者生存的关联分析:采用ESTTIMATE算法对ccRCC样本数据集进行免疫评分和基质评分,并用箱型图描述。根据免疫评分和基质评分中位数,将ccRCC患者分为高分组和低分组,结合临床数据分析其与OS的关系。采用Kaplan-Meier生存曲线,并应用log-rank test验证法比较两组生存曲线的差异。
2.差异表达基因的筛选:利用R语言软件包limma函数功能进行基因表达数据差异分析。根据差异倍数和显著性,以logFC>1和P<0.05为阈值筛选出高分组和低分组之间的差异表达基因。
3.功能富集分析:将差异表达基因列表上传至DAVID数据库,选择GO注释和KEGG通路富集分析,设置P<0.05,阐明这些基因所涉及的生物进程、分子功能和细胞组分,以及参与基因的信号通路。
4.差异表达基因的生存分析:采用Kaplan-Meier法绘制差异表达基因生存曲线,挑选出与ccRCC OS显著相关的基因。
5.蛋白质互作网络构建:STRING数据库(http://www. string-db.org/)包含大量关于蛋白质-蛋白质相互作用(protein-protein interaction, PPI)的信息。为了评估上述差异表达基因之间的关系,我们将差异表达基因列表上传到 STRING数据库,从而获得差异表达基因之间的PPI网络。使用Cytoscape软件构建PPI网络,所得网络为无向网络。
6.数据集验证:应用验证数据集GSE29609的表达矩阵和临床特征进行回归分析,验证所筛选核心差异表达基因与ccRCC之间的关系。
结 果
一、免疫评分和基质评分与ccRCC患者生存的关联分析
从TCGA数据库下载所得534例ccRCC的基因表达数据和相应的临床信息中,有533例符合研究要求,其中435例患者具有DFS信息,患者的具体特征见表1。基于ESTIMATE算法,免疫评分分布在-1 159.0~3 076.0之间,基质评分范围为-1 558.0~2 030.0。平均免疫评分为1 083.0,平均基质评分为617.3(图1A)。
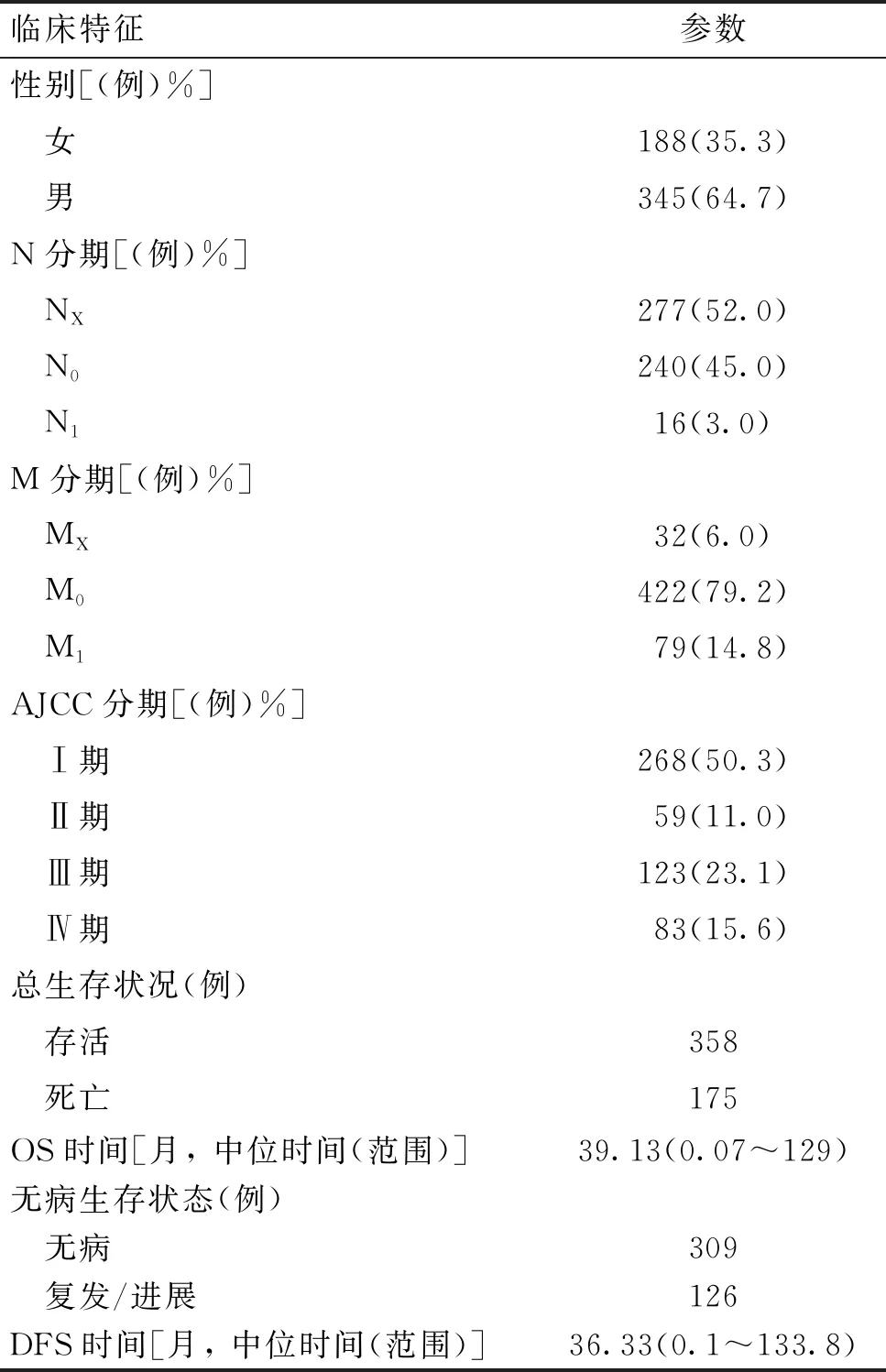
表1 TCGA中533例ccRCC样本的临床特征
为了找出OS与免疫评分或基质评分之间的潜在相关性,本研究根据免疫/基质评分的中位数将ccRCC患者分为高分组和低分组。Kaplan-Meier生存曲线(图1B)显示,高免疫评分组患者的中位OS时间为77个月,低免疫评分组患者的中位OS时间长于高免疫评分组(P=0.011)。此外,基质评分患者的中位OS趋势与免疫评分相似(图1C,116.8个月 vs 74.1个月,P=0.19)。因此,ccRCC患者免疫评分与OS显著相关(P<0.05)。
二、差异表达基因筛选
533例ccRCC样本按照免疫评分中位数划分为高评分组和低评分组,并进行差异基因筛选。在P<0.05和logFC>1条件下,得到804个差异表达基因,包括720个上调基因和84个下调基因,差异表达基因热图如图1D所示。一行代表一个基因,一列代表一个样本。其中样本按照免疫评分由高到低、从左至右排序,左侧粉色组为免疫评分低评分组样本,右侧蓝绿色组为免疫评分高评分组。热图中红色代表基因高表达,蓝色代表低表达;红色或蓝色越深则基因的差异程度越大。
A:ccRCC样本获得的表达数据集生成基质和免疫评分[方框代表中位数(粗线)和四分位数(细线),Whisker表示下四分位数或上四分位数的1.5四分位距];B~C:Kaplan-Meier 生存曲线所示,低免疫评分组的中位OS时间长于高免疫评分组(P=0.011),低基质评分组的中位OS时间长于高基质评分组(P=0.19);D:差异表达基因热图
三、GO注释和KEGG通路富集分析
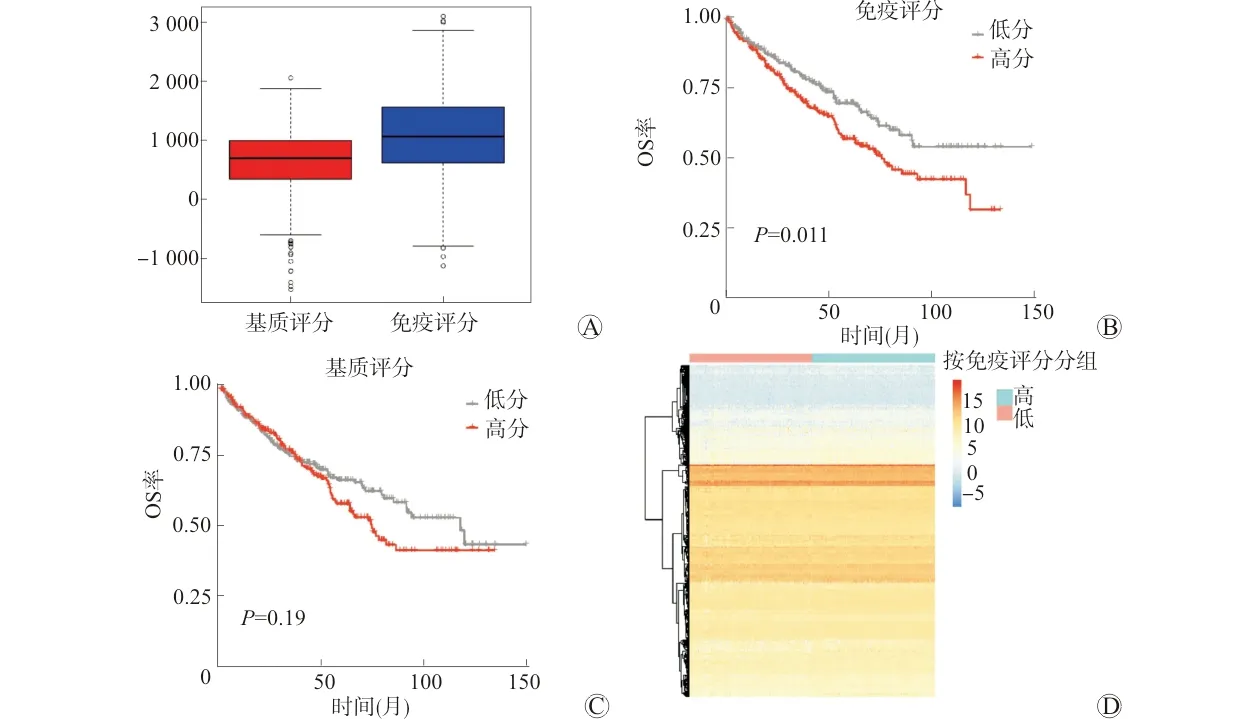
为研究所得到差异表达基因的功能,应用DAVID富集工具对804个差异表达基因进行了富集分析,其中最显著的10个GO注释和KEGG富集结果见图2。如图所示,ccRCC相关差异表达基因的分子功能主要表现于转录因子活性、序列特异性DNA结合、poly(A) RNA结合等,生物进程中涉及的主要因素是前/后模式规范、剪接体复合物组装、RNA聚合酶Ⅱ启动子的转录等。与细胞组分相关的有甲基小体、核质、剪接体复合物等。KEGG信号通路主要包括细胞周期、TGF-β信号通路和Toll样受体信号通路。
A:分子功能;B:生物进程;C:细胞组分;D:KEGG 通路图
四、差异表达基因的生存分析
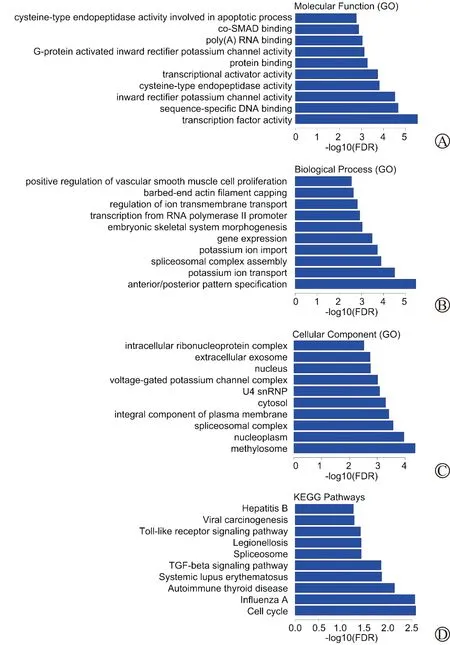
为筛选出与ccRCC预后相关基因,我们绘制了804个差异表达基因的生存分析曲线,其中190个与OS显著相关(P<0.05),图3为部分基因生存分析结果。
A、E:显示基因ABAT和APP高分组生存优于低分组;B、C、D、F、G、H、I:显示基因ACTN1、AMPD3、AMY1A、ADAM12、ALX1、ACTR1A、ANGPTL7低分组生存优于高分组
五、蛋白质互作网络
PPI分析发现88个与生存相关的基因,其中41个是独立的。利用另外47个与生存显著相关的基因构建PPI网络,如图4所示,网络由47个节点和42条边组成,网络的聚类系数、密度和异质性分别为0.064、0.039和0.576。在这个PPI网络中,我们发现RAC3[10]、LEP[11]和RABEP1[12]3个基因已被证明与免疫功能密切相关,其中RAC3基因是网络中的枢纽基因,它可以调控网络中与生存和免疫显著相关的基因PLXNB3。
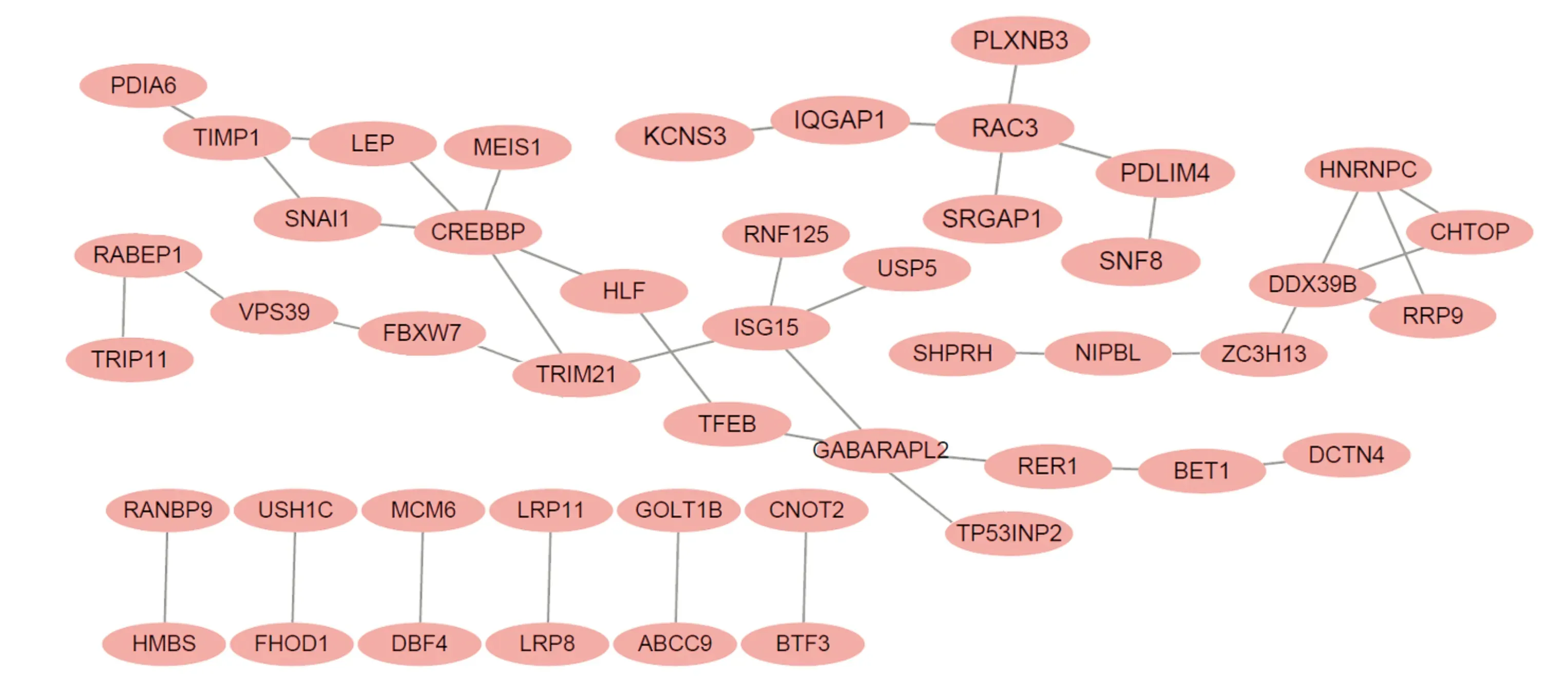
图4 蛋白质互作网络
对190个OS相关基因的生存分析显示,108个基因与DFS显著相关,如表2所示。其中,11个基因(DBF4、RER1、HMBS、SNF8、LRP8、RABEP1、MCM6、BET1、USP5、HLF、RRP9)存在差异表达且在具有预后效能的PPI网络中出现,有望作为ccRCC治疗的潜在靶点,特别是RABEP1被证实是免疫相关重要基因(图5)。

表2 TCGA数据集中与OS和DFS相关基因
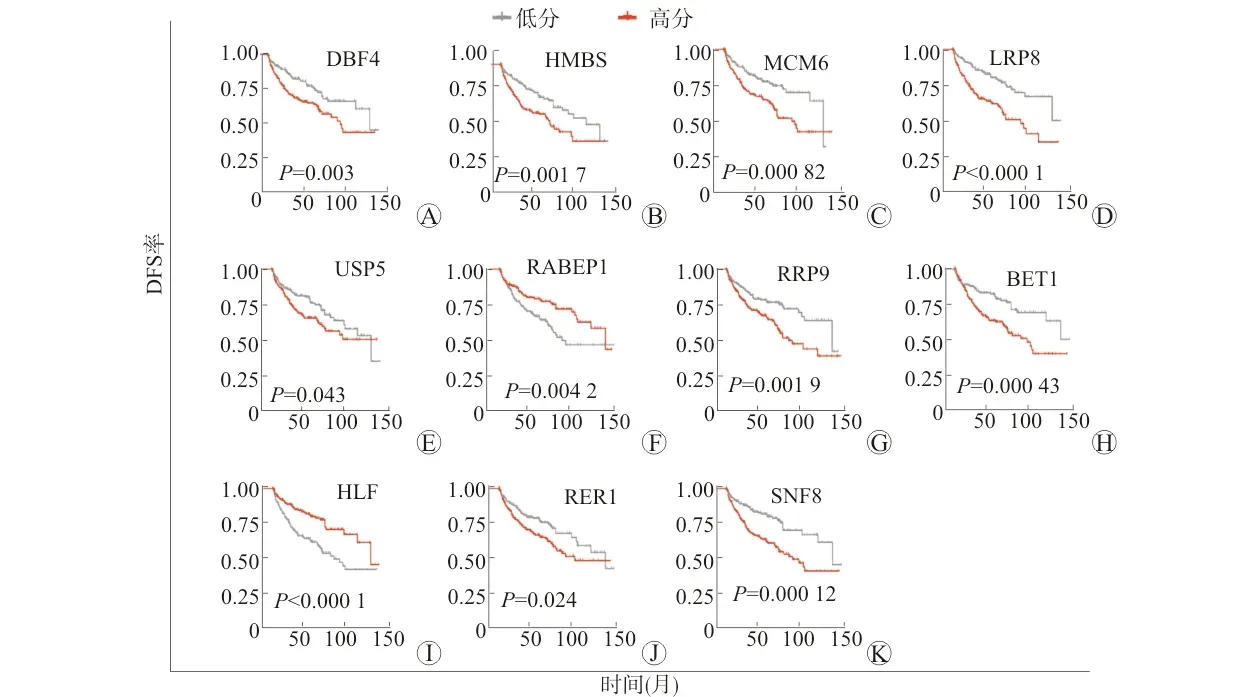
A、B、C、D、E、G、H、J、K:显示基因DBF4、HMBS、MCM6、LRP8、USP5、RRP9、BET1、RER1和SNF8低分组生存优于高分组;F、I:显示基因RABEP1、HLF高分组生存优于低分组
六、GEO数据集验证
为了确定TCGA数据库中鉴定的基因在其他ccRCC病例中是否具有预后意义,将前述所得的190个OS相关基因在GEO数据集(GSE29609)中验证。结果表明,其中134(134/190)个基因在这组GEO数据集中同样表达;生存分析显示,其中16(16/134)个基因与TCGA结果一致,而且其表达水平与OS显著相关(P<0.05),如表3所示。其中,12(12/16)个基因(BCL3、F3、HLF、HNRNPAB、SHOX2、SNRPB2、USP5、ALX1、FAM50A、USP39、DBF4、SPIN1)出现在前述108个与DFS显著相关的差异表达基因中,显示能够预测患者DFS。
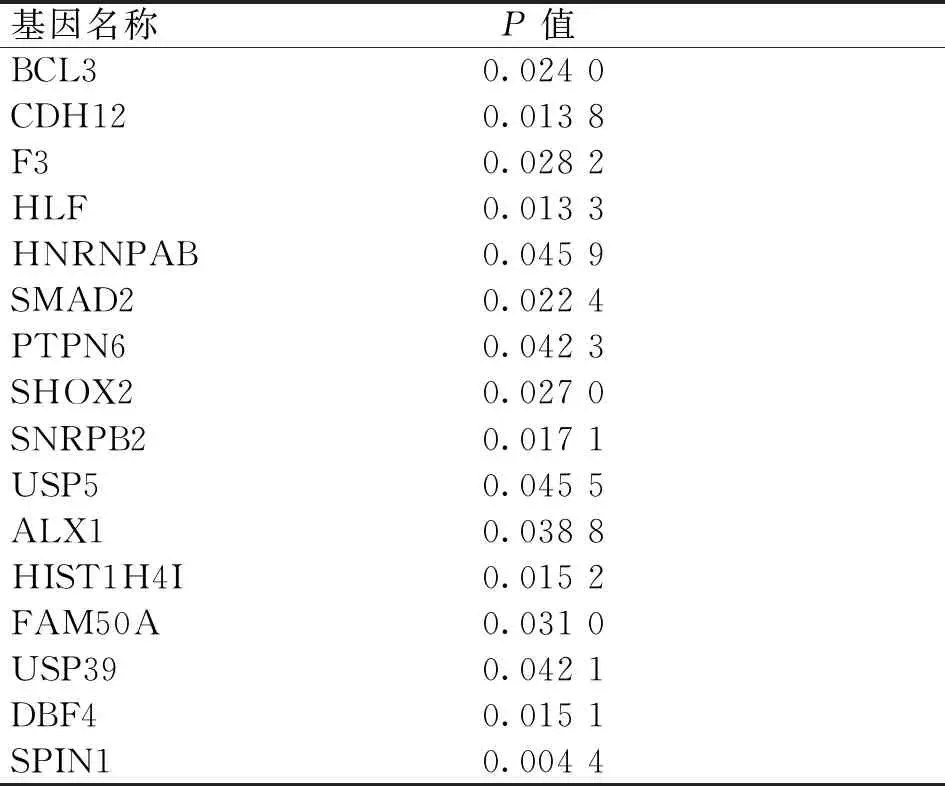
表3 在GEO额外数据集中验证具有预后效能的基因
讨 论
在目前的工作中,我们试图在TCGA数据库中挖掘与肿瘤免疫微环境相关的基因,这些基因对ccRCC患者的DFS具有重要的预后预测价值。通过比较大量免疫评分高低样本的全局基因表达,提取出804个参与免疫反应的差异表达基因,发现其中许多基因与肿瘤微环境有关,这与之前的报道一致,免疫细胞的功能在ccRCC构建肿瘤微环境中是相互关联的[13-15]。
为了找出与OS显著相关的基因,对804个基因进行了生存分析,鉴定出与OS显著相关的190个基因。使用STRING软件在PPI网络中发现88个基因,其中47个是非独立的。这些基因与免疫或炎症反应有关,尤其是RAC3、LEP和RABEP1,已被证明与免疫功能密切相关[16]。RABEP1是肿瘤发生和进展的主要负调节因子[16-18]。图1显示,免疫评分高的患者中位OS时间明显长于免疫评分低的患者。因此,我们推断RABEP1基因也通过某种机制负调控ccRCC的进展,进一步影响患者的DFS。对190个基因的DFS分析显示,108个基因与DFS显著相关。其中,11个基因在PPI网络中差异表达并具有预后预测作用,包括DBF4、RER1、HMBS、SNF8、LRP8、RABEP1、MCM6、BET1、USP5、HLF和RRP9。此外,使用GEO交叉验证,获得了12个基因(BCL3、F3、HLF、HNRNPAB、SHOX2、SNRPB2、USP5、ALX1、FAM50A、USP39、DBF4、SPIN1),可以预测患者的DFS。特别是,在ccRCC样本中筛选出与免疫相关的HLF、USP5和DBF4基因既存在于TCGA和GEO数据库中,也存在于差异表达和预后PPI网络中,推测这3个基因可能在ccRCC免疫微环境中发挥重要作用。
研究表明,髓源性抑制细胞衍生的外泌体蛋白,包括DBF4,可以调节抗肿瘤免疫,促进肿瘤生长[19]。DBF4是细胞分裂周期7相关蛋白激酶(CDC7)的调节亚基[20]。CDC7-DBF4复合物是调节DNA复制起始的必需激酶[21]。Bonte等[22]报道CDC7-DBF4在多种癌症和肿瘤细胞系中的过表达与p53失活相关。p53的失活与ccRCC的发生和进展有关。CDC7-DBF4表达降低可能导致p53激活[22]。因此,我们推断DBF4可能有效控制ccRCC中的肿瘤细胞生长和细胞增殖。
USP5作为DUB的泛素特异性蛋白酶家族的一员,通过与蛋白质结合和双链DNA修复,对调节未锚定的多聚泛素分解和蛋白质去泛素化起到一定作用[23-25]。USP5在恶性程度高的肝细胞癌中高表达并与SLUG表达呈正相关。USP5的敲低抑制SLUG去泛素化并抑制肝细胞癌细胞的增殖、转移和侵袭,而USP5的过表达在体外和体内促进SLUG稳定性和EMT[25]。USP5基因的免疫评分与ccRCC患者的预后呈负相关。我们推测USP5可能是治疗ccRCC的潜在靶点之一。
有数据显示HLF可能是Meis1的关键下游靶基因[26]。Meis1可以通过维持缺氧条件来防止氧化应激,诱导白血病转化。当它缺失时,白血病细胞中的活性氧增加,伴随着白血病细胞凋亡,导致存活率显著提高。HLF的表达可以逆转氧化应激,间接说明Meis1促进白血病的特性可能部分是由HLF辅助的低氧化状态介导。本研究中,与HLF基因相关的Kaplan-Meier生存曲线(图5)显示,高免疫评分组的DFS长于低免疫评分组(相反秩检验P=0.000 1)。推测HLF在ccRCC进展过程中可能具有不一样的调节机制。
在PPI网络中,我们将RAC3确定为唯一的枢纽基因。RAC3在多种恶性肿瘤中高表达,如肺腺癌[27]、脑肿瘤[28]、乳腺癌[29]、前列腺肿瘤[30]和宫颈癌[31],其在癌症发展中发挥了重要作用。有研究表明RAC3通过p38 MAPK通路促进肺腺癌的细胞侵袭、迁移和EMT[27]。尽管RAC3在ccRCC发展中的具体机制鲜有报道,但推测RAC3可能是ccRCC的潜在治疗靶点。
以上相关文献研究证明,肿瘤与肿瘤免疫微环境的相互作用严重影响着肿瘤的发生、发展和整体预后。本研究筛选出的12个基因(BCL3、F3、HLF、HNRNPAB、SHOX2、SNRPB2、USP5、ALX1、FAM50A、USP39、DBF4、SPIN1)可以预测ccRCC患者的DFS。这些基因与免疫/炎症反应、肿瘤细胞的增殖/凋亡、血管生成、肿瘤的侵袭和转移密切相关,尤其是HLF、USP5和DBF4基因,在TCGA、GEO数据库和生存显著相关PPI网络中均显示出潜在价值。此外,RAC3还在PPI网络中起到枢纽基因作用。推测这4个基因在ccRCC免疫微环境中具有重要意义。本研究认为,通过使用TCGA数据库的功能富集分析,并基于ESTIMATE算法,提取了与肿瘤免疫微环境相关的基因列表,可用于预测ccRCC患者的预后。其中,HLF、USP5、DBF4和枢纽基因RAC3可能成为ccRCC潜在的治疗生物靶点。在后续研究中,我们将进一步验证这些具有预后预测价值的基因在ccRCC免疫微环境中的具体分子生物学功能。
