骨髓增生异常综合征临床特点及预后分析
2022-05-30张婷,牟俊宇,孟繁军
张婷,牟俊宇,孟繁军
[摘要]目的 了解骨髓增生異常综合征病人的临床特点、分子遗传学特征及预后。 方法 回顾性分析2010年9月—2020年9月我院收治、初诊且首次基因突变检测结果为阳性的骨髓增生异常综合征病人125例的临床资料,对所有病人进行随访,分析影响预后的因素。 结果 随访1~84个月,生存组(n=94,随访结束仍存活且未转归为急性髓系白血病)与非生存组(n=31,随访结束死亡或转归为急性髓系白血病)性别、年龄、血红蛋白、白细胞、中性粒细胞和乳酸脱氢酶等比较,差异均无显著性(P>0.05);两组血小板、骨髓原始细胞比较,差异有统计学意义(t=2.465、-4.185,P<0.05)。对突变频率>2%的基因进行单因素生存分析及log-rank检验,结果显示,[STBX]ETV6(χ2=4.820,P<0.05)、MPL(χ2=8.807,P<0.01)、NRAS(χ2=4.690,P<0.05)、KIT(χ2=5.727,P<0.05)及[STBX]TP53(χ2=7.289,P<0.01)基因与更短的总生存期显著相关。 结论 基因突变对预测骨髓增生异常综合征病人的预后具有重要意义。
[关键词]骨髓增生异常综合征;DNA突变分析;预后
[中图分类号]R551.3
[文献标志码]A
[文章编号]2096-5532(2022)04-0571-06
doi:10.11712/jms.2096-5532.2022.58.105[HT]
[开放科学(资源服务)标识码(OSID)]
[网络出版]https://kns.cnki.net/kcms/detail/37.1517.r.20220622.1623.009.html;[JY]2022-06-2415:57:39
AN ANALYSIS OF CLINICAL FEATURES AND PROGNOSIS OF PATIENTS WITH MYELODYSPLASTIC SYNDROME
ZHANG Ting, MU Junyu, MENG Fanjun
(Department of Hematology, The Affiliated Hospital of Qingdao University, Qingdao 2666500, China)
[ABSTRACT] Objective[WTBZ] To analyze the clinical features, genetic characteristics, and prognosis of patients with myelodysplastic syndrome.
Methods A retrospective analysis was performed using the clinical data of 125 patients with myelodysplastic syndrome who were newly diagnosed and first tested positive for gene mutation and admitted to our hospital from September 2010 to September 2020. All patients were followed up to analyze the factors affecting prognosis.
Results Comparison of the survival group (n=94, alive without progression to acute myelogenous leukemia at the end of 1-84 months of follow-up) and the non-survival group (n=31, died or progression to acute myelogenous leukemia at the end of follow-up) showed no significant differences in gender, age, hemoglobin, white blood cells, neutrophils, and lactate dehydrogenase (P>0.05), but showed significant differences in platelet (t=2.465,P<0.05) and bone marrow primitive cells (t=-4.185,P<0.05). The univariate survival analysis and log-rank test on genes with mutation frequencies of >2% showed that the genes [STBX]ETV6 (χ2=4.820,P<0.05), MPL (χ2=8.807,P<0.01), NRAS (χ2=4.690,P<0.05), KIT (χ2=5.727,P<0.05), and [STBX]TP53 (χ2=7.289,P<0.01) were significantly correlated with shorter overall survival.
Conclusion Gene mutation is of great significance in predicting the prognosis of patients with myelodysplastic syndrome.
[KEY WORDS] myelodysplastic syndromes; DNA mutational analysis; prognosis
骨髓增生异常综合征(MDS)[1]是一组起源于造血干细胞的异质性髓系克隆性疾病,其特点是髓系细胞发育异常,表现为无效造血、难治性血细胞减少,高风险向急性髓系白血病(AML)转化。随着分子检测技术的进步,基因突变在越来越多的MDS病人中被发现,但各个突变基因的意义尚未全部明确。本文通过回顾性分析125例初诊且首次基因突变检测为阳性病人的临床资料,探讨MDS临床特征、分子遗传学特点及预后。
1资料与方法
1.1一般资料
回顾性分析2010年9月—2020年9月在青岛大学附属医院血液内科住院治疗、初诊为MDS、同时基因突变检测阳性病人125例,通過电子病历系统收集其临床资料,包括性别、年龄、血红蛋白、白细胞计数、中性粒细胞计数、血小板计数、铁蛋白、乳酸脱氢酶、骨髓及外周血原始细胞计数、骨髓病态造血、骨髓环形铁粒幼细胞计数、骨髓阳性突变基因、骨髓染色体、是否有输血依赖以及治疗方案等。按照MDS中国诊断与治疗指南(2019年版)进行诊断,按照MDS 2016年WHO修订分型标准进行分型,按照修订的MDS国际预后积分系统(IPSS-R)进行预后评价[2]。所有病人均为初诊MDS,且第一次骨髓穿刺送检基因检测结果为阳性,排除慢性病性贫血、慢性髓系白血病及继发性MDS等。
1.2基因检测
标本为髂前上棘或髂后上棘骨髓穿刺血样。提取送检标本中基因组DNA,采用超高多重PCR靶向富集技术进行基因测序,分析检测区域的单核苷酸变异和短片段插入/缺失突变,平均测序深度为1 000×,对变异频率超过5%的突变位点检出率>97%。使用Ion Reporter系统和Variant Reporter软件进行基因突变分析;综合千人基因组计划(1 000 Genomes)数据库、 突变数据库、中文数据库、人类基因突变数据库等权威数据库及实验室自建数据库进行变异注释及氨基酸突变分析。
1.3临床治疗方案
根据MDS病人的预后分组、年龄、体能状况及治疗依从性等,低危MDS主要采用支持治疗、促造血药物、去甲基化药物地西他滨等治疗,而中高危MDS主要采用去甲基化药物地西他滨、化疗和造血干细胞移植等治疗。
1.4随访
所有病人均随访至2020年9月,随访资料来自电话联系、门诊及住院病历,随访时间为1~84个月。总生存期(OS)定义为自疾病确诊至死亡或随访截止日期的时间。随访结束后,将病人分为生存组与非生存组,生存组病人指自确诊至随访截止时间(2020年9月)仍存活且未转归为AML的病人,非生存组病人指自确诊至随访截止时间死亡或转归为AML的病人。
1.5统计学方法
应用SPSS 23.0软件进行统计学处理。计量资料以[AKx-D]±s表示,数据间比较采用独立样本t检验;计数资料以例数(百分比)表示,数据间比较采用χ2检验;单因素分析采用log-rank检验。以P<0.05为差异有统计学意义。
2结果
2.1MDS病人临床特征
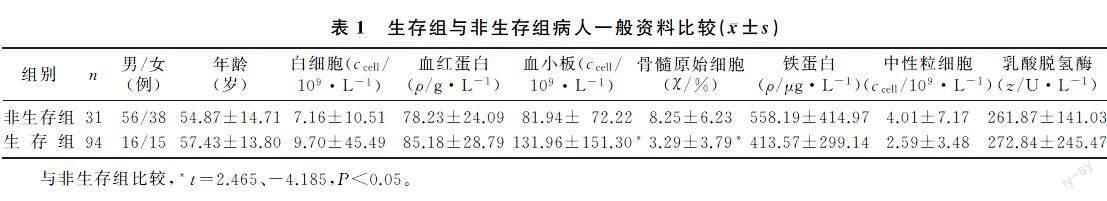
MDS病人125例中,男72例,女53例;年龄15~79岁,中位数60岁;血红蛋白18~180 g/L,平均83.46 g/L;白细胞计数(0.36~46.91)×109/L,平均5.57×109/L;中性粒细胞计数(0~30)×109/L,平均2.94×109/L;血小板计数(3~650)×109/L,平均119.55×109/L;铁蛋白范围13~>2 000 μg/L,乳酸脱氢酶90~1 715 U/L。按照WHO(2016)的分型标准,MDS伴单系血细胞发育异常(MDS-SLD)18例,MDS伴多系血细胞发育异常(MDS-MLD)29例,MDS伴环状铁粒幼红细胞(MDS-RS)3例,MDS伴原始细胞增多(MDS-EB)54例,MDS-不能分类型(MDS-U)21例。按照IPSS-R预后分层,极低危11例,低危28例,中危39例,高危36例,极高危11例。生存组与非生存组病人性别、年龄、血红蛋白、白细胞、中性粒细胞、乳酸脱氢酶等差异均无显著意义(P>0.05),而血小板、骨髓原始细胞数目比较,差异有统计学意义(t=2.465、-4.185,P<0.05)。见表1。
2.2分子遗传学特征
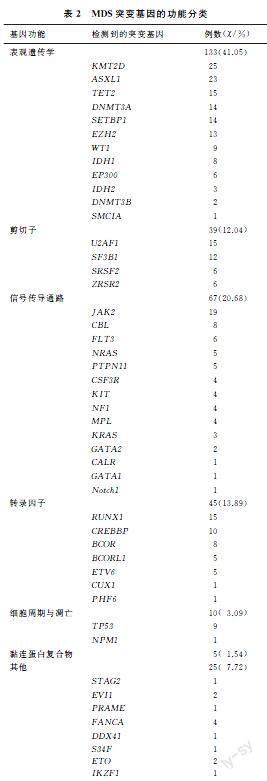
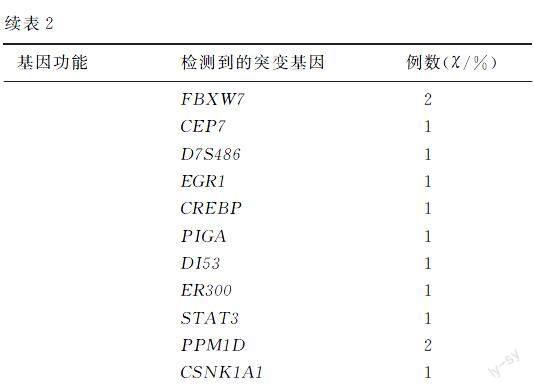
本文125例初诊MDS病人中,40例(32.0%)病人发生1种基因突变,同时发生2种基因突变为30例(24.0%),3种基因突变25例(20.0%),4种基因突变为13例(10.4%),5种基因突变为8例(6.4%),6种基因突变为6例(4.8%),7种基因突变为3例(2.4%)。125例病人中位居前10位的突变基因分别为[STBX]KMT2D、ASXL1、JAK2、U2AF1、RUNX1、TET2、SETBP1、DNMT3A、EZH2及SF3B1。按照基因的功能分类,表观遗传学基因占41.05%,剪切子基因占12.04%,信号传导通路基因占20.68%,转录因子基因占13.89%,细胞周期与凋亡基因占3.09%,黏连蛋白复合物基因占1.54%,其他意义未明的基因占7.72%。见表2。
2.3预后分析
所有病人随访至2020年9月20日,随访时间为1~84个月,共有94例(75.2%)病人存活,21例(16.8%)病人转归为AML,19例(15.2%)病人死亡,中位生存时间为10个月,其中有10例(8.0%)病人死于MDS相关感染或多器官功能障碍综合征,9例(7.2%)病人死于转归为AML。对突变频率>2%的29个突变基因分别进行单因素生存分析和log-rank检验,结果显示,[STBX]ETV6(χ2=4.820,P<0.05)、MPL(χ2=8.807,P<0.01)、NRAS(χ2=4.690,P<0.05)、KIT(χ2=5.727,P<0.05)及[STBX]TP53(χ2=7.289,P<0.01)基因与更短的OS显著相关。见表3和图1。
3讨论
MDS是一组骨髓造血系统恶性肿瘤,以不同程度的血细胞减少和发展为AML的高风险为特征。MDS是成年人中最常见的骨髓性造血肿瘤,西方国家MDS发病人群通常为老年人,HAASE等[3]研究显示,MDS病人中位发病年龄为65.7岁。而亚洲MDS发病年龄通常较小,泰国为56岁[4],韩国为51岁[5]。由于MDS临床和生物异质性显著,基于个体病人准确诊断和预后评估的优化治疗尤为重要。随着分子检测技术的进步,越来越多MDS的基因突变被发现,且不同种类的突变之间常以不同的组合伴随发生,导致了MDS分类及预后的复杂性。相关研究已证实,常见种类的基因突变对MDS分层及预后生存有一定的影响[6-9]。
新一代基因测序技术可以在绝大多数MDS病人中检出至少1个基因突变。据报道约有90%的MDS病人存在基因突變,有超过50种基因突变与MDS的发病机制、转白机制、治疗反应和生存预后相关。来自英国和日本的两项大型研究,通过靶向测序分别在78.0%和89.5%的MDS病人中发现了基因突变。MDS的发病率随着年龄增长而升高,预示造血细胞的老化及其导致的克隆性异常与MDS的发病相关。MDS常见基因突变包括[STBX]DNMT3A、TET2、RUNX1、ASXL1、EZH2及SF3B1等[1]。BEJAR等[6]对439例MDS病人进行了18种基因测序,发现51%的病人至少携带1种基因突变,其中突变率>10%的基因有[STBX]ASXL1、RUNX1以及TET2。HAFERLACH等[10]对944例MDS病人进行了104种基因测序,发现89.5%的病人至少含有1种基因突变,其中突变率>10%的基因包括[STBX]RUNX1、SF3B1、ASXL1、SRSF2、DNMT3A和TET2,较少见的突变基因包括[STBX]U2AF1、ZRSR2、STAG2、TP53、EZH2、CBL、JAK2、BCOR、IDH2、NRAS、MPL、NF1、ATM、IDH1、KRAS、PHF6、BRCC3、ETV6和LAMB4。XU等[11]研究发现,MDS基因突变率最高的是[STBX]ASXL1(16.8%),其次是[STBX]RUNX1(14.4%)和[STBX]TET2(12.0%)。HOU等[12]研究发现,42.7%的MDS病人有2种或2种以上的基因突变,突变率>10%的基因有[STBX]ASXL1(22.5%)、[STBX]TET2(13.8%)、[STBX]SRSF2(13.6%)、[STBX]RUNX1(12.2%)和[STBX]SF3B1突变(11.0%),较少见的基因突变包括[STBX]DNMT3A(9.9%)、[STBX]ZRSR2(9.6%)、[STBX]TP53(9.6%)、[STBX]U2AF1(7.3%)、[STBX]STAG2(6.3%)和[STBX]EZH2(6.3%)等。本文的研究结果显示,其中突变率>10%的基因包括[STBX]KMT2D(20.0%)、[STBX]ASXL1、[STBX]JAK2(14.4%)、[STBX]U2AF1(12.0%)、[STBX]TET2(12.0%)、[STBX]RUNX1(12.0%)、[STBX]SETBP1(11.2%)、[STBX]DNMT3A(11.2%)和[STBX]EZH2(10.4%)等。提示基因突变在初诊MDS病人中较为常见,与上述文献的研究结果基本一致。
突变基因常被分成几个功能通路,如RNA剪切子、表观遗传学、信号传导通路、转录因子、细胞周期与凋亡等。HAFERLACH等[10]研究发现,MDS最常见的功能通路为RNA剪切子(64%),其次为DNA甲基化、染色质重塑、转录因子、RAS信号途径和DNA修复通路相关的基因。XU等[11]研究发现,MDS最常见的功能通路为转录因子(58.4%),其次的通路分别为表观遗传修饰(53.6%)、信号传导(52.0%)、RNA剪切子(20.8%)、黏连蛋白复合物(11.2%)、DNA修复(10.4%)和细胞周期调节(7.2%)等。HOU等[12]的研究发现,MDS最常见的功能通路为RNA剪切子(39.0%),其次为DNA甲基化(24.4%)、染色质重塑(24.2%)以及转录因子(14.3%)等。本文研究显示,MDS最常见的功能通路为表观遗传学基因(41.05%),其次分别为RNA剪切子(12.04%)、信号传导通路(20.68%)、转录因子(13.89%)、细胞周期与凋亡(3.09%)和黏连蛋白复合物(1.54%)等,与相关研究结果不太一致。究其原因,可能与本文研究病例数量较少有关,有待于进一步分析。
常见基因突变检测对MDS的诊断有潜在的应用价值,如[STBX]SF3B1基因突变[13-14]对MDS-RS亚型有重要诊断和鉴别诊断价值,应作为必检基因。部分基因的突变状态对MDS的鉴别诊断和危险度分层有一定价值,推荐作为选做检测项目,这些基因包括[STBX]TP53、TET2、DNMT3A、IDH1/2、SETBP1、ASXL1、SRSF2、RUNX1、U2AF1及EZH2等[1]。MDS有恶性转化的倾向,有研究认为某些基因突变可以促进MDS的发生、发展[15]。CHEN等[16]研究结果显示,[STBX]ASXL1基因突变在MDS病人中是频繁发生的分子畸变,预示病人预后不良。对MDS病人进行[STBX]ASXL1基因突变筛查可能有助于进行临床危险分层和制定治疗决策。相关研究结果也显示,[STBX]TP53基因突变在MDS病人中为独立的预后不良因素;存在[STBX]DNMT3A基因突变的MDS病人OS率低,且是其进展为急性白血病的一个危险因素;伴[STBX]JAK2-V617F基因突变的MDS病人骨髓原始细胞比例、外周血乳酸脱氢酶水平高,脾脏大发生率高,并且约有37.5%的[STBX]JAK2基因突变病人会发生急性白血病转化[17-19]。本研究中2例MDS-RS-MLD病人均携带[STBX]SF3B1基因突变,这2例MDS-RS-MLD病人同时携带[STBX]SETBP1基因突变,尚未找到相关文献证据。
MDS作為一种造血干细胞恶性克隆性疾病,临床具有高度异质性,病人的结局极其不一致,中位生存期6个月~5年以上不等[20]。MDS病人预后不良,约30%转化为AML,但多数死于骨髓衰竭引起的并发症[3]。相关研究结果表明,某些基因突变可影响MDS病人的预后[21-24]。NAZHA等[25]进行的单因素分析显示,年龄、IPSS-R评分和[STBX]BCORL1、EZH2、RUNX1、SF3B1、TP53基因突变与OS显著相关;而多因素分析显示,只有年龄、IPSS-R评分、[STBX]EZH2、SF3B1、TP53与OS相关。STEENSMA[26]对439例MDS病人18种不同基因的突变进行测序发现,[STBX]TP53、ETV6、ASXL1、EZH2和RUNX1与不良预后相关。NAZHA等[27-28]对610名MDS病人进行单因素分析发现,基因[STBX]EZH2、TP53、RUNX1、NPM1和SF3B1的突变对OS有显著影响。相关研究将基因突变与MDS的其他预后参数相结合,提出了新的预后积分系统[29]。本文对突变频率>2%的基因分别进行log-rank检验,结果显示,[STBX]ETV6、MPL、NRAS、KIT以及TP53基因与更短的OS显著相关。本文结果与相关研究不太一致,可能与病例数量较少有关。
综上所述,基因突变对预测MDS病人的预后具有重要意义。但本文总体病例数量偏少,在后续的研究中需纳入更多的病例及进行多中心研究,减少统计偏倚。
[参考文献]
[1]中华医学会血液学分会. 骨髓增生异常综合征中国诊断与治疗指南(2019年版)[J]. 中华血液学杂志, 2019,40(2):89-97.
[2]HAIDER M, DUNCAVAGE E J, AFANEH K F, et al. New insight into the biology, risk stratification, and targeted treatment of myelodysplastic syndromes[J]. American Society of Clinical Oncology Educational Book American Society of Clinical Oncology Annual Meeting, 2017,37:480-494.
[3]HAASE D, GERMING U, SCHANZ J, et al. New insights into the prognostic impact of the karyotype in MDS and correlation with subtypes: evidence from a core dataset of 2 124 patients[J]. Blood, 2007,110(13):4385-4395.
[4]INTRAGUMTORNCHAI T, PRAYOONWIWAT W, SWASDIKUL D, et al. Myelodysplastic syndromes in Thailand: a retrospective pathologic and clinical analysis of 117 cases[J]. Leukemia Research, 1998,22(5):453-460.
[5]JUNG S W, LEE S Y, JEKARL D W, et al. Cytogenetic characteristics and prognosis analysis in 231 myelodysplastic syndrome patients from a single institution[J]. Leukemia Research, 2011,35(6):735-740.
[6]BEJAR R, STEVENSON K, ABDEL-WAHAB O, et al. Clinical effect of point mutations in myelodysplastic syndromes[J]. The New England Journal of Medicine, 2011,364(26):2496-2506.
[7]PAPAEMMANUIL E, GERSTUNG M, MALCOVATI L, et al. Clinical and biological implications of driver mutations in myelodysplastic syndromes[J]. Blood, 2013,122(22):3616-3627.
[8]BEJAR R, STEVENSON K E, CAUGHEY B A, et al. Validation of a prognostic model and the impact of mutations in patients with lower-risk myelodysplastic syndromes[J]. Journal of Clinical Oncology, 2012,30(27):3376-3382.
[9]TIU R V, VISCONTE V, TRAINA F, et al. Updates in cytogenetics and molecular markers in MDS[J]. Current Hematologic Malignancy Reports, 2011,6(2):126-135.
[10]HAFERLACH T, NAGATA Y, GROSSMANN V, et al.plastic syndromes[J]. Leukemia, 2014,28(2):241-247.
[11]XU Y Y, LI Y, XU Q Y, et al. Implications of mutational spectrum in myelodysplastic syndromes based on targeted next-generation sequencing[J]. Oncotarget, 2017,8(47):82475-82490.
[12]HOU H A, TSAI C H, LIN C C, et al. Incorporation of mutations in five genes in the revised International Prognostic Scoring System can improve risk stratification in the patients with myelodysplastic syndrome[J]. Blood Cancer Journal, 2018,8(4):39.
[13]MALCOVATI L, KARIMI M, PAPAEMMANUIL E, et al. [STBX]SF3B1 mutation identifies a distinct subset of myelodysplastic syndrome with ring sideroblasts[J]. Blood, 2015,126(2):233-241.
[14]PATNAIK M M, LASHO T L, HODNEFIELD J M, et al. [STBX]SF3B1 mutations are prevalent in myelodysplastic syndromes with ring sideroblasts but do not hold independent prognostic value[J]. Blood, 2012,119(2):569-572.
[15]GILL H, LEUNG A Y, KWONG Y L. Molecular and cellular mechanisms of myelodysplastic syndrome: implications on targeted therapy[J]. International Journal of Molecular Sciences, 2016,17(4):440.
[16]CHEN T C, HOU H A, CHOU W C, et al. Dynamics of [STBX]ASXL1 mutation and other associated genetic alterations du-ring disease progression in patients with primary myelodysplastic syndrome[J]. Blood Cancer Journal, 2014,4:e177.
[17]STENGEL A, KERN W, HAFERLACH T, et al. The impact of [STBX]TP53 mutations and TP53 deletions on survival varies between AML, ALL, MDS and CLL: an analysis of 3307 cases[J]. Leukemia, 2017,31(3):705-711.
[18]WALTER M J, DING L, SHEN D, et al. Recurrent [STBX]DNMT3A mutations in patients with myelodysplastic syndro-mes[J]. Leukemia, 2011,25(7):1153-1158.
[19]陳婉,张日,晁红颖,等.160例骨髓增生异常综合征患者[STBX]JAK2 V617F突变的检测及临床意义[J]. 中华血液学杂志, 2010,19(1):49-51.
[20]GREENBERG P L, TUECHLER H, SCHANZ J, et al. Revised international prognostic scoring system for myelodysplastic syndromes[J]. Blood, 2012,120(12):2454-2465.
[21]HOU H A, KUO Y Y, TANG J L, et al. Clinical implications of the [STBX]SETBP1 mutation in patients with primary myelodysplastic syndrome and its stability during disease progression[J]. Clinical Lymphoma Myeloma and Leukemia, 2014,14: S153.
[22]MURPHY D M, BEJAR R, STEVENSON K, et al. NRAS mutations with low allele burden have independent prognostic significance for patients with lower risk myelodysplastic syndromes[J]. Leukemia, 2013,27(10):2077-2081.
[23]TAKAHASHI K, JABBOUR E, WANG X, et al. Dynamic acquisition of [STBX]FLT3 or RAS alterations drive a subset of patients with lower risk MDS to secondary AML[J]. Leukemia, 2013,27(10):2081-2083.
[24]CARGO C, BOWEN D. Individual risk assessment in MDS in the era of genomic medicine[J]. Seminars in Hematology, 2017,54(3):133-140.
[25]NAZHA A, NARKHEDE M, RADIVOYEVITCH T, et al. Incorporation of molecular data into the Revised International Prognostic Scoring System in treated patients with myelodysplastic syndromes[J]. Leukemia, 2016,30(11):2214-2220.
[26]STEENSMA D P. The evolving role of genomic testing in assessing prognosis of patients with myelodysplastic syndromes[J]. Best Practice & Research Clinical Haematology, 2017,30(4):295-300.
[27]NAZHA A, BEJAR R. Molecular data and the IPSS-R: how mutational burden can affect prognostication in MDS[J]. Current Hematologic Malignancy Reports, 2017,12(5):461-467.
[28]NAZHA A, AL-ISSA K, HAMILTON B K, et al. Adding molecular data to prognostic models can improve predictive power in treated patients with myelodysplastic syndromes[J]. Leukemia, 2017,31(12):2848-2850.
[29]肖志堅. 骨髓增生异常综合征的分子生物学研究:现况与启示[J]. 白血病·淋巴瘤, 2014,23(9):513-514.
(本文编辑黄建乡)
