SNP微阵列和基因捕获测序联合在智力障碍或精神发育迟缓儿童中的应用价值
2022-05-28季芳
季芳
南京医科大学附属儿童医院 康复科,江苏 南京 210008
引言
据不完全统计,我国智力障碍新生儿发生率为0.43%~0.96%,该病多发生在儿童时期5岁以下,表现为智力低下或生长发育缓慢,其致病原因复杂,遗传不规则性较大。目前该病超过一半的临床人群不能被明确地诊断出病因。本研究选取19例原因不明的智力障碍或发育迟缓的患儿,通过单核苷酸多态性(Single Nucleotide Polymorphism,SNP)微阵列技术和新一代基因捕获测序技术先后对全基因组拷贝数变异(Copy Number Variation,CNV)和发病基因变异进行检测,以期了解流行病学相关的造成儿童智力障碍和精神发育迟缓的遗传性病因不明的潜在影响因子,该技术成果若能应用于诊断和遗传咨询服务中,可减少产前诊断二次筛查的次数,对优生计划来说十分重要,尤其是对原发性病因研究方面意义重大[1-2]。
1 材料与方法
1.1 一般资料
通过资料分析法对2018年9月至2020年3月在南京医科大学附属儿童医院康复门诊接受治疗的非明确性病因所造成的智力障碍或精神发育迟缓患儿19人[3-5],分别采用发育诊断量表和韦氏儿童智力量表中国修订本(WISCRC)对其进行智力评定,病史采集与分析流程如图1所示。本研究得到患儿家属认可并签署知情同意书,并通过院方伦理委员会的审核批准(201907238-1)。

图1 病史采集与分析流程图
纳入标准:① 年龄6个月至14岁;② 发育评分低于49分或智商低于51分;③ 无遗传性疾病和遗传代谢病。
排除标准:① 存在孕期不良因素;② 有围生期损伤史;③ 有生后脑损伤史。
1.2 方法
在受试者同意的情况下,取患儿静脉血5 mL,并相应抽取其父母静脉血5 mL。操作流程:首先,做SNP微阵列检测,若有异常标本用实时定量聚合酶链反应(Polymerase Chain Reaction,PCR)检测芯片进一步处理,并确定其亲本的对应点;若无异常则进行标准的基因捕获测序检测,通过Sanger测序查找突变点,同时也相应测试其亲本的对应位点[6]。
1.2.1 SNP微阵列技术
选用HumanCytoSNP-12 Bead Chip Kits(美国Illumina公司)芯片(内有30万个检查的位点)进行检测,取患儿基因组DNA 200 ng与4 μL NaOH溶液混合,在室温状态下静置10 min再继续DNA变性操作,交替加入68 μL马赫稀释试剂和75 μL标准核膜基液将DNA进行复性处理,放入杂交炉37 ℃环境下静置20 h;之后加入50 μL段化性测试基液在37 ℃状态下等待1 h再继续进行DNA片段化处理,稍后注入100 μL博飞美科缓冲溶液和300 μL的C3H8O,在4 ℃下以14000 r/min离心处理并收集DNA沉淀;然后溶入46 μL的人参皂苷试剂,在48 ℃的环境下静置1 h后再次悬置片段化处理DNA,注意将得到的DNA溶液置入芯片后放入密封的杂交箱中,并在48 ℃下继续杂交16 h。除掉薄层后,用细胞内二硫化物还原剂(Protected Borane Tcep,PB1)溶液清洗掉片上的非交叉性和非特异性杂交的DNA片段,启动单碱基扩增来扩增芯片上的DNA片段。在芯片清洗过程中,从PB1清洗液中除去扩展的多余反应液,继续用包被试剂浸渍芯片,再采用负压干燥,接着使用Isean Rapid System继续采集,后经KaryoStudio 软件模块进行分析[7]。该项技术手段较传统SNP检测法(凝胶电泳进行检测)有更快速、高效、自动化的特点,且针对问题DNA链的二级结构不易产生人工假象,获得的结果可信度高。
1.2.2 新一代基因捕获测序技术
采用最新德国肖特(SCHOTT)NEXTERION®Slide AL型号的基因芯片进行测定,步骤如下:① 针对智力障碍或基因缺陷而引起的生长迟缓问题,本研究设计了1016个基因探针,这些探针目前已被识别或被报道可能与智力障碍或精神发育迟缓有关,本次使用的探针由美国AngelenTech公司提供,能有效地覆盖上述及邻近的所有外基因区域,其中含有亚区域50 bp[8];② 第二代高通量序列:针对智力障碍或发育迟缓基因检测应用SNP微阵列和二代基因捕获测序技术联合的技术方法。基因组DNA获取自外胚层血液,基因组DNA随机中断为250~300 bp的片段,并进行细节修复、连接密封。其经连接介导(Ligation-Mediated,LM)-PCR放大、净化,取得DNA文库,并进行文库的质量检验。接着将LM-PCR产物与捕获芯片进行探针杂交、摄取、包装和洗涤,运用定量PCR检测富集效率[9]。使用Illumina Hiseq 2500高通量测序仪器进行测序,测序长度为2×150 bp;③ 进行必要的DNA生物信息辨证与突变验证:经序列仪获得原始序列再结合SoftGenetics GeneMarker 3.0.0美国版基因分析软件,通过现有的UCSC数据库比对人类基因组提供的HG19参考序列,并评估目标区域的序列覆盖率和质量,这要求每个样本的平均序列深度超过100倍,靶区覆盖率超过99%[10]。利用HGMD基因突变数据库、人类基因组数据库和DHSNP数据库信息对检测到的变异位点进行评价,以识别基因、坐标、RNA位点和氨基酸变化,再结合SIFT和PPLYS2等软件预测单个核苷酸变化对功能蛋白的影响。最后,研究与疾病相关的候选基因的突变点,然后进行Sanger测序和亲代染色体DNA分析。新一代基因捕获测序技术有如下优点:① 高通量;② 高效测序形式;③ 更长读取区域;④ 结果精准并可测出极小的变异相;⑤ 低起始量;⑥ 低成本。
2 结果
2.1 一般情况比较分析
本研究选取的19名智力障碍或精神发育迟缓患儿,其年龄在6个月至14岁,其中男童11人和女童8人,对0~4岁患儿采用发育诊断量表和对所有患儿采用韦氏儿童智力量表中国修订本(WISC-RC )评定,结果如表1所示。其中发育商低于40分或智商值低于51分者均表现出不同程度的智力低下问题或发育不良,且为先天性、静止性病程,其体检中血液、尿代谢检测和生物化学等指标表现正常,家族史中也没有智力障碍现象。

表1 19例患儿的发育诊断量表和韦氏儿童智力量表中国修订本(WISC-RC )评定结果
2.2 SNP微阵列、新一代基因测序诊断指标及实时PCR芯片验证
19例患儿中,发育不良可疑性病例2的SNP微阵列分析显示21q22.2q22.3区间内存在基因微缺失,如图2所示,其红色处为发现的CNV,对应的箭头标示为区域缺失段;由表2联合诊断结果可知,3名患儿临床表现均有发育迟缓体态,且伴有SNP微阵列区位缺失,遗传模式N/A,属新生致病性变异。病例2的检测结果是染色体21号q22.2q22.3区发生了6.1 Mb缺陷,选择PCNT及DSCAM 的Gene E47和E2 外显子使用实时PCR技术检查芯片结果,2-ΔΔct值为0.31、0.35,父亲2-ΔΔct值为0.74、0.67,母亲2-ΔΔct值为0.62、0.82,证明所检测的主体PCNT及DSCAM 的Gene E47和E2外显子出现杂合缺陷,而父母是正常的(通常情况下2-ΔΔct<0.6即代表杂合缺失现象)[11]。

图2 发育迟缓病例2的SNP微阵列分析显示21q22.2q22.3区域缺失
2.3 基因变异与智力障碍或精神发育迟缓关联性
经数据库查询发现以上变异与智力障碍或精神发育迟缓相关,得出的分析结果如表2所示。研究结果显示19名参与者中有16名患儿的SNP微阵列分析结果为阴性,确认了6例患有单一遗传疾病的儿童病例(4例新生儿突变、2例存在复合杂合子突变,突变来自正常父母表型);患儿例7表现为发育迟缓,同时伴有先天性心脏病和精神错乱的疼痛,经检查发现为切割部位斑点复合杂合子突变,图3为该先天性多发异常-肌张力衰减-癫痫综合征I型疾病患者的基因突变验证及父母测序结果。这是一种复合型突变,包括切割点Pig N Gene c.1172+1G>C和c.1434+5G>A复合突变。其中c.1172+1G>C是一个共同的剪切点突变,而c.1434+5G>A则是一个很有可能的剪切点突变,软件预测提示该突变可能影响剪切。另有3个病例存在可疑的基因突变情况(1例为新生儿缺陷移码突变、2例为表型正常的父亲移码突变所致)[12-15]。

图3 患儿例7(阴性及有先天性心脏病及癫痫I型综合征)的基因突变验证及父母测序
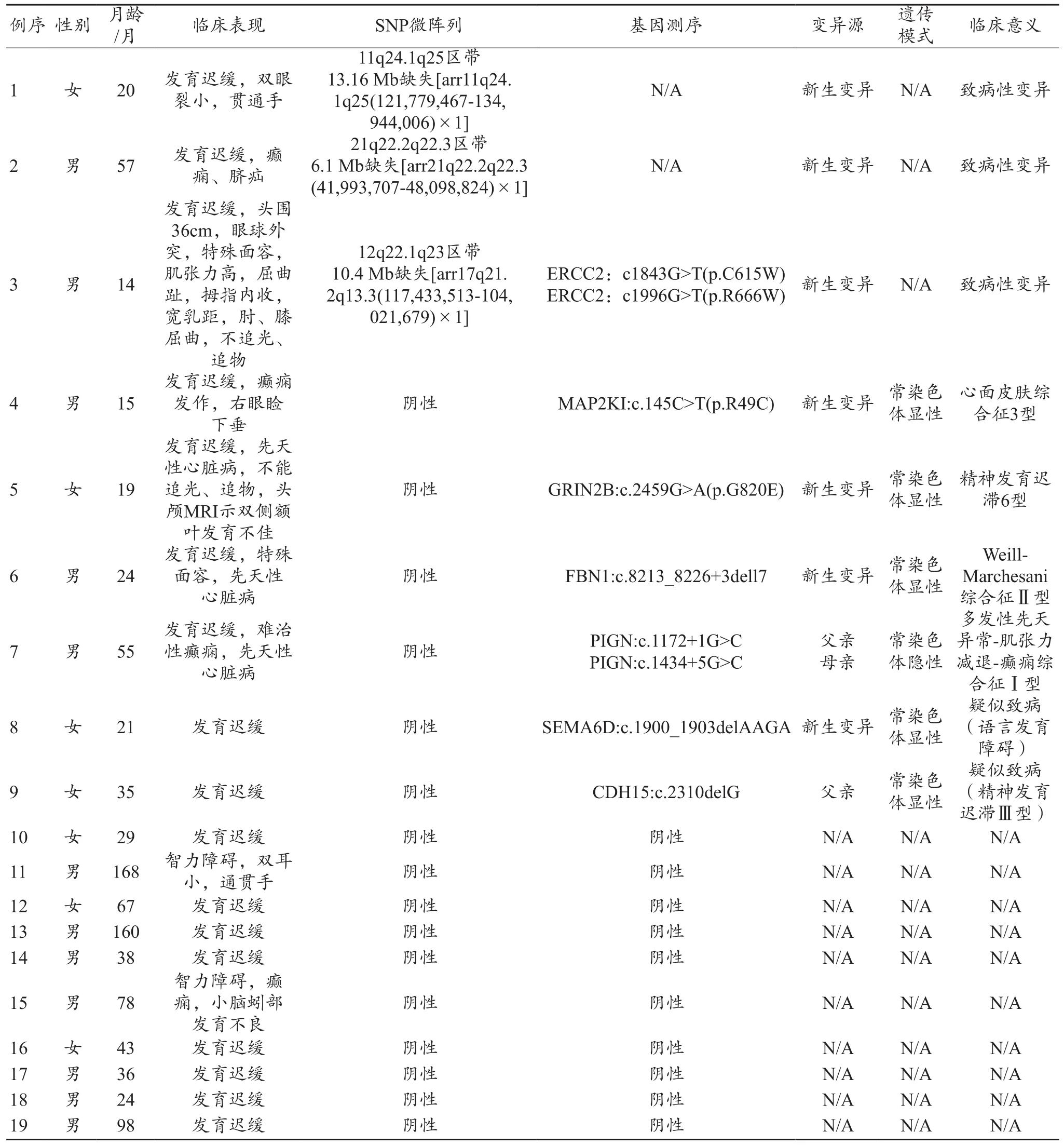
表2 19名患儿的SNP微阵列和新一代基因捕获测序技术联合诊断结果
3 讨论
随着医疗设备及医学技术的高速发展,基因捕获测序技术也不断发展,其中第三代基因捕获测序技术的诞生,将以SNP微阵列和新一代基因捕获测序技术为代表的大规模测序正式推入新时代。SNP微阵列和新一代基因捕获测序技术联合属于更高通量测序技术,其比传统的基因捕获测序检测更具优越性[16]。SNP微阵列联合新一代基因捕获测序技术可同时进行几百万甚至几千万条DNA序列的检测,实现对某一个物种重复组和基因组的整体分析。其具有检测速度快、准确率高、成本低、覆盖面广、扩展性强等特点,特别适合于儿童智力障碍或发育迟缓等遗传性病因不明疾病相关影响因子的筛查。
综上所述,本研究采用SNP微阵列和基因捕获测序技术联合对智力障碍或发育迟缓儿童进行诊断,进而筛查出染色体异常、CNV和个体遗传突变诱发病变后果。研究从3人CNV分析中发现有1人在染色体11号的末端q24.1q25区带存在13.16 Mb缺失,被证实会引起发育迟缓、双眼裂小、贯通手症状;另1人为染色体21号的末端q22.2q22.3区带存在6.1 Mb缺失,被证实会引起发育迟缓、癫痫、脐疝等不良症状;最后1人为染色体12号的末端存在q22.1q23区带10.4 Mb缺失,被证实会引起发育迟缓、头围异样、眼球震颤、特殊面容、肌张力高、屈曲趾、拇指内收、宽乳距、肘膝屈曲、不追光等症状。通过PCR定量检查发现上述3名患儿所携带的CNVs都是新生变异,病原体是明确的,临床和报告的隐性模型是一致的。
鉴于当下国内外基于诊断智力缺陷或单一基因发育迟缓的第二代序列主要包括外部质子组的序列和靶基因捕获序列,其存在着检测成本高、数据分析复杂、难以在临床上广泛应用的缺点;本研究的创新点在于设计了1016个基因探针,这些探针目前已被识别或可能与智力缺陷或生长迟缓有关,通过在微阵列芯片测试并针对阴性结果对基因进行排序,筛选出了6个单纯基因疾病病例、3个病例存在可疑的情况;其中患儿例7属阴性及有先天性心脏病和癫痫存在,检测结果表明在多个前期异常——肌肉衰竭——癫痫中进行的研究提示,在患有I型癫痫痛综合征的患儿中发现了该突变,其临床和遗传分析的结果与过住文献所述的一种癫痫综合征(OMIM:614080)相符[17]。
除了上述6个经证实的病例外,本研究还在1个病例中发现了CDH15,一种智力迟缓基因Ⅲ型,出现移码突变,正常情况下其父本也应存在c.2310delG移码突变,其突变蛋白预测应为病原体,但有关参考文献显示该基因为外显不全可能,因此该突变只能被怀疑为可能与疾病有关。但对该基因的相关研究太少,突变是病原体所致目前尚未得到充分的研究证实。另外研究还发现,有1例患儿携带SEMA6D基因,被检测出为c.1900_1903delAAGA的新生杂合突变,Ercan-Sencicek等[18]研究表明该基因可能与语言发育障碍有关,也不排除与儿童临床表型关联。
另外,类似的神经系统类不明原因智力低下病症可能由CNV或简单的基因突变引起,而不同的CNV突变可能根据所涉及的基因差异而具有不同的临床表现。本研究的技术革新体现在SNP微阵列和新一代测序技术联合可以有效地弥补传统单一技术层面测序技术的不足,其可准确诊断单个基因对智力低下或发育迟缓的影响,该技术创新能很好地提供病因学诊断及遗传机制研究,为遗传咨询、产前诊断、新生儿疾病发作风险评估提供科学的理论依据,具有重要的临床意义。
由于智力障碍或发育迟缓的患儿存在着临床诊断多样性和复杂性等难题,即使在发达国家,也只有约50%的患者有中、重度病理诊断。随着围生期检测技术的发展和孕、产妇生活质量的提高,不良环境因素影响的比例相对大幅下降,而遗传因素的影响却表现得越来越突出,25%~50%的智力缺陷是遗传基因所致,其存在较大的隐蔽性。如何深入绘制患儿完整的基因突变图谱、践行基因干预医治是永恒的课题。如何破译更多致病基因是下一步的探究方向,尤其是不明遗传因素性疾病,包括染色体异常、CNV和个体遗传突变等,尚需深入研究。
4 结语
智力障碍或发育迟缓患儿的致病原因往往较复杂,遗传性、不规则性较大。目前该病症超过一半的临床人群不能被诊断出明确的病因。而采用SNP微阵列和新一代基因捕获测序技术对不明原因导致的智力障碍或发育迟缓患儿的研究,可为病因学诊断及遗传机制研究提供良好的基础信息,并为遗传咨询、产前诊断、新生儿疾病发作风险评估提供科学的理论依据。
