肠杆菌科细菌耐药性的遗传稳定性
2022-05-14王少林
刘 鹏 , 王少林
(中国农业大学动物医学院 , 北京 海淀 100193)
肠杆菌科细菌是社区和医院获得性感染的重要病原体,多重耐药肠杆菌科细菌的相继出现极大地增加了疾病治疗过程中的难度,已经成为人类健康的重大威胁。耐药性的维持通常会对细菌造成一定的适应性代价,表现为细菌生长速率、毒性和传播的减弱,而补偿性进化[1]、无适应性代价和适应性增加的耐药突变的出现[2]、耐药基因与其他基因的遗传连锁和协同选择[3]以及质粒的稳定性机制[4]均在一定程度上促进了肠杆菌科细菌耐药基因稳定遗传。本文综述了肠杆菌科细菌的耐药现状和传播现状、耐药基因适应性代价的研究方法和影响因素以及耐药性稳定遗传的驱动因素,以期为解决肠杆菌科细菌抗生素耐药性的持续存在这一重大公共卫生问题提供新的视角。
1 肠杆菌科细菌的耐药现状和传播现状
肠杆菌科细菌属于革兰阴性菌家族,常在人类或者动物的肠道生存,是不同类型的社区和医院获得性感染的常见病原体之一。
氨基糖苷类抗生素是临床治疗革兰阴性菌感染的老牌抗生素,肠杆菌科细菌主要通过产生氨基糖苷钝化酶和修饰药物作用靶点导致耐药。其中产氨基糖苷钝化酶是导致该类抗生素失活的最常见机制,其包括乙酰转移酶(AACs)、腺苷转移酶(ANTs)和磷酸转移酶(APHs)。16S rRNA甲基化酶通过修饰药物作用靶点,介导肠杆菌科细菌对大多数临床常用氨基糖苷类抗菌药物产生高水平耐药,主要包括ArmA、RmtA和RmtB等,大多数16S rRNA甲基化酶的结构基因均位于可转移的质粒,且与可移动遗传因子相关联,导致水平传播。
氟喹诺酮类耐药决定区(Quinolone resistance determining regions,QRDR)基因的点突变,是肠杆菌科细菌对氟喹诺酮类抗生素产生耐药的主要原因,特别是多个位点同时突变导致高水平耐药[5]。质粒介导的喹诺酮耐药基因(Plasmid-mediated quinolone resistance,PMQR)包括qnrA基因及其等位基因qnrS、qnrB、qnrD和qnrC,能降低环丙沙星活性的氨基糖苷乙酰转移酶的变异体基因aac(6′)-Ib-cr和编码药物外排泵的基因(qepA和oqxAB),虽然大多数PMQR带来低水平的耐药,但是由于PMQR既可以通过垂直传播,也可以通过水平传播的特性,给人类和动物的健康带来了不容忽视的威胁。
β-内酰胺类抗生素是目前应用最广泛的药物,其产生耐药性的常见原因是β-内酰胺酶的产生,包括超广谱β-内酰胺酶(ESBLs)和头孢菌素酶(AmpC酶)。产ESBLs和AmpC酶的肠杆菌科细菌均可以通过染色体和质粒介导的方式传播耐药性,携带ESBLs和AmpC酶的质粒通常也会协同氨基糖苷类、磺胺类、氟喹诺酮类等其他类耐药基因共同转移,扩大了肠杆菌科细菌引起感染的强度。临床上常见的ESBLs主要包括blaTEM、blaSHV和blaCTX-M三大类别,其中blaCTX-M是世界各地普遍存在的ESBLs。
碳青霉烯类抗生素是一类非典型β-内酰胺类抗生素,因其对β-内酰胺酶稳定,通常用于ESBLs和AmpC酶产生菌感染的治疗,被视为治疗多重耐药革兰阴性菌感染的最后一道防线[6]。碳青霉烯酶可以使碳青霉烯类抗生素失活,由碳青霉烯酶介导的耐药机制是导致碳青霉烯类耐药的肠杆菌科细菌(Carbapenem-resistantEnterobacteriaceae,CRE)快速传播的重要原因[7]。迄今为止,CRE中传播最广泛的是肺炎克雷伯菌,它是导致呼吸道等医疗卫生相关感染的重要原因。根据Ambler分类法,碳青霉烯酶可以分为A、B和D三类,KPC、IMP、VIM、OXA-48、NDM是常见的碳青霉烯酶,这些碳青霉烯酶常常位于质粒、整合子、转座子等可移动遗传元件上,极大地增强了碳青霉烯类抗生素耐药基因的迅速蔓延。2009年,新德里金属β-内酰胺酶(NDM)被首次报道,因为NDM对几乎所有的β-内酰胺酶药物(除了多黏菌素和替加环素)耐药,所以携带该基因的细菌被称为“超级细菌”。
近年来,CRE的广泛传播严重影响了碳青霉烯类药物的疗效,多黏菌素和替加环素被认为是最为有效的CRE治疗方案[7]。随着2016年可转移耐药基因mcr-1在大肠杆菌中的发现,黏菌素的临床潜力已经被质粒介导的mcr耐药基因的全球传播显著削弱。当前肠杆菌科细菌中已经有mcr-1和blaNDM共存的报导,甚至Sun等[8]在2016年报道了mcr-1和blaNDM共存于同一个质粒。2019年,质粒介导的高水平替加环素耐药基因tet(X3)和tet(X4)在肠杆菌科细菌中被发现[9-10]。数据库挖掘和回顾性筛选分析证实,tet(X3)和tet(X4)在临床细菌中普遍存在,甚至与blaNDM-1共存的细菌中也存在[9]。同时,tet(X4)阳性的大肠杆菌菌株,包括与mcr-1共存的分离株,已在中国的猪、鸡、土壤和灰尘样品中广泛检测到[10]。这些报道为我们敲响了警钟。试想,如果 CRE同时携带mcr和tet(X),则会成为无敌的“超级细菌”,人类的感染将真正地陷入无药可治的局面。
2 肠杆菌科细菌耐药基因的适应性代价
2.1 适应性代价的产生原因 面对抗生素选择压力时,细菌可以通过新生突变和耐药基因水平转移(结合、转化和转导)2种方式获得耐药性,而当抗生素压力消除后,由于这些耐药突变机制改变了细菌体内在生理生化中起关键作用酶的结构和表达,通常会给菌体的正常代谢带来额外的负担,导致细菌适应性的降低,表现为细菌生长速率、毒性和传播的减弱,从而产生了适应性代价[11]。特别是在缺乏抗生素选择压力下,如果耐药机制的适应性代价较大,耐药细菌将无法与敏感细菌竞争,从而会很快从群体中消失。所以适应性代价是影响耐药性可逆性的重要生物学参数,是决定耐药菌能否在细菌群体中长久维持的重要因素[11]。
2.2 适应性代价的研究方法 测量适应性代价的常见试验方法有2种,包括菌株的生长曲线的测定和竞争性试验。在相同的纯培养条件下,通过测量耐药菌株和敏感菌株在600 nm处的光密度(OD600)随时间的变化曲线,通过比较菌株的最大生长速率(vmax)、最大的OD600(ODmax)、生长曲线下面积(AUC)和延迟期(Lag)的差别来评估适应性代价的大小[12-13],如图1A所示。竞争性试验被认为是研究适应性代价的黄金标准,包括体外竞争试验(图1B)和体内竞争试验(图1C),体外竞争试验是在标准培养基中进行,体内竞争性试验是在分离耐药菌株常见的动物宿主中进行的,将耐药菌株和敏感菌株以1∶1的计数比例共培养,通过监测竞争前后耐药菌株和敏感菌株的计数比值的变化,直观地反应耐药菌株的适应性强弱,如果耐药菌株存在适应性代价,计数的比值将会变小。Lenski等[14]提出的相对适应性(Relative fitness)的计算方法被广泛用来评估耐药菌株的适应性,计算方式:Relative fitness=ln(A1/A0)/ln(B1/B0),A0和B0分别是竞争开始时突变菌株和野生菌株的细胞数量,A1和B1分别是竞争结束后耐药菌株和敏感菌株的细胞数量。

图1 适应性代价的试验性测量方法
2.3 影响耐药性适应性代价的主要因素 不同的遗传背景下,耐药基因的适应性代价有所不同。Vogwill等[15]通过对发表的耐药性适应性代价的文献进行汇总分析,发现相对于染色体突变产生的耐药性,细菌获得介导耐药性的质粒的平均适应性代价较小。这可能是由于染色体突变是细菌基因组上的新生突变,细菌最初不可能拥有适应能力来抵消这些基因突变的代价。相反,当细菌通过获得质粒而进化出耐药性时,它们就获得了一种耐药性决定因素,这种决定因素已经经历了选择,以使其代价最小化[15]。相同的质粒在不同的宿主中的适应性代价不同。Porse等[13]从临床获得的肺炎克雷伯菌(Kp33)中分离出1株巨大的结合质粒pKP33,将其结合转移到不同宿主后,发现大肠杆菌(Ec37和Ec38)结合pKP33产生的适应性代价显著高于肺炎克雷伯菌(Kp08)结合pKP33的适应性代价。
耐药基因的适应性代价受不同环境压力的影响。Lin等[16]通过耐利福平菌株与敏感菌株在不同营养水平下的竞争试验,发现在富营养化条件下,耐药菌株与敏感菌株的绝对数量比值迅速下降,而在贫营养化条件下,耐药菌株与敏感菌株的绝对数量比值缓慢下降或保持稳定,这表明贫营养化条件下,耐药细菌变得更有竞争力。环境温度也是影响适应性代价的重要因素,研究发现相比于37 ℃条件,在42 ℃条件下IncK1质粒的适应性代价更高,而IncK2质粒的适应性代价在37 ℃条件或者在42 ℃条件下没有显著变化,这可能是IncK2主要在家禽中被发现,而IncK1总是在各种哺乳动物中被报道的原因[17]。
耐药基因的适应性代价与基因的异位显性效应(Epistasis)有关,异位显性也称上位性[18],指一个基因突变受其他已存在的基因突变影响的现象,正向异位显性效应能减少适应性代价,负向异位显性效应相应地增加适应性代价。研究发现,对链霉素和利福平耐药的大肠杆菌(RifR和StrR)的适应性代价均远远高于单一耐药的大肠杆菌(RifR或者StrR)[19]。相对于负向异位显性效应,正向异位显性效应较普遍,这在一定程度上也加快了多重耐药性的传播和进化。
3 肠杆菌科细菌耐药基因稳定遗传的驱动因素
3.1 补偿性进化 补偿性进化是细菌为了改善自身适应性代价所发生的适应性突变,是避免耐药菌株被敏感菌株轻易击败的关键因素[1]。补偿性进化可以通过耐药基因的突变积累来恢复耐药菌的适应性。Yang等[20]研究发现,黏菌素耐药基因mcr-3的2个变异体mcr-3.1和mcr-3.5之间仅存在3种氨基酸替换(M23V、A457V和T488I),mcr-3.5通过积累的3个氨基酸的替换,改善了黏菌素耐药基因的适应性。通过适当调控基因表达来调节耐药的适应性代价可能对维持耐药基因至关重要,也是可转移耐药质粒传播抗生素耐药性的重要原因之一[3]。pKpQIL是在肺炎克雷伯菌ST258中发现并携带碳青霉烯类耐药基因blaKPC的大型耐药质粒,研究发现,pKpQIL变体会对宿主细菌的代谢、运输、信号转录等过程中的通路产生普遍的干扰,这些表达的变化可以补偿质粒对细胞的影响,从而降低质粒的适应度代价[21]。
3.2 无适应性代价和适应性增加的耐药突变 大多数耐药突变的出现均会对菌体产生或大或小的适应性代价,但在肠杆菌科细菌进化的无数年历史中,还是出现了罕见的无适应性代价和适应性增加的耐药突变现象。研究发现,大肠杆菌编码核糖体S12蛋白的rpsL基因发生点突变(K42R),导致了对链霉素产生耐药性,突变株和野生株在体外和体内进行竞争试验,发现rpsL基因的突变仅仅在体外对大肠杆菌345-2 RifC有很小的适应性影响,而在体内,rpsL基因的突变没有带来适应性代价[22]。大肠杆菌对氟喹诺酮类药物的耐药性是一个长期选择进化的结果,研究发现,大肠杆菌中有多达5个氟喹诺酮类耐药突变的积累通常与适应性显著降低有关,进化得到的耐药突变体极大地提高了细菌的适应性,同时显著地增加了抗生素耐药性[23]。
3.3 遗传连锁和协同选择 耐药基因与其他耐药基因或毒力基因的遗传连锁决定了它们的协同选择[2],通常意味着这2个或多个基因共处于1条染色体和同一个质粒上,或者较小的遗传单位,包括可移动的转座子和捕获外源基因的整合子。遗传连锁和协同选择的可能结果是,一种抗生素的选择会直接筛选出多重耐药细菌[3]。遗传连锁和协同选择促进了耐药肠杆菌科细菌的流行和传播[24]。大肠杆菌ST131、肺炎克雷伯菌ST258以及肠炎沙门氏菌DT104等流行性肠杆菌科病原菌能在全球占据主要地位的重要原因是它们的基因型将抗生素耐药基因、毒力基因和传播特性基因成功地整合到了一起,形成了一个具有高适应性的生物组合[25]。几乎所有的大肠杆菌ST131都是多重耐药菌,携带blaCTX-M-15的质粒也在该谱系中稳定下来,随后在全球流行传播[26]。肺炎克雷伯菌ST258携带的抗生素耐药基因几乎覆盖了各种类型的抗生素,大多数都携带碳青霉烯类耐药基因blaKPC[21],部分ST258减少了孔蛋白的表达,导致细菌对黏菌素耐药。目前已经报道的NDM阳性肠杆菌科细菌尚无明显的优势流行株,但blaNDM多数定位于质粒上,blaNDM与其他抗生素耐药基因的遗传连锁和协同选择,是促进blaNDM持久性和快速传播的重要原因[27-28]。
3.4 质粒的进化和稳定遗传 作为遗传连锁和协同选择最大的可移动元件,质粒被认为是原核生物进化的主要驱动力,无论有无环境选择压力,携带有益宿主基因质粒都会以一定的传播频率入侵到不同宿主菌群。在某些条件下(例如抗生素环境和重金属环境),质粒可以编码对宿主有益的基因,但质粒对维持宿主基本的生存能力不是必需的,所以质粒的存在往往会对宿主菌造成一定的适应性代价。如图2所示,为确保携带有益基因质粒的稳定遗传,宿主菌或质粒的基因组通常会发生补偿性进化,包括单核苷酸多态性(Single nucleotide polymorphism,SNPs)和大规模结构的改变(插入、删除、倒置和易位),从而逐步达到稳定遗传的结果。质粒自身也会编码稳定性机制,包括多聚体识别系统[29]、质粒主动分区机制和质粒成瘾系统[4]。多聚体识别系统和主动分区机制确保了质粒在子代的可靠遗传[4]。质粒成瘾系统是质粒编码的特定的生存机制,包括限制修饰系统(Restriction modification system,RM)[30]和毒素抗毒素系统(Toxin-antitoxin-like system,TA)[31],RM系统帮助质粒越过宿主的RM系统的屏障,TA系统可以杀死没有遗传母代细胞完整质粒结构的子代细胞。有学者认为通过破坏质粒稳定性来消除质粒可能是解决细菌多重耐药的一种新的有效策略[30]。
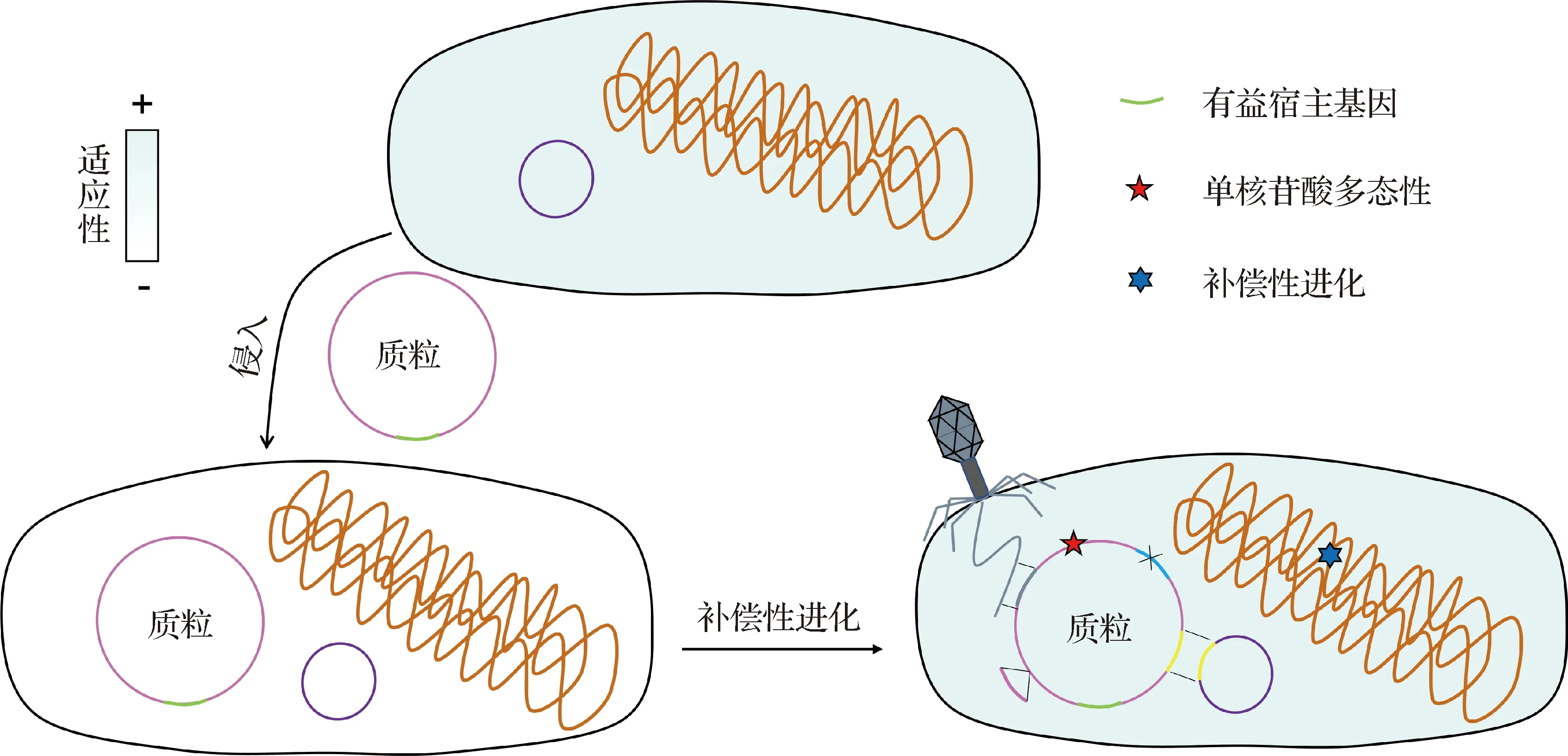
图2 质粒的进化和稳定遗传
4 展望
由于肠杆菌科细菌的高度进化潜力,最初用于治疗肠杆菌科细菌感染的抗生素的黄金时代正迅速转向青铜时代。在肠杆菌科细菌耐药性肆虐的今天,依靠耐药性自身的适应性代价达到耐药性的逆转已经成为了一种奢望。加强对肠杆菌科细菌特定耐药基因[例如blaNDM、mcr和tet(X)]的适应性代价和补偿性进化研究,将有助于更加全面地理解肠杆菌科细菌病原体抗生素耐药性的发展轨迹,同时可以预测潜在病原体在接触药物后可能采取的进化路径。适应性代价和补偿性进化与环境因素有很强的相关性,因此需要使用动物模型对携带特定耐药机制的病原菌的定植、竞争、传播进行进一步的研究,以期找到影响特定耐药基因和耐药质粒转移和稳定遗传的关键基因。进一步可以利用不断进步的基因组学、生理学和抗生素耐药机制的知识,指导新型药物和药物靶点的选择,通过破坏耐药机制基因环境的稳定性,或者将耐药突变关联到一个具有高的适应性代价和不易补偿性进化的基因环境,从而在一定程度上限制肠杆菌科细菌耐药性的传播和进化趋势。这些研究将为开发新的抗菌策略和治疗药物提供理论框架,为解决肠杆菌科细菌耐药问题提供新的思路。
