模块化工程大肠杆菌从头合成丁香酚
2022-05-12赵广荣曹嘉誉马雅婷邱泽天
赵广荣,曹嘉誉,马雅婷,邱泽天,李 嘉
模块化工程大肠杆菌从头合成丁香酚
赵广荣1, 2,曹嘉誉1, 2,马雅婷1, 2,邱泽天1, 2,李 嘉1, 2
(1. 天津大学化工学院,天津 300350;2. 教育部合成生物学前沿科学中心和系统生物工程重点实验室,天津 300350)
丁香酚是一种绿色生物农药,主要用于防治番茄灰霉病、葡萄霜霉病和马铃薯晚疫病等主要的农业病害.目前丁香酚是从丁香和罗勒等植物中提取制备,其规模和产能受到限制,还存在废渣排放影响生态环境的问题.本研究采用合成生物学和模块化工程策略,设计构建工程大肠杆菌,以期微生物从头合成丁香酚.将丁香酚异源合成途径分成3个模块,分别是上游的阿魏酸合成模块、中游的松柏醇合成模块和下游的丁香酚合成模块,对每个模块进行不同来源基因的组合筛选和表达优化.利用His标签、敲除甲基供体合成途径中的全局调控基因,优化了上游模块(PcTAL-HpaBC-AtCOMT),减少了中间产物咖啡酸的积累,提高了阿魏酸合成.筛选了4CL和醇脱氢酶基因来源,研究了表达模式,获得最优的中游模块是双顺反子表达At4CL1-AtCCR1,单顺反子表达YahK,松柏醇产量最高.对3个酰基转移酶基因和4个丁香酚合成酶基因进行组合筛选,获得最优的下游模块是单顺反子PhCFAT和GdlEGS2,丁香酚合成最多.最后,在酪氨酸高产底盘中表达丁香酚合成途径,对初始葡萄糖、酵母提取物和诱导剂浓度等进行优化.在适宜的培养基和发酵条件下,丁香酚的产量为114.67mg/L.本文采用模块化代谢工程策略优化丁香酚异源合成途径,改造底盘菌株,首次实现了微生物从头合成丁香酚,具有重要应用前景.
丁香酚;大肠杆菌;代谢工程;合成生物学
化学农药的应用对土壤、水体和大气等生态和环境造成污染和破坏,同时影响了农副产品的质量.生物农药具有杀灭病虫害的选择性强、对人畜安全性高、易降解对环境生态影响小等特点,其开发和使用成为当前全球的热点[1-3].丁香酚是一种高效的植物源生物农用杀菌剂,其具有抗菌活性是阻断质子泵、主动转运以及对细胞内含物的聚沉作用[4],已被农药管理部门批准,应用于防治番茄灰霉病、葡萄霜霉病和马铃薯晚疫病等主要的农业真菌病害.
丁香酚作为天然生物活性化合物主要是从植物中提取分离制备[5],合成生物学和代谢工程的快速发展为天然产物的规模化生产提供了新思路[6-9].结构上,丁香酚属于苯丙烷类天然产物,通过莽草酸途径合成.在微生物中合成丁香酚主要涉及8个功能酶.酪氨酸在氨基裂解酶(TAL)催化下生成对香豆酸,被3-羟化酶(HpaBC)催化生成咖啡酸.通过甲基转移酶(COMT)催化咖啡酸,生成阿魏酸.辅酶A连接酶(4CL)对阿魏酸进行活化,生成阿魏酰-CoA.在还原酶(CCR)催化下,生成松柏醛,进一步被还原成松柏醇.在酰基转移酶(CFAT)催化下生成松柏醇乙酰酯,最后在丁香酚合成酶催化下,脱除乙酸基,生成丁香酚(图1).
模块化工程是合成生物学的一个重要原则,将代谢途径划分为多个模块,分别进行设计构建和调控优化[10-11],实现各代谢模块之间的最优适配性,从而使整个代谢途径的碳代谢流最大化[12-14].模块化工程策略的优点是降低了冗长途径的复杂度,简化了构建和优化过程,节省时间,因此广泛应用于代谢途径的重构中.本文采用模块化代谢工程策略,将丁香酚生物合成途径分阿魏酸(ferulic acid,FA)模块、松柏醇(coniferyl alcohol,CA)模块和丁香酚(eugenol,EG)模块(图1).对不同生物来源的同工酶进行组合和优化,构建了丁香酚的人工合成途径,以期实现工程大肠杆菌以葡萄糖为碳源发酵合成丁香酚.
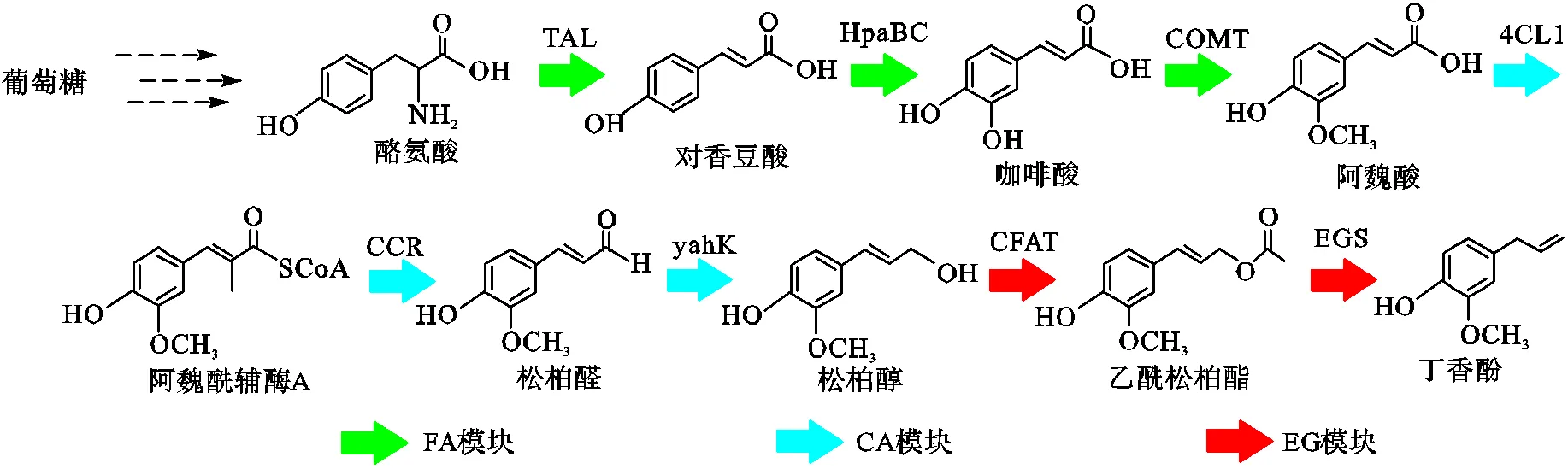
图1 工程大肠杆菌合成丁香酚代谢途径
1 材料与方法
1.1 菌株及质粒
对矮牵牛来源的松柏醇酰基转移酶PhCFAT (GenBank ABG75942.1)基因、三齿拉雷亚灌木的酰基转移酶LtCAAT1(GenBank KF543260.1)基因、拟南芥来源的HXXXD型酰基转移酶AtAT(NCBI Ref-erence Sequence NP_178020.1)基因,来源于啤酒花的丁香酚合成酶CbEGS1(GenBank KF543260.1)基因、手掌参兰花的丁香酚合成酶GdlEGS2(GenBankAKB11750.1)基因、三齿拉雷亚灌木的(异)丁香酚合成酶LtAPS1(GenBank KF543262.1)基因和小茴香的(异)丁香酚合成酶PaAIS1(GenBank ACL13526.1)基因,来源于拟南芥的At4CL1(GenBank CP002684.1)基因、AtCCR1(GenBank AF332459.1)基因、AtCOMT (GenBank CP002688.1)基因,以及来源于克氏杆菌的酪氨酸裂解酶PcTAL(NCBI Reference Sequence XM_007398051.1)基因进行密码子优化,由南京金斯瑞生物科技有限公司合成;来源于大肠杆菌的(GenBank CAQ33376.1)基因、(GenBank CAQ30801.1)基因、(GenBank CAQ34705.1)基因由大肠杆菌BL21(DE3)基因组为模板PCR扩增获得;来源于伯克霍尔德氏菌的基因、天蓝色链霉菌的基因以及幽门螺杆菌的基因皆由实验室保存.大肠杆菌DH5α用于基因克隆和质粒构建,大肠杆菌BL21(DE3)用于丁香酚的合成.本文构建的载体与菌株如表1和表2所示.
表1 菌株

Tab.1 Strains used in this study
表2 质粒

Tab.2 Plasmids used in this study
1.2 主要试剂
丁香酚(纯度≥98%)、松柏醇(纯度≥97%)、阿魏酸(纯度≥98%)、咖啡酸(纯度≥98%)、对香豆酸(纯度≥98%)、酪氨酸(纯度≥98%)购自大连美仑生物科技公司.分析纯甲醇、分析纯乙腈购自康科德科技有限公司.质粒提取试剂盒、凝胶回收试剂盒、无缝克隆试剂盒和DNA聚合酶购自诺唯赞生物科技有限公司,限制性内切酶和T4DNA连接酶购自赛默飞世尔.表3所示的PCR引物由苏州金唯智生物科技有限公司合成.
表3 PCR引物序列

Tab.3 PCR primer sequences
1.3 表达载体构建
本文主要采用PCR扩增、酶切和连接方法或无缝克隆方法,构建的基因表达载体信息如表2所示.
设计同源臂,将His标签设计在引物中,利用无缝克隆试剂盒构建表达载体pMYT4-7.类似地,构建阿魏酸表达载体pMYT12及松柏醇表达载体pMYT23-28.
PCR扩增目的基因,将酶切位点设计在引物中,采用酶切连接方法,将目的基因按需分别连接至pACYCDuet-1、pCDFDuet-1、pETDuet-1和pRSF-Duet-1上,构建表达载体pCJY1-14、pCA1-4.
1.4 培养基与发酵方法
LB培养基:NaCl 10.0g/L,酵母提取物5.0g/L,蛋白胨10.0g/L,pH=7.0.固体LB培养基还需要加入15~20g/L的琼脂粉.使用时按需加入相应工作浓度的抗生素.
M9Y培养基:Na2PO4·12H2O 17.1g/L,KH2PO43.0g/L,NaCl 0.5g/L,NH4Cl 1.0g/L,酵母提取物 1.0g/L,pH=7.2.
发酵培养基配制:向50mL的M9Y培养基加入灭菌的1mol/L MgSO4100μL、1mol/L CaCl25μL、10g/L葡萄糖,使用时按需加入相应工作浓度的抗生素.
发酵:取过夜活化的菌液接种到含有50mL 发酵培养基的250mL锥形瓶中,在37℃、220r/min下培养3~4h至OD600值为0.8~1.0.加入0.1mmol/L IPTG诱导,30℃,250r/min培养24h.发酵结果取3次重复的平均值(标准偏差).
1.5 检测与分析
使用Primaide高效液相色谱(HPLC)测定L-酪氨酸、对香豆酸、咖啡酸等代谢物.使用气相色谱(GC)Agilent 7820A测定阿魏酸、松柏醇、丁香酚等代谢物.
在500μL样品中加入等体积的1mol/L盐酸进行溶解,离心后上清用孔径0.22μm滤膜过滤,用于HPLC分析检测L-酪氨酸.使用4.6×250mm C18色谱柱,流动相组成为95%甲醇∶5%水,再添加15g/L磷酸,流速1mL/min;紫外检测波长230nm,进样量10μL.标准曲线至少利用5个点标定,相关系数2大于0.99(下同).
取发酵液样品3mL,加入等体积乙酸乙酯充分振荡,收集上层有机相.采用旋蒸或者氮吹法去除乙酸乙酯,然后用甲醇重新溶解产物,经0.22μm有机滤膜过滤后,用于HPLC检测对香豆酸及咖啡酸.使用4.6×250mm C18色谱柱,流动相组成为30%乙腈∶30%水,再添加1g/L磷酸,流速1mL/min;紫外检测波长280nm,进样量10μL.
取发酵液样品3mL,加入等体积乙酸乙酯充分振荡后,取少量有机相,利用无水硫酸钠除水后用0.22μm有机滤膜过滤,用于气相检测阿魏酸、松柏醇及丁香酚.升温程序为初始温度60℃,保持1min,20℃/min升到240℃,保持5min,之后 10℃/min升到280℃,然后320℃后运行8min.进样口温度为280℃,检测器温度为320℃.氮气的流速为3mL/min,分流比为10.
2 结果与讨论
2.1 阿魏酸模块构建与优化
由克氏杆菌的酪氨酸氨基裂解酶基因、大肠杆菌的羟化酶基因和拟南芥的甲基转移酶基因构成从酪氨酸到阿魏酸的合成途径,首先对基因表达进行优化.对PcTAL的N端添加His 标签,发现菌株MYT6合成对香豆酸的产量提高了5.47倍(图2(a)).类似地,对AtCOMT 的N端添加His 标签,发现菌株MYT8合成阿魏酸的产量提高了2.47倍(图2(b)).这可能是前缀His标签有利于异源蛋白在大肠杆菌中的表达,提高酶的活性,与文献[15]报道是一致的.
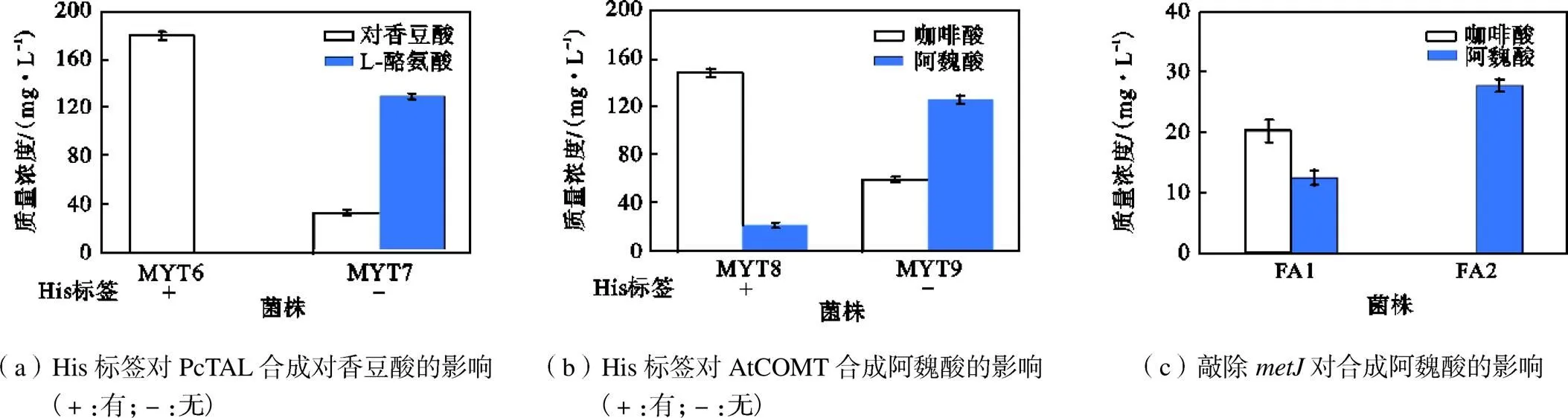
图2 阿魏酸合成模块的构建与优化
用优化的His-PcTAL、His-AtCOMT、HpaBC基因构建pMYT12质粒,导入BL(DE3)中,构建了FA1菌株.虽然合成了阿魏酸,但发现发酵液中存在中间产物咖啡酸的积累(图2(c)).从咖啡酸到阿魏酸的转化为甲基化反应,涉及甲基供体S-腺苷甲硫氨酸(SAM)的参与,我们推测可能为SAM不足导致咖啡酸.基于SAM的合成途径[16],这里敲除全局负调控基因,共表达优化后pMYT12,构建了FA2菌株.发酵结果显示,没有检测到咖啡酸的积累,从头合成27.74mg/L阿魏酸(图2(c)).表明解除阻遏调控,是增强SAM供应的一种策略.
2.2 松柏醇模块构建与优化
松柏醇合成模块涉及到4CL、CCR和脱氢酶3个基因,首先对不同来源的基因进行组合筛选.选择对阿魏酸具有较强特异性的拟南芥At4CL和天蓝色链霉菌Sco4CL以及伯克霍尔德氏菌的Bmfcs[17-19]为候选4CL酶,同样地,拟南芥来源的AtCCR1被证实对阿魏酸酰CoA到松柏醛的反应具有较高催化活 性[20].由于松柏醛到松柏醇为脱氢还原反应,基于序列的同源性[21],选择大肠杆菌对醛基的催化效率较高的醇脱氢酶YahK和YgiB[22-23]、幽门螺杆菌的HpCAD[24]作为候选酶.
为了验证这些酶的功能,在AtCCR1不变的情况下,笔者设计了一套组合实验,构建了9个菌株(图3(a)),以获得松柏醇合成模块的最佳酶组合.表达Sco4CL的菌株仅产生少量的松柏醇,远低于At4CL1和Bmfcs.另一方面,表达YahK和YgiB合成的松柏醇含量在125.73~140.11mg/L之间,远 高于表达HpCAD,而且yahK优于ygiB.因此At4CL1、AtCCR1和yahK是最适合的松柏醇合成 模块.
其次,优化松柏醇合成模块的基因表达模式.以pETDeut-1为骨架载体,对3个基因进行单(双)顺反子表达设计,构建了6种表达载体(图3(b)),并导入大肠杆菌,构建了菌株MYT28-MYT33.发酵结果表明(图3(c)),不同表达模块中松柏醇的产量相差很大.最适宜的表达模式是双顺反子表达At4CL1和AtCCR1,单顺反子表达yahK时,菌株MYT28合 成 145.60mg/L的松柏醇.进一步表明异源基因在 载体上的排列顺序和组合对整个模块功能有较大 影响,这种现象在代谢工程合成紫杉醇前体中也被 报道[10].
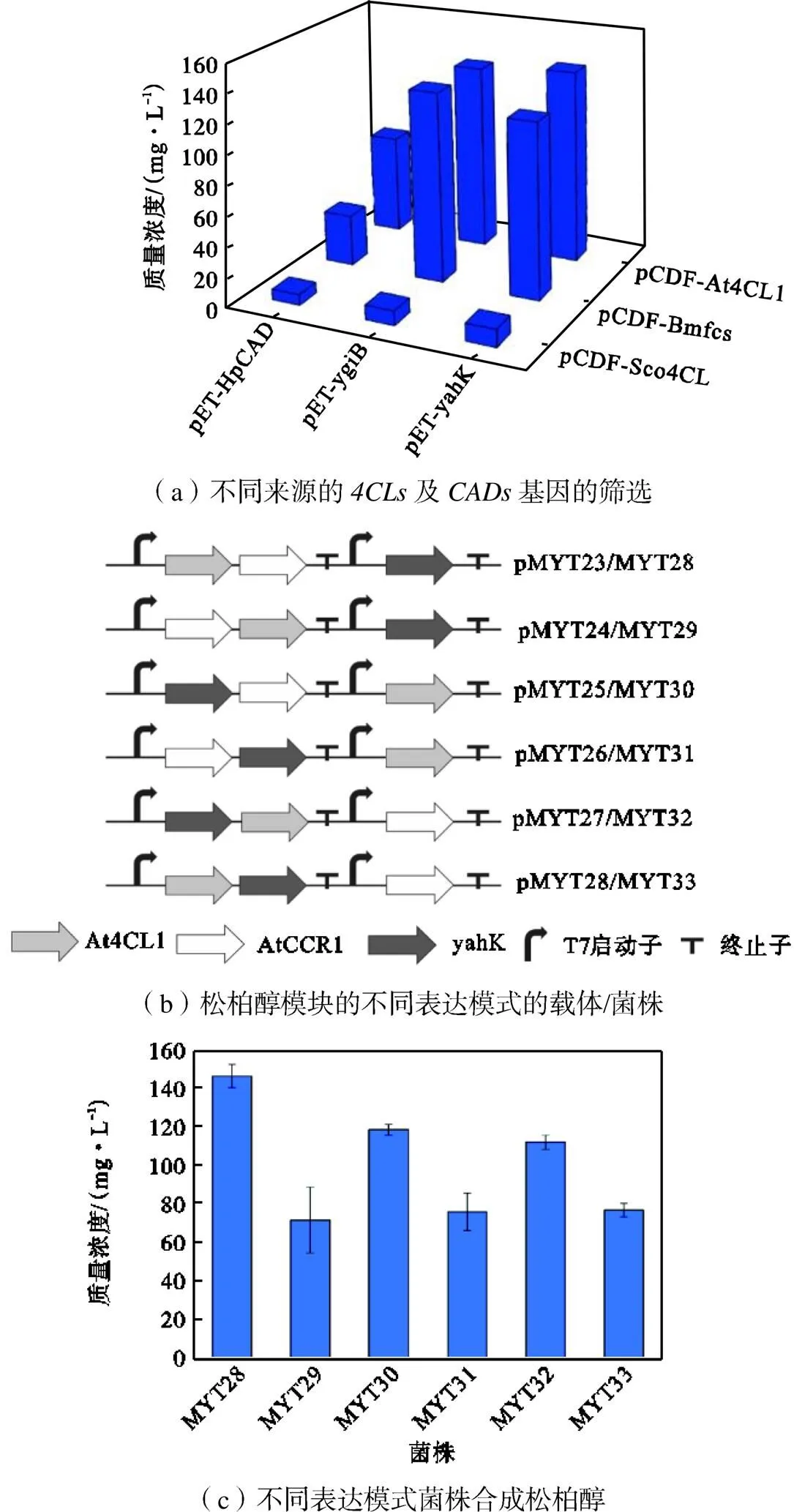
图3 松柏醇合成模块的构建与优化
2.3 丁香酚合成模块的构建与优化
丁香酚合成模块由两个酶组成,松柏醇酰基转移酶和丁香酚合成酶.以文献报道最高活性的PhCFAT[25]氨基酸序列为模板,通过NCBI smart blast(http:// blast.ncbi.nlm.nih.gov/smarblast/smartBlast.cgi)搜索,找到同源性较高的拟南芥的乙醇羟基转移酶(AtAT).LtCAAT1也具有松柏醇酰基转移酶活 性[26],作为候选酶之一.已经有多种植物来源的丁香酚合成酶被鉴定,通过比较Km等酶学动力参数,选取CbEGS1、GdlEGS2、LtAPS1和PaAIS1[27-29]作为丁香酚合成酶候选酶.按单顺反子模式,将3个酰基转移酶基因和4个丁香酚合成酶基因克隆到pRSFDuet-1骨架载体上,构建12个表达载体,并导入BL21(DE3)细胞,获得12个菌株CJY1-CJY12.培养基中添加松柏醇,进行发酵.如图4(a)所示,所有菌株在11.23min出现一个新峰,与丁香酚标准品具有相同的保留时间.经过质谱鉴定,新峰产物为丁香酚.表明所选择的12个基因在大肠杆菌中表达后具有催化功能,能合成丁香酚.以共表达PhCFAT和GdlEGS2的菌株CJY10合成最多的丁香酚,为67.20mg/L.
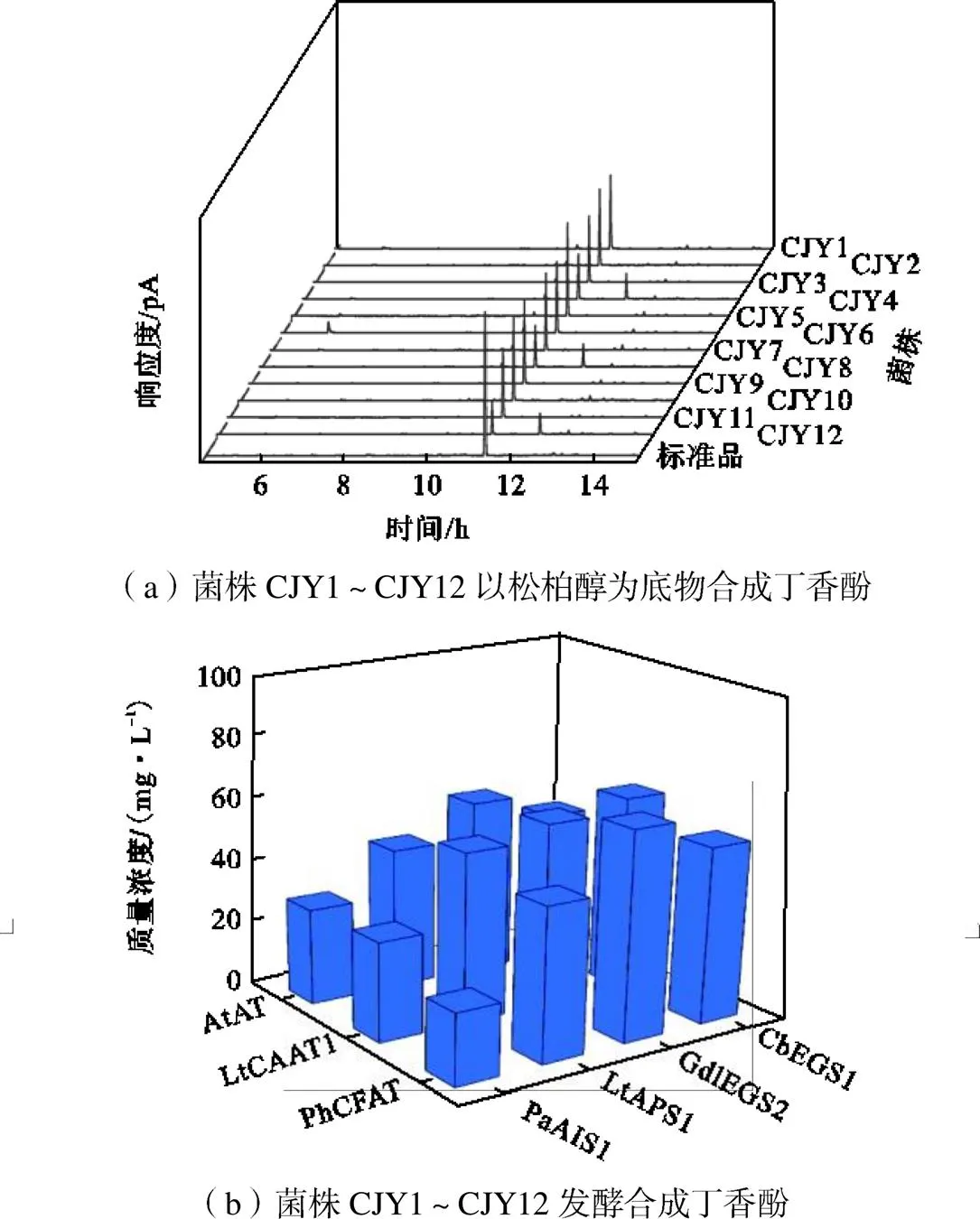
图4 丁香酚合成模块的构建与优化
2.4 葡萄糖发酵合成丁香酚
丁香酚合成前体酪氨酸受到基因的全局负调控[30],为了增强酪氨酸的供给,在MYT1的基础上敲除基因,得到一株底盘菌株MYT2,酪氨酸产量达到138.76mg/L(图5(a)).为了验证中低拷贝数质粒对丁香酚合成模块表达的影响,将阿魏酸合成模块pMYT12、松柏醇合成模块pMYT23、丁香酚模块pCJY13(或pCJY14)导入MYT2中,得到丁香酚合成菌株EG1和EG2.用葡萄糖发酵,结果如图5(b)所示,菌株EG2可以从头合成更多的丁香酚,为43.64mg/L.
不同的发酵参数在不同程度上会影响大肠杆菌合成异源产物的能力[31].为了提高丁香酚的产量,进行递进式优化发酵工艺.首先对IPTG诱导剂工作浓度进行优化,结果如图6(a)所示.IPTG浓度越高,丁香酚产量呈下降趋势,同时生物量减少,表明高浓度的诱导不利于丁香酚的合成和细胞生长.其次对发酵培养基的碳源浓度进行优化,结果如图6(b)所示,5g/L葡萄糖的产量最低,但超过10g/L葡萄糖,丁香酚产量和生物量没有得到提高.最后对酵母提取物浓度进行优化,结果如图6(c)所示,1g/L酵母提取物的产量最高,进一步提高酵母提取物浓度,虽然有利于细胞生长,但丁香酚产量显著下降.为此得到最优的发酵条件为0.1mmol/L IPTG、10g/L葡萄糖和1g/L酵母提取物.控制发酵起始OD600为1,发酵18h,从头合成114.67mg/L丁香酚(图6(d)).
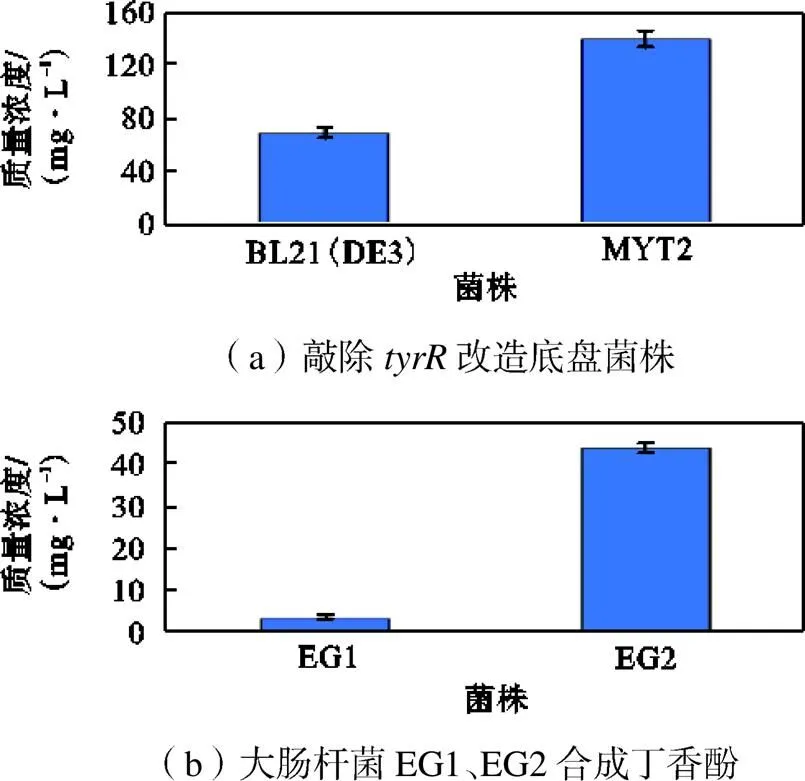
图5 工程大肠杆菌从头合成丁香酚
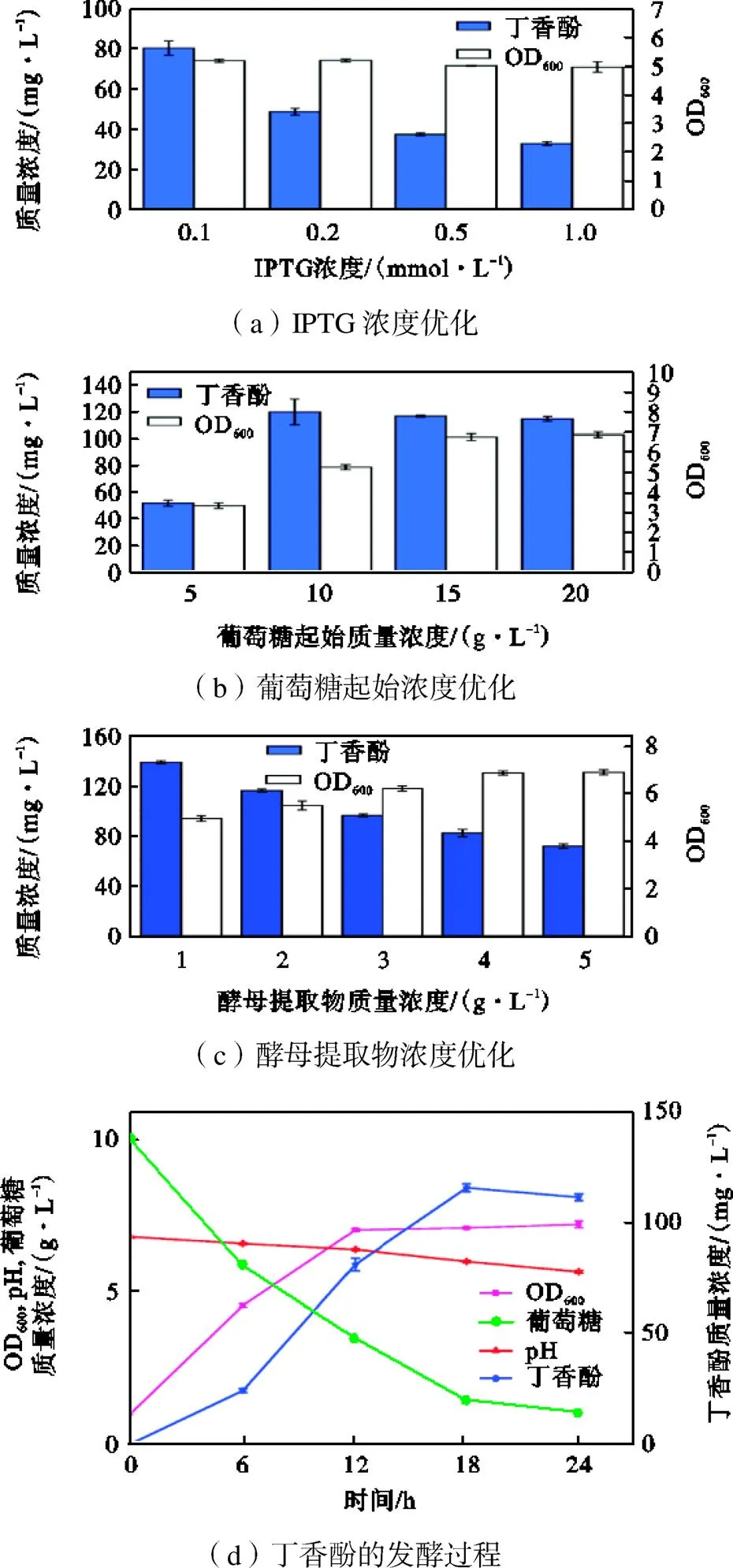
图6 优化发酵参数从头合成丁香酚
3 结 语
在丁香酚的生物合成中,碳骨架来源于酪氨酸和S-腺苷甲硫氨酸,敲除阻芳香氨基酸合成途径的全局遏基因以及腺苷甲硫氨基酸途径中的全局负调控基因,增强了前体酪氨酸合成和甲基供体的供给,减少了中间副产物,提高了产物的代谢流通量,得到丁香酚合成底盘菌株.
采用模块化代谢工程策略,将丁香酚生物合成途径分成3个模块并对每个模块进行不同来源基因筛选和表达优化,构建了工程大肠杆菌,首次实现了丁香酚的从头微生物合成.
农业真菌病害较难防治,其造成的作物损失受到全球的广泛关注,对生物农药的需求日益剧增.本文研发了丁香酚的微生物合成技术,有望在未来具有较大的应用前景.
[1] Lamberth C,Jeanmart S,Luksch T,et al. Current challenges and trends in the discovery of agrochemichals [J]. Science,2013,341(6147):742-746.
[2] Glare T R,Gwynn R L,Moran-Diez M E. Develop-ment of biopesticides and future opportunities[J]. Microbial-Based Biopesticides,2016,1477:211-221.
[3] Pavela R,Benelli G. Essential oils as ecofriendly biopesticides? Challenges and constraints[J]. Trends in Plant Science,2016,21(12):1000-1007.
[4] 孔晓军,刘希望,李剑勇,等. 丁香酚的药理学作用研究进展[J]. 湖北农业科学,2013(3):21-24.
Kong Xiaojun,Liu Xiwang,Li Jianyong,et al. Advance in pharmacological research of eugenol[J]. Hubei Agricultural Science,2013(3):21-24(in Chinese).
[5] Da Silva F F M,Monte F J Q,de Lemos T L G,et al. Eugenol derivatives:Synthesis,characterization,and evaluation of antibacterial and antioxidant activities[J]. Chemistry Central Journal,2018,12(1):34-42.
[6] Cravens A,Payne J,Smolke C D. Synthetic biology strategies for microbial biosynthesis of plant natural products[J]. Nature Communications,2019,10(1):2142-2153.
[7] Mitchell W. Natural products from synthetic biology[J]. Current Opinion Chemical Biology,2011,15(4):505-515.
[8] Nielsen J,Keasling J D. Engineering cellular metabo-lism[J]. Cell,2016,164(6):1185-1197.
[9] Chen Z,Sun X,Li Y,et al. Metabolic engineering offor microbial synthesis of monolignols [J]. Metabolic Engineering,2017,39:102-109.
[10] Ajikumar P K,Xiao W H,Tyo K E,et al. Isoprenoid pathway optimization for Taxol precursor overproduction in[J]. Science,2010,330(6000):70-74.
[11] Qin J,Zhou Y J,Krivoruchko A,et al. Modular pathway rewiring ofenables high-level production of L-ornithine[J]. Nature Commu-nications,2015,6:8224-8235.
[12] Nakagawa A,Matsumura E,Koyanagi T,et al. Total biosynthesis of opiates by stepwise fermentation using engineered[J]. Nature Communica-tions,2016,7:10390-10397.
[13] Fang H,Li D,Kang J,et al. Metabolic engineering offorbiosynthesis of vitamin B12[J]. Nature Communications,2018,9(1):4917-4928.
[14] Srinivasan P,Smolke C D. Engineering a microbial biosynthesis platform forproduction of tropane alkaloids[J]. Nature Communications,2019,10(1):3634-3648.
[15] Kang S Y,Choi O,Lee J,et al. Artificial biosynthesis of phenylpropanoic acids in a tyrosine overproducingstrain[J]. Microbial Cell Factories,2012,11(1):153-161.
[16] Kunjapur A M,Hyun J C,Prather K L. Deregulation of s-adenosylmethionine biosynthesis and regeneration improves methylation in thede novo vanillin biosynthesis pathway[J]. Microbial Cell Factories,2016,15(1):61-77.
[17] Costa M A,Bedgar D L,Moinuddin S G,et al. Characterization in vitro and in vivo of the putative multigene 4-coumarate:CoA ligase network in arabi-dopsis:Syringyl lignin and sinapate/sinapyl alcohol derivative formation[J]. Phytochemistry,2005,66(17):2072-2091.
[18] Salas-Navarrete C,Hernández-Chávez G,Flores N,et al. Increasing pinosylvin production inby reducing the expression level of the gene fabI-encoded enoyl-acyl carrier protein reductase[J]. Electronic Journal of Biotechnology,2018,33:11-16.
[19] Rashamuse K J,Burton S G,Cowan D A. A novel recombinant ethyl ferulate esterase from[J]. J Appl Microbiol,2007,103(5):1610-1620.
[20] Goujon T,Ferret V,Mila I,et al. Down-regulation of the atccr1 gene in:Effects on phenotype,lignins and cell wall degradability[J]. Planta,2003,217(2):218-228.
[21] Youn B,Camacho R,Moinuddin S G,et al. Crystal structures and catalytic mechanism of thecinnamyl alcohol dehydrogenases AtCad5 and AtCad4 [J]. Organic & Biomolecular Chemistry,2006,4(9):1687-1697.
[22] Pick A,Ruhmann B,Schmid J,et al. Novel cad-like enzymes fromK-12 as additional tools in chemical production[J]. Applied Microbiology & Biotechnology,2013,97(13):5815-5824.
[23] Koma D,Yamanaka H,Moriyoshi K,et al. Produc-tion of aromatic compounds by metabolically engineeredwith an expanded shikimate pathway [J]. Applied & Environment Microbiology,2012,78(17):6203-6216.
[24] Mee B. Characterisation of the cinnamyl alcohol dehydrogenase from[J]. Dublin Institute of Technology,2005,272(5):1255-1264.
[25] Dexter R,Qualley A,Kish C M,et al. Characteriza-tion of a petunia acetyltransferase involved in the biosynthesis of the floral volatile isoeugenol[J]. Plant J,2007,49(2):265-275.
[26] Kim S J,Vassao D G,Moinuddin S G,et al. Allyl/propenyl phenol synthases from the creosote bush and engineering production of specialty/commodity chemicals,eugenol/isoeugenol,in[J]. Archives of Biochemistry & Biophysics,2014,541:37-46.
[27] Koeduka T,Louie G V,Orlova I,et al. The multiple phenylpropene synthases in bothandrepresent two distinct protein lineages [J]. The Plant Journal,2008,54(3):362-374.
[28] Araguez I,Osorio S,Hoffmann T,et al. Eugenol production in achenes and receptacles of strawberry fruits is catalyzed by synthases exhibiting distinct kinetics[J]. Plant Physiology,2013,163(2):946-958.
[29] Gupta A K,Schauvinhold I,Pichersky E,et al. Eugenol synthase genes in floral scent variation inspecies[J]. Functional & Integrative Genomics,2014,14(4):779-788.
[30] Yao Y F,Wang C S,Qiao J,et al. Metabolic engineering offor production of salvianic acid A via an artificial biosynthetic pathway[J]. Metabolic Engineering,2013,19(5):79-87.
[31] Zhao Y,Wu B H,Liu Z N,et al. Combinatorial optimization of resveratrol production in engineered[J]. Journal of Agricultural and Food Chemistry,2018,66(51):13444-13453.
Modular Engineering offorProduction of Eugenol
Zhao Guangrong1, 2, Cao Jiayu1, 2,Ma Yating1, 2,Qiu Zetian1, 2,Li Jia1, 2
(1. School of Chemical Engineering and Technology,Tianjin University,Tianjin 300350,China;2. Frontier Science Center for Synthetic Biology and Key Laboratory of System Bioengineering (Ministry of Education),Tianjin 300350,China)
Eugenol is a green biological pesticide that is mainly used to control agricultural diseases,such as tomato gray mold,grape downy mildew,and potato late blight. Since eugenol has been extracted only from cloves,basil,and other plants so far,its production rate is poor and is unfriendly to the environment in terms of waste residue discharge. In this study,in order to synthesize eugenolusing microorganisms,synthetic biology and modular engineering strategies were used for designing,constructing,and engineering. The eugenol heterogeneous synthesis pathway was divided into three modules,namely the upstream module for ferulic acid synthesis,midstream module for coniferyl alcohol synthesis,and downstream module for eugenol synthesis. Combinatorial screening of enzymes from different sources in these modules and expression optimization of each module were performed. By using His-tag and knocking out the global regulatory gene,the upstream module(PcTAL-HpaBC-AtCOMT)was optimized to reduce the accumulation of intermediate caffeic acid and increase the synthesis of ferulic acid. 4CL and alcohol dehydrogenase genes were screened,and their expression patterns were studied.The optimal midstream module was the one having a bicistronic expression of At4CL1-AtCCR1 and monocistronic expression of YahK,which maximized the yield of coniferyl alcohol. Combinatorial screening of three acyltransferase genes and four eugenol synthase genes resulted in the most eugenol synthesis,with monocistronic PhCFAT and GdlEGS2. The eugenol synthesis pathway was expressed in a tyrosine-over-producing chassis. By optimizing the initial concentration of glucose,yeast extract,and inducer,the eugenol yield reached 114.67mg/L within a suitable medium and fermentation conditions. By modularly optimizing the eugenol heterogeneous synthesis pathway and transforming the chassis strains,the firstsynthesis of eugenol by microorganisms was realized herein with a promising application prospect.
eugenol;;metabolic engineering;synthetic biology
10.11784/tdxbz202103018
Q815
A
0493-2137(2022)07-0728-09
2021-03-08;
2021-05-26.
赵广荣(1966— ),男,博士,教授.Email:m_bigm@tju.edu.cn
赵广荣,grzhao@tju.edu.cn.
广东省重点领域研发计划资助项目(2020B0303070002).
the Key-Area Research and Development Program of Guangdong Province(No. 2020B0303070002).
(责任编辑:田 军)
