桑树HSP基因的生物信息学分析
2022-05-03冯胜泽
王 敬 冯胜泽 王 鹏 王 晖*
(1.承德医学院 蚕业研究所/河北省高校特产蚕桑技术应用研发中心,河北承德 067000;2.河北地质职工大学,河北石家庄 050086;3.承德应用技术职业学院,河北承德 067000)
桑(拉丁名:MorusalbaL.)是桑科、桑属落叶乔木或灌木,在我国有着广泛的分布。桑树在自然环境下生长,就不可避免的会受到外界环境例如极端高温、干旱、土壤酸碱度变化等不利因素的胁迫[1]。在漫长的自然选择过程中,桑树进化出了一套能够抵御这种胁迫的能力[2],其中诱导热激蛋白表达就是抵御外界胁迫的一种方式。热激蛋白(heat shock proteins,HSP),广泛存在于真核生物的细胞质中,是当植物受到逆境胁迫时诱导而表达提高的蛋白[3],其功能为防止蛋白质变性[4],并使已变性的蛋白恢复原有的空间构象和生物活性。热激蛋白是一种保守性很高的抗逆性蛋白,因而在生物进化的过程中具有高度保守性[5]。本文拟通过对HSP的氨基酸序列进行理化性质、亲水性/疏水性、信号肽、跨膜结构域、二级结构、亚细胞定位、聚类分析、结合区等进行预测分析,以期为基于HSP基因研究桑属植物的分类提供理论基础。
1 材料与方法
桑树HSP基因(登录号:NW_010362230.1),HSP的氨基酸序列(登录号:XP_024023540.1)。用在线软件对HSP进行分析,应用的分析软件如下表:
表1 软件名称及网址
最后,使用DNAman软件对7个物种进行同源树构建。
2 结果与分析
2.1 对桑树HSP基因开放阅读框的分析
开放阅读框,是指从起始密码子开始的DNA序列中具有能够编码蛋白质潜质的序列,且结束于终止密码子的连续碱基序列。通过使用OFR Finder对HSP基因进行分析(图1),获得了7条开放阅读框,其长度分别为597 bp、426 bp、138 bp、123 bp、111 bp、96 bp和78 bp;起始密码子分别位于1 bp处、440 bp、187 bp、210 bp、485 bp、396 bp和347 bp处;终止密码子分别位于597 bp、15 bp、50 bp、332 bp、595 bp、301 bp和424 bp处;翻译的氨基酸数目分别为198、141、45、40、36、31和25。
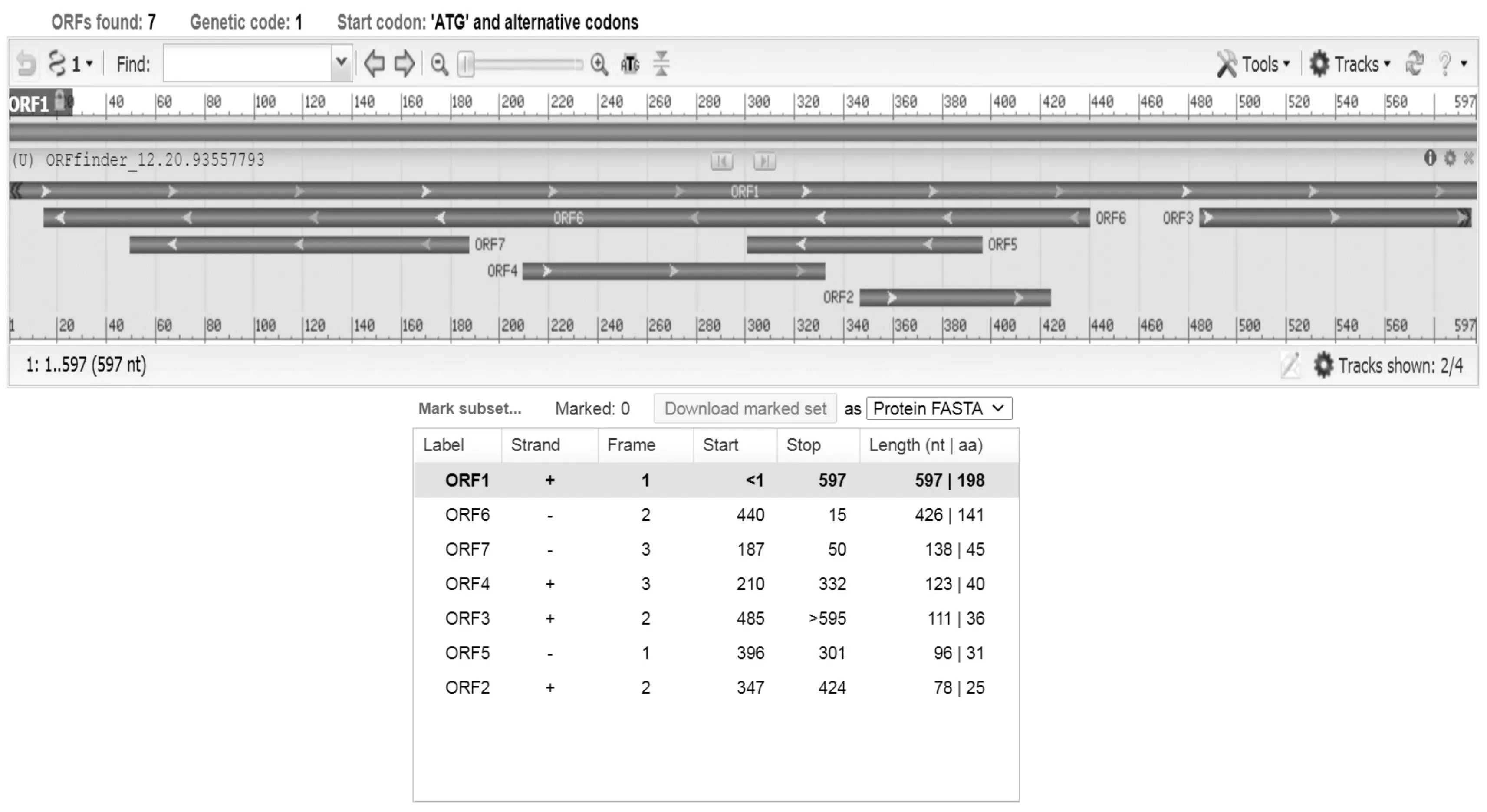
图1 HSP基因的开放阅读框分析
2.2 桑树HSP氨基酸序列的理化性质分析
Prot Param分析发现,桑树叶绿体HSP的氨基酸序列共有198个氨基酸,分子式为C1017H1626N296O295S5,分子量22880.20,等电点为8.88,不稳定指数为35.19,属于稳定蛋白。氨基酸序列N末端是蛋氨酸。氨基酸的组成中,亮氨酸(Leu)占比最高为11.6%,其次为精氨酸(Arg)、天冬氨酸(Asp)、赖氨酸(Lys)、谷氨酸(Glu)等,其中酪氨酸(Tyr)占比最低(图2)。
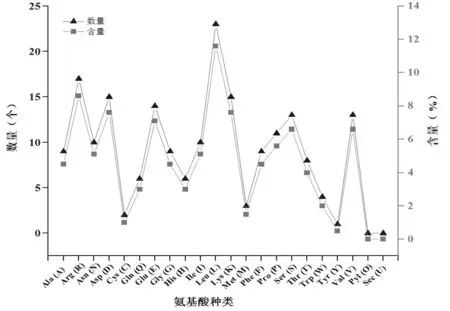
图2 HSP基因各氨基酸的种类及含量分析
2.3 桑树HSP的氨基酸序列亲水性/疏水性的分析
Prot Scale对HSP氨基酸序列亲水性/疏水性进行预测,发现得分为负值的峰数量明显多于正值的峰。因此,桑树HSP氨基酸序列表现出亲水性(图3)。
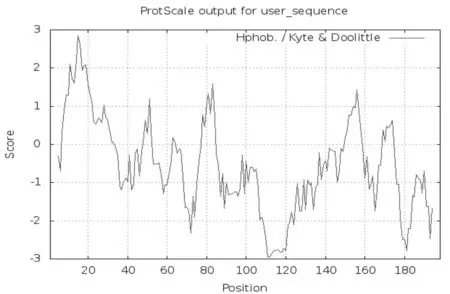
图3 HSP氨基酸序列的亲水性/疏水性分析
2.4 桑树HSP氨基酸序列的信号肽预测
用SignalP-5.0预测桑树HSP氨基酸序列的信号肽,其存在信号肽的可能性为0.9436,接近于1(图4)。因此,桑树HSP氨基酸序列存在信号肽,推断桑树HSP氨基酸序列为分泌蛋白。
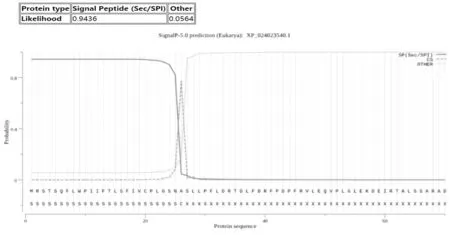
图4 桑树HSP氨基酸序列的信号肽预测
2.5 桑树HSP氨基酸序列跨膜结构域的预测
TMHMM Server.v.2.0对氨基酸序列的跨膜结构域进行预测,发现第1位到第6位氨基酸及第30位到198位氨基酸均不存在跨膜结构域;第7位到第29位氨基酸存在跨膜结构区域(图5)。
2.6 桑树HSP基因氨基酸序列的二级结构
PBIL软件分析结果表明,HSP氨基酸序列的组成分别由28.61%的螺旋、29.29%的延伸链及66.67%的无规则卷曲组成(图6)。
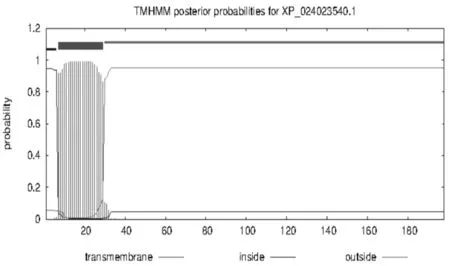
图5 桑树HSP氨基酸序列跨膜结构域的预测
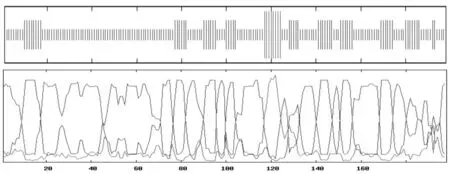
图6 HSP氨基酸序列二级结构的组成
2.7 桑树HSP氨基酸序列的亚细胞定位、GO功能注释、结合区分析
使用PredictProtein软件对HSP氨基酸序列进行亚细胞定位分析发现:HSP氨基酸序列存在于真核生物的细胞质中,这与目前对于HSP的研究结果一致。HSP氨基酸序列共有14个蛋白结合位点,分别位于:25-26、27-44、45-48、67-71、72-72、73-74、126-130、132-132、2-2、116-118、119-122、123-127、128-131、132-134位(图7)。
HSP氨基酸序列的GO功能注释分为三类,分别为细胞组分、分子功能、生物学进程。细胞组分包括内质网腔、内质网;分子功能包括未折叠蛋白结合、蛋白质自缔合;生物学进程包括热反应、蛋白质折叠、对活性氧的反应、蛋白质复合物寡居化、对盐胁迫的反应。(图8)。
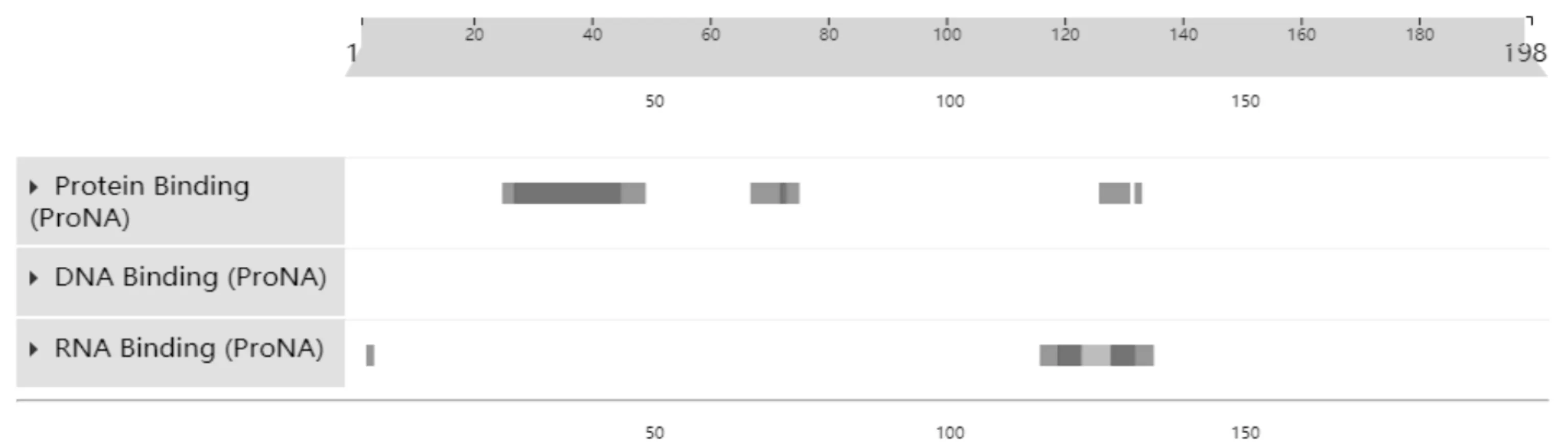
图7 HSP氨基酸序列结合区分析

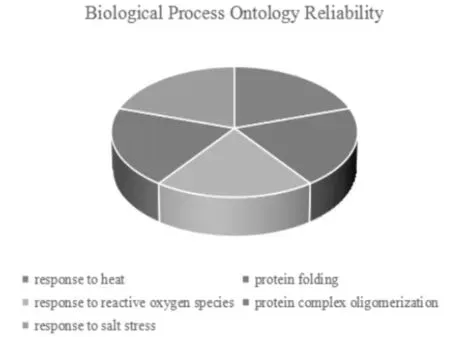
图8 HSP氨基酸序列GO功能注释
2.8 桑树等物种HSP基因序列的亲缘关系分析
用DNAMAN软件对桑树等4个物种的HSP氨基酸序列构建同源树(图9)。图9中桑树与木豆、花生、雷公藤等的HSP氨基酸序列高度相似,这说明HSP氨基酸序列在不同的物种间有着高度的保守性及同源性,可能有相近的生物学功能。

图9 桑树等4个物种HSP氨基酸序列的同源树
3 结论与讨论
桑树的HSP基因是经过自然选择而进化出的、桑树抵御外界生物或者非生物的逆境胁迫而产生的一种防御性功能基因[6],它对桑树的生长和发育有着极其重要的意义。当桑树受到外界高温刺激时[7],某些蛋白质的合成受到抑制或者失活。这时编码HSP基因的表达量增加,使热激蛋白的合成提高[8],从而提升桑树抵抗外界高温的侵害。
本研究对桑树的HSP基因进行生物学分析,发现HSP基因存在于真核生物的细胞质中,这与陈立松[9]的研究一致。HSP基因存在7条开放阅读框,氨基酸序列共有198个氨基酸,有信号肽的存在,为亲水性跨膜蛋白。蛋白的不稳定指数为35.19,属于稳定蛋白。在亲缘分析中发现桑树HSP氨基酸序列与其他物种的HSP序列相似度很高,说明HSP基因是一种具有很高保守性的抗逆性蛋白,在生物进化的过程中具有高度保守性。本研究使用生物信息学对桑树的HSP基因进行分析,而对于桑属植物分类的潜在研究方式还有很多,需要今后作进一步的探索。
