CNTNAP3B基因单核苷酸多态性与199例食管鳞癌患者生存关联分析
2022-04-18陈亚杰赵学科徐瑞华魏梦霞范宗民胡景峰谢叶真马琳琳乔佳欣王立东
陈亚杰,赵学科,徐瑞华,宋 昕,魏梦霞,李 贝,范宗民,胡景峰,王 苒,孙 琳,谢叶真,马琳琳,乔佳欣,王立东
食管鳞癌是全球最常见的六大恶性肿瘤之一(发病率和死亡率均居第六位),中国是食管鳞癌发病率和死亡率最高的地区(发病率居第六位,死亡率居第四位)[1]。研究显示遗传多态性是影响食管癌易感性的重要因素。近年来分子流行病学资料表明汉族人群食管癌遗传易感基因主要包括抑癌基因、癌基因、代谢酶相关基因、DNA 切除修复相关基因和细胞因子基因等,深入了解易感基因的表达变化,有助于明确食管癌的发病机制[2-3]。目前已发现一些单核苷酸多态性(single nucleotide polymorphism,SNP)位点,如rs671对食管鳞癌的早期筛查具有一定的临床意义[4]。SNP与多种恶性肿瘤的遗传易感性已被相关研究证实[5-7]。因此,本研究探讨人类CNTNAP3B基因rs3739620位点SNP不同基因型与食管鳞癌患者的相关性,为食管鳞癌患者的预后生存提供新思路,现报道如下。
1 材料与方法
1.1 样本来源
1.1.1 研究对象的采集共收集来自高发地区医院199位食管鳞癌患者的癌组织。所用样品均为新鲜组织样本,在手术切除后30 min内进行取样,并保存在-80 ℃下。样本采集前未接受放化疗等其他治疗。以半定量的方式对肿瘤细胞进行病理学评分,仅选择恶性细胞>70%的肿瘤样品进行全基因组外显子测序(WES)。所有患者都签署了独立的知情同意书,用于取样和测序分析。本研究已获得了相关机构的审查和批准。
1.1.2 研究对象的随访本研究主要通过电话、家访和村医与患者或患者家属直接联系,或通过新合作医疗数据库、医疗保障局数据库和公民死亡信息登记管理等系统查询方式进行随访,每半年进行一次随访,记录去世时间和主要死因等。本研究随访截止到2021年12月1日,随访成功199例。
1.1.3 组织样本的制备将199例食管鳞癌患者的癌组织分装到准备好的已经标记的冻存管内,样本编号为001~199号。收集到的样品在液氮中快速冷冻并保存,同时保证每份样品有足够的DNA可供提取。
1.2 WES测序SNP分析
1.2.1 DNA样品检测利用琼脂糖凝胶电泳分析DNA样本是否有蛋白、RNA污染及其降解程度。并用Qubit3.0测出DNA样品浓度,选择0.6 μg以上的样品进行后续建库。
1.2.2 建库捕获及上机测序采用Agilent液相芯片捕获系统对人全外显子区的DNA进行富集,Agilent SureSelectHumanAllExonV6试剂盒进行建库及捕获,后在Illumina平台上进行高通量测序。
1.2.3 数据质量控制高通量测序获得的原始图像数据经过转化后形成原始Raw Data测序序列。将原始Raw Data数据进行过滤,并且控制平均碱基位置测序错误率低于0.1%,样本的测序reads达到95%以上的比对率,且碱基覆盖深度达到10×以上时该点检测的SNP比较可信。
1.2.4 SNP检测和功能注释使用bcftools软件识别SNP,并利用ANNOVAR软件对多态性位点用进行功能注释。注释包括dbSNP数据库、千人基因组计划,包含变异的位置、类型,保守性及有害性预测等。
1.3 蛋白质组学分析与鉴定
采用液相色谱—质谱(liquid chromatography-tandem mass spectrometry,LC-MS/MS)技术对199例食管癌的癌组织样本进行分析。
1.4 eQTL分析整合eQTL分析所获得的SNP位点,用HaploView 软件排除冗余,采用Sequenom MassARRAY中通量基因分型平台,对来自高发地区医院的199位食管鳞癌患者进行基因分型检测并且建立线性模型推测CNTNAP3B基因对MRPL18蛋白表达的影响。
1.5 统计学分析
统计学处理采用SPSS 21.0进行分析。高发区食管鳞癌组织的SNP影响蛋白的表达从而导致肿瘤的发生采用双重线性回归检验,采用Kaplan Meier方法进行2 a生存分期。检验标准α=0.05。
2 结果
2.1 CNTNAP3B基因rs3739620位点不同基因型对食管鳞癌生存期的影响
统计学分析显示SNP不同基因型食管鳞癌患者的生存之间有显著差异(P<0.02)。在单个SNP 位点分析时,发现C/T基因型比C/C基因型生存率高,T/T基因型比C/T基因型生存率高,说明rs3739620位点T/T基因型是患者的保护因素。此外rs3739620位点T/T、C/T、和C/C基因型频率分别为4.0%、56.2%和39.6%,见图1。
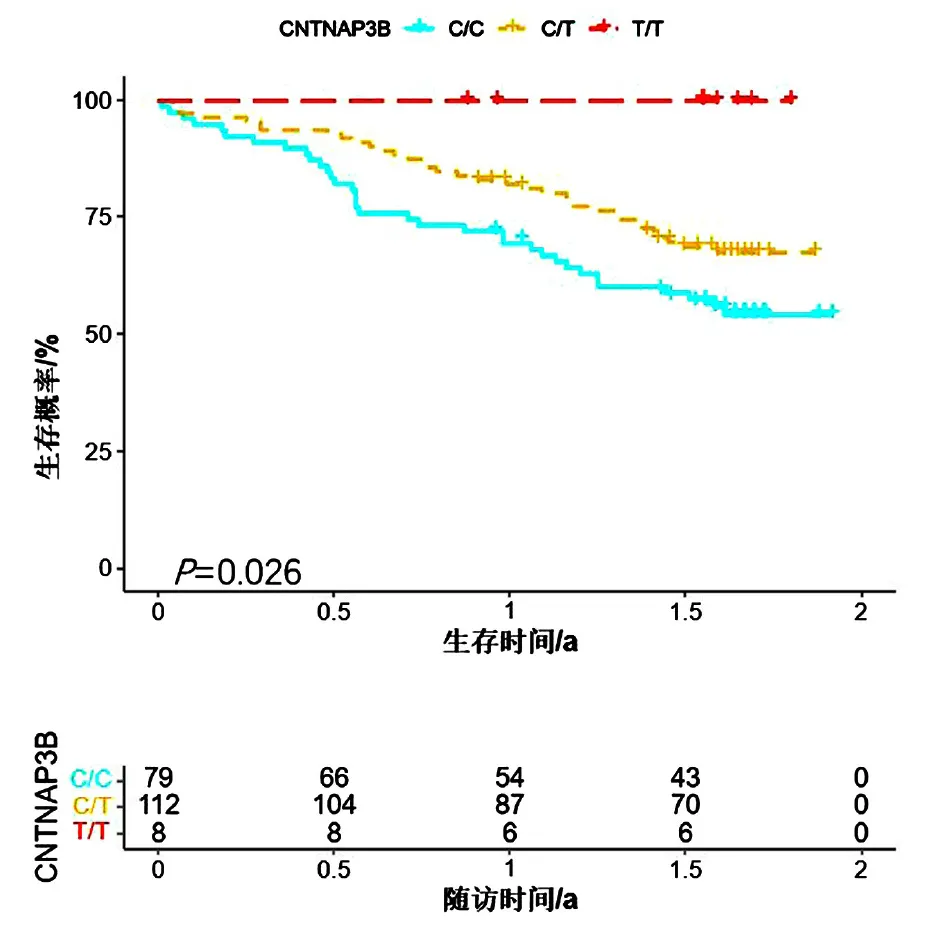
图1 CNTNAP3B基因生存分期
2.2 CNTNAP3B基因rs3739620位点不同基因型对MRPL18蛋白表达水平的影响
为了探讨rs3739620位点不同基因型影响食管鳞癌患者生存的可能机制,本研究对该位点3种不同基因型患者的蛋白质组学数据进行了比较分析,结果显示:3组不同基因型患者间的MRPL18蛋白表达水平存在显著差异(P<0.001),其中C/C基因型组的MRPL18蛋白表达水平最高,C/T 基因型组次之,T/T基因型组最低,见图2。

图2 rs3739620位点不同基因型患者MRPL18蛋白的表达情况
为了矫正其他混杂因素的影响,作者应用表达数量性状位点(expression quantitative trait locus,eQTL)算法进一步分析了rs3739620位点不同基因型与MRPL18蛋白表达水平的关系,结果显示:C/C基因型组、C/T 基因型组、T/T基因型组食管鳞癌患者MRPL18蛋白的表达水平呈线性下降趋势(P<0.001),其在拟合方程中对应的系数(β)为-0.32,即当某患者rs3739620位点上携带的突变基因(T)数量较其他患者每多出1个时,其MRPL18蛋白的表达水平约降低0.32个单位。
3 讨论
食管癌具有明显的地域分布差异,但是在相同环境因素下的人群只有少数罹患食管癌,可见个体基因型在食管癌的发生发展中起重要的作用[8-9]。从基因水平判断患者的病变发展情况以及预后的评估正逐渐成为研究的热点[10]。单核苷酸多态性主要是指DNA序列上变异频率大于>1%的单个核苷酸变异[11],可以认为这些SNP是人群间异质性的来源之一,而且SNP在DNA序列上分布广泛,有研究认为每100~300个碱基就能发现一个SNP[12]。在NCBI网站中查询发现,CNTNAP3B基因即接触相关蛋白家族成员3B(contactin associated protein family member 3B),位于人类第9号染色体,突变点是rs3739620。该基因8号外显子1 316位碱基C突变成了T,439位氨基酸由脯氨酸变成了亮氨酸。
本研究探讨CNTNAP3B基因rs3739620位点SNP不同基因型与食管鳞癌患者生存期之间的相关性。在单个基因位点分析rs3739620位点SNP不同基因型与食管鳞癌患者的生存有显著的差异,有统计学意义。在不同基因分型中,发现T/T基因型的生存率最高,推测rs3739620位点突变对食管鳞癌患者预后生存具有保护作用。提示CNTNAP3B基因单核苷酸多态性与食管鳞癌患者生存有关,可能作为预测食管鳞癌术后患者生存的潜在预后标志物。
CNTNAP3B基因rs3739620位点突变导致MRPL18蛋白表达下调,尤其是T/T基因型蛋白表达量最低。研究发现MRPL18蛋白在肿瘤中高表达[13],推测这个靶蛋白可能是肿瘤中潜在的靶基因,再次说明rs3739620突变可能是影响食管鳞癌患者生存的机制。SNP造成靶蛋白表达量的改变,本研究目前仅阐述了二者之间的相关性,可能是一种潜在的Trans eQTL调控作用,需要后期进一步研究证明。
综上所述,CNTNAP3B基因rs3739620位点T/T基因型生存期最好,并且CNTNAP3B基因影响MRPL18蛋白表达,对食管鳞癌患者预后生存具有一定的临床指导意义。
