基于IBS算法预测亲缘关系准确性研究*
2022-03-31管珊珊张文杰魏以梁李鹰翔赵雯婷
管珊珊 张文杰 魏以梁 李鹰翔 赵雯婷 范 虹 刘 京
(1)陕西师范大学计算机科学学院,西安710119;2)公安部物证鉴定中心,北京100038;3)江苏师范大学,徐州221116;4)安澜智能(深圳)有限公司,深圳510630;5)中国政法大学,证据科学教育部重点实验室,北京100088)
短串联重复序列(short tandem repeat,STR)基因座一直是司法领域中鉴定个体身份和亲缘关系的主要遗传标记,但由于使用的位点数目有限,常将 单 核 苷 酸 多 态 性 (single nucleotide polymorphism,SNP)遗传标记作为STR标记的补充。近年来,测序技术的进步发展带来了更密集的遗传标记集,由于SNP位点分布广泛、突变率低、相比STR 等重复序列标记具有更高的遗传稳定性等特点,使得利用全基因组高密度SNP 分型数据预测亲缘关系成为新的研究热点。法医系谱推断是指通过遗传谱系分析解决涉及司法实践中的身份识别问题,早在2005年,美国科学家Fitzpatrick提出此概念[1]。“金州杀手”案是第一宗使用法医系谱学技术破获的悬案[2],该技术被誉为2018 年度十大科学突破之一,此案告破后,警方利用该技术为200 余例案件提供侦查线索[3-4]。2020 年12 月,国内利用法医系谱推断技术为14 年前的一起命案积案锁定重点家系[5],为案件侦破提供了直接线索。研究表明,高密度SNP 技术结合传统STR 技术将会成为法医DNA服务案件侦查和诉讼的新模式[6]。
个体间的共祖片段(identity-by-descent,ⅠBD)长度算法或者等位基因频率估计的状态一致性(identity-by-state,ⅠBS)共享统计量算法是目前预测亲缘关系的主要方式[7]。前者通过检测个体之间从一个共同祖先继承的相同DNA 片段长度和数量,判断亲缘关系远近。该算法适用于已进行基因型定相的单倍型,需要较大的参考人群数据。并且对法医样本的质量非常敏感,当使用来自低质量DNA 样本的少量SNP 基因型时,很难实现可靠的ⅠBD检测。基于等位基因频率估计ⅠBS共享统计量预测亲缘关系的算法,在假设各标记间独立的情况下,通过估计整个样本中每个SNP 的等位基因频率,计算基因组中共享的等位基因比例确定亲缘关系。该算法虽然只能准确预测1~4 级内的亲缘关系,在5级以上的远亲关系中预测准确率低于ⅠBD方法,但其受位点检出率影响较小。
本文描述的ⅠBS 算法依赖于高密度SNP 数据,通过计算每个SNP标记等位基因频率和ⅠBS的共享等位基因数量估计两两个体之间的共享统计量,并转化为亲缘关系系数得出亲缘关系等级。该算法在项目组开发的亲缘关系预测系统(kinship prediction system version 1.0,KPS v1.0)[8]中实现,可准确预测4级以内的亲缘关系,并且能在几分钟内对数百万对个体进行关系推断[9]。
1 材料与方法
1.1 样本来源
采集中国中部地区5个家庭共253个汉族样本,其中包含4 184对1~7级亲缘关系(图1显示各等级数量分布,包括双胞胎(MZ)、亲子(PO)、全同胞(FS)、2 级(2nd)、3 级(3rd)、4 级(4th)、5级(5th)、6 级(6th)、7 级(7th)亲缘关系),26 325 对无亲缘关系(UN)。所有样本在采集前均签署知情同意书,本研究通过了公安部物证鉴定中心伦理委员会审查(编号:2020-022)。
1.2 DNA提取与检测
所有样本均使用QⅠAamp DNA Midi 试剂盒(QⅠAGEN 公 司, 德 国) 提 取DNA, 使 用NanoDrop 2000c 超微量分光光度计(Thermo Scientific公司,美国)进行DNA定量和纯度检测。使 用 美 国Ⅰllumina Ⅰnfinium Global ScreeningArray(GSA)芯片进行全基因组SNP检测,获得约70万个常染色体SNP 位点分型(安澜智能公司,中国)。
1.3 亲缘关系推断方法
使用亲缘关系预测系统KPS v1.0 进行亲缘关系预测,该系统通过ⅠBS算法估计的亲缘关系系数Φ和零ⅠBD 共享统计量π0推断亲缘关系等级。具体来说,亲缘关系系数Φij表示从个体i、j中随机抽取的两个等位基因来源于同一祖先的概率。

其中NAA,aa为个体i,j基因型都为纯合子的标记数,NAa,Aa为个体i、j基因型都为杂合子的标记数,N(x)Aa是个体x的基因型为杂合子的标记数。零ⅠBD共享统计量π0表示从个体i,j在一个SNP 位点上共享同一祖先零个等位基因的概率。

其中pm为标记m的估计等位基因频率个体。
表1 是对Manichaikul 等[9]文献中亲缘关系系数Φ和零ⅠBD 共享统计量π0的推理标准的扩展。根据系统预测的所有个体间的亲缘关系系数与此表中亲缘关系系数的推理标准范围比对,可进行个体间亲缘关系等级推断。由于亲子与全同胞关系的亲缘关系系数范围一致,可使用零ⅠBD共享统计量作进一步区分。
1.4 位点筛选
使用高密度SNP 标记集进行亲缘关系预测时通常包含一定程度的冗余信息,故本研究分别使用连锁不平衡、最小等位基因频率对标记进行过滤,评估不同位点组合的预测准确率。并且考虑到在真实案例样本中,检材质量不一,可能会导致位点随机丢失,故本研究还进一步通过随机减少位点数模拟真实低质量样本,以检验该算法的适用性。

Fig.1 The quantity distribution of each kinship degree among samples

Table 1 The criteria for inference of kinship
1.4.1 连锁不平衡
等位基因间的关联(连锁不平衡)会增加相邻遗传标记上等位基因共享的程度,从而高估个体间的亲缘关系程度,甚至将无关个体推断为有亲缘关系。考虑到全基因组SNP 数据中存在大量连锁不平衡位点,本文使用PLⅠNKv1.9 软件[10]通过连锁不平衡的筛选标准R2参数对原始数据进行位点过滤,以检验连锁不平衡是否对准确性产生影响。
1.4.2 最小等位基因频率
最小等位基因频率(minor allele frequencies,MAF)较低的位点对亲缘关系的信息贡献小,甚至增加假阳性,本文使用PLⅠNK v1.9 软件根据MAF 值对原始数据进行位点过滤,去除冗余和非信息量标记位点,从而保留最大的信息量,以此评估位点信息量大小是否对预测结果造成影响。
1.4.3 随机筛选位点
由于法医学中常遇到陈旧、微量及降解检材等造成的位点检出率低的情况,为了探究SNP 位点数量的减少对该算法预测效能影响,我们对位点进行随机的梯度下降筛选,将筛选的位点组合进行亲缘关系预测的结果与原始数据结果进行比较,检验不同密度SNP 位点组合对预测准确性的影响,以及位点数量减少到何种程度,准确率会大幅下降。
2 结 果
2.1 关系推断的准确性评估
使用KPS v1.0系统对253份测序数据进行亲缘关系计算,将所有个体间预测的亲缘关系等级与实际调查的亲缘关系进行比较,评估亲缘关系预测准确性。
表2 中展示了253 份样本数据进行亲缘关系预测的准确性,由于亲缘关系系数Φ在(0,0.002 76)范围的个体对亲缘关系无法确定,将不确定关系的样本对定义为7 级以上或未知关系(>7th/UnK)。从表中可以看出,1 级亲缘关系的预测准确率为100%,3级亲缘关系预测准确率为89.8%,随着亲缘关系等级的增加,预测准确率也随之降低,4级开始出现假阴性,5级之后的亲缘关系准确率明显下降。

Table 2 The evaluation of the accuracy of genetic relationship prediction in 253 samples
2.2 亲缘关系等级系数分布
基于调查的真实亲缘关系所估计的亲缘关系系数分布(图2),1~3 级亲缘关系都比较清楚地分开,而4级以后的亲缘关系分布开始出现重叠,并且越远的关系与无关分布有更高程度的重叠。表3中为各亲缘关系等级系数的分布范围。
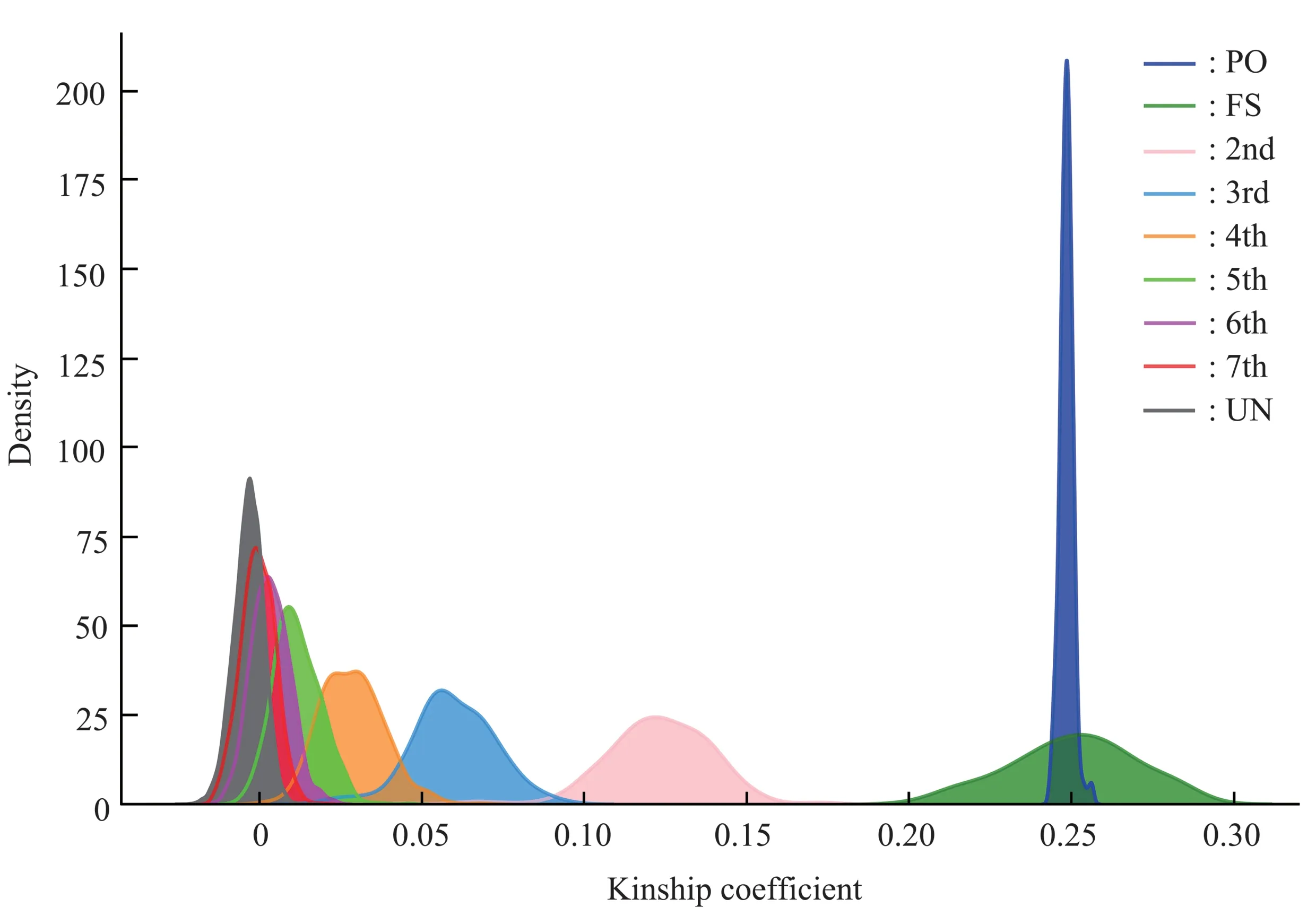
Fig.2 The distribution map of kinship coefficient of each kinship degree

Table 3 The distribution range of kinship coefficient of each kinship degree
2.3 不同SNP标记组合的关系预测
2.3.1 连锁不平衡
为研究连锁不平衡的对于亲缘关系预测的影响,本文根据连锁不平衡的度量参数R2对位点进行过滤,使得保留的所有位点间的相关性都低于给定的R2值。根据集合[0.1,0.125,0.15,0.175,0.2,0.225,0.25,0.275,0.3]中的值筛选位点,表4 为不同R2值筛选的位点组合预测准确性结果。图3中显示了不同位点组合在各亲缘关系等级的预测准确性分布。与原始数据预测准确性比较发现,R2值越大,该算法的预测准确性越高,尤其对于4级以上的亲缘关系更为明显,例如5级的绝对准确率由40.9%升至56.8%,并且当R2≥0.125 时,消除了4级上唯一的一对假阴性结果。虽然筛选的位点在一定程度上提高了预测准确率,降低了总体的假阴性,但同时也增加了假阳性,并且在4级关系上出现假阳性结果。

Table 4 The predictive accuracy of locus combinations screened by different R2-values
2.3.2 最小等位基因频率
本文根据MAF 值[0.000 1,0.01,0.05,0.1,0.2]对原始数据进行位点过滤,在筛选的结果数据集中,SNP标记数范围在222 770~514 962之间。使用过滤后的SNP 位点组合进行亲缘关系预测(表5),SNP 位点数量随MAF 参数值增大而减少,预测准确率也随之降低。本文使用F检验分别将5组数据的准确性与原始数据的准确率进行计算,均得出F值在0.05 的水平上无显著性差异(F>F0.05)。因此可得,虽然不同的SNP 位点组合对预测的结果会产生影响,但这种影响不显著。

Fig.3 The predictive accuracy of locus combinations screened by different R2 values

Table 5 The predictive accuracy of locus combinations screened by different MAF-values
2.3.3 随机筛选位点
案件现场的生物检材受时间和环境等因素影响,DNA 会发生降解,从而降低样本检出率。因此本文通过随机筛选不同数量的位点组合,模拟低质量样本的预测结果。从253 份样本数据的699 537个SNP位点中,随机筛选40万、5万、1万和5 000 各10 组数据,使用ⅠBS 算法预测亲缘关系,预测准确性以平均值和标准差反映。表6结果显示,准确性随位点数量的减少而轻微降低,对3级内的亲缘关系准确性影响很小。但需要注意的是,当位点减少到5 万个SNP 时,4 级亲缘关系预测开始出现假阳性,位点数量下降至1万时,少量无关样本被预测为3级。

Table 6 The predictive accuracy of random screening of different number of locus combinations
3 讨 论
在法医遗传学领域,利用密集SNP 标记数据预测亲缘关系的应用研究受到越来越多的关注,但目前缺乏针对中国人群的系统研究,包括从大规模SNP基因型数据集中筛选适合中国人群亲缘关系预测的位点组合,建立预测算法并对算法相关参数进行研究分析。本文中描述的ⅠBS算法基于全基因组SNP数据进行关系推理,其框架核心是将一对个体之间的遗传距离作为其等位基因频率和亲属关系系数的函数进行建模,从而预测亲缘关系等级。该算法能够快速准确预测4级以内的亲缘关系,平均准确率可达99%以上。与ⅠBD 算法相比,此算法不需要特殊的计算资源,能在几分钟内对数百万对个体进行关系推断[9]。
本研究采用高密度SNP芯片对253份汉族样本进行检测,采用项目组前期开发的KPS v1.0 系统进行亲缘关系预测,该系统将ⅠBS算法的整体分析流程进行集成,实现了程序自动化。预测结果(表2)表明,ⅠBS在4级以内的准确率极高,在1级误差内,4级预测准确率高达98.1%。
基于253份样本真实亲缘关系的亲缘关系系数分布(图2)显示,1~3 级关系能明显分离开来,而4 级以上的亲缘关系系数会出现重叠,最远的7级关系与无关关系重叠最大。该结果与预期一致,由于亲缘关系越远的个体间共享的等位基因数量相对较少使得亲缘关系系数降低,且从表3中可以看出,4级以上的亲缘关系系数的均值开始与对应的推理标准值出现明显偏差,因此该方法对于此类关系难以准确区分。
本研究还进一步探讨了连锁不平衡、最小等位基因频率以及位点数量对该算法的影响,以筛选适合中国人群的系谱分析位点组合。首先考虑到密集SNP遗传标记数据中存在大量连锁位点、冗余信息等现象,可能对预测结果产生影响,研究中通过连锁不平衡度量参数R2值对原始数据进行位点筛选,与原始数据预测准确性比较发现,随着R2值越大,该算法的预测准确性越高,尤其对于4级以上的亲缘关系更为明显(表4和图3)。虽然筛选的位点在一定程度上提高了预测准确率,降低了总体的假阴性,但在4级关系上出现假阳性结果。因此,使用此参数时,预测结果中的假阳性和假阴性率需要均衡。其次,MAF 使用近似0 值过滤了不提供信息的位点,预测结果(表5)与原始结果一致,并减少了计算时间(本文中未体现)。其他参数值的预测结果(表5)显示,预测准确性随MAF 值的增加而轻微降低,该结果很有可能由于位点数减少的影响。虽然最小等位基因频率对预测的结果会产生影响,但这种影响不显著。由此可知,该算法对此参数并不敏感。最后,在法医工作中,标记密度不一定能得到保证,比如脱落细胞、腐败组织等微量降解的案件检材。本研究进一步通过随机筛选位点数量模拟低质量DNA 样本的少量SNP 位点进行亲缘关系预测的准确性。结果表明(表6),预测准确性随位点数量的减少而降低,但影响较小。并且所有结果显示,位点数量对近亲缘关系影响更小,比如亲子、全同胞以及2 级关系。但值得注意的是,使用5 000 个SNP 位点进行计算时,3 级关系的预测准确性能达到92.7%,误差在1 级内,与Kling 等[11]研究结果(至少需要5.6 万个SNP 来确定一代堂表兄妹(3级关系))相比,该算法预测效能很好,在较低密度SNP 中也能以较高准确率预测4级以内关系。
4 结 论
本文探索研究了基于高密度SNP数据利用ⅠBS算法进行亲缘关系预测的可行性,研究结果表明,该算法在4级以内亲缘关系的预测效能很好,并且此算法受SNP 位点数量减少的影响较小,对于陈旧降解等低质量检材,也能保持较高的准确性。因此该方法可辅助物证鉴定工作,为刑事犯罪、灾难受害者身份识别(disaster victims identification,DVⅠ)、冷案积案等疑难案件的侦破提供科技支撑。
