空间转录组技术研究进展*
2022-03-31张小丹胡苗苗吴忠琴
危 莹 张小丹 胡苗苗 吴忠琴 程 酩 郭 妍*
(上海交通大学生物医学工程学院,上海200240)
细胞作为机体的基本单元,在特定的空间位置与微环境协同,发挥其特有的生物学功能。生物学研究的重要目标是理解正常和病理状态下细胞的特点和功能。近十几年转录组技术飞速发展,已可从全基因组尺度对基因表达进行定量测量,极大地推进了研究人员对细胞功能的理解。转录组技术的发展主要经历了三个重要的阶段。第一个阶段是对大量 混 合 细 胞 进 行 转 录 组 测 序 (bulk transcriptome)[1],该方法获得的是大量细胞的基因平均表达水平,虽然推进了对细胞群体的特性认识,但无法获知群体内特定细胞的基因表达情况。第二个阶段是对单细胞进行转录组测序(single cell transcriptome),该方法将对细胞基因表达的认识推进到单细胞的层面,目前已在发现新细胞类型、揭示细胞异质性等方面展现出优势。但是目前的单细胞转录组测序方法仍存在一定局限性,由于单细胞悬液的获得需要对组织进行酶解,悬液中的单细胞失去了组织空间位置信息,而且已有研究表明,有些单细胞在酶解过程中或离开特定微环境后,基因表达谱会发生变化[2]。除此以外,某些特殊形态的细胞,如大脑中结构复杂的神经元和神经胶质细胞,难以通过组织酶解获取。近年来,转录组研究进入空间转录组(spatial transcriptome)阶段,该技术可以同时获得细胞的空间位置信息和基因表达数据,进一步推进了对组织原位细胞真实基因表达的研究,为组织细胞功能、微环境互作、发育过程谱系追踪、疾病病理学等多个领域提供了重要的研究手段。多种新的空间转录组技术得到快速发展,成为生物技术研究领域的前沿热点,本文就近年来空间转录组技术的发展和应用进行综述。
1 空间转录组技术的发展
1.1 基于原位杂交的空间转录组技术
1.1.1 单分子荧光原位杂交(smFISH)
空间转录组技术最早可追溯到单分子荧光原位杂 交 (single molecule fluorescencein situhybridization,smFⅠSH)技术[3]。原位杂交技术于1960 年代产生,Gall 等[4]将细胞经过碱处理使得DNA变性,而后与放射性同位素标记的RNA探针一同孵育,利用放射自显影技术检测了青蛙卵母细胞中高度扩增的核糖体DNA。随后,Bauman等[5]于1980 年将3'端带有荧光基团标记的RNA 用作特定DNA 序列的探针,首次实现了荧光原位杂交(fluorescencein situhybridization,FⅠSH)技术的应用。FⅠSH 对识别目标DNA 或RNA 具有高分辨率、高灵敏度和高特异性等特点,可直接应用于中期染色体和间期细胞核的目标序列检测[6-7]。除此以外,FⅠSH 还可以在单细胞水平上根据可视化的杂交信号区分不同的转录本[8]。1998年,Femino等[9]将数字成像显微镜应用于FⅠSH,并且合成了带有5个荧光基团的寡核苷酸探针,校准了单个探针发出的光,使得检测单个RNA分子成为可能。smFⅠSH一般通过多个标记有荧光团的20 bp 短寡核苷酸探针来进行(通常每个探针都标记有一个荧光团),这些荧光探针在组织原位与特定的mRNA 进行特异性杂交(图1a),以此反映细胞内转录本丰度并同时进行空间定位[10]。
1.1.2 组合标记
由于smFⅠSH 能够同时使用的荧光团种类有限,一次识别的转录本数量很少,通常为3或4个,因此smFⅠSH 还不能实现基因组范围的大通量分析[4]。2002年,Levsky等[11]发现可以使用组合标记来增加可识别的转录本数量,并且可以减少单色假阳性信号的出现。实验中,通过将探针用不同条形码(barcode)标记来进行分组,分别与4个荧光基团相结合,在显微镜下识别荧光团的颜色。之后通过检测算法进行图像处理,根据所识别的不同颜色下的像素点的强度,分析出该位点是哪些颜色的结合,以此分析出不同的转录本。当使用的荧光基团的种类为n时,可鉴定的转录本的数量为2n-1个。Jakt 等[12]将组合标记方法应用到体外分化的小鼠胚胎干细胞中,结果表明组合标记可定量单个细胞的多个转录本,还可以追踪细胞分化过程中细胞身份的变化。虽然组合标记方法增加了转录本的检测数量,但是对于基因组范围的分析还是远远不够,而且由于受到组织自发荧光的影响,组合标记的方法应用于组织切片还有一定的困难。
2012年,Lubeck等[13]通过结合超分辨显微镜(super-resolution microscopy,SRM)和组合标记,识别单个mRNA 上不同荧光寡核苷酸探针的空间排序,大幅提高了检测的转录本数量。SRM 的分辨率高达10~20 nm,20 个寡核苷酸探针长度约为7 nm,转录本长度约为100 nm,在SRM 下可以看到不同荧光寡核苷酸探针的空间排序,因此可解析出某个感兴趣的mRNA 分子的序列。在SRM 的帮助下,可以根据不同颜色荧光团的排列组合来区分转录本,例如某红、黄、蓝顺序的转录本与蓝、黄、红顺序的转录本是不同的转录本(图1b),因此在可使用的荧光团数量有限的情况下,可以通过不同的荧光团顺序来鉴定多个转录本。
1.1.3 顺序杂交
虽然组合标记增加了可识别的转录本数量,但是还无法检测全基因组的转录本。2014 年,Lubeck等[14]开发了顺序杂交技术。由于固定的细胞中转录本的位置不会发生改变,即使通过多轮杂交,相同的荧光点还是可以保留在原位,所以能通过顺序杂交进行单细胞原位的RNA 分析。该技术在每一轮杂交中,使用一组标记了单一类型荧光团的探针,结合转录本后进行成像。之后用DNA 酶处理,清除杂交链上结合的探针,再将转录本与另一组不同荧光染料标记的、但序列相同的探针杂交(图1c)。理论上,这种方法可以检测Fn个不同的转录本(F=荧光团数量,n=杂交回合)[15]。因此,原则上,4 种荧光染料和8 个杂交回合可以检测一个细胞中的所有转录本。这种方法的好处在于即使只有2种荧光染料,也可以通过多次杂交使得检测规模迅速扩大。但是,如果想要将这种方法的应用范围扩大到整个转录组,价格较昂贵。除此以外,虽然顺序杂交可检测的转录本数量随杂交回合的增加而呈指数增长,但同时检测误差也呈指数增长。
1.1.4 MERFISH
为了克服检测误差,2015年,Chen等[16]开发了一种名为多重错误鲁棒荧光原位杂交(multiplexed error-robust FⅠSH,MERFⅠSH) 的 方法。该方法利用二进制条形码,在检测时会将单个检测错误进行自动校正,只有当检测错误为多个时,才会识别为另一个转录本。除此以外,该方法还结合了组合标记、顺序杂交和成像等方法,可检测单个细胞中数千种RNA 的拷贝数和空间定位。Moffitt 等[17]通过对MERFⅠSH 进行一系列改进,包括使用化学裂解代替光漂白来去除连续两轮smFⅠSH 成像之间的荧光信号,增加成像视野并使用多色成像。通过这些改进,在一次18 h的测量中就检测了多达40 000个细胞[18]。除此以外,Wang等[19]开发了一种结合MERFⅠSH 和扩展显微镜(expansion microscopy)的方法,扩展显微镜可以有效扩大相邻分子之间的距离,显著增加了可测量的RNA 的总密度,使得检测效率提高近1 倍,单日可测量数以万计的细胞,可测到的转录本密度是以前的10倍以上,并且可与免疫荧光技术相结合。这些改进大大扩展了MERFⅠSH可解决的生物学问题的范畴。

Fig.1 The principle of spatial transcriptome technologies based on in situ hybridization图1 基于原位杂交的空间转录组技术原理
总而言之,smFⅠSH 及其衍生方法可应用于组织切片和体外培养细胞的基因表达测量。该类技术已成功应用于细胞间异质性的研究[20]和疟原虫感染的红细胞基因表达的研究[21],还应用于一些模式动物如果蝇、秀丽隐杆线虫和斑马鱼等的相关研究中[22]。但是该方法在组织中的应用效果不如在培养细胞中的应用效果好,这是由于组织中的应用受到组织自发荧光的影响。而且随着复用探针次数的增加,组织背景的强度也增加,同时背景强度也随着组织复杂程度的增加而增加。Moffitt等[23]开发了一种降低组织背景的smFⅠSH 方法,smFⅠSH实验操作过程中背景的主要来源是探针与非RNA的细胞成分(例如蛋白质)非特异性结合。该方法的关键在于通过将RNA 分子锚定在不可膨胀的聚丙烯酰胺基质上,再去除不需要的非RNA 成分(蛋白质和脂质等)。研究人员在成年小鼠下丘脑的冰冻切片上使用MERFⅠSH方法对130种RNA进行检测,所得到的成像结果证明,该方法大大降低了成像时的组织背景干扰信号,能够清晰地观察到代表单个RNA分子的荧光点。
1.2 基于高通量测序 (high-throughput sequencing)的空间转录组技术
1.2.1 LCM-RNA-seq技术
1996 年,Emmert 等[24]开发了激光捕获显微切割技术(laser capture microdissection,LCM)。该技术可以在保留空间位置信息的前提下,可视化地从切片中快速且准确地捕获目标细胞[25],现已被广泛用于生物学研究中。该技术原理是将组织切片贴在特殊薄膜覆盖的玻璃载玻片上,组织切片经过染色后,在显微镜下可视化地进行特定细胞的选择、激光切割和分选,收集组织中特定的细胞样品。分选获得的细胞可以进行特定基因表达的分析,结合基因芯片或测序技术(图2a),对分选细胞的基因表达谱进行解析[26]。该技术分选的细胞可来源于活细胞培养物、新鲜冷冻组织和福尔马林固定的石蜡包埋组织等。
目前基于激光显微切割的空间转录组技术,已应用于非常少量的细胞,甚至单细胞的分选。2016年,Peng 等[27]通过整合与优化LCM 和单细胞转录组测序(single cell RNA-seq)技术,构建了一种能解析少量细胞转录组信息,同时保留细胞原有位置信息的方法。通过LCM 分区捕获,可以得到小鼠胚胎E7.0 时期的转录组信息,以及各个区域的标志基因,将这些标志基因作为“邮政编码(zipcode)”,用于推断空间坐标,从而将未知来源的胚胎细胞“映射”到体内胚胎的特定位置。
最近,Moor等[28]利用基于激光显微切割的空间转录组技术获得组织空间区域特异性的标志基因,通过标志基因的表达将单细胞转录组数据定位到组织空间,获得单细胞分辨率的空间转录组信息。他们使用激光显微切割技术将小鼠小肠绒毛底部到顶部之间5个等间隔的区域进行切割,分别捕获各个区域的上皮细胞并进行转录组测序。他们通过差异表达分析获得每个区域的肠上皮细胞标志性基因,建立不同空间的基因坐标,并利用这些坐标将单细胞“映射”到小肠绒毛的对应区域。该方法也在其他器官的空间单细胞转录组研究中得到成功应用,如肝小叶组织[29]。
目前基于激光显微切割的空间转录组技术已在肿瘤生物学、发育生物学以及各类疾病生物学研究领域发挥积极的作用。通过激光显微切割获得细胞的具体空间位置,根据研究目的将分选的细胞构建不同的测序文库,通过一次高通量转录组测序,就能获得大量已知和未知基因的表达信息,甚至可以得到可变剪切的基因信息。随着测序成本的不断降低,该方法用于研究空间转录组具有很高的性价比。
1.2.2 HDST和Slide-Seq
2019 年Vickovic 等[30]提出了高分辨空间转录组(high-definition spatial transcriptomics,HDST)测序方法,通过在载玻片表面雕刻2.05 μm的大量小孔,将直径为2 μm的硅胶磁珠分配到这些孔里,通过在磁珠表面连接带有特定barcode 序列以及多种唯一分子标识符(unique molecular identifiers,UMⅠs)和poly-dT的寡核苷酸链,来捕获对应位置细胞的mRNA 进行反转录,并进行文库构建和转录组测序。同一时期,Rodriques等[31]发明了类似的Slide-Seq 方法,该方法用带已知barcode 修饰的直径为10 μm磁珠覆盖的玻片(磁珠同时连有不同的UMⅠs 以 及poly-dT),捕 获 带poly(A)尾 的mRNA,进行cDNA 合成后建库测序(图2b)。这两种方法通过结合测序数据和图像处理,都可以将基因表达情况可视化地比对到原始的组织切片中,得到非常直观的结果,通量大且分辨率高。但其存在一定比例的磁珠会同时比对到两种细胞类型的情况,且磁珠的捕获效率仍然有待提高。

Fig.2 The principles of space transcriptome technologies based on high-throughput sequencing图2 基于高通量转录组测序的空间转录组技术原理
1.2.3 10×Genomics Visium空间转录组测序技术
2019 年,10× Genomics 公司推出了Visium 空间转录组技术,结合显微成像技术和RNA 测序技术,将染色后的组织切片与不同空间位置细胞的mRNA 表达数据相结合。利用Visium 技术,可以从整个组织切片获得高通量的转录组数据,研究人员可以通过此技术研究正常和疾病组织的细胞组成及其与基因表达的关系,可用于发现新的组织生物标志物。基于此技术,研究人员还开发了整合可视化基因表达和形态学数据的软件,可以在一个工作日之内完成从对样品切片到建好测序文库。
Visium技术的原理是利用载有poly(A)和空间barcode 标记的载玻片,原位捕获对应细胞的mRNA 并进行反转录,通过高通量测序结合原位的barcode 信息,绘制复杂组织样品的空间基因表达图谱。该技术所使用的一张玻片可容纳4片组织切片,将组织切片贴在具有5 000个空间捕获区域的载玻片上,每个空间捕获区域中包含数百万个含有poly-dT 的寡核苷酸链,这些寡核苷酸链可以捕获mRNA,寡核苷酸链连接有唯一的分子标识符UMⅠ(计数mRNA 分子)和barcode(每个barcode对应特定的捕获区域)。每个空间区域的直径为55 μm,可以检测到1~10个细胞[32-33]。Visium技术的操作流程是首先将样品进行冰冻切片,贴在载玻片上的捕获区域。利用标准的固定和染色技术对切片进行染色,然后对切片进行透化,释放mRNA,让其与空间区域中带有poly-dT 序列的引物进行结合,然后合成cDNA。之后,将每个空间区域反转形成的cDNA合并回收,进行最终文库的构建以及测序,通过数据分析软件即可获得细胞在组织原始的空间位置所发生的基因转录情况[34]。
2019 年,Maniatis 等[35]使 用10× Genomics Visium 空间转录组技术,研究肌萎缩侧索硬化(amyotrophic lateral sclerosis,ALS)模型小鼠以及ALS 患者死后脊髓组织的基因表达。ALS 导致的瘫痪是由于运动神经元退化造成骨骼肌神经支配失调,但是对完整的脊髓组织中分子发生过程的时空顺序仍然知之甚少。该团队通过10× Genomics Visium 空间转录组技术表征了鞘脂信号在多种细胞类型、脊髓区域和疾病阶段的动力学,为之后药物的设计及治疗策略提供了方向。
空间转录组技术此前已成功应用于小鼠大脑复杂的结构与功能关系研究。大脑的空间结构与其功能是紧密相关的,研究大脑的空间结构有助于更好地理解神经发育、神经精神病学和神经退行性疾病。 2020 年, Maynard 等[36]首 次 使 用10×Genomics Visium 技术获得了人类背外侧前额叶皮层(dorsolateral prefrontal cortex,DLPFC)的空间基因表达图谱。根据组织学和细胞结构,DLPFC可以分为6层(L1~L6),Maynard等通过Visium技术构建了DLPFC 空间基因表达图谱,发现了很多以前未得到充分认识的、但可能具有更高保真度的空间分层特有标记基因,包括HPCAL1(L2)、KRT17(L6)和TRABD2A(L5),这些基因可用于人类大脑单细胞RNA测序的分层注释。
10× Genomics Visium 空间转录组测序可以对测序数据进行可视化分析,与组织切片形貌进行结合,深入探究细胞在特定空间位置上的生物学功能。下一步如果将免疫荧光标记和10× Genomics Visium 技术结合起来,可以把蛋白质检测和整个转录组分析相结合,在空间范围下探索转录组和蛋白质的关系。
1.3 基于原位测序的空间转录组技术
1.3.1 原位测序(ⅠSS)
2013 年,Ke 等[37]开发了原位测序(in situsequencing,ⅠSS)方法,该方法在固定的组织或细胞中先将mRNA 在原位逆转录成cDNA,然后用RNA 酶H 使cDNA 上的RNA 链降解,再通过锁式探针(padlock probe)与cDNA 杂交。由于探针末端之间有缺口,可以通过DNA聚合和DNA连接使锁式探针形成一个完整的环,称为“DNA球”,通过滚环扩增(rolling-circle amplification,RCA)形成滚环产物(rolling-circle product,RCP)(图3a)。之后,在显微镜下观察荧光团偶联的寡核苷酸杂交,这是一个循环。在进行下一个循环之前,要将之前的探针冲洗干净,再重复连接、成像、冲洗的步骤来进行下一个寡核苷酸的测序,直至几个循环完成(图3b)。通过图像分析获得的碱基顺序来实现原位测序(图3c)。组织中可分辨的基因数量与荧光团的数量以及测序周期数有关,F个荧光团和n个测序周期可解码Fn个不同的序列条码,如4 个荧光团和4个测序周期(得到4个核苷酸长度的序列),则可以解码44=256 个不同的序列条码。除此以外,ⅠSS首次实现了对小RNA片段的组织原位测序。
利用该技术,Ke等[37]对人类乳腺癌组织切片中的突变位点进行了原位测序,并揭示了其基因表达特征。2014 年,Pacureanu 等[38]开发了一种基于图像的方法,每个文库都用荧光染料标记,该染料为每个RCP 生成特定的颜色(对应于匹配的碱基),并使用染料专用滤光片通过荧光显微镜获取不同焦平面的图像,可对细胞和组织中的大量RNA片段进行测序。他们对3个HER2阳性新鲜冷冻乳腺癌组织切片进行了原位RNA 表达和定位分析,通过成像区分肿瘤与非肿瘤之间的界限。
ⅠSS适用于复杂结构的组织或细胞,它可以对单个RNA 分子进行测序,分辨率高,为研究复杂的生物学事件提供了新的途径,例如原位异质细胞群体中的细胞分化[37]。然而,ⅠSS的主要缺点在于一次实验所能识别到的RNA 数量较少,且使用的锁式探针可能会产生大量的探针特定偏差,即使结合了图像的方法,增加了识别到的RNA 数量,但还需要处理大量图像数据,且需要高分辨率的显微成像才能够准确获得碱基对信息,扫描出来的图像数据通常非常大,这对图像分析有不小的挑战。

Fig.3 The principle of in situ sequencing图3 靶向原位测序原理
1.3.2 荧光原位测序(FⅠSSEQ)
ⅠSS 技术中,锁式探针可能会产生大量的探针特定偏差,而且ⅠSS技术通常仅限于有明确注释基因的检测。针对这些问题,2015 年,Lee 等[39]开发了荧光原位测序(fluorescencein situsequencing,FⅠSSEQ)技术,利用随机六聚体引物在固定细胞中逆转录RNA,然后通过cDNA 的环化以非靶向方式生成测序文库。该方法进一步提高了原位测序技术的检测通量,能获得全基因组范围的基因表达图谱,包括基因表达,RNA剪接和转录后修饰等,同时保留了它们的空间位置信息。
FⅠSSEQ 技术首先将细胞固定在载玻片上,然后原位进行逆转录。逆转录后,将残留的RNA 降解以防止其竞争性抑制双链DNA 环化连接酶(CircLigase)。cDNA片段在60°C环化,CircLigase将环化后的cDNA 连接起来,之后进行滚环扩增(rolling-circle amplification,RCA),将每个cDNA环形扩增为包含多个原始cDNA 序列的滚环产物(RCP)(图4)。该过程中,在第k个循环引入荧光标签,对应于该cDNA 链的第k个碱基,然后再进行成像。之后冲洗掉荧光标签,再进行下一个循环。FⅠSSEQ 技术适合有细胞显微操作经验的研究人员,文库构建和测序可以在14 d内完成,该技术对计算处理能力的要求较低,图像分析大约只需2 d[39]。
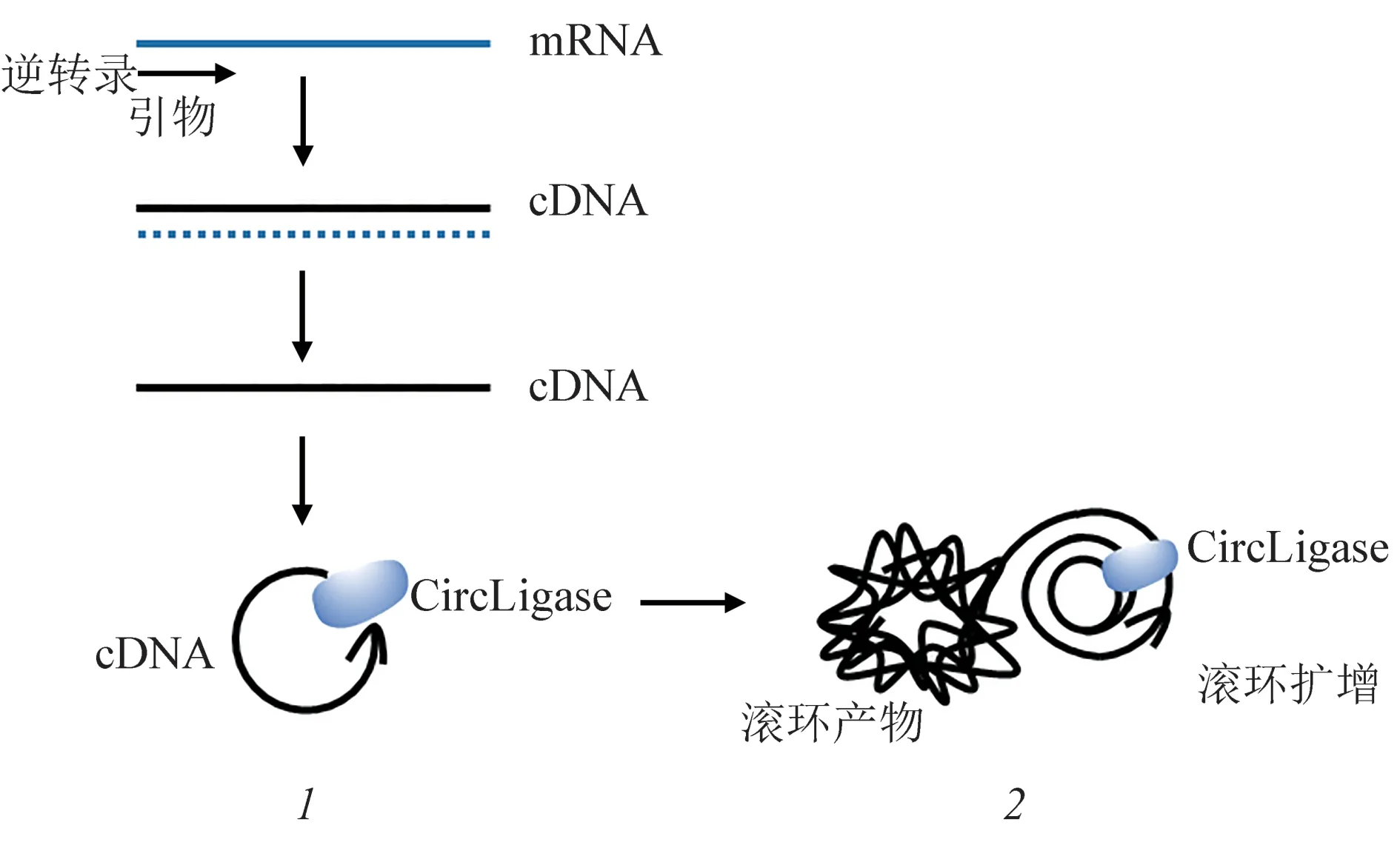
Fig.4 The principle of rolling-circle amplification in FISSEQ图4 荧光原位测序的滚环扩增原理
FⅠSSEQ 技术的优势在于,对于功能重要的转录本,其富集程度是单细胞RNA-seq的10倍以上。FⅠSSEQ 方法的局限性在于获得的基因数远少于RNA-seq,每个细胞仅获得约200 个mRNA 片段,而单细胞RNA-seq 则约为40 000 个mRNA 片段。该方法测序深度低,即使最初去除了rRNA,但rRNA 读数仍占总读数的40%~80%。除此以外,RCP 的大小又会限制每个细胞所能得到的读数,从而限制了该方法的灵敏度[34]。FⅠSSEQ无法提供单个细胞内RNA 的完整信息,会丢失低丰度的转录本[40]。
FⅠSSEQ 技术适用于大多数培养细胞和组织切片的样品(包括石蜡切片和冰冻切片),也适用于果蝇胚胎、诱导的多能干细胞及类器官等组织。FⅠSSEQ 测序得到的每个read 均有空间坐标,可以用于分析转录本在原位的分布情况。FⅠSSEQ 技术还可以与锁式探针技术结合,来检测癌组织中特异的RNA标志物。除此以外,FⅠSSEQ还可以对单个细胞的转录本进行测序[34]。
1.3.3 STARmap技术
2018 年, Wang 等[41]开 发 了STARmap(spatially-resolved transcript amplicon readout mapping)方法,将水凝胶的组织化学方法和原位测序相结合,可应用于完整的组织。STARmap 技术首先去除组织中的蛋白质和脂质,将组织转化为3D 水凝胶,降低成像背景,再用锁式探针与cDNA 结合,进行酶促扩增,形成“DNA 球”,这一过程相当于原位测序的滚环扩增。接着进行成像,成像一轮后使用60%甲酰胺将探针从组织水凝胶中洗脱下来,再开始下一个循环,从而实现对完整组织的3D原位测序。
1.4 基于活细胞标记的空间转录组技术
1.4.1 TⅠVA
上述几类空间转录组技术,需要对组织进行切片,而组织一旦切片,就无法获得完整单细胞的基因表达信息,当前的捕获方法难以从单个细胞中分离出mRNA 而不损害相邻组织。2014 年,Lovatt等[42]开发了一种新的空间转录组方法——TⅠVA(transcriptomein vivoanalysis),第一次非破坏性地从活细胞中捕获mRNA。该方法设计了一种光激活的标签(TⅠVA-tag),TⅠVA-tag包括一段poly(U)序列和两个poly(A)片段,该标签通过二硫键连接的细胞穿透肽穿透细胞膜,以使TⅠVA-tag 进入细胞,之后在细胞中进行光裂解,使得poly(U)序列暴露出来,并与细胞中mRNA的poly(A)尾退火连接。组织裂解后,用链霉亲和素和磁珠纯化得到TⅠVA-tag-mRNA 结合产物。之后,将mRNA 洗脱下来,进一步用于转录组分析(图5)。
TⅠVA适用于活细胞或活组织,Lovatt等[42]对比了TⅠVA-tag 方法和移液管吸取方法捕获的单个神经元细胞的转录组相关性,结果显示无显著差异(P= 0.2396,t检验)。除此以外,Lovatt 等[42]还在小鼠大脑的海马体活组织切片中使用了TⅠVA-tag方法,证明了光活化的TⅠVA-tag 可以保留在目标细胞内,不会迁移到邻近细胞。TⅠVA-tag可从活组织切片中的单个细胞中捕获大小和丰度不同的mRNA,而不会造成明显的细胞损伤。利用TⅠVAtag 方法,他们还鉴定了人脑组织切片中单个细胞的基因表达,证明TⅠVA 可用于表征人类细胞在原位组织环境中的转录组。2017年,Yeldell等[43]对原始TⅠVA 探针进行改进,增强了光解前的热稳定性,提高了光解后对mRNA的捕获亲和力。
TⅠVA 方法与RNA-seq 的结合能获得具有空间信息的完整单细胞基因表达信息,而不是某个切片或者部分单细胞的基因表达信息,从而为研究复杂的组织微环境对完整单细胞转录组的影响提供了一种可靠途径。但是该方法目前不适用于大量细胞的分析,且因其只能穿透活细胞,对已处理过的组织样品(例如固定后或冰冻后的组织样品)不适用。
1.4.2 ZipSeq
2020 年,Hu 等[44]发明了ZipSeq 方法,用于活细胞和活组织切片的空间转录组研究。该方法利用光控空间编码进行实时的活组织单细胞空间转录组研究。该技术将连有双链DNA 片段的高亲和力抗体或者脂质修饰过的寡核苷酸(lipid-modified oligonucleotide,LMO)锚定到细胞膜表面,用特定模式照明,释放特定组织区域细胞上锚定的双链DNA 片段上所连接的笼锁序列。再利用与该笼锁序列相匹配的,且带有不同zipcode,以poly(A)序列以及Ⅰllumina 读取序列结尾的单链杂交。该zipcode 作为组织细胞空间位置的识别序列,poly(A)序列作为反转录时poly-dT 引物的捕获对象,对其进行放大作为细胞空间位置鉴定的依据。该方法对活组织切片进行位置标记后,将细胞解离成单个细胞,再结合10×Genomics单细胞测序的方法进行转录组测序。

Fig.5 The principle of TIVA图5 TIVA原理
利用ZipSeq 技术,他们检测了体外伤口愈合过程中细胞基因表达的变化情况、淋巴结活组织切片的空间转录组情况以及肿瘤活组织内空间转录组表达情况。每种模型中都发现了一些与组织结构紧密相关的新的基因表达模式。在肿瘤微环境中,基因表达模式表明了骨髓和T细胞由外围向内的分化轨迹。该方法还提供了两种ZipSeq 的组合变体,可以有效缩放所定义区域的数量,从而为实时的或干扰之后的活组织基因表达模式的完整绘制提供了有效的途径,但该方法在空间分辨率上仍然有待进一步提高。
2 展 望
目前发展的各类空间转录组技术,都存在各自的优缺点(表1)。总的来说,基于原位杂交技术的smFⅠSH 及其衍生的空间转录组方法在不断地提高检测的转录本数目。但是检测的通量和灵敏度还受到组织空间的限制,所获得的基因表达信息只在一个组织层面上,还无法反映组织内完整单细胞的基因表达情况,价格也相对昂贵。基于激光显微切割的空间转录组方法,可以直观地分选出任何形态的细胞或细胞群,通过高通量测序获得已知和未知的基因表达信息,具有较好的性价比。但是由于组织需要切片,同样无法获得组织内完整单细胞的基因表达信息。基于组织原位测序的空间转录组方法,空间分辨率较高,可以直观地在较大组织切片尺度观察基因表达的丰度情况,但是获得的也是某个组织层面的细胞基因表达信息,检测通量不高,且捕获效率目前仍然非常有限。最近发展的活细胞空间转录组方法,可以获得具有空间信息的完整单细胞的基因表达情况,但对于细胞的精准定位以及细胞通量还需要进一步提高。
可以预见,这些空间转录组的方法将不断克服各自的不足,包括防止RNA 的降解、提高检测的通量和效率、降低成本和获取完整空间单细胞转录组等。同时,在获得细胞空间信息的基础上,未来空间转录组技术将加入时间的维度,从时空转录组的角度进一步提升转录组的研究层次,不断加深对组织细胞真实特性的理解,从而推进对发育过程和癌症等恶性疾病发生机制的理解和新治疗方案的研究。

Table 1 Comparison of spatial transcriptome technologies表1 空间转录组技术比较
