致病性大肠杆菌携带原噬菌体的预测及耐药性与毒力研究
2022-03-17祝希辉庞喆羽王志伟裴兰英曹胜亮薛希娟李玉保
祝希辉,庞喆羽,王志伟,裴兰英,曹胜亮,薛希娟,李玉保
(1.聊城大学农学院,聊城 252059,2.山东信得科技股份有限公司,青岛 266000)
大肠杆菌大量存在于环境中,可分为共生菌株和致病性菌株,致病性菌株又分为肠道致病性菌株和肠道外致病性菌株[1]。肠道致病性菌株可引起人和动物的肠炎和腹泻,某些血清型甚至会导致死亡[2]。肠道外致病性菌株可导致人或动物的尿路感染、新生儿脑膜炎与败血症,以及禽的呼吸系统疾病,可引起禽的急性死亡[3]。
致病性大肠杆菌对养殖业造成巨大的经济损失,同时对人类和公共卫生安全构成威胁。近年来,由于抗生素长期不科学使用,导致大肠杆菌耐药性快速攀升[4]。为有效控制细菌耐药性的产生,中国在饲料端禁用抗生素,在养殖端限用抗生素。因此,亟需寻找一种新型抗生素替代物用于大肠杆菌病的防治。噬菌体(bacteriophage)是能感染细菌、真菌、放线菌或螺旋体等微生物的病毒的总称[5],可特异性杀灭细菌而不破坏机体的菌群平衡[6],具有广阔的应用前景[7]。根据生活周期,噬菌体可分为烈性噬菌体和温和性噬菌体。在短时间内连续完成吸附、侵入、增殖、装配和裂解释放5个阶段而实现其增殖的噬菌体称为烈性噬菌体;温和噬菌体侵入宿主菌后并不发生裂解,而是整合到细菌基因组中,整合到细菌基因组的噬菌体称为原噬菌体(prophage)。研究证明,原噬菌体可通过水平转移等方式影响细菌的耐药性、毒力、生物膜形成及环境耐受力等特性,如霍乱弧菌的霍乱毒素(cholera toxin,CT)的编码基因(ctxA和ctxB)位于原噬菌体CTXφ上,通过构建霍乱弧菌CTXφ缺失突变株发现,原噬菌体CTXφ缺失使染色体上的调控基因aphA的转录水平降低,进而影响到形成生物被膜的基因表达,使其丧失生物被膜形成能力[8]。刘云等[9]通过构建原噬菌体缺失突变株发现,相较于野生株,缺失突变株的耐酸、耐碱、耐氧化应激能力下降,表明原噬菌体提高了菌株的环境耐受性。此外,原噬菌体还可通过编码细菌毒素和调控毒素的产生与释放来调控细菌的毒力[10]。本试验对NCBI中公布的不同大肠杆菌序列进行原噬菌体预测及分析,了解大肠杆菌携带原噬菌体及原噬菌体携带耐药基因、毒力基因情况,以期为大肠杆菌噬菌体的基础研究及噬菌体制剂研发提供一个新的视角与借鉴。
1 材料与方法
1.1 大肠杆菌全基因组来源
自NCBI中查询大肠杆菌全基因组数据,最后访问时间为2021年6月6号,本研究主要考虑致病性大肠杆菌携带原噬菌体情况,而NCBI中公布的大肠杆菌全基因组截至2021年6月6号有26 084株,考虑到工作量巨大及大肠杆菌人畜共患的特点,因此,试验随机下载了2010-2021年公布的来自12个国家的112株致病性大肠杆菌进行分析(表1)。由于NCBI中收录的样品地域性较强,美国的样品占41.07%,中国的样品占30.36%,而其他10个国家的数据占比相对较少(占28.57%)。
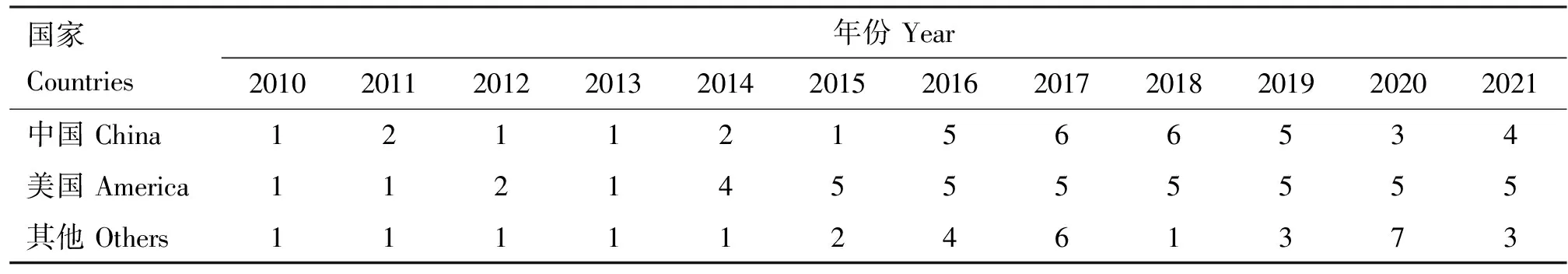
表1 大肠杆菌分离年份与地区
1.2 原噬菌体预测
PHASTER(http:∥phaster.ca/)用于快速识别和注释细菌基因组和质粒中的噬菌体序列,功能较全,且采用近几年最新的识别和处理数据策略,是一种实用的前噬菌体在线预测工具[11]。将获得的不同地域不同时间的大肠杆菌全基因序列统一整理为FASTA文件,使用原噬菌体在线预测工具PHASTER预测大肠杆菌基因组中的原噬菌体序列,PHASTER根据细菌中CDS的数量及含有的噬菌体序列进行打分,预测结果分为完整型噬菌体(>90分)、疑似型噬菌体(90~70分)、缺陷型噬菌体(<70分)3种。下载网站预测结果,获得所测菌株中预测的原噬菌体数量、在染色体上的具体位置、GC含量等信息,留存并用于分析。
1.3 原噬菌体分类
PHASTER网站对每一个原噬菌体给出了相似性最高的噬菌体,将其作为一种噬菌体类型,统计所有完整性原噬菌体所对应的最相似噬菌体数目,可对原噬菌体进行初步分类,同时在NCBI中查询每个最相似噬菌体所属的种属,对原噬菌体的种属分布进行统计分析。
1.4 原噬菌体携带抗生素耐药基因预测
将从NCBI中下载并命名整理好的大肠杆菌FASTA格式文件,输入到抗生素耐药基因(antibiotic resistance genes,ARGs)数据库[12](https:∥card.mcmaster.ca),预测大肠杆菌完整型原噬菌体所携带的耐药基因、耐药基因家族、耐药表型、耐药机制并加以分析。
1.5 原噬菌体携带毒力基因预测
将从NCBI中下载并命名整理好的大肠杆菌FASTA格式文件,输入到毒力因子数据库[13](http:∥www.mgc.ac.cn/VFs/main.html)中,预测大肠杆菌完整型原噬菌体中携带的毒力基因数目及毒力基因家族分布情况。
2 结 果
2.1 原噬菌体预测情况
2.1.1 112株大肠杆菌的原噬菌体预测情况 预测结果显示,112株大肠杆菌携带完整型原噬菌体1 024个,疑似型原噬菌体287个,缺陷型原噬菌体505个(图1)。每株大肠杆菌平均携带完整型原噬菌体9.1个,疑似型原噬菌体2.6个,缺陷型原噬菌体4.5个(图2)。
2.1.2 完整型原噬菌体分类情况 PHASTER统计结果表明,1 024个完整型原噬菌体共有63种噬菌体类型,其中BP4795(登录号:NC_004813.1)类型的占比最高,达到19.24%(197/1 024),其次是DE3(登录号:NC_042057.1),占比11.72%(120/1 024),LAMBDA(登录号:NC_001416.1)、MEP460(登录号:NC_019716.1)、P88(登录号:NC_026014.1)、CDTL(登录号:NC_009514.1)分别占比8.89%(91/1 024)、6.74%(69/1 024)、6.15%(63/1 024)和5.47%(56/1 024)。另外还有12种噬菌体类型占比在1%~5%(对应的完整型前噬菌体数目占总完整型原噬菌体数目的比例),30种噬菌体类型占比在1%以下,16种噬菌体类型仅有1个完整型原噬菌体对应。占比超过5%的噬菌体类型除P88 外均为长尾噬菌体科,在占比5%以下的噬菌体类型中长尾噬菌体占比最高。因此本研究中的完整型前噬菌体所对应的噬菌体类型大多是长尾噬菌体科,而短尾与肌尾噬菌体科相对较少,具体分布见表2。
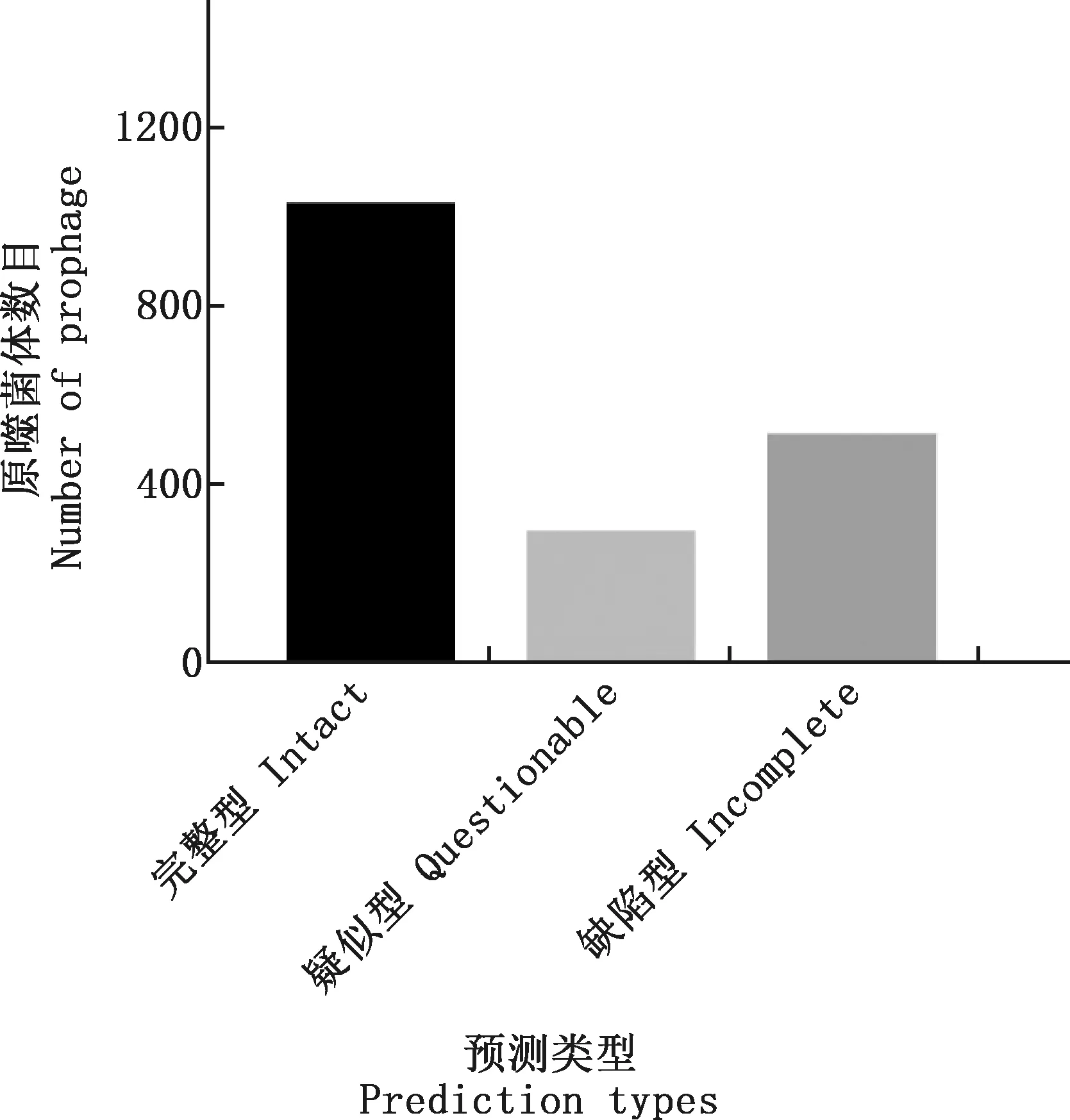
图1 112株大肠杆菌原噬菌体预测数目统计Fig.1 Statistics of predicted number of prophage of 112 strains of E. coli
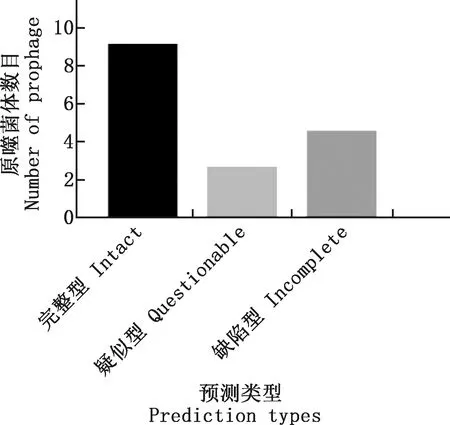
图2 平均每株大肠杆菌携带原噬菌体数目Fig.2 Average number of prophage per strain of E. coli

表2 完整型原噬菌体类型分布
2.2 原噬菌体的基因组分析
大肠杆菌原噬菌体基因组大小为3.1~152.6 kb,具体分布见表3,其中80 kb以内的原噬菌体占97.03%(1 762/1 816)。大肠杆菌携带原噬菌体的GC含量在38%~57%之间(表4),其中完整型原噬菌体基因组较大,且差别也较大,大小在5.6~152.6 kb,基因组大小在20~80 kb之间的占比88.97%(911/1 024),基因组GC含量在44%~52%之间的占比93.07%(953/1 024);疑似型原噬菌体基因组较小,在3.6~63.8 kb之间,40 kb以内占比91.29%(262/287),GC含量在44%~52%之间占比82.58%(237/287);缺陷型原噬菌体基因组最小,在3.1~47.7 kb之间,大部分在40 kb以内,占比98.61%(498/505),GC含量在44%~52%之间,占比84.56%(427/505)。大肠杆菌原噬菌体占细菌全基因组的比例在0.05%~3.01%之间,其中大部分处于0.1%~1.2%之间(图3)。

表3 不同类型大肠杆菌原噬菌体基因组大小分布
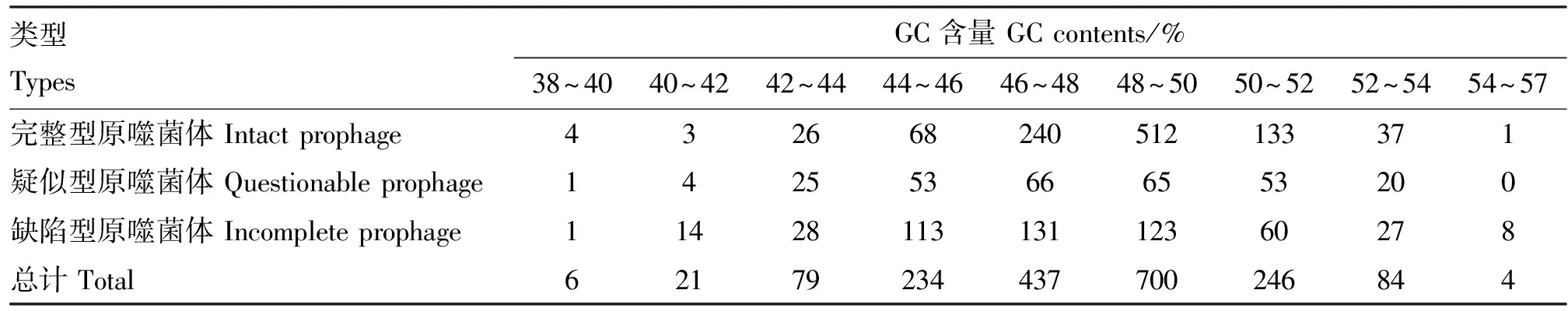
表4 不同类型大肠杆菌原噬菌体GC含量分布
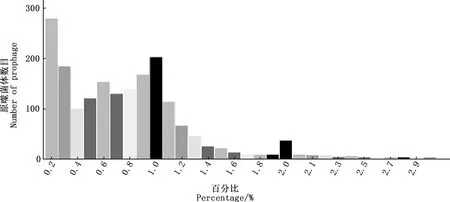
图3 原噬菌体基因占细菌基因组的比例Fig.3 Percentage of prophage in bacterial genome
2.3 大肠杆菌原噬菌体携带AGRs的预测统计
通过CARD耐药基因数据库对大肠杆菌原噬菌体进行检测,探讨AGRs在大肠杆菌原噬菌体中的分布情况,结果表明,1 024个完整型原噬菌体中携带1个或多个AGRs的有65个,携带率为6.35%,65个完整型原噬菌体共携带耐药基因253个,其中以大环内酯类(13.83%)、香豆素类(8.70%)、头孢菌素类(8.30%)、四环素类(7.11%)、利福霉素(6.72%)、氨基糖苷类(6.32%)为主,具体分布情况见图4。
253个耐药基因分别来自11个抗生素耐药基因家族,其中以耐药结节细胞分化(resistance-nodulation-cell division,RND)家族、小多重耐药(small multidrug resistance,SMR)家族、β-内酰胺渗透性(general bacterial porin with reduced permeability to beta-lactams)家族、主要协同转运蛋白超家族 (major facilitator superfamily,MFS)为主,具体分布情况见图5。
65个完整型原噬菌体耐药机制主要显示出5种模式,包括抗生素外排、抗生素渗透性降低、抗生素灭活、靶点代替、靶点改变,其中以抗生素外排为主(93.80%),具体分布情况见图6。
2.4 原噬菌体携带毒力基因预测
将1 024个完整型原噬菌体的基因序列提交到VFDB毒力因子数据库中,了解大肠杆菌原噬菌体中携带的毒力因子情况,数据统计结果显示,1 024个完整型原噬菌体中,携带1个或多个毒力基因的共438个,携带率高达42.78%,438个含有毒力基因的完整型原噬菌体共携带毒力基因1 274个,平均每个携带2.9个毒力基因,这1 274个毒力基因分别来自9个不同毒力因子家族(图7),包括NLE家族(793/1 274)、毒素相关家族(253/1 274)、铁转运相关家族(148/1 274)、黏附相关家族(33/1 274)、自动转运相关家族(20/1 274)、分泌相关家族(10/1 274)、铁/锰运输相关家族(12/1 274)、LEE家族(2/1 274)及沙门氏菌应激蛋白相关家族(3/1 274)。
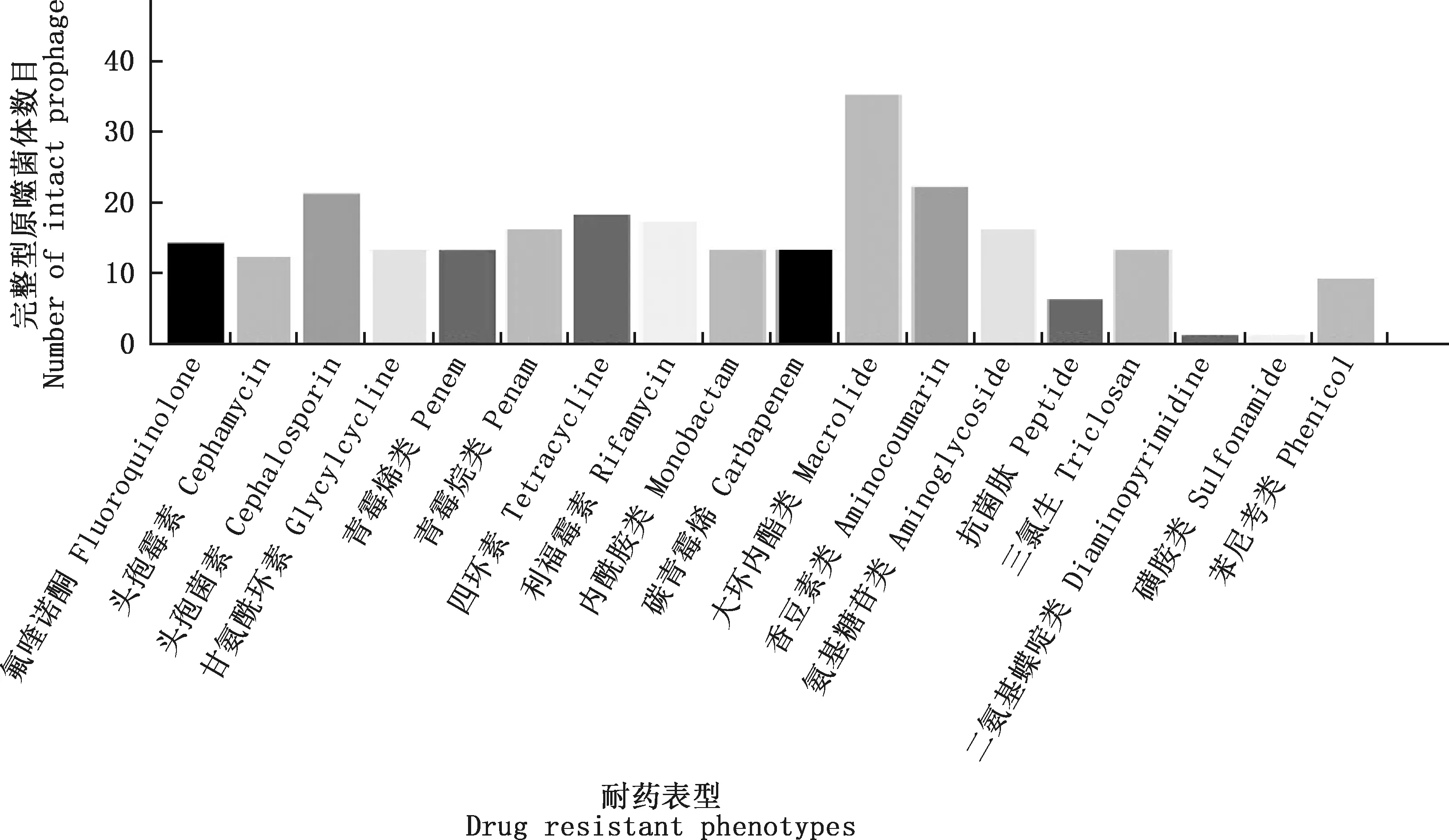
图4 完整型原噬菌体耐药表型分布Fig.4 Distribution of drug resistance phenotypes among intact prophage

图5 完整型原噬菌体携带耐药基因家族分布Fig.5 Distribution of drug resistance gene family carried by intact prophage

图6 完整型原噬菌体携带耐药基因耐药机制分布Fig.6 Distribution of drug resistance mechanism of drug resistance gene carried by intact prophage

图7 原噬菌体携带毒力基因家族分布Fig.7 Distribution of virulence gene family carried by prophage
3 讨 论
通过数据分析,112株大肠杆菌携带原噬菌体数目高达1 816个,平均每株大肠杆菌仅完整型原噬菌体就可携带9.1个,表明大肠杆菌普遍携带原噬菌体。对大肠杆菌原噬菌体的分析表明,大肠杆菌原噬菌体可携带耐药基因与毒力基因,完整型原噬菌体耐药基因携带率为6.35%,毒力基因携带率为42.78%。目前在多种细菌的原噬菌体中发现了抗生素耐药基因,如在大肠杆菌和沙门氏菌中发现了P1-like噬菌体携带blaSHV-2和blaCTX-M-27β-内酰胺酶基因[14-15];在猪源大肠杆菌和人源临床肺炎克雷伯菌中发现P7-like噬菌体携带多黏菌素耐药基因MCR-1[16-17];在西班牙某医院分离到的多个肠杆菌科细菌中发现超广谱β-内酰胺酶基因blaCTX-M-10与原噬菌体存在偶联关系[18]。 原噬菌体携带耐药基因可能会增强细菌的耐药性,使其更容易适应环境[19]。
本试验发现,完整型原噬菌体携带大量毒力基因,提示原噬菌体可能会影响细菌的毒力。研究发现,携带毒力基因的温和性噬菌体侵入宿主细菌后成为原噬菌体,原噬菌体中的毒力基因被表达使菌株产毒,因此病原菌就依赖原噬菌体编码的基因产生更多毒素[10]。大多数情况下,原噬菌体整合的位置在非编码RNA,但少数温和性噬菌体整合到细菌编码蛋白质的基因中,从而破坏了该蛋白质的功能,如果这个蛋白质是毒力因子,那么细菌的毒力则会下降,因此,原噬菌体对于细菌的毒力不仅仅是增强,少数情况也会降低[20]。同一种原噬菌体对一种细菌中的不同菌株毒力的影响效果不同,有的使其毒力增强,而有的则使其毒力减弱。研究发现,原噬菌体BTP1上的bstA基因编码一个功能尚未知的蛋白,其在不同血清型的同种细菌上表现出增强或减弱毒力两种不同的效果,这些现象体现出原噬菌体对细菌毒力影响的复杂性[21-23]。
另外,原噬菌体中还携带有一部分黏附相关基因,提示原噬菌体的存在可能与细菌黏附能力的高低相关。原噬菌体可以影响细菌的黏附能力,这与本试验预测到的原噬菌体中携带黏附相关毒力基因的发现相符,细菌感染的第一步是黏附到机体组织细胞表面,原噬菌体能够增强宿主细菌对组织细胞的黏附性[10]。研究发现,缺失原噬菌体phiv142-3的大肠杆菌菌株的菌落变小,对DF-1细胞的黏附力降低,对血清杀伤作用的抵抗力明显降低,在酸、碱和氧化应激下的存活率分别降低95.6%、71.6%和99.6%[24]。原噬菌体编码的蛋白还可以增强细菌对宿主血小板和免疫球蛋白的黏附能力,以增强细菌的毒力[25-26]。值得注意的是,在本次数据分析中发现,大肠杆菌原噬菌体中含有沙门氏菌应激蛋白相关基因,提示原噬菌体可能是毒力基因跨菌种传播的一个途径,相关研究还有待进一步深入,但噬菌体及原噬菌体在同种细菌、不同种细菌甚至不同物种之间介导水平基因转移的作用不可忽视[10,27-28]。
大肠杆菌携带原噬菌体及原噬菌体携带耐药基因与毒力基因的现象提示,大肠杆菌原噬菌体可影响大肠杆菌自身的耐药性、毒力及环境适应性等一系列特性,改变大肠杆菌的变异频率,这些影响多数是对人类有害的,如增强毒力、提高耐药性等,为临床防治大肠杆菌感染增加难度。也有部分研究表明,有些原噬菌体可以降低大肠杆菌对头孢菌素和氟喹诺酮的敏感性,同时减少变异频率[29]。另外在噬菌体生物制剂的研发时,要充分了解菌种和噬菌体的生物学信息,敲除其自身所携带的耐药基因和毒力基因等,防止或减少细菌水平基因转移和进化,保证噬菌体产品的安全高效。
4 结 论
大肠杆菌普遍携带原噬菌体,但原噬菌体在大肠杆菌基因组中所占比例不高,在0.05%~3.01%之间,部分完整型原噬菌体携带耐药基因,以大环内酯类、香豆素类及头孢菌素类耐药表型为主,耐药机制有5种,以抗生素外排为主。另外,42.28%的完整型原噬菌体携带毒力基因,分别来自9个毒力因子家族。
