RNA测序分析OCT4诱导人毛囊间充质干细胞去分化的作用机制
2022-03-13阮迎春彭森孙鹏鹏姜维娜刘志晶陈桦
阮迎春 彭森 孙鹏鹏 姜维娜 刘志晶 陈桦

[摘要] 目的 检测转导Octamer转录因子4(OCT4)的人毛囊间充质干细胞(hHFMSC)衍生的贴壁和悬浮亚群转录组去分化差异,探讨OCT4诱导hHFMSC去分化的作用机制以及去分化程度对细胞形态及黏附性的影响。方法 收集hHFMSC、贴壁亚群和悬浮亚群3组细胞并提取总RNA,进行转录组测序,分析差异基因的组织特异性生物功能,并检测组织特异性基因的表达变化。结果 OCT4转导后hHFMSC中下调基因显著富集于毛囊、表皮及皮肤附属器发育的生物过程,且毛囊发育、皮肤分化相关基因(MYSM1、KRT15及RBPJ等)显著下调;与贴壁亚群相比,悬浮亚群中MYSM1及FZD6等毛囊发育基因进一步下调。结论 过表达OCT4可能通过调控毛囊、表皮及皮肤附属器发育基因的表达诱导hHFMSC去分化,且细胞形态及黏附性可能与组织特异性基因表达谱有关。
[关键词] 八聚体转录因子3;间质干细胞;毛囊;细胞重新编程;细胞去分化
[中图分类号] R329.2
[文献标志码] A
[文章编号] 2096-5532(2022)01-0019-05
doi:10.11712/jms.2096-5532.2022.58.036
利用基因工程技术重编程成体细胞向特定谱系细胞分化是近年来再生医学领域的研究热点,单独转导Octamer转录因子4(OCT4)即可触发体细胞的重编程[1-3]。有研究将OCT4转导入人毛囊间充质干细胞(hHFMSC)后细胞由长梭形变为多边形,且逐渐出现一类小而圆的易悬浮细胞,其经造血因子诱导可转分化为成熟红细胞 [4];转录组测序分析基因表达谱发现,贴壁细胞中上调基因富集于胚胎发育,悬浮细胞中上调基因富集于造血谱系分化[5]。本研究利用转录组测序检测OCT4对毛囊、表皮以及皮肤附属器发育等组织特异性基因表达的调控作用,分析贴壁和悬浮细胞去分化程度的差异,探讨OCT4对hHFMSC的去分化作用,以及去分化程度对细胞形态及黏附性的影响,为hHFMSC在再生医学等领域广泛应用提供理论依据。现将结果报告如下。
1 材料和方法
1.1 细胞培养和试剂
hHFMSC为前期工作中从人毛囊真皮乳头和真皮鞘中分离并鉴定获得,用携带OCT4的慢病毒载体pLV-EF1α-OCT4-IRES-EGFP及含pVSVG、gag-pol和rev的包装质粒共同转染293T细胞。然后再用包装成慢病毒颗粒感染hHFMSC而获得hHFMSCOCT4。经传代培养12 d后获得贴壁生长细胞亚群hHFMSCOCT4-adherent,收集hHFMSCOCT4-adherent上层培养液,以800 r/min离心5 min,用完全培养液重悬获得悬浮细胞亚群hHFMSCOCT4-floating。hHFMSCOCT4-adherent和hHFMSCOCT4-floating加含体积分数0.10的FBS、10 μg/L bFGF和100 kU/L青霉素/鏈霉素的H-DMEM/F12培养液,移入37 ℃、含体积分数0.05 CO2的细胞培养箱中培养。
1.2 总RNA提取及转录组测序
用Trizol提取各细胞样品总RNA,每组设3个重复样本。用带有Oligo(dT)的磁珠富集mRNA;以mRNA短片段为模板合成cDNA,经过纯化、末端修复以及加A尾等处理后进行PCR扩增。用Agilent 2100 Bioanalyzer对构建好的文库进行质检,使用Illumina HiSeq X Ten测序仪进行测序。采用Trimmomatic软件对原始数据进行质量预处理,并对整个质控过程中的reads数进行统计汇总,htseq-count软件获取每个样本中比对到蛋白编码基因上的reads数,用cufflinks软件计算蛋白编码基因的表达量(FPKM值)。
1.3 差异基因(DEG)筛选及富集计算
DEG筛选条件为差异倍数(FC)>2或<-2,且P<0.05。利用DAVID数据库分别对两两细胞对比的DEG进行GO功能富集分析,计算Enrichment score。Enrichment score=(m/n)/(M/N)。其中,N为所有基因中具有GO注释的基因数目;n为DEG中具有GO注释的基因数目;M为所有基因中注释为某特定GO条目的基因数目;m为注释为某特定GO条目的DEG数目。
1.4 统计学分析
用basemean值估算各个样本基因表达量,计算FC;负二项分布检验对reads数进行差异显著性检验;利用超几何分布检验计算GO功能中显著富集的差异蛋白编码基因/转录本列表的P值,对P值进行多重检验纠正后得到FDR。以P<0.05为差异有统计学意义。
2 结 果
2.1 DEG的功能富集分析
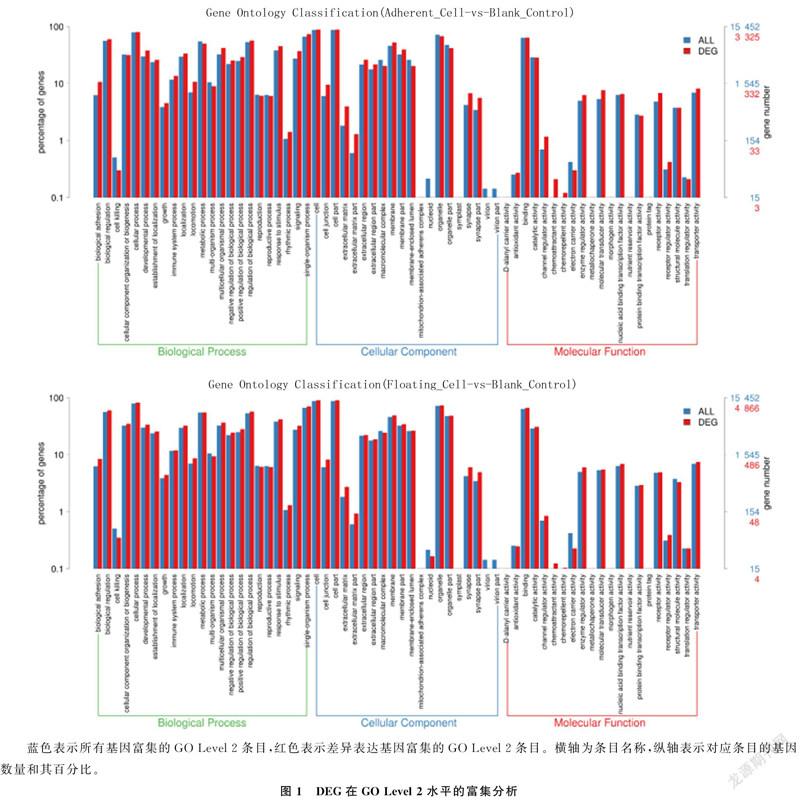
与hHFMSC进行比较,hHFMSCOCT4-adherent中上调基因共有2 401个,下调基因共有1 882个;而hHFMSCOCT4-floating中上调基因3 107个,下调基因有2 999个。与hHFMSCOCT4-adherent相比,hHFMSCOCT4-floating中筛选出1 940个DEG,其中上调基因有833个,下调基因有1 107个。表明hHFMSCOCT4-floating与hHFMSC的转录本差异更为显著,且贴壁细胞与悬浮细胞是两种相对独立的亚群。利用DAVID数据库对DEG的生物功能进行GO Level 2水平的富集分析,包括生物过程(BP)、细胞组分(CC)和分子功能(MF)。结果显示,与hHFMSC相比,hHFMSCOCT4-adherent中DEG显著富集于生物黏附、生物调节、细胞过程以及发育过程等生物过程,并发现DEG主要分布在细胞连接、细胞外基质部分及细胞膜等细胞成分,主要具有结合、通道调节因子活性、酶调节因子活性、分子转导活性及受体活性等分子功能(图1);与hHFMSC相比,hHFMSCOCT4-floating中DEG富集的生物过程主要包括生物黏附、生物调节、多器官过程和单器官过程等,DEG主要分布在细胞连接、细胞外基质部分、大分子复合物、细胞膜等细胞成分中,并具有结合、催化活性、酶调节因子活性、核酸结合转录因子活性及转运活性等分子功能(图1)。说明OCT4转导后细胞黏附性、细胞连接组分、器官发育潜能、信号转导及转录活性均发生改变,提示细胞骨架可能发生重塑,黏附生长的方式可能改变,组织特异性基因转录失活及多潜能性基因转录激活共同协调器官或组织发育潜能,从而实现去分化与重编程。
2.2 组织特异性基因的富集分析
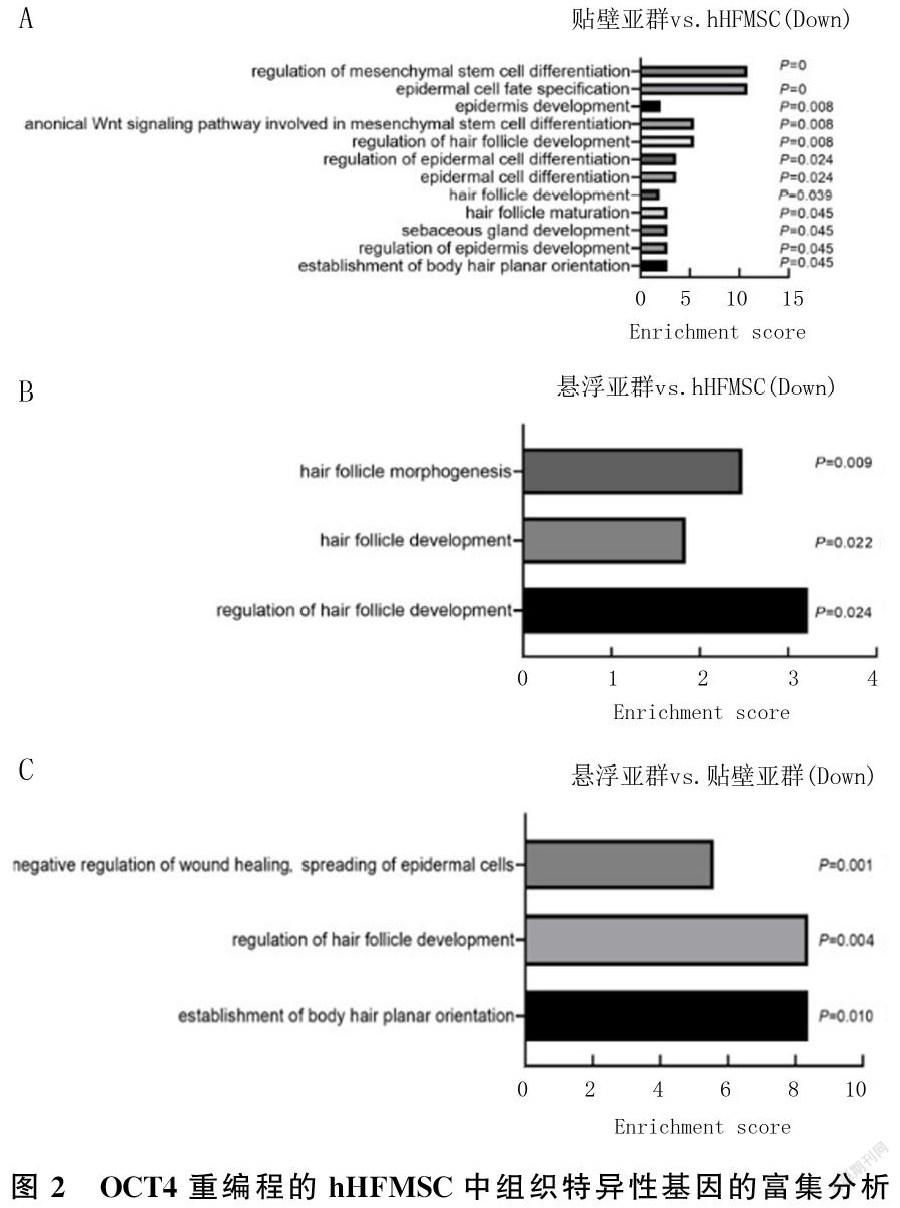
本文GO分析OCT4转导对hHFMSC中组织特异性基因表达调控情况,结果显示,与hHFMSC相比较,hHFMSCOCT4-adherent中下调基因富集的组织特异性生物过程有12个(图2A),包括间充质干细胞分化、表皮细胞命运特异化、表皮发育、毛囊发育与成熟、皮脂腺发育等;hHFMSCOCT4-floating中下调基因富集的组织特异性生物过程有3个,包括毛囊形态发生、毛囊发育以及毛囊发育的调节。
转导OCT4后hHFMSC中毛囊以及皮肤组织的特异性基因表达下调,OCT4可能通过抑制组织特异性基因的表达而促使hHFMSC去分化(图2B)。与hHFMSCOCT4-adherent相比,hHFMSCOCT4-floating中下调基因显著富集于损伤修复与表皮延伸的负调控、毛囊发育的调节以及体毛水平定向建立的生物过程(图2C)。hHFMSCOCT4-floating的低黏附性、细胞骨架蛋白表达的改变及紧密连接通路基因的下调可能与组织特异性基因下调有关,从而失去了hHFMSC原有的细胞形态及生长方式。
2.3 毛囊、表皮及皮肤附属器等组织特异性基因的表达情况
与hHFMSC进行比较,hHFMSCOCT4-adherent中有14个特异性基因显著下调,其中KRT15、KRT34与表皮发育及角化相关,GLI2参与表皮细胞分化与基底细胞癌发生, COL17A1与基底膜形成有关,FZD3、ALX4等与毛囊发育有关,RBPJ则在毛囊成熟、皮脂腺发育、表皮细胞命运的特异化中发挥重要作用;下调最显著的基因为KRT15、FZD3与COL17A1,下调倍数分别为-6.92、-4.60和-5.74(图3A);而hHFMSCOCT4-floating中下调最显著的特异性基因包括毛囊形态发生相关基因FOXQ1、FGF7及毛囊发育相关基因ALX4,下调倍数分别为-9.61、-6.23和-5.43(图3B)。提示过表达OCT4后,hHFMSC中部分组织特异性基因表达下降,可能在一定程度上消除了基因组定向分化的记忆,抑制了hHFMSC毛囊及皮肤的特异性分化发育潜能,实现了重编程;与hHFMSCOCT4-adherent相比较,hHFMSCOCT4-floating中体毛水平定向建立的相关基因FZD6和ASTN2、表皮延伸负调控的相关基因CLASP1以及毛囊发育的相关基因MYSM1显著下调(图3C)。3组细胞两两比较均显示MYSM1基因表达显著下调,MYSM1基因可能是OCT4对hHFMSC发挥去分化作用的潜在靶点。
3 讨 论
OCT4作为一种具有强大重编程作用的多潜能转錄因子,主要存在于胚胎干细胞(ESC)细胞核内,在维持ESC的多潜能性和自我更新中发挥核心作用[6-8]。OCT4不仅可以维持生殖细胞和肿瘤细胞的“干性”,其异位表达还可提高骨髓间充质细胞的“干性”[9]。细胞去分化是指细胞从高度专能性分化状态转变到细胞重新获得多潜能性的过程;细胞的去分化、转分化以及重编程对于新细胞或组织的再生有重要意义[10]。重编程研究发现,OCT4、SOX2、KLF4和cMyc(OSKM)可将体细胞重编程为诱导性多能干细胞(iPSC),还可以将子宫内膜息肉与宫颈息肉来源的间质干细胞去分化成胚胎样细胞[11]。
转分化研究发现,OCT4可以促使类胚体中肝细胞向胆管上皮细胞转分化,还可在内皮诱导体系中促使肝癌干细胞向内皮样细胞转分化[12]。
毛囊是可再生的器官并且可进行自我更新[13]。hHFMSC为存在于毛发真皮乳头和真皮鞘中的间充质干细胞,其免疫原性低且来源广泛,在体外经诱导具有成骨、成软骨、成脂肪、成神经和成肌肉等多向分化潜能[14]。前期研究将OCT4单独转导入hHFMSC,经传代培养后细胞形态从长梭形变成多边形,并逐渐由贴壁生长变成低黏附生长;通过转录组测序显示基因表达谱发生显著改变,且贴壁细胞与悬浮细胞具有显著不同的转录本;二者多潜能性生物过程和多潜能性基因的表达亦不同,即贴壁细胞倾向于胚胎发育,而悬浮细胞获得一定造血谱系分化潜能[6]。结合本文研究结果,OCT4转导后下调基因显著富集于毛囊、表皮及皮肤附属器发育等生物过程,说明转导OCT4使hHFMSC丢失了一部分组织特异性分化发育专能,从而实现去分化;由于转导OCT4后细胞形态发生改变,推测OCT4对hHFMSC的去分化作用可能影响了细胞形态和骨架结构,且去分化程度的差异可能导致了贴壁细胞和悬浮两种细胞亚群的产生。还有研究结果证实,RhoA/ROCK通路可通过肌动蛋白细胞骨架聚合参与平滑肌的去分化[15];microRNA-138可以通过抑制F-肌动蛋白聚合导致软骨细胞去分化[16];此外,黏着斑的组装可诱导软骨细胞表型改变并发生去分化,说明细胞骨架蛋白及黏附分子与细胞去分化之间关系密切[17]。
本文对DEG进一步深入分析显示,OCT4可能通过调控KRT15、KRT34、ALX4及RBPJ等基因的下调使hHFMSC去分化,特别是毛囊发育基因MYSM1在各组细胞表达均下调,推测其可能是OCT4抑制hHFMSC组织特异性分化的潜在靶点;而悬浮细胞的类圆形形态以及低黏附性则有可能与hHFMSC更高程度的去分化状态相对应,或许与此亚群细胞更易诱导成造血细胞有关。但去分化与细胞骨架重塑及黏附性改变之间的因果关系尚不明确,且OCT4如何调控组织特异性基因表达的具体分子途径尚需进一步研究。
综上所述,过表达OCT4对hHFMSC转录组进行了重编程,可能通过下调组织特异性基因的表达在一定程度上抑制了其向毛囊、表皮及皮肤附属器等组织分化的专能性,从而赋予hHFMSC向造血谱系等多胚层组织及器官的分化潜能,且去分化程度及向造血谱系转分化潜能可能与不同细胞亚群对应的组织特异性基因表达谱有关。本文结果可为hHFMSC在再生医学领域应用提供了理论依据。
[参考文献]
[1]KIM J B, GREBER B, ARAZO-BRAVO M J, et al. Direct reprogramming of human neural stem cells by OCT4[J]. Nature, 2009,461(7264):649-643.
[2]LI Y Q, ZHANG Q, YIN X L, et al. Generation of iPSCs from mouse fibroblasts with a single gene, Oct4, and small molecules[J]. Cell Research, 2011, 21(1):196-204.
[3]KIM J B, SEBASTIANO V, WU G M, et al. Oct4-induced pluripotency in adult neural stem cells[J]. Cell, 2009,136(3):411-419.
[4]WANG B, LIU X M, LIU Z N, et al. Human hair follicle-derived mesenchymal stem cells: isolation, expansion, and differentiation[J]. World Journal of Stem Cells, 2020,12(6):462-470.
[5]YU X Z, SUN P P, HUANG X G, et al. RNA-seq reveals tight junction-relevant erythropoietic fate induced by OCT4 in human hair follicle mesenchymal stem cells[J]. Stem Cell Research & Therapy, 2020,11(1):454.
[6]WADKIN L E, OROZCO-FUENTES S, NEGANOVA I, et al. OCT4 expression in human embryonic stem cells: spatio-temporal dynamics and fate transitions[J]. Physical Biology, 2021,18(2): 026003.
[7]APOSTOLOU E, STADTFELD M. Cellular trajectories and molecular mechanisms of iPSC reprogramming[J]. Current Opinion in Genetics & Development, 2018,52:77-85.
[8]STREBINGER D, DELUZ C, FRIMAN E T, et al. Endogenous fluctuations of OCT4 and SOX2 bias pluripotent cell fate decisions[J]. Molecular Systems Biology, 2019,15(9):e9002. doi:10.15252/msb.20199002.
[9]SONG B M, KIM D K, SHIN J, et al. OCT4 directly regulates stemness and extracellular matrix-related genes in human germ cell tumours[J]. Biochemical and Biophysical Research Communications, 2018,503(3):1980-1986.
[10]BROCKES J P, KUMAR A. Plasticity and reprogramming of differentiated cells in amphibian regeneration[J]. Nature Reviews Molecular Cell Biology, 2002,3(8):566-574.
[11]DING D C, CHU T Y, LIU H W. Dedifferentiation of human uterine polyp stem cells into embryo-like cells during inducing pluripotency[J]. International Journal of Biological Sciences, 2018,14(11):1586-1598.
[12]LIU H L, TANG H T, YANG H L, et al. Oct4 regulates the transition of cancer stem-like cells to tumor endothelial-like cells in human liver cancer[J]. Frontiers in Cell and Developmental Biology, 2020,8:563316.
[13]PAUS R, COTSARELIS G. The biology of hair follicles[J]. The New England Journal of Medicine, 1999,341(7):491-497.
[14]LIU X J, SONG L, LIU J Y, et al. miR-18b inhibits TGF-β1-induced differentiation of hair follicle stem cells into smooth muscle cells by targeting SMAD2[J]. Biochemical and Biophysical Research Communications, 2013,438(3):551-556.
[15]QI Y, LIANG X Y, DAI F, et al. RhoA/ROCK pathway activation is regulated by AT1 receptor and participates in smooth muscle migration and dedifferentiation via promoting actin cytoskeleton polymerization[J]. International Journal of Molecular Sciences, 2020,21(15):E5398.
[16]ZHENG H J, RAMNARAIGN D, ANDERSON B A, et al. MicroRNA-138 inhibits osteogenic differentiation and mineralization of human dedifferentiated chondrocytes by regulating RhoC and the actin cytoskeleton[J]. JBMR Plus, 2019,3(2):e10071. doi:10.1002/jbm4.10071.
[17]SHIN H, LEE M N, CHOUNG J S, et al. Focal adhesion assembly induces phenotypic changes and dedifferentiation in chondrocytes[J]. Journal of Cellular Physiology, 2016, 231(8):1822-1831.
(本文編辑 黄建乡)
3063500338222
