基于PSY2基因单碱基突变的木薯薯肉颜色分子标记开发与利用
2022-03-11朴朴森尚小红许丰收廖明馨严华兵王文泉
朴朴森 尚小红 许丰收 廖明馨 王 明 严华兵,* 陈 新 王文泉
(1 中国热带农业科学院热带作物生物技术研究所,海南 海口 571101;2 广西农业科学院经济作物研究所, 广西 南宁 530007;3 海南大学热带作物学院,海南 海口 570228;4 中国热带农业科学院热带作物品种 资源研究所,海南 海口 571101;5 缅甸教育部研究与创新部生物技术研究院,缅甸 曼德勒 05151)
木薯(ManihotesculentaCrantz)是大戟科木薯属的高淀粉块根作物,广泛应用于淀粉生产,工业原料,以及作为亚非拉地区人民的日常口粮[1],为世界近6亿人口提供粮食保障,尤其在非洲,主要以木薯为食[2]。自然界中木薯块根肉质颜色以白色为主,长期以白肉木薯为主食易导致维生素A缺乏并引发相应疾病,如夜盲症等。根据类胡萝卜素含量的差异,木薯可分为粉红色、浅黄色、黄色和深黄色等不同块根肉质颜色的种质资源[3],这些颜色的木薯比白肉木薯具有更高的营养价值,因此需要培育具有高类胡萝卜素含量的木薯新品种以改善非洲人的膳食结构,减少维生素A源缺乏现象。木薯块根中的类胡萝卜素不仅可以帮助富集维生素A源,提高蛋白质含量,还可延缓采后生理衰变(postharvest physiological deterioration,PPD)[4],而且类胡萝卜素的累积量与PPD的延迟存在显著正相关关系[5-6]。
近年来木薯分子标记辅助育种技术的发展取得了巨大的进步。木薯的分子标记遗传连锁图从以同工酶、限制性内切酶片段长度多态性(restriction fragment length polymorphism,RFLP)和随机扩增多态性DNA标记(random amplified polymorphic DNA,RAPD)为主[7-8],过渡到基于PCR技术的简单重复序列标记(simple sequence repeats,SSR)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)、相关序列扩增多态性(sequence-related amplified polymorphism,SRAP)和少数基因特异性标记[9-13],到现阶段通过新一代测序技术产生的SNP构成[14]。遗传连锁图的标记类型和标记数量都有极大的改善,如在国际木薯遗传图谱委员会2014年发表的超高密度的遗传连锁图中,标记总数达22 403个,其中18个遗传连锁群与染色体组完全对应[15]。随着木薯基因组精细图的陆续发表[16-20],针对控制关键性状的分子标记定位已经达到单碱基突变的程度,如抗木薯花叶病毒位点被锁定在6个关键的SNP位点,木薯的Waxy位点被确定为GBSSI基因第11个内含子上的C→G突变[21],已开发成基于PCR技术的分子标记。
木薯块根类胡萝卜素合成途径中,八氢番茄红素合成酶(phytoene synthase,PSY)是类胡萝卜素合成起始的关键酶,木薯基因组中含有3个编码八氢番茄红素合成酶的基因,分别为PSY1、PSY2、PSY3,其中PSY2基因编码序列第572个碱基C→A的突变,导致编码氨基酸由丙氨酸(Ala)变成天冬氨酸(Asp),氨基酸的改变使PSY2活性增加3倍以上,从而提高了块根中类胡萝卜素的含量[22]。通过检索植物基因组数据库(JoinTGenome Institute,JGI)发现,该基因的突变位点为木薯特有,鲜出现在其他物种中[23]。该突变位点存在限制性内切酶AluI的酶切位点,根据该特性开发了一对酶切扩增多态性序列(cleaved amplified polymorphism sequences,CAPS)分子标记,并用于鉴定该位点基因型,结合基因型和田间块根颜色表明该标记的选择准确率达100%[24]。然而,该CAPS分子标记的应用需要一些酶切的步骤,相对于简易的分子标记过程复杂。此外,在已开发的CAPS分子标记检测结果中,黄肉与白肉分子标记的条带仅相隔22 bp,条带在琼脂糖凝胶电泳图中太近而不易分辨。以上原因导致该CAPS分子标记存在弊端,限制了其实用性。因此,需要开发出一种简易、快捷且价格低廉的分子标记来提高木薯分子标记辅助育种的效率。
单核苷酸扩增多态性(single-nucleotide amplified polymorphism,SNAP)标记技术是通过将一条PCR引物的3′末端安排在SNP(single nucleotide polymorphism)位点上,并在第3个碱基(3′→5′)引入1个错配的碱基,另一条PCR引物按照传统方法设计;当利用这种引物对进行PCR扩增时,仅在第3个碱基存在错配等位基因的PCR产物是第1和第3个碱基都存在错配等位基因的1 000倍左右,因此通过标准的琼脂糖凝胶电泳可以明确判定PCR扩增条带的“有”和“无”,进而呈现出单碱基变异[25]。SNAP具有操作简单、快速便捷等优点,已先后应用于水稻(OryzasativaL.)[26]、大豆[Glycinemax(Linn.) Merr.][27-28]、玉米(ZeamaysL.)[29]、油菜(BrassicanapusL.)[30]、黄瓜(CucumissativusL.)[31]等作物的分子标记辅助助育种。本研究利用SNAP技术对前期开发的薯肉颜色CAPS标记进行升级[24],直接通过PCR产物的有无来判断不同薯肉颜色品系PSY2的等位基因类型,并用于食用木薯培育的亲本选择和子代基因型筛选。
1 材料与方法
1.1 试验材料
白色薯肉木薯品系:华南205、KU50;黄色薯肉木薯品系:SC9、18R和新选048/XX048;XX048自交一代28个株系,总计33个试验材料,于广西壮族自治区农业科学院里建科研基地正常田间种植,常规管理。
木薯杂交种实生苗株系62个:以黄色薯肉品种SC9、CH12和18R为母本,与泰国优良品种HB60以及国内主推品种SC205、SC124、SC8、18R和GR6在2016年配制F1代杂交组合8个,2017年8月育苗529株并于当年11月移栽大田(澄迈大丰农场),经2018和2019年自然淘汰和人工选择,于2020年4月保留62个株系(表1)。
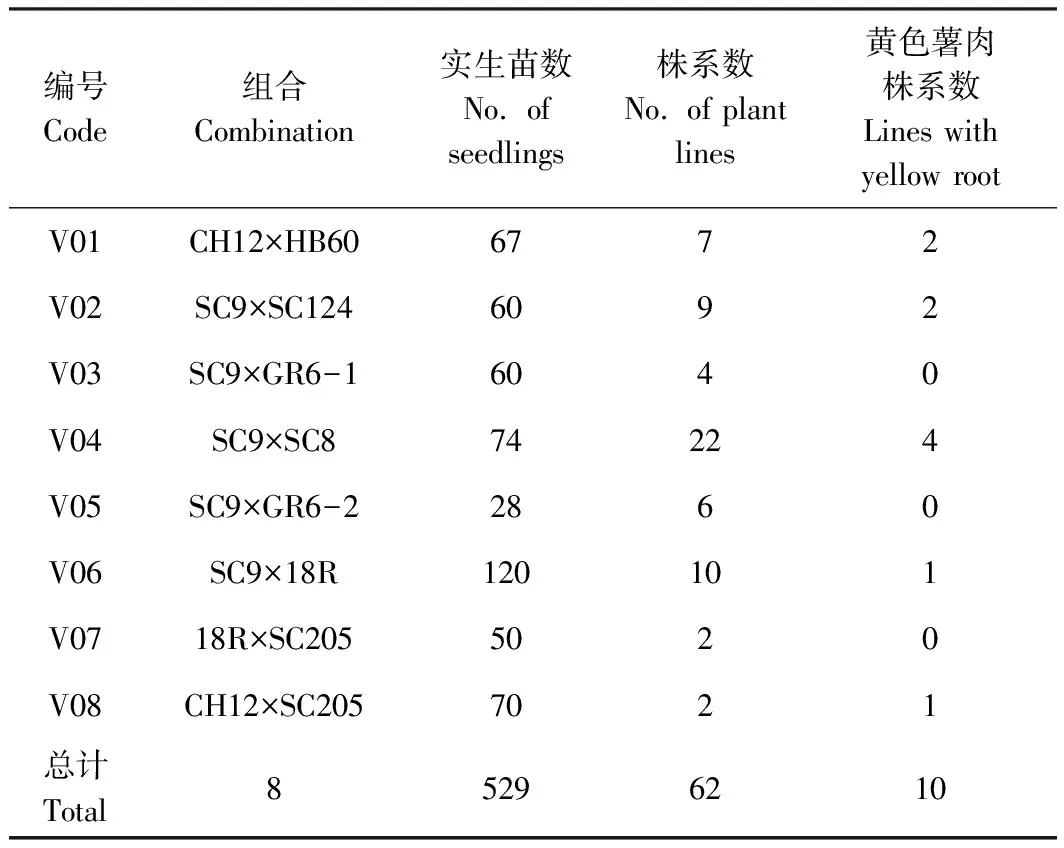
表1 杂交组合配制与实生苗数量Table 1 Hybrid combination and the number of their seedlings
国家木薯种质资源圃(儋州)可食用木薯种质45份,种植于儋州宝岛新村,常规管理。
1.2 试验方法
1.2.1 DNA提取 采集木薯植株幼嫩叶片,用液氮研磨成细粉状,然后参照植物DNA提取试剂盒(成都福际生物)的程序提取基因组DNA,通过琼脂糖凝胶电泳检测DNA质量,并用UV-2700紫外分光光度计(日本岛津)检测浓度,稀释成50 ng·L-1,于-20℃保存备用。
1.2.2 木薯块根颜色评估 田间收获块根时,每个株系选取3个来自不同单株的块根,横切后记录块根颜色并拍照留存。统计各株系块根颜色,并利用Excel软件进行卡方测验。
1.2.3 SNAP引物设计PSY2基因的完整序列从美国能源部生物信息网站(www.phytozome.com)下载,并确定目标突变位点的位置,利用网络版的SNAPER软件(http://pga.mgh.harvard.edu/cgibin/snap3websnaper3.cgi)进行引物设计,并将正向引物3′端倒数第3个碱基依据SNAP引物设计的原则将A错配为G,以保证引物对能够特异地区分2个不同的等位基因。引物序列详见表2。

表2 本试验中PSY2-SNAP引物序列Table 2 Sequence of PSY2-SNAP primer set used in the experiment
1.2.4 PCR扩增和产物多态性检测 通过优化模板DNA浓度和PCR反应退火温度及循环数,确定SNAP引物PCR扩增体系20 μL,包括50 ng·μL-1模板DNA 2 μL,2× PCR master mixture (Vazyme,南京) 10 μL,正向引物0.5 μL,反向引物0.5 μL,ddH2O 7μL;PCR反应条件为:94℃预变性 3 min;94℃变性30 s,61℃退火30 s,72℃延伸1 min,32个循环;72℃终延伸5 min,4℃保存。每个PCR反应重复2次,确保试验结果的一致性和准确性。PCR产物经过1.5%琼脂糖凝胶电泳,并用Tanon4000凝胶成像系统(上海天能公司)进行拍照。
2 结果与分析
2.1 SNAP标记的引物设计及其可靠性评价
在获得PSY2基因全序列并确定变异位点的基础上,依据SNAP技术引物设计的基本原则,设计能够区分变异位点的PCR引物,并依据预试验结果将正向引物3′端倒数第3个碱基A特异地错配为G,对5个薯肉颜色存在差异的木薯品种进行基因型分析。结果表明,SC9、18R和XX048在该位点的等位基因均为杂合型AC,即这3个木薯品种基因组中存在两种不同的等位基因;SC205和KU50在该位点为纯合型CC,在基因组中仅存在一种且变异位点碱基为C的等位基因(图1-a、b)。综上,本研究设计的引物组合通过PCR扩增和产物电泳检测,可明确区分不同薯肉颜色木薯品系的基因型,PCR产物电泳条带特异且清晰,是一个潜在的食用木薯辅助育种分子标记。

注:a:5个木薯品系的薯肉颜色;b:5个木薯品系SNAP 标记展示的基因型;c:3种不同基因型CC/CA/AA 所对应的薯肉颜色。Note: a: The rooTColors of five cassava lines. b: The genotype of five cassava lines by SNAP markers. c: The different rooTColors correspondinGTo CC/CA/AAGenotypes.图1 PSY2-SNAP标记的准确性评价Fig.1 Evaluation on the accuracy of the PSY2-SNAP markers
2.2 薯肉颜色SNAP标记准确性的进一步验证
为了进一步验证本研究开发的PSY2-SNAP标记的可靠性,研究组构建了XX048的自交一代群体,最终获得28个自交株系。经实生苗扩繁成株系后,按照前述方法调查薯肉颜色,发现薯肉颜色存在分离(图1-c),有白色(8株系)、黄色(16株系)和深黄(4株系),经卡方测验发现符合1∶2∶1的遗传分离比例(χ2=1.71<3.84,P<0.05;群体偏小)。为了确定自交系群体薯肉颜色与基因型的对应关系,利用PSY2-SNAP标记对28个株系的基因型进行分析,发现在自交群体中确实存在CC型的纯合位点,而且不同基因型分离比为8CC∶16AC∶4AA(图2),卡方测验结果表明基因型也符合1∶2∶1的遗传分离比,进一步表明薯肉颜色为显性单基因控制。

注:a:引物组合PSY-F1/PSY-R的PCR产物;b:引物组合PSY-F2/PSY-R的PCR产物。M:DL2000;P:木薯品种XX048; 1~7和11:白色薯肉株系CC;8~10和12~14:深黄薯肉株系AA;15~28:黄色薯肉株系AC。Note: a: PCR products with PSY-F1/PSY-R. b: PCR products with PSY-F2/PSY-R. M: DL2000 Marker. P: XX048. 1 to 7 and 11: Lines with white root. 8 to 10 and 12 to 14: Lines with deep-yellow root. 15 to 28: Lines with yellow root.图2 基于PSY2-SNAP标记的XX048及其自交群体的基因型Fig.2 The genotypes of XX048 and its self-bred lines basing on the PSY2-SNAP markers
2.3 薯肉颜色SNAP标记的应用
以62个木薯杂交种实生菌株系为材料验证PSY2-SNAP标记的实用性,首先记录了62个株系的薯肉颜色,然后利用PSY2-SNAP标记对这62个株系的基因型进行分析,结果显示基因型与表型完全匹配(部分结果见图3),表明PSY2-SNAP标记可对育种中间材料的基因型进行准确的检测。

注:a:引物组合PSY-F1/PSY-R的PCR产物;b:引物组合PSY-F2/PSY-R的PCR产物。M:DL2000; 1~2、4~13、15~16、19~20和22~25是白色薯肉株系CC;3、14、18和21是黄色薯肉株系AC。Note: a: PCR products with PSY-F1/PSY-R. b: PCR products with PSY-F2/PSY-R. M: DL2000 Marker. 1~2, 4~13,15~16, 19~20 and 22~25 are lines with white root. 3, 14, 18 and 21 are lines with yellow root.图3 基于PSY2-SNAP标记的部分Ⅴ系列株系基因型Fig.3 The genotypes of Ⅴ-series lines basing on the PSY2-SNAP markers
2.4 黄色薯肉特异种质和新类型的发现
为了在更大范围内检验PSY2-SNAP标记的可靠性,并对现有可食用木薯种质资源进行基因型检测,本试验从国家木薯种质圃抽选出45份木薯品系(表3),利用PSY2-SNAP标记进行基因型分析,并与薯肉颜色进行对应,发现29份薯肉颜色与基因型一致,其中黄色薯肉品系19份,白色薯肉品系10份,其余16份薯肉颜色与基因型不符,薯肉均为黄色(图4,如ZME333),但呈白色薯肉基因型(表3)。
在薯肉颜色与基因型一致的19个黄肉品系中,ZMC723和泰引1号薯肉颜色深黄,但ZMC723基因型为纯合AA,泰引1号基因型为杂合AC(杂合基因型一般是黄色,类似ZMC608,图4),因此泰引1号薯肉颜色为深黄的内在调控机制,和ZMC723的育种利用价值值得进一步挖掘。

图4 4个不同的黄色薯肉品系Fig.4 Four cassava lines with different yellow-roots
3 讨论
3.1 PSY2-SNAP标记是一个具有育种价值的分子标记
SNAP分子标记自研发以来,在多种作物中开展应用。Lestari等[26]基于水稻数据库中的SNP位点成功设计出能够识别水稻籼粳品种的SNAP分子标记,可对水稻材料进行快速选择;Kim等[27]研究发现,大豆野生型与突变型在基因GmNARK的959 bp处存在一个SNP突变,能调控大豆根结瘤的合成,根据这
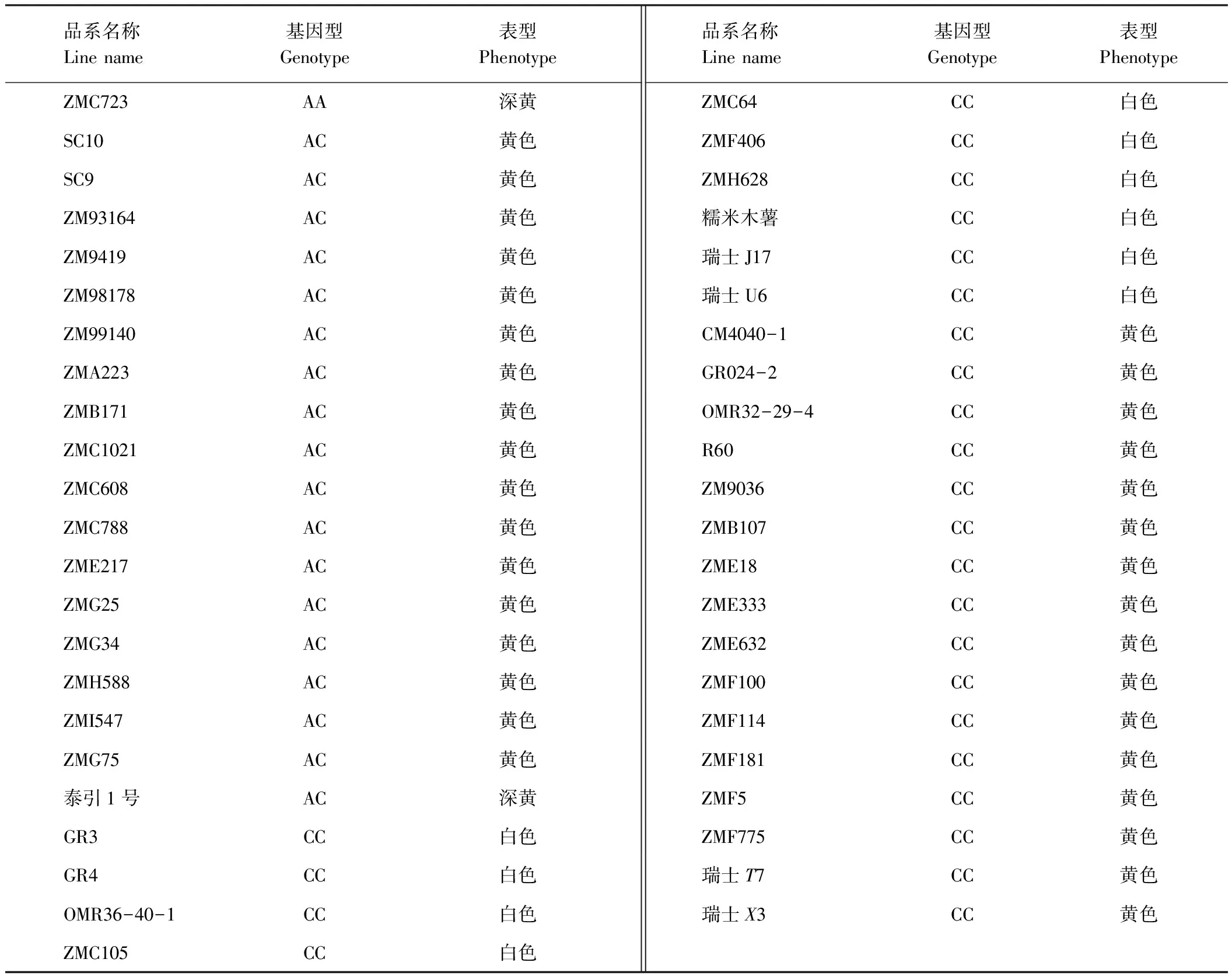
表3 45份可食用木薯品系SNAP标记的基因型和薯肉颜色Table 3 The rooTColors and SNAP based genotypes of 45 edible cassava lines
一位点设计的SNAP分子标记可快速检测出超结瘤的大豆材料。由此可见,在明确SNP位点功能的基础上,设计SNAP分子标记,可以快速准确地区分作物的性状。本研究在前期开发的PSY2-CAPS标记基础上[24],针对其操作繁琐、费用较高的缺点,提出简化方案,开发出仅需进行PCR检测的PSY2-SNAP标记,该标记的准确度与PSY2-CAPS标记相当,且育种中间材料的基因型鉴定结果准确。利用该标记对国家木薯种质圃中的可食用木薯资源进行基因型鉴定,发现了更多由该位点控制的黄色薯肉种质资源,不仅解释了这些种质资源薯肉颜色的成因,也拓宽了今后开展食用木薯育种可选择的亲本范畴,而且杂交子代可以使用PSY2-SNAP标记进行早期选择,提高育种效率。
在作物颜色分子标记的研发中,葛宇等[32]研究发现成熟南瓜黄色果肉受一对完全显性基因控制,黄肉为显性,并获得一个与黄色果肉紧密连锁的SSR标记PU132712,遗传距离为6.72 cM。许芸梅等[33]通过BSA-Seq方法,把调控马铃薯薯肉花青素合成的主效位点定位在第10号染色体上,并利用分子标记将该基因定位于51.47~51.85 Mb的377 kb区间内,包含5个候选基因。上述研究中开发出的颜色相关的分子标记均为区段,未能精准地对作物的颜色性状进行区分。本研究开发的PSY2-SNAP分子标记,可以针对关键突变位点,对木薯黄白薯肉颜色进行准确的区分,具有良好的育种价值。
3.2 木薯块根薯肉颜色变异的分子机理亟待挖掘
木薯块根薯肉颜色除常见的白色外,还有黄色和粉红色等。除PSY2基因编码序列第572个碱基C→A的突变,导致八氢番茄红素合成酶活性增强进而提高块根类胡萝卜素的含量外[13],还有位点PSY2_549和lcyE_1066可能影响类胡萝卜素的含量[14]。本研究在对国家木薯种质圃抽选出的45份木薯品系进行PSY2-SNAP分子标记检测中发现,16个薯肉颜色与基因型不符,其中ZMC723和泰引1号薯肉颜色均为深黄,但ZMC723的基因型为纯合AA,而泰引1号的基因型为杂合AC(其他基因型杂合AC品系薯肉颜色均为黄色),因此深入挖掘泰引1号薯肉颜色深黄的分子机制,可以帮助选育块根类胡萝卜素含量更高的木薯新品系。
利用PSY2-SNAP标记对食用木薯种质资源进行鉴定,发现部分黄色薯肉品系不受C→A突变模式控制,推测为PSY2基因内其他位点突变,或为类胡萝卜素合成途径中其他基因突变导致存在其他的控制位点,为新类型的黄色薯肉种质资源。需要设计试验找到新的提高类胡萝卜素含量的突变位点或基因,并开发成相应的分子标记,从而对多个提高类胡萝卜素含量的位点进行聚合,进一步改良木薯块根类胡萝卜素的合成效率。
4 结论
本研究开发的PSY2-SNAP分子标记可快速鉴定木薯块根肉质颜色,如PSY-F1/PSY-R引物对能扩增出388 bp的条带,则木薯材料为白肉;如PSY-F2/PSY-R引物对能扩增出388 bp的条带,则木薯材料为深黄色;如PSY-F1/PSY-R和PSY-F2/PSY-R均能扩增出388 bp的条带,则木薯材料为黄色。同时,本研究还发现存在受其他基因突变位点控制的黄色薯肉种质资源,有待进一步开发利用。
