非洲猪瘟实时荧光RPA诊断方法建立及应用
2022-02-22张静远缪发明陈腾李敏扈荣良
张静远,缪发明,陈腾,李敏,扈荣良
非洲猪瘟实时荧光RPA诊断方法建立及应用

1宁夏大学生命科学学院,银川 750021;2军事科学院军事医学研究院军事兽医研究所,长春 130122
【】非洲猪瘟(African swine fever,ASF)自2018年首次暴发于我国沈阳后,迅速扩散至全国,对我国养猪业造成了沉重打击。研究针对其病原非洲猪瘟病毒(African swine fever virus,ASFV)建立一种快速核酸检测技术,为及时发现、准确处理ASF疫情提供一种快速诊断方法。针对ASFV保守的B646L(p72)基因,设计并筛选合适的引物、探针组合,利用基于重组酶-聚合酶扩增(recombinase polymerase amplification,RPA)的实时荧光RPA方法,对反应体系、反应条件、样品处理步骤等进行优化;利用质控品,对检测方法的特异性和灵敏度进行评价。对1 009份临床样品进行检测,并利用OIE推荐的实时荧光定量PCR方法(qPCR)和病毒分离,进一步验证该方法的检测结果。筛选得到一套合适的引物、探针,建立了针对ASFV p72基因的实时荧光RPA检测方法,优化后的反应体系总体积为25 µL,在实时荧光定量PCR仪器上,最适反应条件为39℃ 10 s,39℃ 20 s,40个循环,扩增反应时间约20 min;室温裂解法可取代传统核酸提取方法作为本检测的样品处理方法;使用优化后的条件,可在30 min内实现样品处理、核酸扩增和结果判定的整个过程,特异性评价结果显示本方法对猪细小病毒(PPV)、伪狂犬病毒(PRV)、圆环病毒1型/2型(PCV1/2)、猪瘟病毒(CSFV)、猪繁殖与呼吸综合征病毒(PRRSV)样品扩增结果均为阴性;敏感性评价结果显示本方法对基因I型、II型、IX型ASFV的模拟样品均可检出,对II型ASFV阳性模拟血样品可检测至10 拷贝/µL,对已知阳性临床组织样品可检至1﹕103.0稀释度,与OIE推荐的实时荧光定量PCR(qPCR)方法的检测灵敏度一致。通过对1 009份临床样品进行检测,实时荧光RPA诊断方法检出17份阳性,其结果与qPCR方法完全一致;病毒分离获得17份阳性培养物。建立的实时荧光RPA核酸检测方法操作简便,耗时短,具有较高的灵敏度和特异性,为临床提供了一种新型、简单、特异、快速诊断非洲猪瘟的方法。
非洲猪瘟病毒;重组酶聚合酶扩增; 实时荧光RPA;核酸检测;诊断技术
0 引言
【研究意义】非洲猪瘟(African swine fever,ASF)是由非洲猪瘟病毒(African swine fever virus,ASFV)引起的猪的一种高度接触性急性出血性传染病[1],被OIE规定为需要通报的动物疫病,我国明确该病为一类动物传染病。ASFV是非洲猪瘟病毒科(asfarviridae)非洲猪瘟病毒属(asfivirus)的唯一成员[2],是一种大的DNA病毒,其基因组全长170—190 kb,含160—175个ORFs。根据p72(B646L)基因序列的差异,可将ASFV毒株划分为I—XXIV个基因型[3-4]。其中基因II型自2007年从东非地区传入格鲁吉亚的高加索地区后,广泛传播至东欧、西亚以及包括俄罗斯联邦在内的邻国[5-6],2018年8月确认传入中国[7-8],后亚洲其他养猪国家也相继报告疫情[9-10]。由于尚无有效疫苗进行预防,针对非洲猪瘟疫情只能通过确诊后采取扑杀和净化的措施。虽然目前疫情态势有所放缓,但病毒已在全国形成较大污染面。为尽可能有效降低该病对我国养猪业、国民经济和国际贸易的影响和损失,快速准确的诊断仍是有效控制本病扩散的必要手段。【前人研究进展】传统的非洲猪瘟病原学诊断方法有红细胞吸附试验[11]、免疫组化[12]、酶联免疫吸附试验(ELISA)[13]等,但这些方法耗时较久,操作复杂。随着分子生物学技术的发展,核酸检测技术逐渐建立:早在1995年,孙书华等[14]建立了针对ASFV的PCR方法,而LUO等[15]2017年建立的PCR方法检测敏感性达60拷贝/反应;KING等[16-17]报道的针对ASFV p72基因建立的实时荧光定量PCR(qPCR)检测敏感性达10—100拷贝/反应;JAMES等[18]报道的环介导等温扩增技术(LAMP)操作流程较PCR更为简便,检测敏感度为330拷贝,最近WANG等[19]针对ASFV p10基因建立的LAMP可检测至30拷贝/µL的质粒,并适用于多个基因型。相比传统方法,核酸检测方法不再需要原代细胞、单克隆抗体等昂贵的材料,且具有较高的检测敏感性,但这些常用的核酸扩增方法在实际应用中也存在一定缺陷,如样品处理过程复杂,常需对核酸进行多步纯化提取;需要较高反应温度,常在60℃以上;同时整个检测过程耗时较长,急需更为便捷的替代方式。【本研究切入点】针对II型ASFV的保守的B646L(p72)基因,利用重组酶聚合酶扩增(recombinase polymerase amplification,RPA)技术,建立了一种实时荧光RPA(real-time RPA)核酸检测方法;结合实际需要,针对RPA反应体系和条件及核酸提取步骤进行了优化,简化操作程序,为临床非洲猪瘟检测提供便捷诊断工具。【拟解决的关键问题】筛选合适的引物探针,优化反应条件,简化样品处理过程,为非洲猪瘟临床诊断提供一种简单、特异、快速的方法。
1 材料与方法
试验于2018年9月至2019年10月于军事科学院军事医学研究院军事兽医研究所进行。
1.1 引物和探针
RPA引物和探针为军事科学院军事医学研究院军事兽医研究所实验室设计和筛选;PPV、PRV、PCV1/2、CSFV、PRRSV引物参考国家标准[20-24];ASFV实时荧光定量PCR(即qPCR)引物参考OIE推荐[16],序列为OIE-F:5′-CTGCTCATGGTATCAAT CTTATC GA-3′,OIE-R:5′-GATACCACAAGATCAGC CGT-3′,OIE-P:5′-FAM-CCACGGGAGGAATACCAACCCAGTG-TAMRA-3′;所有引物探针均由上海生工生物工程股份有限公司合成。
1.2 质粒与样品
1.2.1 pMD19-ASFV-P72质粒是以pMD19T为载体,携带有ASFV SY18株B646L(p72)全基因的阳性质粒,为军事科学院军事医学研究院军事兽医研究所实验室构建并保存。pUC-P72-I、pUC-P72-IX分别为携带基因I型和基因IX型ASFV P72基因的阳性质粒,由上海生工生物工程股份有限公司合成。
1.2.2 阴性抗凝血采集自健康猪,按照OIE推荐方法和国标检测方法检测证明其不含ASFV、PPV、PRV、PCV1/2、CSFV、PRRSV。
1.2.3 ASFV临床样品为军事科学院军事医学研究院军事兽医研究所实验室自2018年8月以来收集并保存的病料,共1 009份。主要为来源于猪场的临床样品及疫苗研究攻毒试验中的组织样品,按照国家要求处理后冷冻保存。
1.2.4 特异性样品:PPV、PRV、PCV1/2、CSFV、PRRSV样品分别为军事科学院军事医学研究院军事兽医研究所实验室分离保存的病毒株或购买的市售活疫苗。
1.2.5 敏感性样品:M1为含pUC-P72-I质粒的抗凝血,浓度为1×105拷贝/μL;M2为含pUC-P72-IX质粒的抗凝血,浓度为1×105拷贝/μL;M3—M8为含人为加入的不同浓度(10倍梯度)pMD19-ASFV-P72质粒的抗凝血,质粒浓度分别为M3:1×105拷贝/μL至M8:1×100拷贝/μL。
1.3 试剂与仪器
RPA TwistAmp® exo试剂盒购自TwistDx,核酸提取试剂盒(离心柱法)购自Axygen,DNA纯化试剂盒购自碧云天,磁珠法DNA提取试剂盒购自天津美菱生物,Lysis1(主成分为NaOH溶液)、Lysis2(主成分为Tris-HCl溶液)及FITC的p30单抗为实验室制备。Eco实时荧光定量PCR仪器,购自Illumina。
1.4 引物探针的筛选
根据p72基因序列设计筛选3套引物-探针(分别命名为Pa,Pb,Pc),使用TwistAmp® exo试剂盒,根据表1配制实时荧光RPA(下称实时RPA)反应体系,将1—5号成分预先混合后,再加入冻干管中,充分溶解后等分至两个反应管,再依次加入1 μL作为模板的阳性质粒或4d水,1.25 μL MgOAc。最终反应体积为25 μL。设置荧光定量PCR仪的程序。步骤1:39℃ 10 s,步骤2:39℃ 20 s,并在步骤2收集荧光信号;如此反应50个循环(约25 min)后,根据结果筛选最佳引物-探针组合。

表1 实时RPA反应体系
1.5 反应条件优化
1.5.1 反应时间和温度优化 将pMD19-ASFV-P72质粒稀释至浓度为1×106拷贝/μL、1×103拷贝/μL,作为强阳性、弱阳性对照样品。分别在33℃、36℃、39℃、42℃恒温条件下,用荧光定量PCR仪对阴性(4d水)、强阳性、弱阳性对照进行扩增,反应程序设置同1.4,50个循环(约25 min),观察扩增曲线。同时以5、10、15、20、25 min为节点,观察扩增曲线。
1.5.2 反应体系优化 为尽可能减少操作步骤,提高操作便捷性,对反应体系进行调整,比较两种反应体系的检测效果。①体系A:配制与操作同1.4;②体系B(表2):将工作浓度的引物F、引物R、探针P及Rehydration buffer、4d水按照2.1﹕2.1﹕0.6﹕29.5﹕5.7的比例混合,配制成Rehydration Mix;同时将浓度为280 mmol·L-1的MgOAc与水按照2.5꞉5.5的比例配制成稀释后的MgOAc-B。检测时向每个RPA冻干管加入40 μL Rehydration Mix,充分溶解混匀后分装至2个反应管,每管20 μL,即为1个反应体系。向反应管中依次加入1 μL待检模板,4 μL MgOAc-B。放入荧光定量PCR仪中,程序设置同体系A。③使用体系A、B检测阴性、强阳性、弱阳性对照(同1.5.1)。比较检测结果。

表2 实时RPA反应优化体系
1.6 核酸提取方法优化
将pMD19-ASFV-P72质粒加入阴性抗凝血中,制备成含1×106拷贝/μL、1×103拷贝/μL的模拟强阳性、弱阳性临床样品,同时设阴性抗凝血为阴性临床样品。裂解法和煮沸法均为基于传统DNA提取法改良的适用于多类型样品的核酸提取方法,其操作步骤如下:裂解法—将待检样品与Lysis 1等体积混匀后,室温放置10 min,离心后的上清即为粗提核酸;煮沸法—将待检样品与Lysis 2等体积混合,煮沸5 min后4℃放置5 min,离心后的上清即为粗提核酸。分别以柱提取法、磁珠法、裂解法、煮沸法,对模拟的阴性、强阳性、弱阳性临床样品进行核酸提取,同时设不处理原液对照,按优化后的反应体系和条件进行实时RPA反应。比较检测结果。
1.7 实时荧光RPA诊断方法的特异性
使用优化后的实时RPA体系和反应条件,对特异性样品进行检测。同时与OIE推荐的qPCR方法进行比较。
1.8 实时荧光RPA诊断方法的敏感性和灵敏度
使用优化后的实时RPA体系和反应条件,对敏感性样品M1—M8进行检测,同时与OIE推荐的qPCR方法进行比较。此外,取已知ASFV阳性的脾脏、淋巴结、血清样品,分别进行10、100、1 000、10 000倍稀释后,使用实时RPA和qPCR方法进行检测。通过以上两种指标,评价实时RPA检测方法的对不同基因型ASFV的检测敏感性及检测灵敏度。
1.9 实时荧光RPA诊断方法对临床样品的检测
使用优化后的实时RPA体系和反应条件,针对实验室采集的来源于猪场的临床样品和疫苗研究攻毒试验中的组织样品共1 009份分别进行实时RPA检测,并与实时荧光定量PCR结果比较。利用病毒分离方法对检出的阳性样品进行进一步验证:将研磨后的阳性样品接种单层原代肺泡巨噬细胞(PAM),37℃孵育1 h后换液,继续培养3 d后,观察细胞病变。同时使用FITC标记的p30单抗进行免疫荧光(IFA),根据荧光的有无判定结果。
2 结果
2.1 引物探针的筛选结果
如图1所示,3组引物探针对目的样品扩增后,阳性样品均出现扩增曲线,阴性样品无扩增。但3组引物探针中,Pa组强阳性和弱阳性样品均起峰早,荧光信号强,故选择Pa组为最佳引物探针组合。确定的引物探针序列及相应标记如表3。

Pa:组合a;Pb:组合b;Pc:组合c;1:强阳性对照;2:弱阳性对照;3:空白对照
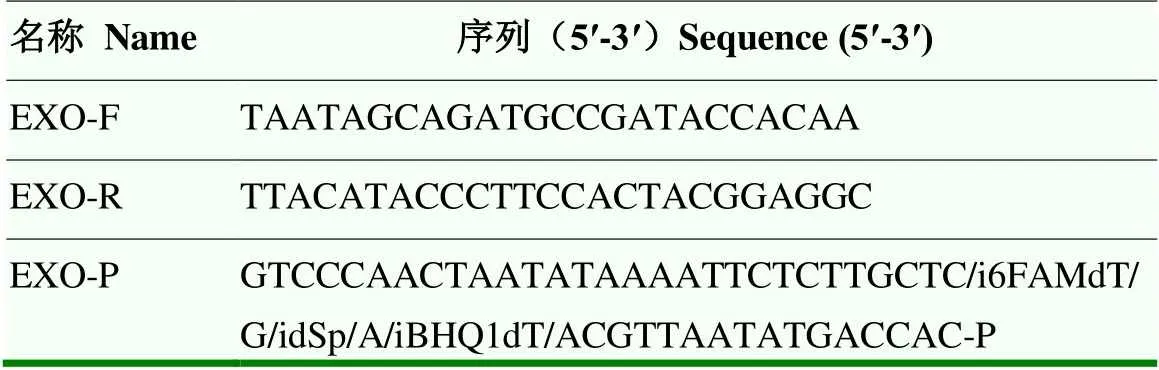
表3 实时RPA引物探针序列
2.2 反应条件优化
2.2.1 反应温度与时间的优化 结果如图2所示,在33℃、36℃、39℃、42℃条件下,阴性对照样品均未出现扩增,强阳性对照样品均可在10 min内(Ct值<20)呈现典型扩增曲线。而对于拷贝数较低的弱阳性样品,在39℃、42℃反应时,其扩增曲线起峰较早,当反应温度降低,在36℃或33℃条件下,弱阳性对照样品至反应15 min后才出现扩增曲线(Ct值30—45),同时荧光信号较低。以上结果显示在39℃、42℃条件下,实时RPA的扩增效率较高。结合试剂盒关键酶的最佳活性温度,考虑实际操作的可行性,选择39℃为最佳反应温度。而就反应时间来看,39℃条件下,弱阳性样品在反应15 min内(约30循环)即出现明显扩增,说明本实时RPA在15 min内即可检测至103拷贝/µL的样品。为保证后续试验的灵敏度和可重复性,最终选择40循环、20 min为实时RPA的反应时间。
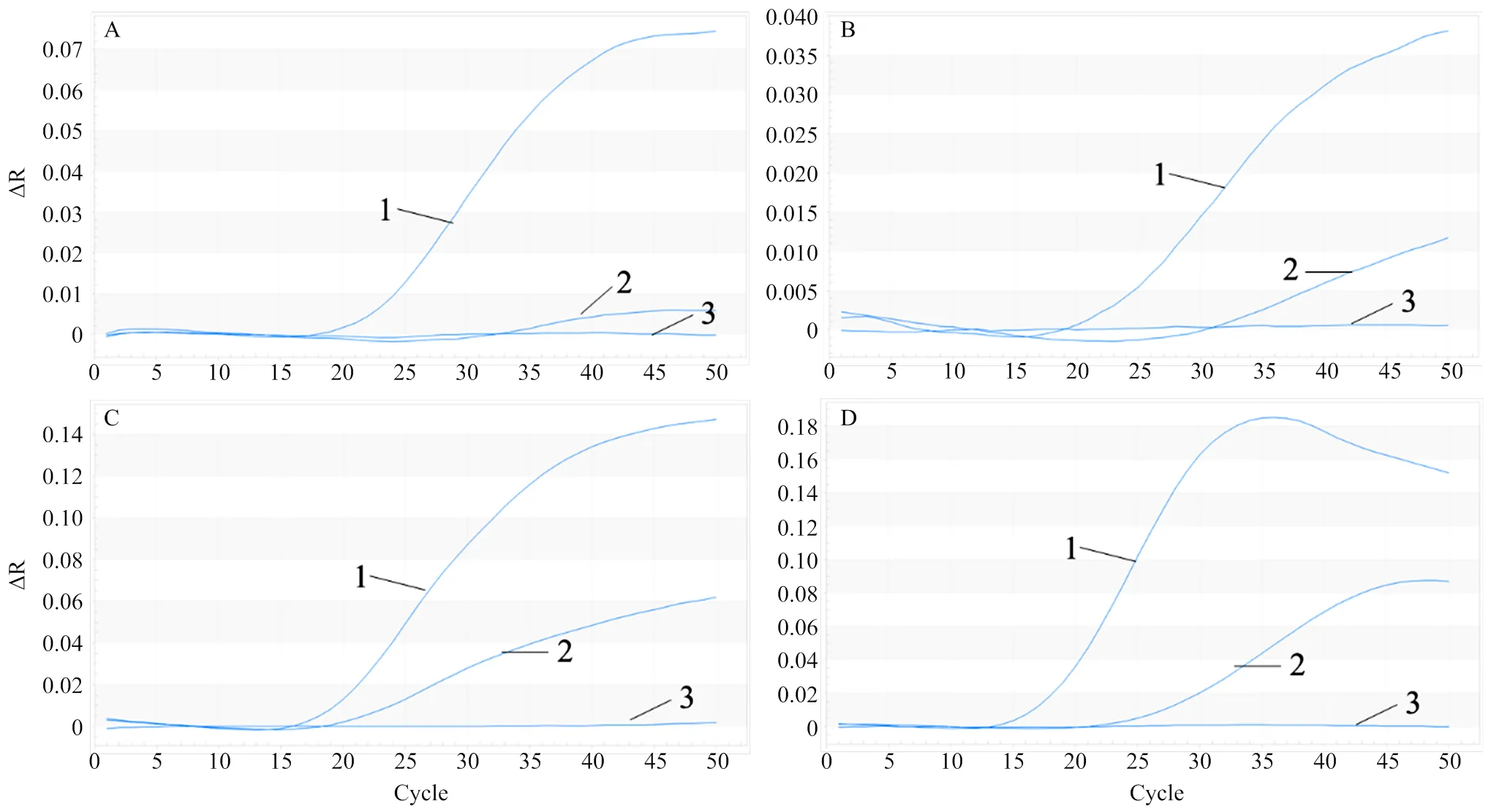
A 33℃;B 36℃;C 39℃;D 42℃
2.2.2 反应体系优化 优化前的反应体系A与使用了Rehydration mix的体系B相比,在检测强阳性、弱阳性对照样品均出现典型扩增曲线,而阴性对照无扩增曲线,说明两种反应体系对本试验中的阳性样品均可检出(图3)。考虑实际应用的便捷性及经济性原则,最终选择反应体系B作为本实时RPA试剂盒的反应体系。
2.3 核酸提取方法的优化
模拟的临床血样品经不同核酸提取方法处理后,进行实时RPA扩增(表4),结果显示,强阳性血样经不同方法处理或不处理时,均可在10 min内出现扩增曲线(Ct值<20),阴性样品均无扩增(无Ct值);但是对弱阳性样品,在不作处理时起峰较晚,在反应15 min后才出现扩增曲线;而其他4种方法处理后的性样品均能在15 min内出现扩增信号(Ct值<30)。以上结果说明,虽然在不处理的条件下RPA也可实现对弱阳性样品的扩增,但血液成分一定程度影响了扩增反应的进行。由表4可见,在几种可选的提取方法中,裂解法仅需加入一种裂解液,并在室温条件即可进行,成分单一,步骤简单,减少了操作中污染的风险,节省操作时间,最终选择该方法作为实际应用的核酸提取方法。
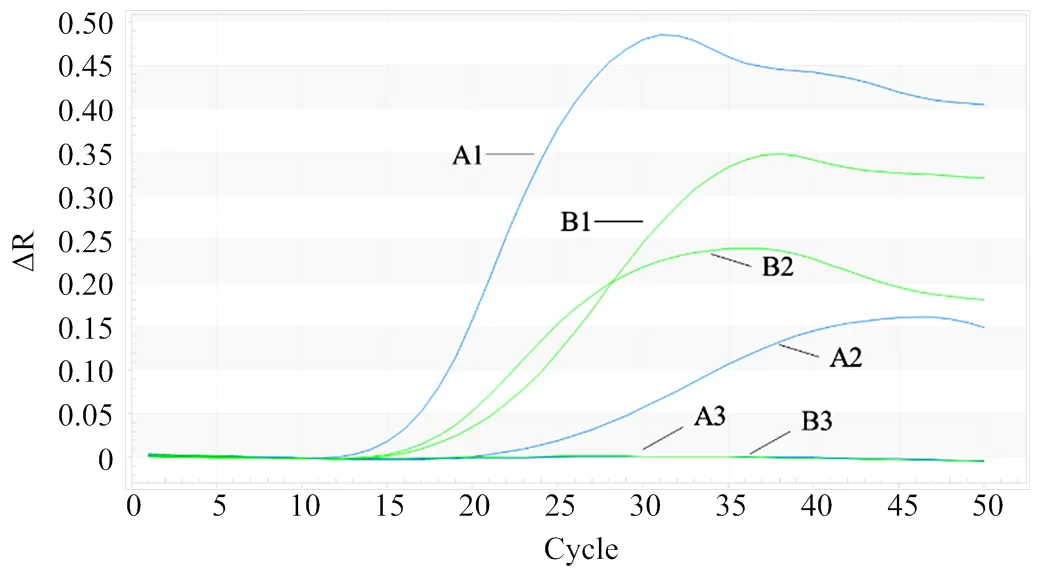
A:优化前的反应体系The amplification system before optimization;B:优化后的反应体系The amplification system after optimization
2.4 实时荧光RPA诊断方法的特异性
使用优化后的反应体系和反应条件,对建立的实时RPA检测方法进行特异性评价,结果显示,本实时RPA对PPV、PRV、PCV1/2、CSFV、PRRSV样品检测均为阴性;同时阴阳性对照成立。其结果与qPCR结果一致(图4)。说明本方法特异性良好。

表4 不同样品处理方法比较
RT:室温Room temperature;N:阴性对照Negative Control;WP:弱阳性对照Weak positive control;SP:强阳性对照Strong positive control; /:无Ct值 No Ct value

A实时RPA对特异性样品检测结果Results of specific samples tested by real-time RPA;B qPCR对特异性样品检测结果Results of specific samples tested by qPCR。1:阳性对照 Positive Control;2:阴性对照 Negative Control;3:PPV;4:PRV;5:PCV1/2;6:CSFV;7:PRRSV
2.5 实时荧光RPA诊断方法的敏感性及灵敏度
使用实时RPA检测方法分别对敏感性样品进行检测,结果显示,阴、阳性对照成立,基因I型、基因IX型样品M1、M2均呈阳性;在含不同浓度的ASFV基因II型质粒的样品中,除了约含1拷贝/μL的样品呈现阴性外,10拷贝/μL以上浓度的样品检测结果均为阳性。说明实时RPA对基因I型、II型、IX 型的阳性样品均可检出,最低检测限为10拷贝/μL,同qPCR方法的灵敏度一致(图5)。而两种方法对不同稀释的脾、淋巴结、血清等临床阳性样品均可检测至1﹕103.0稀释度(表5)。
2.6 实时荧光RPA诊断方法对临床样品的检测
分别使用本研究建立的实时RPA方法与OIE推荐的qPCR方法对本研究室收集和保存的1 009份临床样品进行检测,结果显示,实时RPA方法共检测出17份阳性样品,992份阴性样品,与qPCR结果完全一致,符合率100%。将检测为阳性的样品接种PAM细胞,72 h后均观察到典型的细胞病变,感染细胞折光率降低,少数细胞出现胞内空泡,体积增大,培养基pH下降。以FITC标记的p30蛋白单抗做IFA,感染细胞出现特异性荧光,而对照正常细胞未观察到如上现象(图6)。17份阳性样品的检测结果见表6,两种方法的操作流程比较如表7。
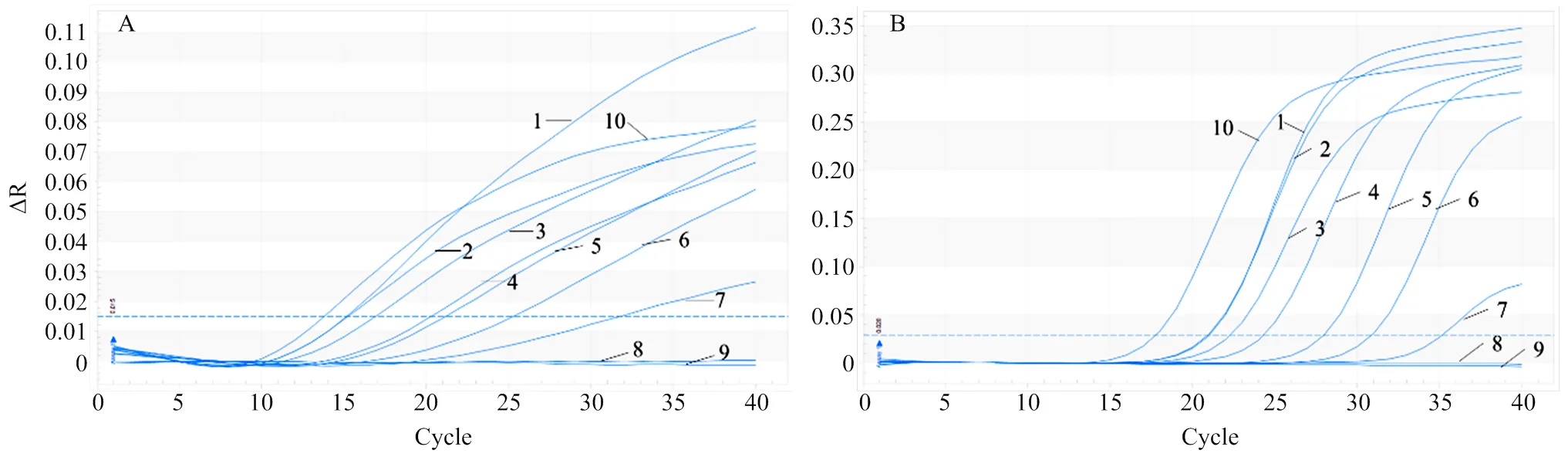
A:实时RPA对敏感性样品检测结果Results for sensitivity samples tested by real-time RPA;B:qPCR对敏感性样品检测结果Results for sensitivity samples tested by qPCR。1:阳性对照Positive Control;2:阴性对照 Negative Control;3—10:M1—M8

表5 实时RPA和qPCR对不同稀释的临床样品检测
“/”:无Ct值 No Ct value;“+”:阳性 Positive;“-”:阴性 Negative
3 讨论
非洲猪瘟自1921年首次发现于非洲以来,已在非洲、欧洲、南美洲、亚洲、加勒比海地区等数十个国家和地区发生过流行,部分国家和地区通过扑杀和净化的方式,已经完成对该病的消除[25]。2018年8月,我国确认了首例家猪非洲猪瘟疫情。由于非洲猪瘟临床症状与经典猪瘟等其他出血性疾病症状和病变极为相似,急性型、亚急性型和慢性型在临床中较难区分,因此,建立及时、快速、准确的诊断方法是精准清除非洲猪瘟必不可少的手段。

A:正常PAMs Normal PAMs;B:出现病变的PAMs Infected PAMs with CPE;C:正常PAMs免疫荧光IFA of normal PAMs;D:感染PAMs免疫荧光 IFA of infected PAMs

表6 阳性临床样品检测结果汇总
“+”:阳性 Positive;“-”:阴性 Negative

表7 实时RPA和实时PCR操作流程比较
RPA技术是由英国公司TwistDx Inc开发的一种可以替代传统PCR的新型核酸检测技术。该技术主要依赖重组酶、单链结合蛋白(SSB)和链置换DNA聚合酶[26];将exo探针技术与核酸外切酶III结合起来,即可实现类似荧光定量PCR仪的实时读取,同时对仪器的要求更低,可实现在37—42℃条件下等温快速扩增,目前也已应用于多个领域[27-28]。RPA分析的关键在于扩增引物和探针设计。一般认为,RPA引物应比一般PCR引物长,达30—38个碱基, RPA引物过短会降低重组率,影响扩增速度和检测灵敏度,同时需要多次条件摸索进行优化[29-30]。但在本研究引物筛选时发现,RPA扩增效率与多种因素有关,引物过长更易导致非特异性增加,缩短引物长度可在一定程度减少非特异反应的产生,从而保证检测的特异性。目前已有将较短的引物应用于RPA技术的报道[31]。最终,本研究通过多组引物的筛选和测定,明确了一组较优引物对和探针,具有较好的扩增效果。
在针对临床样品检测时,待检样品除血清、咽拭子等成分较为简单的体液以外,还常见组织、全血液、饲料等样品。当进行传统PCR、荧光定量PCR时,样品中的杂蛋白可显著抑制PCR反应活性,因此通常需对样品中的核酸进行提取纯化后再作为扩增模板。但常规的柱提取法提取核酸过程成本高,耗时久,同时也增加了环境中或其他样品交叉污染的风险,易造成假阳性结果。磁珠法相对于层析柱提取法更为便捷,不依赖离心机,但同样需要吸附、淋洗和洗脱等多步操作。RPA技术是基于常温扩增的一种技术,其反应中的关键酶对反应微环境的耐受性较高[29],同时,尽可能去除样品中的杂蛋白无疑可提高扩增反应的灵敏性和特异性。因此,尝试对样品进行不处理或粗处理的方法,以期在保证准确性的前提下,减少操作步骤,缩短操作时间。结果证实,采用本课题组摸索的室温裂解法提取后的产物可直接用于RPA扩增,该方法不仅大大降低了样品处理的时间和成本,也提高了检测的敏感性和特异性。
前期报道了针对p72基因建立的RPA-LFD检测方法[32],该方法可在10 min实现对核酸的扩增,不需要特殊的仪器,仅需水浴条件即可完成,适合于临床现场快速检测。但该方法在结果读取时需打开扩增管,将产物滴加到试纸条上,这一步骤容易产生气溶胶,增加了扩增产物污染的风险,因此其应用范围仅限于新发病猪场,在范围上受到一定限制。本研究建立的实时荧光RPA检测方法,对实验仪器的兼容性强,既可采用配套的便携式恒温实时荧光检测仪,也可采用目前基层实验室或中大型养殖场普遍采用的实时荧光定量PCR仪,使用者可根据自身条件选择合适的仪器。同时,本方法给出了可在实时荧光定量PCR仪上进行实时荧光RPA扩增操作程序和判定标准,可固定条件,作为ASF临床诊断的优先选择方法。
4 结论
建立了一种改进的实时荧光RPA检测方法,可在30 min内实现核酸提取、核酸扩增和结果判定,检测灵敏度达10拷贝/μL,对临床样品的检测证实与OIE推荐的实时荧光定量方法、病毒分离具有完全的一致性。该方法操作便捷、耗时短、对仪器设备兼容性强,为ASF临床快速诊断提供了一种新的实用方法。
[1] DIXON L K, STAHL K, JORI F, VIAL L, PFEIFFER D U. African swine fever epidemiology and control. Annual Review of Animal Biosciences, 2020, 8: 221-246. doi:10.1146/annurev-animal-021419- 083741.
[2] DIXON L, SUN H, ROBERTS H. African swine fever. Antiviral Research, 2019, 165:34-41. doi:10.1016/j.antiviral.2019.02.018.
[3] QUEMBO C J, JORI F, VOSLOO W, HEATH L. Genetic characterization of African swine fever virus isolates from soft ticks at the wildlife/domestic interface in Mozambique and identification of a novel genotype. Transboundary and Emerging Diseases, 2018, 65(2): 420-431. doi:10.1111/tbed.12700.
[4] BASTOS A D S, PENRITH M L, CRUCIÈRE C, EDRICH J L, HUTCHINGS G, ROGER F, COUACY-HYMANN E, R THOMSON G. Genotyping field strains of African swine fever virus by partial p72 gene characterisation. Archives of Virology, 2003, 148(4): 693-706. doi:10.1007/s00705-002-0946-8.
[5] GARIGLIANY M, DESMECHT D, TIGNON M, CASSART D, LESENFANT C, PATERNOSTRE J, VOLPE R, CAY A B, VAN DEN BERG T, LINDEN A. Phylogeographic analysis of African swine fever virus, western Europe, 2018. Emerging Infectious Diseases, 2019, 25(1): 184-186. doi:10.3201/eid2501.181535.
[6] PEJSAK Z, TRUSZCZYŃSKI M, NIEMCZUK K, KOZAK E, MARKOWSKA-DANIEL I. Epidemiology of African Swine Fever in Poland since the detection of the first case. Polish Journal of Veterinary Sciences, 2014, 17(4): 665-672. DOI:10.2478/pjvs-2014- 0097.
[7] ZHOU X, LI N, LUO Y, LIU Y, MIAO F, CHEN T, ZHANG S, CAO P, LI X, TIAN K, QIU H J, HU R. Emergence of African swine fever in China, 2018. Transboundary and Emerging Diseases, 2018, 65(6): 1482-1484. doi:10.1111/tbed.12989.
[8] GE S, LI J, FAN X, LIU F, LI L, WANG Q, REN W, BAO J, LIU C, WANG H, LIU Y, ZHANG Y, XU T, WU X, WANG Z. Molecular characterization of African swine fever virus, China, 2018. Emerging Infectious Diseases, 2018, 24(11): 2131-2133. doi:10.3201/eid2411. 181274.
[9] LE V P, JEONG D G, YOON S W, KWON H M, TRINH T B N, NGUYEN T L, BUI T T N, OH J, KIM J B, CHEONG K M, VAN TUYEN N, BAE E, VU T T H, YEOM M, NA W, SONG D. Outbreak of African swine fever, Vietnam, 2019. Emerging Infectious Diseases, 2019, 25(7): 1433-1435. doi:10.3201/eid2507.190303.
[10] KIM H J, CHO K H, LEE S K, KIM D Y, NAH J J, KIM H J, KIM H J, HWANG J Y, SOHN H J, CHOI J G, KANG H E, KIM Y J. Outbreak of African swine fever in South Korea, 2019. Transboundary and Emerging Diseases, 2020, 67(2): 473-475. doi:10.1111/tbed. 13483.
[11] ENJUANES L, CARRASCOSA A, MORENO M, VIñUELA E. Titration of African swine fever (ASF) virus. The Journal of General virology, 1976, 32(3): 471-477.DOI:10.1099/0022-1317-32-3-471.
[12] PéREZ J, RODRíGUEZ F, FERNáNDEZ A, MARTíN DE LAS MULAS J, GóMEZ-VILLAMANDOS J, SIERRA M. Detection of African swine fever virus protein VP73 in tissues of experimentally and naturally infected pigs. Journal of Veterinary Diagnostic Investigation: Official Publication of the American Association of Veterinary Laboratory Diagnosticians, Inc, 1994, 6(3): 360-365. doi:10.1177/104063879400600314.
[13] VIDAL M I, STIENE M, HENKEL J, BILITEWSKI U, COSTA J V, OLIVA A G. A solid-phase enzyme linked immunosorbent assay using monoclonal antibodies, for the detection of African swine fever virus antigens and antibodies. Journal of Virological Methods, 1997, 66(2): 211-218. doi:10.1016/s0166-0934(97)00059-1.
[14] 孙书华, 孙淑芳, 蒋正军, 吴时友.多聚酶链反应技术检测非洲猪瘟. 中国兽医科技, 1995, 11(25): 29. doi: 10.16656/j.issn.1673- 4696.1995.11.016.
SUN S H, SUN S F, JIANG Z J, WU S Y.Detection of African swine fever by polymerase chain reaction. Chinese Journal of Veterinary Science and Technology, 1995, 11(25): 29. doi: 10.16656/j.issn. 1673-4696.1995.11.016. (in Chinese)
[15] LUO Y Z, ATIM S A, SHAO L N, AYEBAZIBWE C, SUN Y, LIU Y, JI S W, MENG X Y, LI S, LI Y F, MASEMBE C, STÅHL K, WIDÉN F, LIU L H, QIU H J. Development of an updated PCR assay for detection of African swine fever virus. Archives of Virology, 2017, 162(1): 191-199. doi:10.1007/s00705-016-3069-3.
[16] KING D P, REID S M, HUTCHINGS G H, GRIERSON S S, WILKINSON P J, DIXON L K, BASTOS A D, DREW T W. Development of a TaqMan PCR assay with internal amplification control for the detection of African swine fever virus. Journal of Virological Methods, 2003, 107(1): 53-61. doi:10.1016/s0166-0934 (02)00189-1.
[17] FERNÁNDEZ-PINERO J, GALLARDO C, ELIZALDE M, ROBLES A, GÓMEZ C, BISHOP R, HEATH L, COUACY-HYMANN E, FASINA F O, PELAYO V, SOLER A, ARIAS M. Molecular diagnosis of African Swine Fever by a new real-time PCR using universal probe library. Veterinary Medicine and Science, 2013, 60(1): 48-58. doi:10.1111/j.1865-1682.2012.01317.x.
[18] JAMES H E, EBERT K, MCGONIGLE R, REID S M, BOONHAM N, TOMLINSON J A, HUTCHINGS G H, DENYER M, OURA C A, DUKES J P, KING D P. Detection of African swine fever virus by loop-mediated isothermal amplification. Journal of Virological Methods, 2010, 164(1/2): 68-74. doi:10.1016/j.jviromet.2009.11.034.
[19] WANG D, YU J, WANG Y, ZHANG M, LI P, LIU M, LIU Y. Development of a real-time loop-mediated isothermal amplification (LAMP) assay and visual LAMP assay for detection of African swine fever virus (ASFV). Journal of Virological Methods, 2020, 276: 13775. DOI:10.1016/j.jviromet.2019.113775
[20] 全国动物卫生标准化技术委员会. 2019, 伪狂犬病诊断方法GB/T 18641-2018.
National Technical Standardization Committee of Animal Health. 2019, Diagnostic method for pseudorabies GB/T 18641-2018. (in Chinese)
[21] 全国动物防疫标准化技术委员会.2011, 猪瘟病毒实时荧光RT-PCR检测方法GB/T 27540-2011.
National Technical Standardization Committee of Animal Epidemic Prevention. 2011, Method of the real-time QPCR for the detection of classical swine fever virus GB/T 27540-2011. (in Chinese)
[22] 全国动物防疫标准化技术委员会.2008, 猪圆环病毒聚合酶链反应试验方法GB/T 21674-2008.
National Technical Standardization Committee of Animal Epidemic Prevention. 2008, Detecting porcine circovirus with polymerase chain reaction GB/T 21674-2008. (in Chinese)
[23] 全国动物防疫标准化技术委员会.2011, 鉴别猪繁殖与呼吸综合征病毒高致病性与经典毒株复合RT-PCR方法 GB/T 27517-2011 .
National Technical Standardization Committee of Animal Epidemic Prevention. 2011, A multiplex QPCR method to differentiate the highly pathogenic and classical porcine reproductive and respiratory syndrome virus GB/T 27517-2011. (in Chinese)
[24] 国家认证认可监督委员会.2007, 猪细小病毒病聚合酶链反应操作规程SN/T 1874-2007.
Certification and Accreditation Administration of the P.R.C. 2007, Protocol of polymerase chain reaction for porcine parvovirus SN/T 1874-2007. (in Chinese)
[25] COSTARD S, WIELAND B, DE GLANVILLE W, JORI F, ROWLANDS R, VOSLOO W, ROGER F, PFEIFFER D, DIXON L. African swine fever: How can global spread be prevented?Philosophical Transactions of the Royal Society of London Series B, Biological Sciences, 2009, 364(1530): 2683-2696.DOI:10.1098/rstb. 2009.0098.
[26] PIEPENBURG O, WILLIAMS C H, STEMPLE D L, ARMES N A. DNA detection using recombination proteins. PLoS Biology, 2006, 4(7): e204. doi:10.1371/journal.pbio.0040204.
[27] CHAO C C, BELINSKAYA T, ZHANG Z, CHING W M. Development of recombinase polymerase amplification assays for detection ofor. PLoS Neglected Tropical Diseases, 2015, 9(7): e0003884. doi:10.1371/ journal.pntd.0003884.
[28] LI Y, LI L, FAN X, ZOU Y, ZHANG Y, WANG Q, SUN C, PAN S, WU X, WANG Z. Development of real-time reverse transcription recombinase polymerase amplification (RPA) for rapid detection of peste des petits ruminants virus in clinical samples and its comparison with real-time PCR test. Scientific Reports, 2018, 8(1): 17760. doi:10.1038/s41598-018-35636-5.
[29] JAMES A, MACDONALD J. Recombinase polymerase amplification: Emergence as a critical molecular technology for rapid, low-resource diagnostics. Expert Review of Molecular Diagnostics, 2015, 15(11): 1475-1489. doi:10.1586/14737159.2015.1090877.
[30] LIU X Q, YAN Q Y, HUANG J F, CHEN J, GUO Z Y, LIU Z D, CAI L, LI R S, WANG Y, YANG G W, LAN Q X. Influence of design probe and sequence mismatches on the efficiency of fluorescent RPA. World Journal of Microbiology and Biotechnology, 2019, 35(6): 95. doi:10.1007/s11274-019-2620-2.
[31] WU L, YE L, WANG Z, CUI Y, WANG J. Utilization of recombinase polymerase amplification combined with a lateral flow strip for detection ofbeihaiensis in the oyster.Parasites & Vectors, 2019, 12(1): 360. doi:10.1186/s13071-019-3624-3.
[32] MIAO F, ZHANG J, LI N, CHEN T, WANG L, ZHANG F, MI L, ZHANG J, WANG S, WANG Y, ZHOU X, ZHANG Y, LI M, ZHANG S, HU R. Rapid and sensitive recombinase polymerase amplification combined with lateral flow strip for detecting African swine fever virus. Frontiers in Microbiology, 2019, 10: 1004. doi:10.3389/fmicb.2019.01004.
Development and Application of a Real-Time Fluorescent RPA Diagnostic Assay for African Swine Fever

1School of Life Sciences, Ningxia University, Yinchuan 750021;2Veterinary Research Institute, Institute of Military Medical Sciences, Academy of Military Sciences, Changchun 130122
【】After the first outbreak of African Swine Fever (ASF) in Shenyang, China in 2018, it has rapidly spread to the whole country, severely hitting the pig industry. This study aimed to establish an optimized nucleic acid testing technique for African Swine Fever Virus (ASFV), so as to provide a fast and accurate method for early diagnosis and accurate treatment of ASF outbreaks. 【】 Appropriate primers and probes were designed and screened for the conserved gene B646L (p72) of ASFV, and a real-time fluorescent RPA assay based on recombinase polymerase amplification (RPA) was established. The reaction system, reaction conditions and sample treatment steps were optimized. Specificity and sensitivity of the optimized detection method were evaluated by using quality controls. In addition, 1 009 clinical samples were tested by the optimized real-time RPA, after which the results were further confirmed by the real time PCR recommended by OIE and through virus isolation. 【】 A pair of primers-probe combinations was successfully screened, and a real-time fluorescence RPA for detection of ASFV p72 gene was developed. The total volume of optimized reaction system was 25 μL. The reaction conditions were set as 39℃ 10 s, 39℃ 20 s, 40 cycles on the fluorescence quantitative PCR instrument, and the whole amplification reaction needs about 20 min. The analysis method at room temperature could replace the traditional nucleic acid extraction method, thus the whole process of sample treatment, nucleic acid amplification and result reading could be completed in 30 min. Specific evaluation showed that the real-time RPA was negative for porcine parvovirus (PPV), pseudorabies virus (PRV), circovirus type1/2 (PCV1/2), classical swine fever virus (CSFV) and porcine reproductive and respiratory syndrome virus (PRRSV); the sensitivity evaluation showed that the assay could detect type I/II/IX ASFV samples, and could detect 10 copies/μL of ASFV positive simulated blood samples and 1﹕103.0dilution of positive clinical samples, which was as sensitive as the OIE-recommended qPCR method. Seventeen out of 1 009 clinical samples were tested positive using the real-time RPA, with the same results as by qPCR, 17 positive cultures were obtained from virus isolation. 【】 A real-time RPA diagnostic method for ASF was developed, which was proved to be simple, less time consuming with high sensitivity and specificity, providing a new, simple, specific and rapid diagnostic method for ASF.
African swine fever virus; recombinase polymerase amplification; real-time fluorescent RPA; nucleic acid detection; diagnosis

10.3864/j.issn.0578-1752.2022.01.016
2020-11-16;
2021-02-03
国家重点研发计划(2017YFD0502300)、国家自然科学基金专项(31941016)
张静远,E-mail:zjyuanff27@163.com。通信作者李敏,E-mail:lim@nxu.edu.cn。通信作者扈荣良,E-mail:ronglianghu@hotmail.com
(责任编辑 林鉴非)
