新型冠状病毒非结构蛋白3结构和功能的生物信息学分析
2022-02-21丁一宋峰刘烁江亚娟王小羽焦伯延
丁一,宋峰,刘烁,江亚娟,王小羽,焦伯延
济宁市疾病预防控制中心,山东 济宁 272000
0 引言
新型冠状病毒肺炎(corona virus disease 2019,COVID-19)是由新型冠状病毒(2019 novel coronavirus,2019-nCoV)引起的一类呼吸道传染病,新型冠状病毒属于冠状病毒科β属冠状病毒属,基因组为单股正链RNA,全长约30kb,含有12个开放读码框(open reading frame,ORF)[1-2]。主要合成非结构蛋白(nonstructural protein,NSP)的ORF1a/b可翻译2种多聚蛋白(polyprotein,pp)1a和1ab,pp1a和pp1ab在NSP3和NSP5催化下分解成16个非结构蛋白[3]。NSP3是新型冠状病毒最重要的非结构蛋白之一,不仅含有的木瓜样蛋白酶(Papain-Like proteases,PLpro)活性,参与pp1a和pp1ab蛋白的裂解,还可以结合NSP4参与病毒复制,结合核衣壳蛋白参与病毒组装,并且可以抑制宿主干扰素(interferon,IFN)信号通路,抑制宿主对新冠病毒的免疫清除[3-5]。然而NSP3的结构功能仍需要进一步阐明。本研究利用生物信息学方法,分析NSP3的性质,为新冠肺炎的致病研究和药物研发提供思路。
1 材料与方法
1.1 材料
在NCBI数据库下载2019-nCoV的NSP3序列,序列号NC_045512.2。
1.2 方法
利用ProtParam分析NSP3的理化性质;利用ProtScale分析亲水性和疏水性;利用TMHMM分析跨膜结构域;利用POLYVIEW-2D分析二级结构和相互作用位点;分别利用NetPhos 3.1、NetNGlyc、NetOGlyc、NetCGlyc、iUbiq-Lys、GPSSUMO、HybridSucc、CSS-Palm、GPS-PAIL分析NSP3的磷酸化位点、N糖基化位点、O糖基化位点、C甘露糖基化位点、泛素化位点、类泛素蛋白修饰分子(small ubiquitin-like modifier,SUMO)化位点、琥珀酰化位点、棕榈酰化位点、乙酰化位点;利用SignalP-5.0分析信号肽;利用Cyscon分析二硫键;利用 PSORT分析亚细胞定位;利用pfam和NCBI服务器CDD分析结构域;利用phyre2进行同源建模构建三级结构;利用IEBD服务器Bepipred Linear Epitope Prediction 2.0和CD4 Immunogenicity prediction分析B细胞表位和CD4细胞抗原表位,利用NetCTL-1.2分析细胞毒性T淋巴细胞(cytotoxic T lymphocyte,CTL)抗原表位。
2 结果
2.1 NSP3的理化性质
NSP3由1945个氨基酸组成,含量最高的是亮氨酸(占9.2%),含量最低的是色氨酸(占0.9%),带正电荷氨基酸(精氨酸和赖氨酸)185个,带负电荷氨基酸(天冬氨酸和谷氨酸)222个,见图1。分子式为C9722H15175N2489O2969S88,相对分子量217252.61,理论等电点为5.56,不稳定系数为36.56,半衰期为4.4h。说明NSP3是稳定蛋白质。

图1 NSP3 氨基酸组成分析
2.2 NSP3的亲/疏水性和跨膜结构域分析
利用 ProtScale软件分析NSP3的亲/疏水性,位于第409位的谷氨酰胺亲水性最强,为-3.744;第1415位的苯丙氨酸疏水性最强,值为2.889;平均亲水性系数为-0.175。利用TMHMM分析NSP3的跨膜结构域,发现NSP3含有4个跨膜结构域,分别是:1413-1435,1500-1522,1532-1554,1561-1583(见图2)。说明NSP3是含跨膜结构域的亲水蛋白。
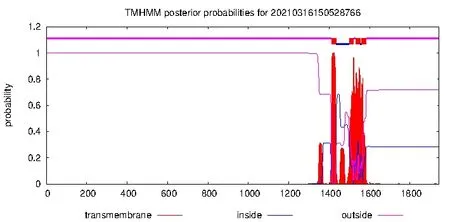
图2 NSP3 跨膜结构域分析
2.3 NSP3二级结构和相互作用位点分析
利用POLYVIEW-2D软件分析NSP3二级结构和相互作用位点,结果显示NSP3共含有57个α-螺旋,91个β-折叠,138个无规卷曲;其中α-螺旋氨基酸占34.0%(661/1945),β-折叠氨基酸占20.9(406/1945),无规卷曲氨基酸占45.1%(878/1945)。此外,NSP3含有93个主要的蛋白相互作用氨基酸位点。
2.4 NSP3翻译后修饰分析
利用NetPhos 3.1分析NSP3的磷酸化位点,发现NSP3共有199个潜在磷酸化位点,包括79个丝氨酸、83个苏氨酸、37个酪氨酸可能被磷酸化(见图3)。利用NetNGlyc、NetOGlyc、NetCGlyc分析NSP3的N糖基化、O糖基化、C甘露糖基化位点,发现NSP3含有11个N糖基化位点、14个O糖基化位点,无甘露糖基化位点(见表1)。
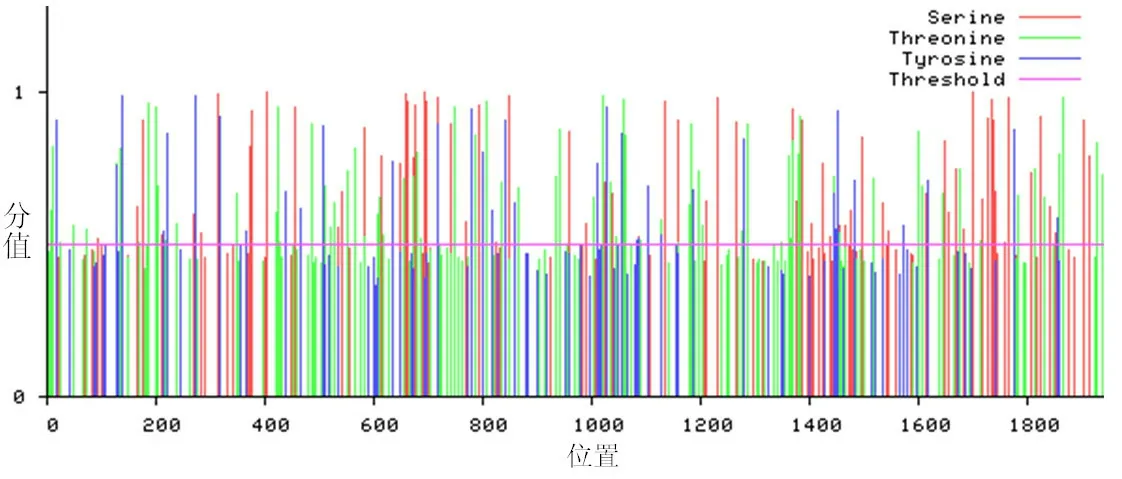
图3 NSP3 磷酸化位点分析
此外,NSP3含有3个泛素化位点、15个SUMO化位点、6个琥珀酰化位点、1个棕榈酰化位点、2个乙酰化位点(见表1)。利用GPS-Lipid分析肉豆蔻酰化位点、法尼基化位点、牻牛儿酰牻牛儿基化位点,发现NSP3无肉豆蔻酰化位点、无法尼基化位点、无牻牛儿酰牻牛儿基化位点。
2.5 NSP3信号肽、二硫键和细胞定位分析
SignalP-5.0分析NSP3的信号肽,显示NSP3无信号肽序列。Cyscon分析二硫键,发现NSP3含有15个二硫键(见表1)。PSORT分析亚细胞定位,NSP3主要分布于内质网。
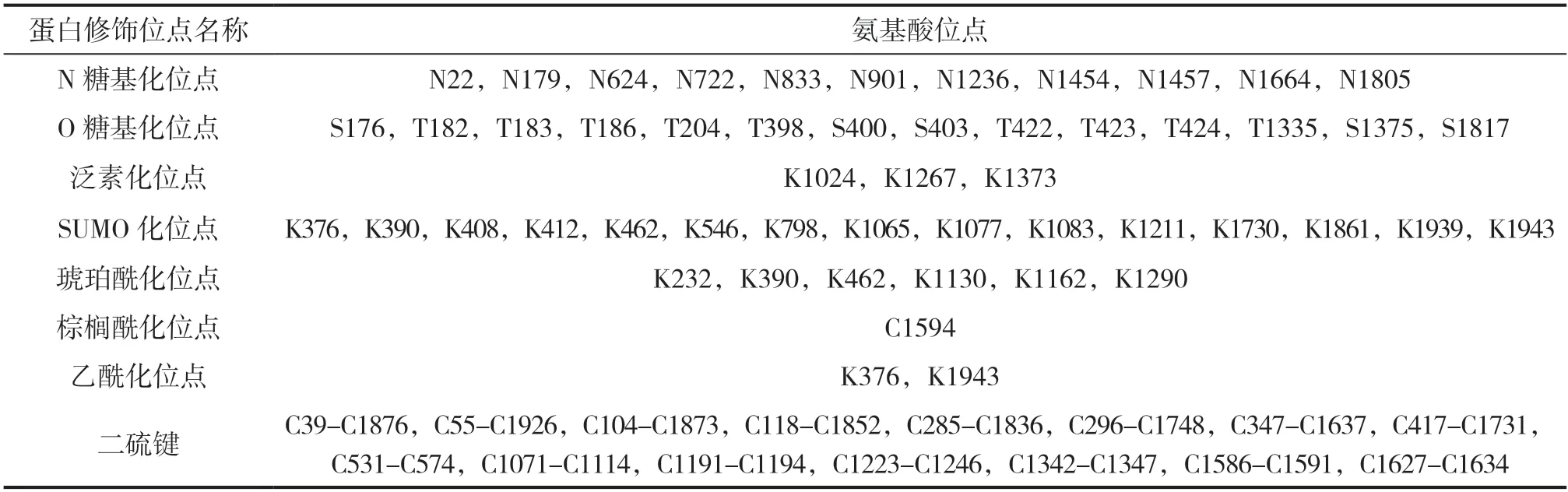
表1 NSP3 翻译后修饰分析
2.6 NSP3的结构域和三级结构分析
利用NCBI服务器CDD软件和pfam软件分析NSP3的结构域,两种软件分析结果基本一致,NSP3含7个保守结构域,见图4。利用phyre2预测NSP3的三级结构,NSP3 675-1053与c4yptA序列相似度100%,覆盖率19%,能够以c4yptA为模板进行同源建模构建NSP3三级结构(见图5)。

图4 NSP3 结构域分析
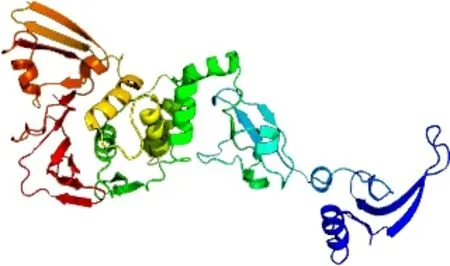
图5 NSP3 三级结构分析
2.7 NSP3抗原表位分析
利用IEBD服务器Bepipred Linear Epitope Prediction 2.0分析NSP3的B细胞抗原表位,N端B细胞抗原表位较丰富,特别是109-217区域抗原性最强。利用IEBD服务器CD4 Immunogenicity prediction分析CD4 T细胞抗原表位,大于90分区域19个,其中1746-1760区域抗原性最强,得分为99.39。利用NetCTL-1.2分析NSP3 CTL抗原表位,大于1.25分区域24个,其中503-511区域抗原性最强,得分为3.72。说明NSP3含有丰富的抗原表位。
3 结论
本研究利用生物信息学方法获得2019-nCoV NSP3的理化性质、跨膜结构域、亚细胞定位、翻译后修饰位点、三级结构、抗原表位等分析结果。理化性质分析显示,NSP3全长1945个氨基酸,其中亮氨酸含量最高,带负电荷氨基酸较多,含4个跨膜结构域,是可以定位于胞膜的稳定的亲水蛋白。二级结构无规卷曲含量最高,多达45.1%。无规卷曲常位于蛋白质表面,是蛋白质结合抗体、行使功能的主要结构[6]。NSP3含有15对二硫键,二硫键能够促进蛋白正确折叠和维持蛋白结构的稳定[7],NSP3的二硫键对其三级结构的折叠形成和稳定可能至关重要。
NSP3无信号肽,主要定位于内质网,但囊泡、线粒体、细胞核、液泡和细胞质也有一定的分布。蛋白质翻译后通常需要修饰后才能形成成熟蛋白质[8]。磷酸化、糖基化、脂酰化、泛素化等修饰对蛋白质的亚细胞定位、调节、代谢、功能和降解至关重要[9]。NSP3含有199个磷酸化位点、25个糖基化位点、15个SUMO化位点、2个乙酰化位点和7个脂酰化位点,说明NSP3的成熟需要复杂的修饰,NSP3可能具有较强的生物学活性。此外,NSP3含有3个泛素化位点,蛋白酶体能够降解泛素化修饰蛋白[10],说明宿主可以通过泛素-蛋白酶体途径降解NSP3。
NSP3是新型冠状病毒氨基酸含量最多的成熟蛋白质,含有7个功能结构域,其中746-1064是NSP3的PLPro结构域,是裂解pp1a和pp1ab蛋白的核心区域。病毒蛋白酶具有与宿主蛋白酶不同的结构,针对病毒蛋白酶的抑制剂是治疗RNA病毒感染的最重要特异性药物[11-12]。PLPro结构域是治疗COVID-19药物研究的最重要靶点之一[13]。本研究对NSP3三级结构的分析和木瓜样蛋白酶结构域的确定将为PLPro抑制剂的研发制备提供研究依据。
本研究利用多参数分析方法分析NSP3含有多个B细胞抗原表位和T细胞抗原表位,说明NSP3具有较强的免疫原性。109-217区域B细胞抗原性最强,这一区域可能能够作为新冠肺炎抗体检测的靶标和疫苗研发的候选序列。此外,NSP3含有数量丰富的抗原性较强的CTL抗原表位,说明NSP3可能能够引起较强的T细胞免疫反应,宿主T细胞免疫反应的激活能够使感染2019-nCoV的细胞凋亡,但同时可能引起宿主发生炎症反应和细胞因子风暴[14-17]。NSP3丰富的抗原表位可能与宿主的免疫清除和肺炎的发生相关。
综上所述,本研究对NSP3生物信息学分析,将有助于进一步了解NSP3的生物学功能,为深入了解2019-nCoV的生活史、致病性及宿主免疫提供理论基础,为检测试剂和抗病毒药物的研究提供参考数据。
