DNA甲基化修饰的定位分析方法研究进展
2022-02-18陈梦园游雪娇袁必锋冯钰锜
陈梦园,游雪娇,袁必锋,2*,冯钰锜,2
(1.武汉大学 化学与分子科学学院,湖北 武汉 430072;2.武汉大学 公共卫生学院,湖北 武汉 430071)
DNA 是一种由磷酸脱氧核糖主链和含氮碱基腺嘌呤(Adenine,A)、鸟嘌呤(Guanine,G)、胞嘧啶(Cytosine,C)和胸腺嘧啶(Thymine,T)组成的天然聚合物,用来储存、编码、传递和表达遗传信息[1]。除了正常碱基外,DNA 中还包含许多化学修饰[2-4]。这些修饰不会改变DNA 的序列,但会调节转录、细胞分化等生理过程,与疾病的发生密切相关[5-6]。其中,5-甲基胞嘧啶(5-Methylcytosine,5mC)作为DNA 中含量最高的甲基化修饰,存在于大多数植物、动物和真菌中,是最具代表性的表观遗传修饰之一[7-9]。
5mC是一种甲基化发生在胞嘧啶第五位碳上的修饰,该修饰参与多种生理过程,比如基因组印记、X 染色体失活和细胞分化等[10-13]。研究发现,异常的高甲基化可能会使正常细胞中的潜在抑癌基因沉默,而异常的低甲基化则可能会导致原癌基因激活,从而引起癌症的发生[14-16]。Dnmt1(DNA methyltransferase 1)、Dnmt3a 和Dnmt3b 是哺乳动物细胞中常见的3 种5mC 的DNA 甲基转移酶[17-18],它们以S-腺苷甲硫氨酸(S-Adenosylmethionine,SAM)为甲基供体,在DNA 胞嘧啶的五位碳上加上甲基基团构成。5mC 的形成过程如图1所示[19]:DNA 甲基转移酶通过具有活性的半胱氨酸残基进攻胞嘧啶C6位形成中间体,该中间体的C5 位对SAM 亲核进攻从而甲基化,随后C6 位的半胱氨酸残基离去得到5mC。

图1 DNA甲基化过程示意图Fig.1 Schematic illustration of DNA methylation
DNA 甲基化修饰在体内是动态平衡的,随着5mC甲基化过程的发现,其去甲基化过程最近几年逐渐清晰和完善。DNA 去甲基化的方式一般有两种:被动去甲基化和主动去甲基化[20]。被动去甲基化是指在基因组DNA的半保留复制过程中,已存在的5mC含量因被动“稀释”而下降,从而达到被动去甲基化的目的[20]。碱基切除修复(Base excision repair,BER)介导的DNA 主动去甲基化是目前研究得较透彻的生物途径(图2)[21]。在该DNA 去甲基化途径中,5mC 在TET(Ten-eleven translocation)蛋白的作用下,逐步被氧化成5-羟甲基胞嘧啶(5-Hydroxymethylcytosine,5hmC)、5-醛基胞嘧啶(5-Formylcytosine,5fC)和5-羧基胞嘧啶(5-Carboxylcytosine,5caC)[22]。胸腺嘧啶DNA 糖苷酶(Thymine DNA glycosylase,TDG)能将5fC和5caC上的碱基切除,形成一个无碱基位点;随后无碱基位点通过BER途径被修复成正常的胞嘧啶,从而实现DNA 的主动去甲基化[21,23]。除此之外,还有另外两种可能的途径:(1)5mC 经TET 蛋白氧化生产的5hmC 经AID(Activation-induced cytidine deaminase)和APOBEC(Apolipoprotein B mRNA editing enzyme,catalytic polypeptide-like)蛋白脱氨成5-羟甲基尿嘧啶(5-Hydroxymethyluracil,5hmU),5hmU 随后被TDG 或SMUG1(Single-stranded-selective monofunctional uracil DNA glycosylase 1)蛋白切除,形成无碱基位点,再由BER 途径修复成正常胞嘧啶;(2)TET 蛋白氧化5mC 生产5fC 和5caC,5fC和5caC可以直接脱醛基或脱羧基生产未修饰的胞嘧啶(图2)。目前已证实在哺乳动物基因组中确实存在直接脱醛基[24-25]和脱羧基[21,26-27]途径,这些研究为后续脱醛基酶和脱羧酶的鉴定奠定了基础。

图2 DNA主动去甲基化机制Fig.2 Mechanism for active DNA demethylation
5mC在生物体内发挥着重要生理功能,其甲基化的异常与各种疾病密切相关。研究DNA 甲基化和去甲基化过程中的相关蛋白和调节因子,能促进对发育和细胞稳态的理解,为临床应用提供参考[28-29]。为了深入探索DNA 甲基化修饰的功能,需要了解其在基因组中的具体位置信息。近些年高通量测序技术的进步推动了DNA甲基化修饰领域的研究进展。本文总结了基于高通量测序技术的5mC定位分析方法的研究和应用情况(表1)。

表1 基于高通量测序对5-甲基胞嘧啶(5mC)进行定位分析的方法Table 1 Summary of analytical methods for localizing 5-methylcytosine(5mC)based on high-throughput sequencing technology
1 5mC的定位分析方法
1.1 免疫沉淀富集介导的定位分析
由于核酸修饰的丰度通常较低,故将含有碱基修饰的片段进行富集有助于测序。Ruike等[30]证明使用特异性抗体富集含有碱基修饰的DNA 片段可以加强对核酸修饰的定位分析。基于此,研究者开发了多种基于免疫沉淀富集的方法,结合高通量测序,对基因组DNA甲基化进行定位分析,即DNA免疫沉淀测序(DNA immunoprecipitation sequencing,DIP-seq)。常用两种方法对甲基化DNA 进行免疫沉淀:甲基化DNA 免疫共沉淀测序(Methylated DNA immunoprecipitation and sequencing,MeDIP-seq)[31]和甲基化DNA 特异性结合蛋白富集甲基化测序(Methyl-CpG binding domain protein-enriched genome sequencing,MBD-seq)[32-33]。
MeDIP-seq 是利用5mC 抗体富集含有5mC 的单链DNA 片段,将富集后的片段进行高通量测序的一种全基因组甲基化检测手段[31,34]。类似地,基于抗体富集的测序方法也适用于DNA 中的其他修饰[35]。由于甲基化CpG结合(Methyl-CpG-binding domain,MBD)蛋白对甲基化序列有很强的亲和性,能被用来对DNA 甲基化进行分析,因此研究者开发了一种将MBD 与高通量测序相结合的方法,即MBD-seq[33]。在MBD 蛋白家族中,MBD1、MBD2、MBD3 和MeCP2(Methyl-CpG-binding protein-2)4 种蛋白有相似的结构域,能识别双链DNA 中CpG 双核苷酸上的对称甲基[35]。MBD2b(Methyl-CpG-binding domain protein 2b)作为MBD2 的一种亚型蛋白,与MBD3L1(Methyl-CpG-binding domain protein 3-like-1)蛋白的作用对调节甲基化DNA 复合物的形成至关重要,因此常用MBD2蛋白进行MBD-seq分析[32-33]。基于MBD蛋白的发现和应用,发展了一系列全基因组DNA 甲基化的检测方法。除MBD-seq 外,还有MIRA-seq(Methylated-CpG island recovery assay sequencing)、MiGS(MBD-isolated genome sequencing)、MethylCapseq(Methylation DNA capture sequencing)等,均能用于5mC的定位分析[35-38]。
基于免疫沉淀的测序方法可以发现基因组中甲基化的区域,但无法实现甲基化修饰的单碱基分析[29]。抗体的高度特异性对测序结果的准确性至关重要。此外,商品化的抗体种类较少,且批次间抗体质量不一致,因此,基于免疫沉淀富集介导的5mC 定位分析对用来富集的抗体或者蛋白要求很高[35]。
1.2 亚硫酸氢盐介导的定位分析
亚硫酸氢盐测序法(Bisulfite sequencing,BS-seq)是指将DNA 甲基化修饰经亚硫酸氢盐转化后进行高通量测序的一种定位分析方法,已被广泛应用于全基因组分析中[39]。在传统的亚硫酸氢盐测序方法中,C、5fC 和5caC 经亚硫酸氢盐处理后脱氨变成尿嘧啶(Uracil,U),在测序过程中被读成T;而5mC和5hmC 经处理后不脱氨,仍被读成C(图3)。将亚硫酸氢盐处理和未处理的测序样本进行比较可以发现甲基化的位点,但是该方法无法区分5mC 和5hmC[40-41]。因此,为了对5mC 进行定位分析,需要对传统的亚硫酸氢盐测序方法进行改进。
氧化亚硫酸氢盐测序法(Oxidative bisulfite sequencing,oxBS-seq)[40]和TET 辅助的亚硫酸氢盐测序法(TET-assisted bisulfite sequencing,TAB-seq)[41]能实现5mC 的单碱基定位分析。在oxBS-seq 中,5hmC被KRuO4氧化成5fC,经亚硫酸氢盐处理后脱氨读成T,而5mC 依旧被读成C[40],从而可在单碱基水平分辨5mC和5hmC(图3)。
此外,还可以用传统亚硫酸氢盐测序的方法间接确定5mC 的位点(图3)。在TAB-seq 中,首先在β-葡萄糖基转移酶(β-Glucosyltransferase,β-GT)的作用下将葡萄糖基转移至5hmC,形成的β-葡萄糖基-5-羟甲基胞嘧啶(β-Glucosyl-5-hydroxymethylcytosine,5gmC)不被TET 蛋白氧化,在测序时被读成C;而5mC 经TET 蛋白氧化成5caC,在测序时被读成T,从而可实现5hmC 的单碱基定位分析。将传统亚硫酸氢盐测序法确定的5mC 和5hmC 总位点减去5hmC 的位点,即可实现对5mC 位点的间接分析[41]。

图3 亚硫酸氢盐测序原理图Fig.3 Schematic diagram of bisulfite sequencing
亚硫酸氢盐介导的定位分析方法存在两个不足:一是该反应需要的条件苛刻,使得DNA 大量断裂,不利于后续的高通量测序[42];二是未修饰胞嘧啶在人类总基因组胞嘧啶中所占比例超过95%,将这部分胞嘧啶转化成胸腺嘧啶使得序列复杂度、测序质量以及覆盖率降低,给后续分析造成较大挑战[28]。此外,未修饰的胞嘧啶如果脱氨不完全,可能产生假阳性结果。因此,需要寻找更合适的方法对5mC进行定位分析。
1.3 酶介导的定位分析
为了克服亚硫酸氢盐对DNA 的损伤,研究人员开发了一种比较温和的方法,即酶促甲基化测序法(Enzymatic methyl-seq,EM-seq)[42-43]。该方法依赖于酶分析定位DNA 胞嘧啶的甲基化状态,避免了过于剧烈的化学反应条件[42-43]。APOBEC3A(Apolipoprotein B m RNA editing enzyme catalytic polypeptidelike 3A)是细胞的抗病毒功能因子之一,可使单链DNA 的胞嘧啶残基脱氨基转化为尿嘧啶[42]。在单链DNA 中,APOBEC3A 能将C 和5mC 脱氨,在随后的聚合酶链式反应(PCR)中二者均会被读成T[42]。为了对5mC 进行单碱基定位分析,在APOBEC3A 脱氨前对5mC 和5hmC 进行选择性保护,以在碱基水平上间接分析5mC[42]。具体来说,先使用β-GT 将5hmC 糖基化成5gmC,再用TET 蛋白将5mC 氧化成5caC,5gmC 和5caC 不被APOBEC3A 脱氨,在PCR 中依旧被读成C,而C经历脱氨被读成T[44-45](图4)。如果只将5hmC 进行糖基化保护,经APOBEC3A 处理后,只有5gmC 被读成C,其余均被读成T。将两种方法相结合即可间接确定5mC的位点[42,44]。

图4 酶促甲基化测序原理图Fig.4 Schematic diagram of enzymatic methyl sequencing
相对于亚硫酸氢盐测序法而言,EM-seq具有条件温和、可维持DNA完整性的优点[43],进一步扩大了应用范围。然而,该方法同样存在脱氨不完全造成假阳性、测序质量较差的问题[46]。
1.4 吡啶硼烷介导的定位分析
Liu 等[46]开发了TET 辅助吡啶硼烷测序法(TETEM-seqassisted pyridine borane sequencing,TAPS):利用TET 酶将5mC 和5hmC 氧化成5caC,再通过吡啶硼烷将5caC 还原形成二氢尿嘧啶(Dihydrouracil,DHU),DHU 在之后的测序中被读成T,从而实现5mC 和5hmC 从C 到T 的转变[35]。该方法可以用来定位分析基因组中的5mC 和5hmC,但无法将二者区分[46]。为此需要改进TAPS 方法以实现5mC 的定位分析。基于此,Liu 等[28,46]开发了β-葡萄糖基转移酶阻断的TAPS(TAPS withβ-glucosyltransferase blocking,TAPSβ)和化学辅助吡啶硼烷测序(Chemical-assisted pyridine borane sequencing,CAPS)。
在TAPSβ中,将葡萄糖基引入5hmC 使其糖基化成5gmC 后,进行TET 氧化和吡啶硼烷还原反应,该过程中5mC经历了从5mC到5caC再到DHU,最后被读成T的过程,而5gmC不参与氧化和还原过程,依旧被读成C[28,35]。除了还原5caC 外,吡啶硼烷也能将5fC还原成DHU,基于此开发了CAPS方法。使用KRuO4将5hmC 氧化成5fC,再经吡啶硼烷还原成DHU,测序时被读成T,而5mC 不参与任何一步反应,依旧被读成C[28](图5)。
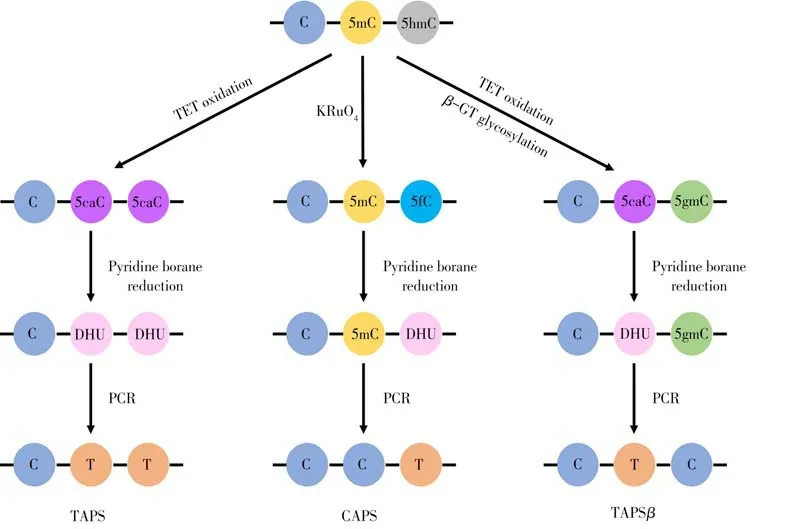
图5 吡啶硼烷测序原理图Fig.5 Schematic diagram of TET-assisted pyridine borane sequencing
与亚硫酸氢盐测序和酶促甲基化测序方法相比,TAPS 方法显著降低了DNA 的损伤,提高了测序质量,避免了未修饰胞嘧啶的转化。此外,也降低了分析的成本[28,46-47]。TAPSβ是一种不依赖亚硫酸氢盐、碱基分辨率高、能定量5mC 特异性位点的分析方法[46],但是,该方法中吡啶硼烷的还原效率为90%多,可能会造成测序结果存在一定的误差。
1.5 纳米孔测序定位分析
纳米孔测序已经成为一种有前景的DNA甲基化修饰分析方法[48-49]。不同的核苷通过纳米孔会产生不同的电流,根据电流的变化可以分辨正常核苷和修饰核苷[50]。目前,纳米孔测序已成功用于区分DNA中的5mC和正常胞嘧啶[50-51],从而实现5mC的单碱基定位分析。此外,该方法也可用于DNA分子中5hmC的定位分析[48,52]。
与其他高通量测序方法相比,纳米孔测序具有无需扩增、可减少聚合酶和连接酶的使用等优点[49],但该方法目前仍然处于发展阶段,需要进一步优化维持酶活性和稳定性的条件[53]。
1.6 单分子实时测序定位分析
除纳米孔测序外,单分子实时(Single molecule real-time,SMRT)测序作为另一种有前景的DNA测序方法,能直接对DNA分子上的5mC进行定位分析[54-55]。在单分子实时测序中,DNA聚合酶催化核苷酸进入DNA 中,通过监测每种核苷酸独特的动力学特征,可以对DNA 中的不同碱基进行区分[29,48,55-56]。由于未修饰碱基和修饰碱基在核苷酸掺入时间上存在差异,该方法可被用于对5mC[55]、5hmC[57]、6-甲基腺嘌呤[58]和细菌硫代磷酸[59]的直接检测。使用TET1将5mC氧化成5caC能增强动力学特征,提高对5mC的检测能力[60]。
单分子实时测序方法的出现为微量样品的分析提供了可能,具有测序通量高、成本低、时间短、操作简单的优点,而如何降低荧光信号对背景噪音的影响是一个需要解决的问题。
2 结论与展望
目前已在生物体DNA和RNA中发现了多种化学修饰,其中,5mC作为DNA分子中的“第五碱基”,参与多种生理过程,如胚胎发育、转录异常、基因沉默。现有分析方法的开发推动了5mC的研究进展,破译5mC 的位置信息有助于深入阐明5mC 的生物学功能,增强对生物生长发育、疾病发生等问题的理解。而对5mC生物学功能的研究需要灵敏度高、准确度高以及稳定性好的方法,这些方法的开发也将促进对应技术和软件的改进与提升。
已报道的化学试剂或酶结合高通量测序对5mC进行定位分析的方法在选择性标记DNA修饰方面具有良好的应用前景,但存在反应条件较为剧烈、易造成假阳性等局限性,这对精确定位5mC 来说是一个挑战。未来可探索新的化学方法、开发新的化学试剂或采用更温和的反应条件来提高5mC 的单碱基分辨率以及实现对单个5mC 位点的量化。使用第三代测序技术对5mC 进行定位分析是一个很有前景的方向,纳米孔测序和单分子实时测序为直接对5mC 进行定位提供了可能。未来可通过研究如何维持酶的活性和稳定性以及降低荧光信号的干扰对纳米孔测序和单分子实时测序进行改进。此外,现有技术精度、灵敏度的改进也可能促进对其它低丰度化学修饰的测序研究,甚至推进新修饰的发现。修饰位点的确定以及修饰种类的扩充有助于在基因层面加深对疾病的研究,从而对DNA甲基化修饰谱图进行更加具体的阐述。
