中国地方品种黄鸡线粒体DNA D-loop遗传多样性研究
2022-02-01黄勋和翁茁先李威娜王庆何丹林罗威张细权杜炳旺
黄勋和,翁茁先,李威娜,王庆,何丹林,罗威,张细权,杜炳旺
中国地方品种黄鸡线粒体DNA D-loop遗传多样性研究

1嘉应学院/广东省山区特色农业资源保护与精准利用重点实验室/广东省五华三黄鸡科技创新中心,广东梅州 514015;2华南农业大学动物科学学院,广州 510642
【目的】地方品种黄鸡是中国重要的家禽遗传资源,系统评估其遗传多样性水平,为提高利用率和制定科学的保护策略提供依据。【方法】对新测的694份样本和下载的589份样本,合计28个中国南方地方品种黄鸡共1 283份的线粒体DNA片段(D-loop,519 bp)遗传多样性进行整合分析,构建单倍型中介网络图,通过单倍型地理分布格局、主坐标分析、分子变异分析和中性检测,确认其内部的群体遗传结构和群体历史。通过分析亚洲和太平洋地方鸡线粒体DNA地理分布格局,推测中国南方地方品种黄鸡稀有单倍型类群的来源。【结果】从1 283份样本检测到101个变异位点,其中92个为多态位点。定义了169种单倍型,归属于6个单倍型类群A-E和G,其中A-C和E为优势单倍型类群,D和G为稀有单倍型类群,占总体样本比例分别为15.43%、49.26%、18.55%、16.37%、0.31%和0.08%。河南省单倍型类群最丰富(6个);广东省单倍型数量最多(61),浙江省(19)和海南省(12)较少。单倍型类群A和B在28个黄鸡品种中均有分布;D只分布于淮南麻黄鸡、固始鸡、宁都黄鸡和霞烟鸡中;G只分布于固始鸡中。从单倍型类群D的分布频率和单倍型数量来看,中国南方地方品种黄鸡单倍型类群D可能来源于东南亚地区。从单倍型类群G的地理分布和中介网络图来看,河南和南亚的单倍型类群G可能来源于中国西南地区。中国南方地方品种黄鸡总体单倍型多样性和核苷酸多样性分别为0.903±0.005和0.01269±n.d.。河南省和湖南省的单倍型多样性和核苷酸多样性最高,分别为0.916±0.011、0.01358±0.00039和0.913±0.012、0.01345±0.00042;海南省的单倍型多样性最低(0.736±0.076),江西省的核苷酸多样性最低(0.00981±0.00072)。在不同地方品种间,淮南麻黄鸡、固始鸡和郧阳大鸡的单倍型多样性最高,江汉鸡、淮南麻黄鸡和黄郎鸡的核苷酸多样性最高,洪山鸡、广西三黄鸡和萧山鸡的单倍型多样性和核苷酸多样性最低。文昌鸡与河田鸡的遗传关系最近,与历史记载相吻合。地方品种黄鸡群体遗传分化不明显,分子变异主要来源于品种内。中性检测显示黄鸡在品种水平上近期未经历明显种群扩张,但单倍型类群A、B和E经历明显的扩张历史。【结论】结果表明地方品种黄鸡总体上保留着较高的遗传多样性水平,处于较好的保种状态,但洪山鸡、萧山鸡和广西三黄鸡应加强保护。地方品种黄鸡存在杂交现象,整体上经历了群体扩张历史。东南亚和西南地方鸡对中国南方地方品种黄鸡有一定的遗传贡献。
黄鸡;线粒体DNA D-loop;遗传变异;遗传分化;群体历史
0 引言
【研究意义】中国独特的地理环境和多样的风俗文化形成了众多形态各异的地方鸡品种,仅在《中国畜禽遗传资源志:家禽志》记载的就有107个,满足了不同地区、不同消费习惯及不同消费层次的需求[1]。地方品种黄鸡是指产于当地、历史悠久、品种纯正、具有“三黄”(毛黄、脚黄、皮黄)或黄麻羽的美丽外观、独特的肉质风味、广受消费者欢迎的地方品种,早在公元540年前北魏时期的农学专著《齐民要术》上就有记载[1-2]。地方品种黄鸡是祖先留给我们的宝贵财富,是我国重要的畜禽遗传资源和极具产业优势的特色肉鸡品种,也是我国畜牧业可持续发展的种源基础。在当今世界各国把种质资源挖掘保护作为国家战略高度之际,谁占有资源,谁就占领了制高点[1]。科学认识和正确评估遗传多样性水平是今后制定家禽遗传资源保护规划和开展利用工作的重要基础和依据。【前人研究进展】线粒体DNA(mitochondrial DNA,mtDNA)因母系遗传、核苷酸突变速率快、无重组等特点,被广泛用于动物遗传多样性和群体母系历史研究[3-4]。Wu等[5]分析了中国14个地方鸡品种的mtDNA控制区(D-loop,片段长度360 bp)遗传多样性,结果显示这些品种处于较高的遗传变异水平,不存在明显的遗传结构。基于黄郎鸡mtDNA D-loop(片段长度524 bp)的研究显示其处于较高的遗传变异水平,起源于中国北方和西南地区,同时受邻省地方鸡的影响[6]。贾晓旭等[7]评估了华东地区11个地方鸡品种(含白耳黄鸡、崇仁麻鸡、仙居鸡、琅琊鸡等黄鸡)的mtDNA D-loop全序列(片段长度1 232 bp)核苷酸多样性,探讨了品种的母系起源,指出部分品种可能受到欧美高产品系的遗传渗入。基于mtDNA D-loop全序列(片段长度1 232 bp)研究表明江苏省鹿苑鸡、溧阳鸡和太湖鸡等3个黄鸡的遗传多样性较低,主要单倍型类群为A和B[8]。黄勋和等[9]分析了广东省及邻省共12个黄鸡品种的mtDNA D-loop(片段长度520 bp)核苷酸多样性,结果显示广东省黄鸡的遗传多样性水平较高,品种的形成受到邻省和北方家鸡,以及东南亚红原鸡的影响。【本研究切入点】然而,这些基于mtDNA D-loop的地方鸡遗传多样性研究所用的序列长短不同,不同研究结果存在的差异使得不同研究比较难以进行平行比较分析,同时研究材料(品种)数量较少及覆盖面较低,不利于摸清中国地方品种黄鸡的遗传多样性水平及其群体历史。【拟解决的关键问题】本研究整合中国南方28个地方品种黄鸡样本及mtDNA D-loop数据,以统一的分析序列长度大尺度对中国南方地方品种黄鸡的遗传多样性进行整体评估,探讨品种的群体历史,为中国家禽遗传资源保护和创新利用提供科学依据。
1 材料与方法
1.1 试验材料
新采集26个中国南方地方品种黄鸡694份样本,同时从GenBank(https://www.ncbi.nlm.nih.gov/ nuccore)数据库下载17个中国南方地方品种黄鸡589条mtDNA D-loop序列数据,合计28个地方品种共1 283份样本(表1)。五华三黄鸡之外的27个品种均入选了《中国畜禽遗传资源志:家禽志》[1],五华三黄鸡入选了《中国禽类遗传资源》[10]。采集地为保种场。根据系谱记录采集无血缘关系的个体,肱静脉取血100 μL,置于70%乙醇于-80℃保存。采用标准的酚氯仿方法提取基因组DNA,-20℃保存备用。
1.2 PCR扩增与序列测定
mtDNA D-loop的PCR扩增引物为:L16750,5¢-AGGACTACGGCTTGAAAAGC-3¢[11];H522,5¢-ATG TGCCTGACCGAGGAACCAG-3¢[12]。反应体系为30 μL,含3 μL10×PCR Buffer(含Mg2+),2.4 μLdNTP mixture(2.5 mmol·L-1),正、反向引物(20 μmol·L-1)各0.3 μL,1 μL模板DNA,0.3 μLDNA聚合酶(宝生物,大连)(5 U/ μL)。PCR扩增条件:94℃预变性4min;35个循环(94℃变性30 s,63℃复性1 min,72℃延伸50 s);最后72℃延伸10 min。PCR扩增产物用1.5%琼脂糖凝胶电泳检测到目标条带后,送广州艾基生物技术有限公司双向测序。
1.3 序列分析
使用软件ChromasPro 2.1.8(Technelysium,澳大利亚)检查测序质量;以红原鸡mtDNA基因组全序列(NC_007235)作为参考序列,用MEGA 7.0[13]比对序列后,基于序列长度的分布情况和方便不同研究结果的平行比较分析,本文统一截取序列长度为519 bp(对应NC_007235核苷酸序列位置nps:1—519)作进一步分析。提取线粒体的SNP位点,利用Structure 2.3.4[14]分析品种间的群体遗传结构。使用DnaSP 6.12.03[15]定义单倍型,提取变异位点信息(variable sites),计算单倍型多样性(haplotype diversity)、核苷酸多样性(nucleotide diversity)和错配分布分析(mismatch distribution analysis)。利用Arlequin 3.5[16]以省份和品种为划分依据,分别计算群体间分化指数(genetic differences among population,ST)、组间分化指数(genetic differences among groups defined a priori,CT)、组内群体间分化指数(genetic differences among population within groups,SC),进行群体内和群体间的分子方差分析(Analysis of Molecular Variance,AMOVA)以及中性检测(Tajima’s和Fu's检验)。ST数值用于主坐标分析(principal coordinate analysis,PCoA),该计算由GenAlEx 6.503[17]执行。应用软件MitoToolPy(http://www.mitotool.org/)获得序列变异信息,然后划入特定的单倍型类群[18-19]。中介网络图(median-joining network)由NETWORK 10.2.0.0[20]绘制。
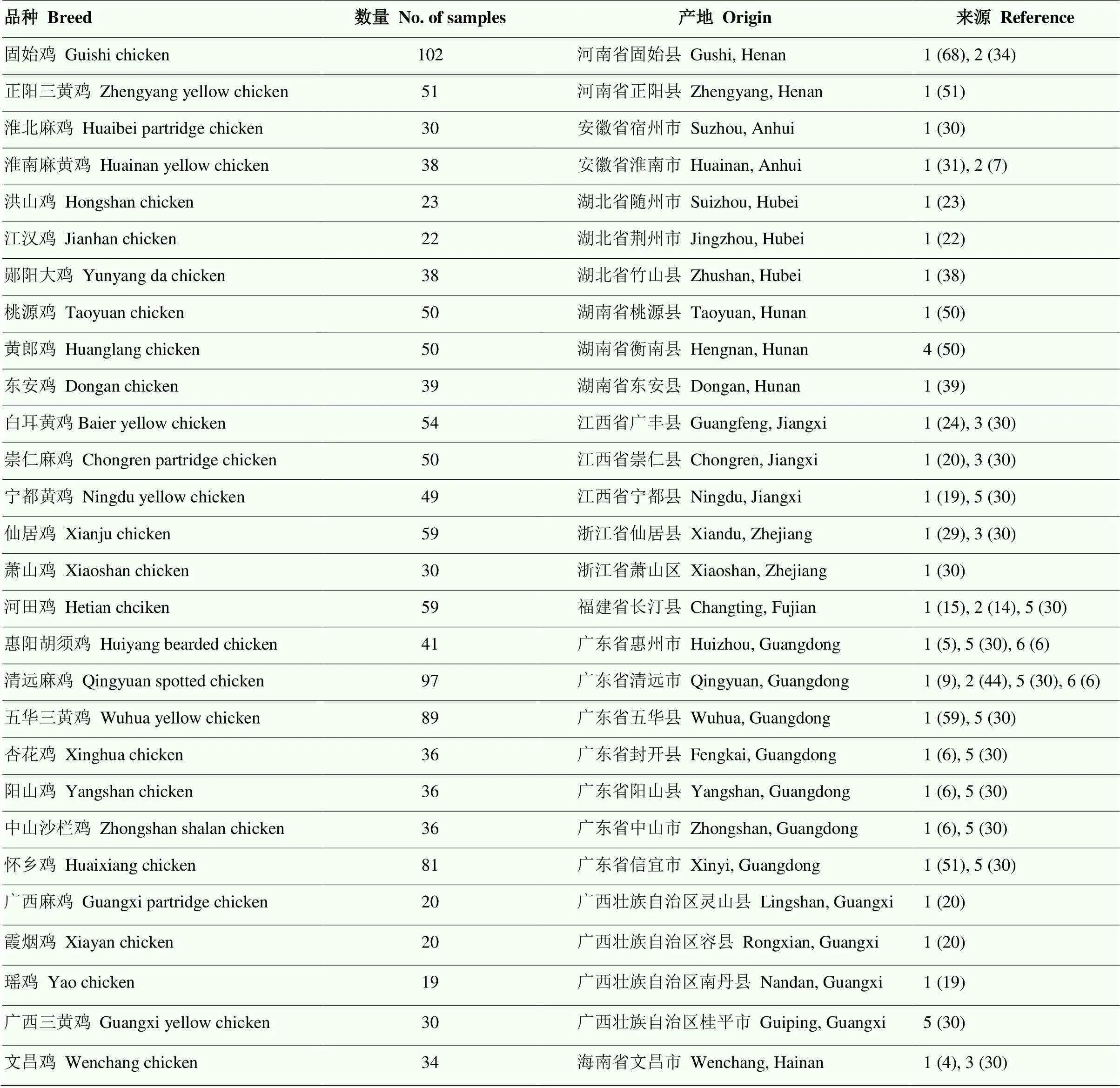
表1 样品信息
样品/数据来源:1:本研究;2:Miao et al,2013;3:Gao et al,2017;4:黄勋和等,2016;5:黄勋和等,2018;6:Liu et al,2006
Sample or data references: 1: This study; 2: Miao et al, 2013; 3: Gao et al, 2017; 4: Huang et al, 2016; 5: Huang et al, 2018; 6: Liu et al, 2006
2 结果
2.1 遗传多样性
获得了1 283条519 bp的mtDNA D-loop序列,检测到101个变异位点,占总分析位点的19.46%,其中92个为多态位点,定义了169种单倍型。28个黄鸡群体的总体单倍型多样性为0.903±0.005,总体核苷酸多样性为0.01269±n.d.。河南省和湖南省黄鸡的单倍型多样性和核苷酸多样性均排在10个省自治区中的前二位,而海南省的单倍型多样性最低,江西省的核苷酸多样性最低(表2)。在地方品种中,淮南麻黄鸡、固始鸡和郧阳大鸡的单倍型多样性位列28个品种的前三位,江汉鸡、淮南麻黄鸡和黄郎鸡的核苷酸多样性位列前三位,而洪山鸡、广西三黄鸡和萧山鸡的单倍型多样性和核苷酸多样性均排在后三位。值得注意的是,萧山鸡的核苷酸多样性远低于其他黄鸡品种。
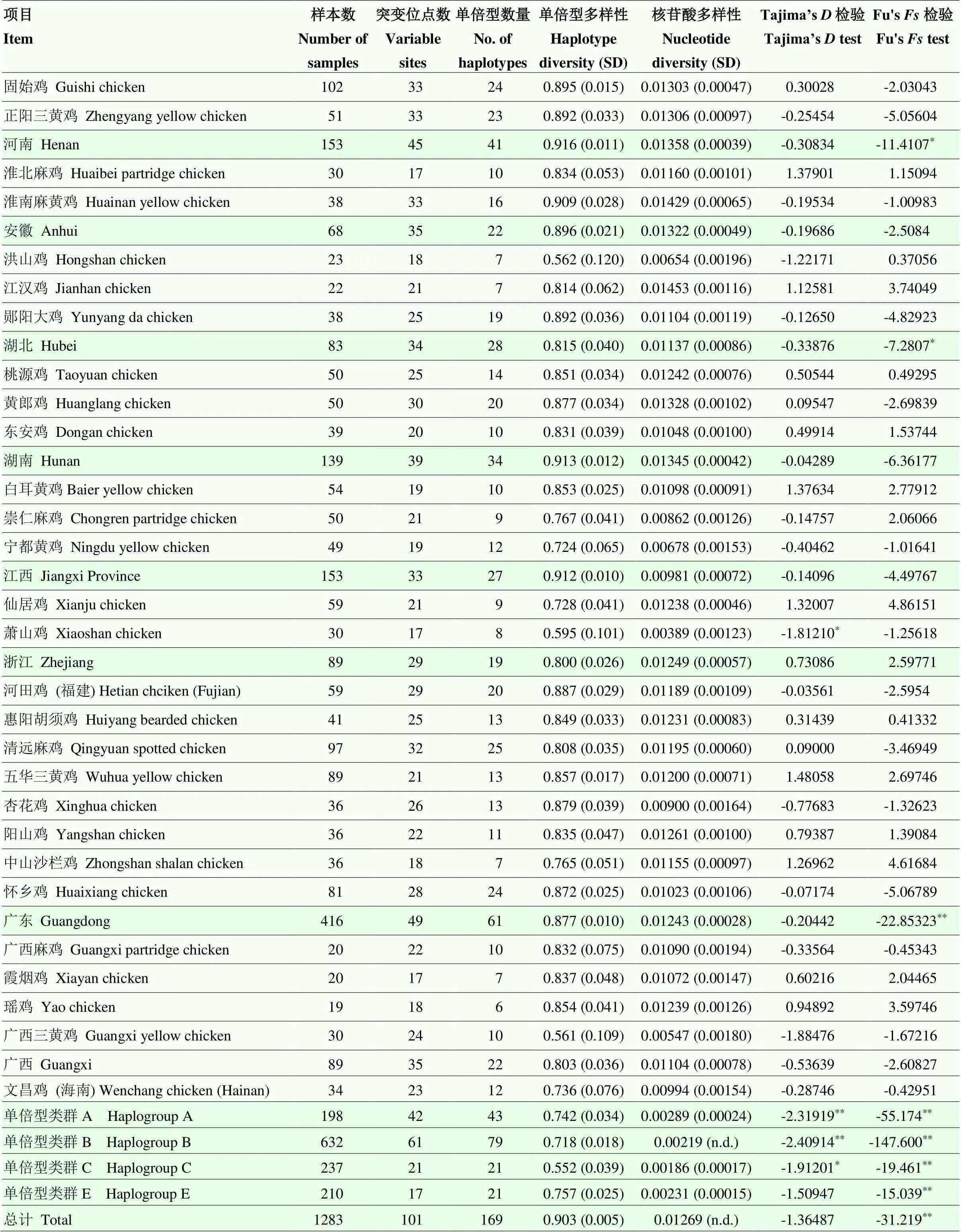
表2 28个中国南方地方品种黄鸡的线粒体DNA D-loop遗传多样性
*:在<0.05水平显著差异;**:在<0.01水平显著差异*: Significant at<0.05;**: Significant at<0.01
2.2 单倍型地理分布格局
169种单倍型划分为6个单倍型类群A-E和G。单倍型类群B为优势单倍型类群,占总样本数的49.26%,其次为C(18.55%)、E(16.37%)和A(15.43%),D和G分别只检测到4个(0.31%)和1个(0.08%)(表3)。广东省黄鸡的单倍型数量最多(61),河南省次之(41),浙江省(13)和海南省(12)较少。河南省黄鸡的单倍型类群组成最为丰富,覆盖6个单倍型类群,安徽省、江西省和广西壮族自治区有5个单倍型类群,而其他省份只有4种单倍型类群(表3)。中介网络图显示单倍型类群A、B、C和E为优势单倍型类群,在10个省份的黄鸡品种均有分布;并呈星状分布模式,即每个单倍型类群由一个主导单倍型和一些衍生单倍型组成(图1)。单倍型类群A和B在28个黄鸡品种中均有分布。除宁都黄鸡和霞烟鸡外,单倍型类群C在其他26个鸡品种均有分布;淮北麻鸡、洪山鸡、宁都黄鸡、萧山鸡缺失单倍型类群E。单倍型类群D和G为稀有单倍型类群,D分布于淮南麻黄鸡、固始鸡、宁都黄鸡和霞烟鸡中,G只分布于固始鸡中。
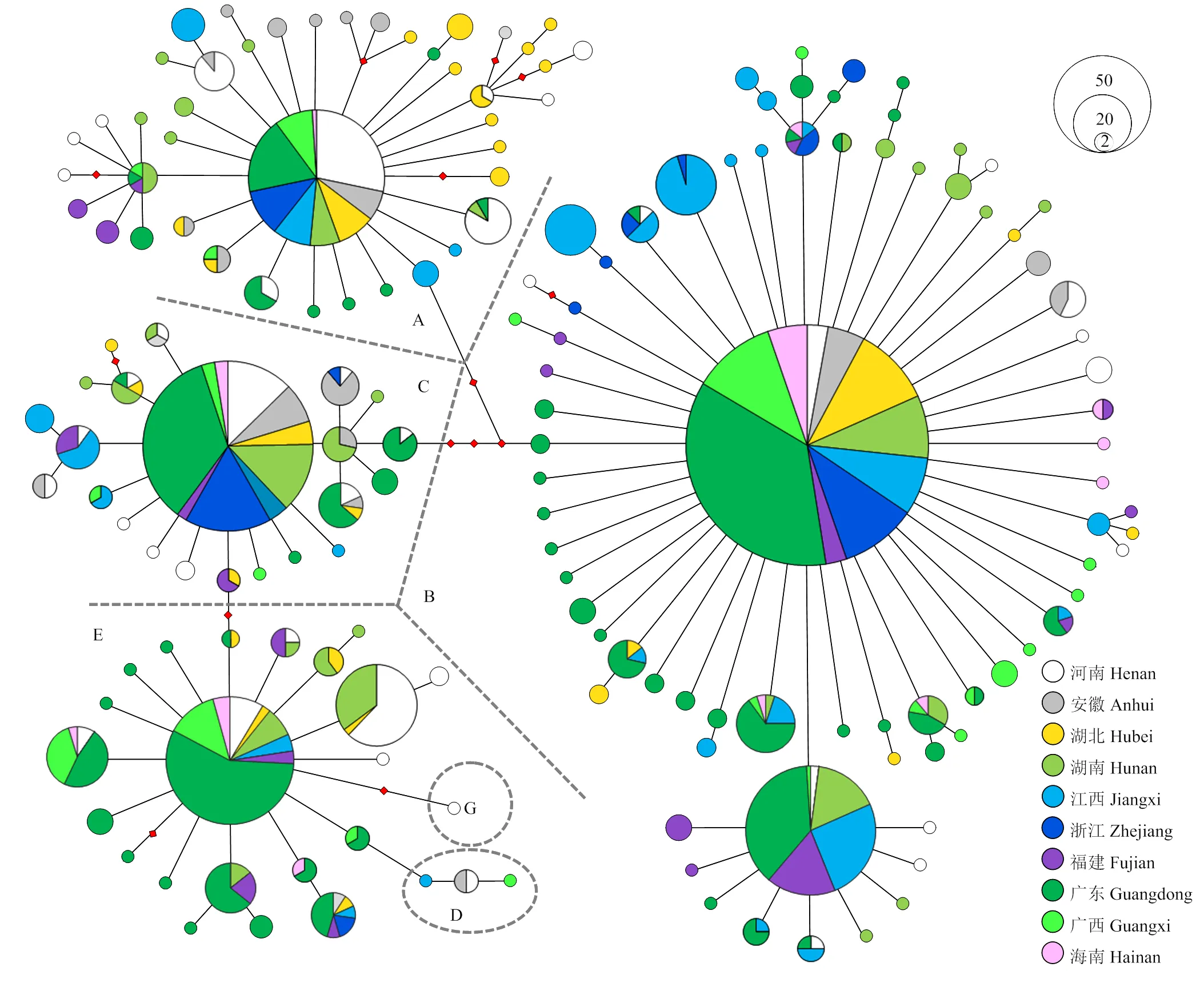
连接点数字表示核苷酸转换的位置,圆的大小对应单倍型频率,不同省份用不同颜色标注
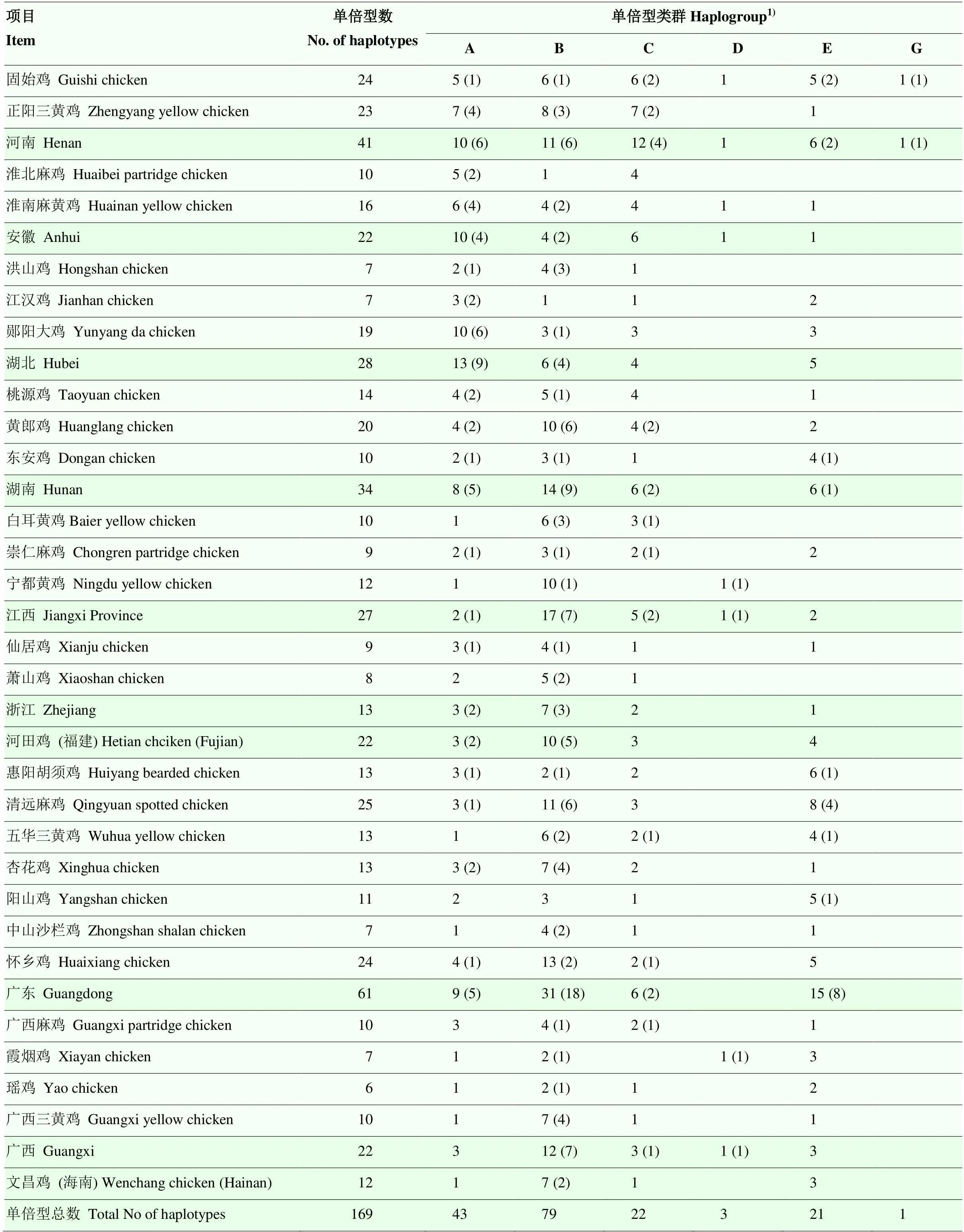
表3 28个中国南方地方品种黄鸡线粒体DNA D-loop的单倍型及其在品种中的分布
1):单倍型数量及该品种或地区的独享型单倍型(括号数值)Numbers in this bracket indicate the private haplotypes to a given breed or region
为进一步探讨黄鸡稀有单倍型类群D和G的来源,我们分析了已发表的8 076份亚洲(含本研究数据)和太平洋地区的地方鸡线粒体D-loop数据,定义了1 345个单倍型类群D个体和288个单倍型类群G个体。中国和东亚的单倍型类群D的分布频率最低,分别为1.55%和1.94%;太平洋和东南亚地区的最高,分别为74.38%和54.00%;其中东南亚的单倍型数最为丰富(附表1)。在4 953份中国地方鸡样品中,检测到76个单倍型类群D,主要分布于斗鸡、黑鸡、乌鸡和藏鸡中,其中藏鸡分布频率最高,达17.73%(50/282)(附表2)。定义的亚洲单倍型类群G个体有288个,主要分布在云南(164),其次是四川(46)、南亚(11)和东南亚(11),而广西(4)、贵州(2)和河南(1)只有少量分布。值得注意的是,藏鸡的单倍型类群G分布频率较高,达17.38%(49/282)。中介网络图显示河南和南亚的单倍型类群G可能来源于西南地区(附图1)。
2.3 遗传分化
主坐标分析显示品种间的遗传距离主要与地理位置相关,但浙江、广西壮族自治区和湖南的黄鸡品种遗传距离较为分散,如萧山鸡与仙居鸡、霞烟鸡与其他广西黄鸡、黄郎鸡与桃源鸡和东安鸡,而河田鸡与文昌鸡的遗传距离最为接近(图2)。分子变异分析显示遗传变异主要来源于品种内,其次是品种间,均显著差异;省份之间的变异很小,差异不显著(表4)。在群体遗传结构分析中,= 2时的Δ值远大于= 3或其他值(附图2)。Δ值与群体分化信号的强度呈正相关,推测= 2为最优聚类。然而,黄鸡品种内部分个体虽含有混合组分,但品种间群体分化不明显,未形成明显的群体遗传结构(图3)。
2.4 群体历史
中性检测显示只有河南、湖北和广东黄鸡的Fu’s数值达到显著水平,但Tajima’s检验数值均不显著;在品种水平上,只有萧山鸡的Tajima’s检验数值达到显著水平,而其他品种数值均不显著(表2)。基于省份(图4)和品种(附图3)的黄鸡核苷酸错配分布图均为多峰,不符合种群扩张的单峰模式。优势单倍型类群A、B和D中性检测数值达到显著水平(表2),并且核苷酸错配分布图为单峰,但单倍型类群C不显著(图4)。
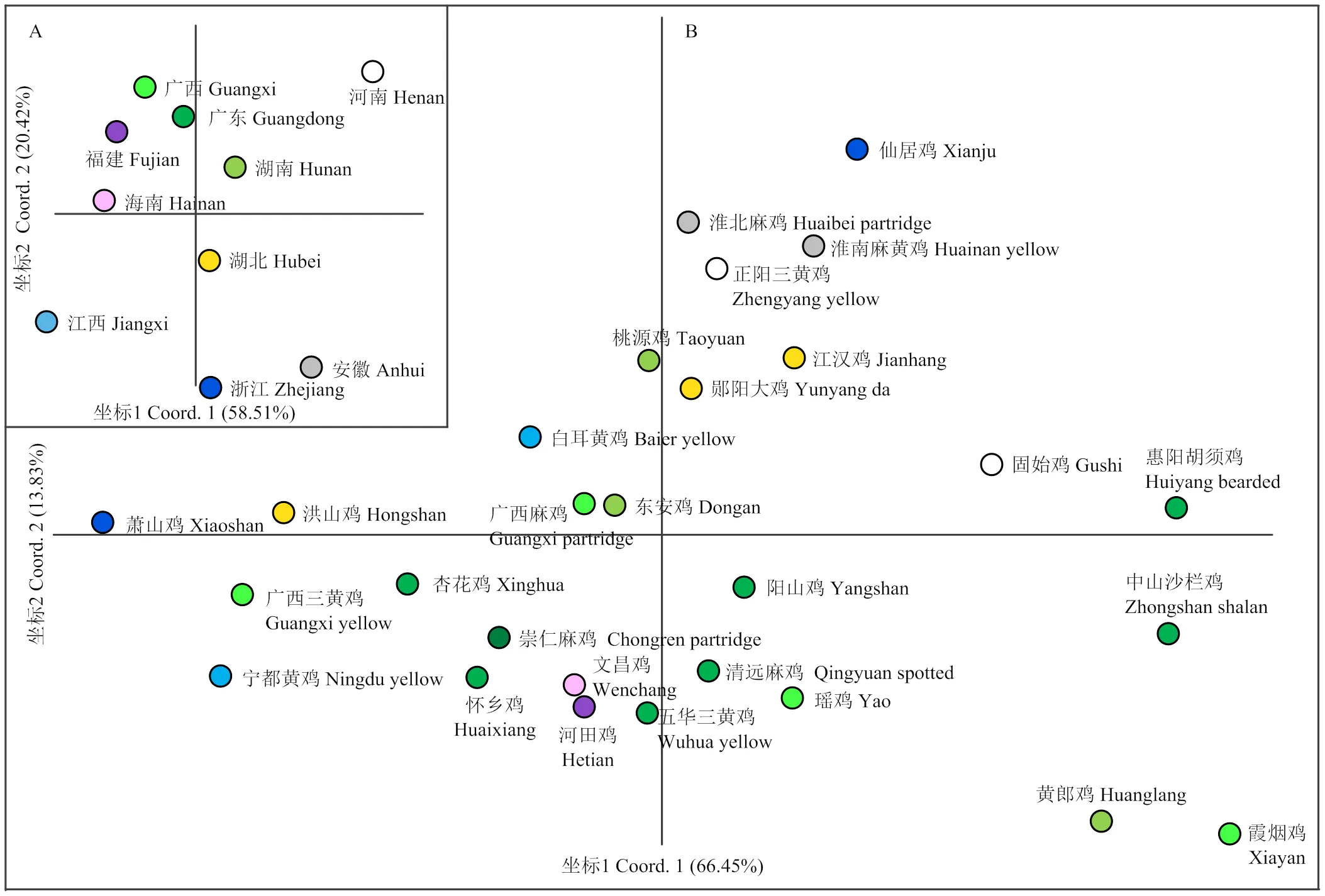
A:基于省区;B:基于品种 A: Based on provinces; B: Based on breeds
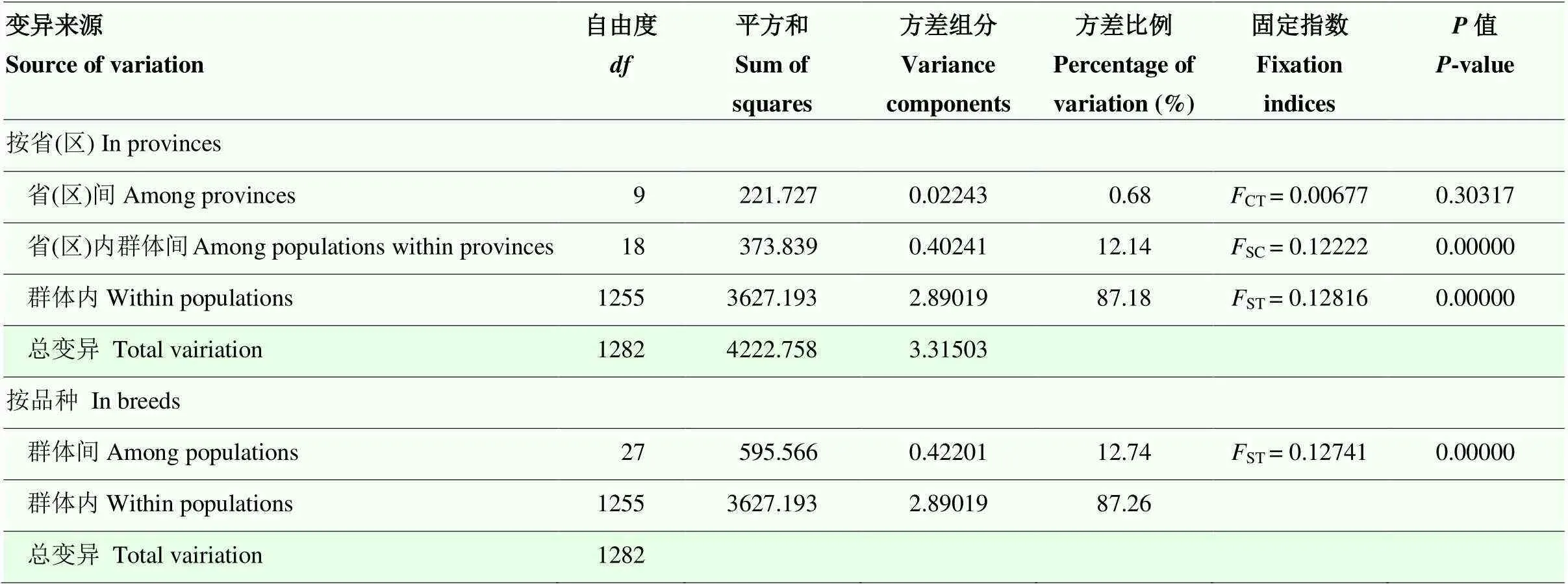
表4 28个黄鸡品种的群体遗传结构分子变异分析
CT、SC和ST分别反映组间、群体间和群体内的遗传变异
CT,SCandSTrepresent genetic differences among groups defined a priori, among populations within group, and within populations, respectively
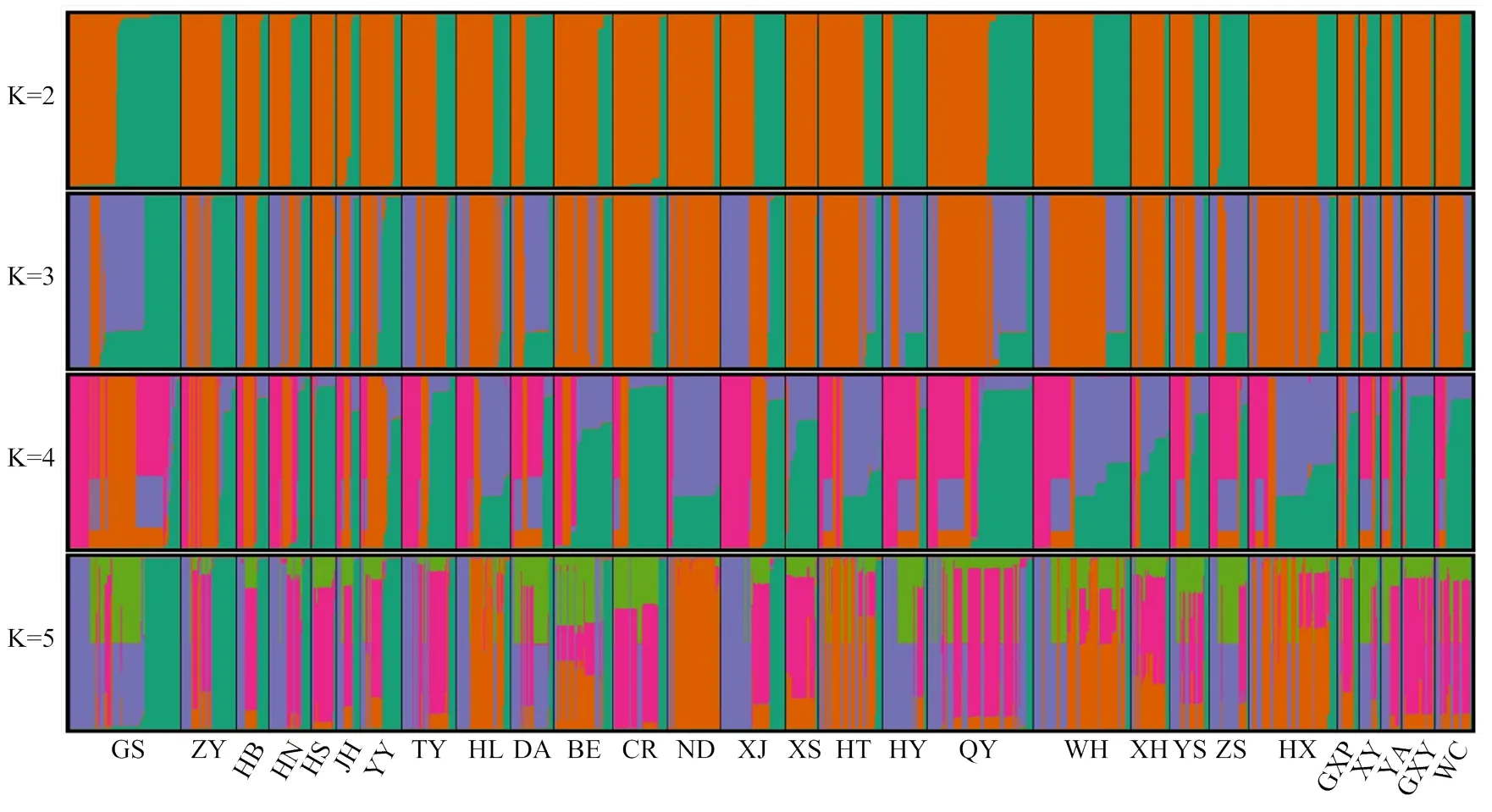
每条竖线表示每个个体被分配到给定聚类中的一个,每个品种用黑色线条分开。GS:固始鸡;ZY:正阳三黄鸡;HB:淮北麻鸡;HN:淮南麻黄鸡;HS:洪山鸡;JH:江汉鸡;YY:郧阳大鸡;TY:桃源鸡;HL:黄郎鸡;DA:东安鸡;BE:白耳黄鸡;CR:崇仁麻鸡;ND:宁都黄鸡;XJ:仙居鸡;XS:萧山鸡;HT:河田鸡;HY:惠阳胡须鸡;QY:清远麻鸡;WH:五华三黄鸡;XH:杏花鸡;ZS:中山沙栏鸡;HX:怀乡鸡;GXP:广西麻鸡;XY:霞烟鸡;YA:瑶鸡;GXY:广西三黄鸡;WC:文昌鸡
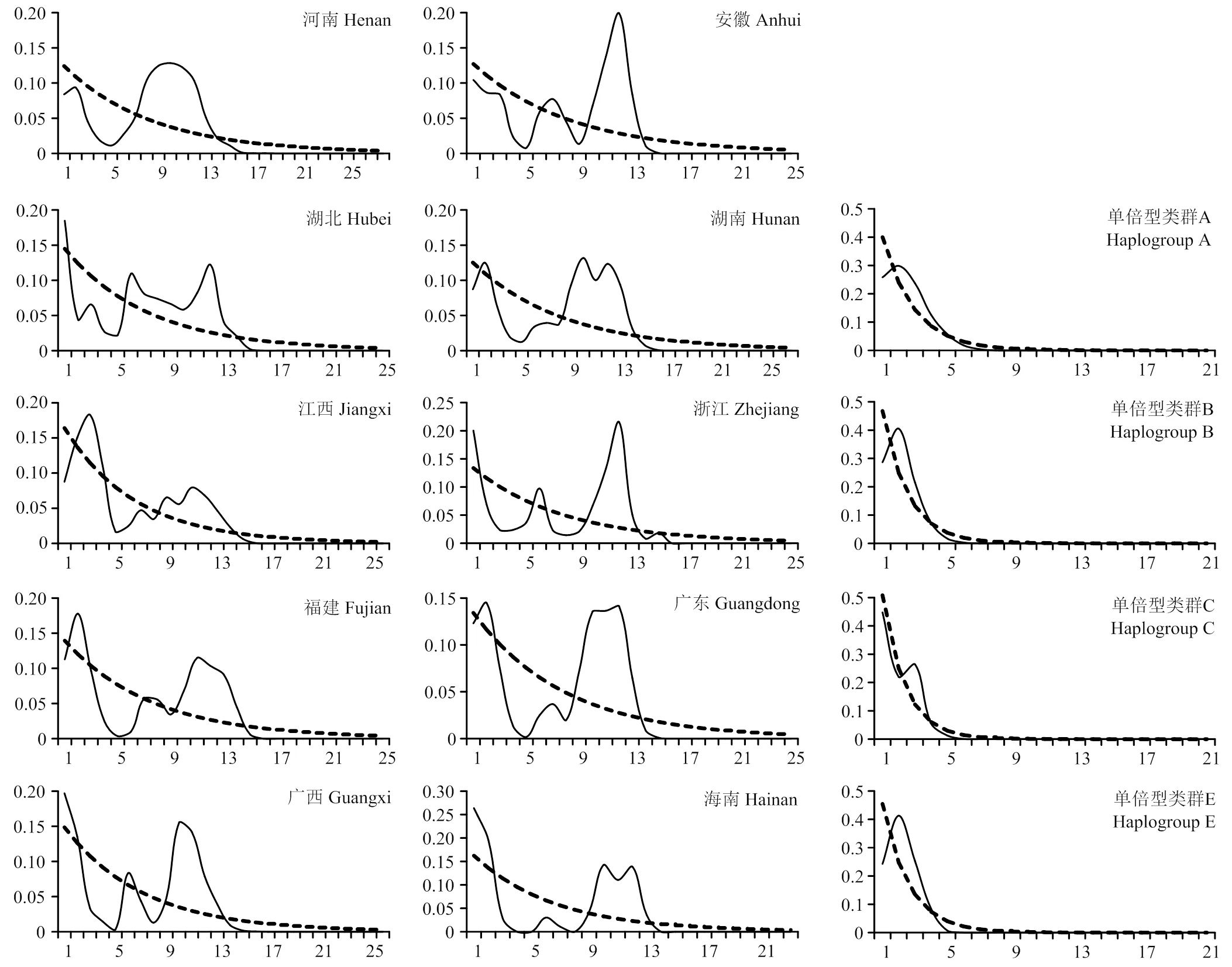
横坐标为成双配对差异,纵坐标为频率;实线表示观察值,虚线表示预测值
3 讨论
本研究应用mtDNA D-loop分子标记分析了中国南方28个地方品种黄鸡的遗传多样性和单倍型地理分布格局,探讨了品种间的遗传分化和群体历史。总体而言,这些黄鸡品种保持着较高的遗传多样性水平,但洪山鸡、萧山鸡和广西三黄鸡核苷酸多样性明显较低。洪山鸡规模较小,开发利用不充分;而广西三黄鸡分布较广,开发应用程度较高;萧山鸡近期群体数量急剧减少,在Tajima’s检验结果也得以体现,这些因素可能与这3个品种遗传多样性较低有关[1]。因此,这3个品种尤其是萧山鸡,保种工作应引起足够的重视,应采取提升保种群体数量、家系选育等措施,以逐渐提高其遗传多样性水平。
不同的样本来源、样本数量或分子标记长度差异会对研究结果造成一定的影响。如广西地方鸡,虽然在LIAO等[21]的研究中核苷酸多样性均高于本研究,但当取相同D-loop长度(nps 189—509)分析时,除了广西三黄鸡之外,其他3个品种均高于LIAO等[21]的研究。在江西地方鸡遗传多样性研究中,GAO等[22]采用D-loop全序列分析,其遗传多样性均低于本研究,这可能与D-loop的突变位点分布频率的差异有关,因为D-loop的突变位点大部分集中核苷酸序列167—446的位置;而当取相同长度分析时,仍然低于本研究的结果,如白耳黄鸡(0.00739)、崇仁麻鸡(0.00770),这可能与GAO等[22]的样本来源有关。类似的差异还有贾晓旭等[7]的研究等。这些差异不利于不同研究的平行比较分析。因此,本研究统一了D-loop分子标记长度,从整体上对中国南方主要的地方品种黄鸡进行了遗传多样性水平评估,分析结果对制定针对性的保种策略,提高保种利用率具有积极的参考作用。
单倍型类群地理格局分析有助于追溯动物的群体历史[3,23-24]。本研究来源于28个地方品种黄鸡的1 283份样本共检测到6个单倍型类,其中A、B、C和E是黄鸡常见单倍型类群,在黄鸡品种中较均匀分布,而D(0.31%)和G(0.07%)则是稀有单倍型类群,这与有关研究结果相一致[7,9,18,21-23]。单倍型类群D在中国的分布频率较低(1.53%),主要分布于斗鸡和藏鸡群体[4,18,23],但在东南亚、太平洋等海岛地区,以及马达加斯加等部分地区分布广泛且单倍型丰富,甚至可达89.13%[24-26],并且中性检测显示东南亚地区家鸡群体曾经历了显著的历史扩张。虽然太平洋等海岛地区的家鸡单倍型类群D分布频率高于其他地区,但其主要来源于东南亚[18,26-27]。中国沿海地区如广东、福建等地自古以来与东南亚交流频繁。基于此,推断黄鸡单倍型类群D可能是通过商贸等途径从东南亚地区携带而来。有趣的是,在河南固始鸡中检测到1例单倍型类群G的个体,另外两个研究中也有报道[18,28]。该单倍型集中分布在四川、云南地方鸡和藏鸡群体中,并且单倍型类群G在西南地区尤其是云南普遍分布,而在中国其他地区的地方鸡品种中均未检测到。因此,固始鸡的单倍型类群G可能是从西南地方鸡种中获得,但具体途径和时间还有待查证。
分子变异分析显示变异主要来自于品种内,品种间的遗传变异也达到显著水平,说明品种的遗传特性明显。但黄鸡的遗传结构不清晰,地区之间分化也不显著。一方面,这是持续多年保种努力的结果,说明黄鸡种群处于稳定较好的保种状态。国家于2005年12月通过了《中华人民共和国畜牧法》,并于2015年4月进行了修正,将49个家禽品种列为国家级保护品种,97个列为省级保护品种。广东省早在20世纪50年代就开展了地方鸡的种质资源调查工作,以及后续的一系列复查、再核查、反复论证和补充调查等大量工作,挖掘出一批优质鸡品种并建立了相应的保种场,制定了科学的保护策略,取得了较好的效果[9,29-30]。另一方面,由于地方鸡没有严格的特定分布/饲养范围,不同品种间可能存在混养/杂交现象,该假设得到了来自28个品种的核苷酸错配分布(典型双峰分布)和遗传结构分析的支持。并且不同品种建立保种场的时间以及重视程度也存在差异,因此地方品种黄鸡的遗传分化不明显。商品鸡在新品种(配套系)的应用对黄鸡品种的遗传特性也具有一定的影响。比如,岭南黄鸡有隐性白洛克的血统,而岭南黄鸡在华南地区的大量饲养对地方鸡有一定的渗透作用[31];鸡60K SNP芯片检测研究发现中国地方鸡存在广泛的商品鸡基因渗透现象[32]。此外,由于大部分地方品种黄鸡的分布范围和饲养规模有限,以及悠久的品种形成历史和复杂的基因交流,因此未检测到以品种/省份为单元的明显扩张现象。然而,单倍型类群A、B和E错配分布分析和中性检测表明地方品种黄鸡整体上经历了显著的群体扩张历史,形成了目前丰富的地方品种资源分布格局,但地方品种黄鸡群体历史的具体细节还需进一步研究。
由于不同分子标记的分辨率不同,对结果解析度也存在差异。D-loop突变速率较快,基因覆盖度低,遗传分化解析度不如微卫星分子标记或全基因组。本研究的线粒体DNA群体遗传分化不明显,但相同的样本在微卫星或全基因组研究中却可以明确地划分群体遗传结构。HUANG等[31]运用微卫星分子标记将12个华南家鸡分为3个明显的群体遗传结构,主要与地理位置有关。HUANG等[33]全基因组重测序分析了10个传统黄鸡的遗传结构,主成分分析(Principal component analysis)、混合分析(ADMIXTURE)和邻接系统发生树分析(Neighbor-joining phylogenetic tree)均显示这些鸡品种可明显分为北部、中部和南部,与微卫星标记分析的趋势一致[31,34]。在主坐标分析中,文昌鸡与河田鸡、五华三黄鸡等客家地区的品种关系较近,与全基因组分析结果一致[33],也与历史记录相符[1]。据记载,文昌鸡早在清代由广东、福建地区的客家移民带入杂交选育而成。因此,遗传分析结果与历史记录相结合,是对了解品种形成历史的有效方法之一。
4 结论
中国南方地方品种黄鸡线粒体DNA D-loop数据整合分析显示,大部分黄鸡群体保留着较高的遗传多样性和群体间遗传变异水平,处于稳定的保种状态,但洪山鸡、萧山鸡和广西三黄鸡应加强保种工作。中国南方地方品种黄鸡遗传组分混杂,缺乏明显的遗传结构,反映了其复杂的杂交起源历史。东南亚和中国西南地方鸡对中国南方地方品种黄鸡有一定的遗传贡献。
[1] 国家畜禽遗传资源委员会. 中国畜禽遗传资源志-家禽志. 北京: 中国农业出版社, 2011.
China National Commission of Animal Genetic Resources. Animal Genetic Resources in China: Poultry. Beijing: Chinese Agriculture Press, 2011. (in Chinese)
[2] 孟祥兵, 徐廷生, 杜炳旺, 滕小华. 鸡艺——中国古代养鸡智慧附书法艺术[M]. 北京: 中国农业出版社, 2017.
Meng X B, Xu T S, Du B W, Teng X H. Chicken art - ancient Chinese wisdom on chicken-raising with an appendix of the Chinese art of calligraphy. Beijing: Chinese Agriculture Press, 2017. (in Chinese)
[3] Lan D, Hu YD, Zhu Q, Liu Y P. Mitochondrial DNA study in domestic chicken. Mitochondrial DNA Part A: DNA Mapping, Sequencing, and Analysis, 2017, 28: 25-29.
[4] Zhang L, Zhang P, Li Q Q, Gaur U, Liu Y P, Zhu Q, Zhao X L, Wang Y, Yin H D, Hu Y D, Liu A P, Li D Y. Genetic evidence from mitochondrial DNA corroborates the origin of Tibetan chickens. PLoS ONE, 2017, 12: e0172945.
[5] WU Y P, HUO J H, XIE J F, LIU L X, WEI Q P, XIE M G, KANG Z F, JI H Y, MA Y H. Phylogeography and origin of Chinese domestic chicken. Mitochondrial DNA, 2014, 25(2): 126-130. doi:10.3109/ 19401736.2013.786704.
[6] 黄勋和, 翁茁先, 李威娜, 陈洁波, 钟福生, 唐寿桂, 赵宇旗. 基于线粒体DNAD-loop序列的黄郎鸡遗传多样性与品种起源研究. 湖南农业大学学报(自然科学版), 2016(1): 75-80.
HUANG X H, WENG Z X, LI W N, CHEN J B, ZHONG F S, TANG S G, ZHAO Y Q. Genetic diversity and breed origin of Huanglang chicken inferred from mitochondrial DNA D-loop sequence. Journal of Hunan Agricultural University (Natural Sciences), 2016(1): 75-80. (in Chinese)
[7] 贾晓旭, 唐修君, 樊艳凤, 陆俊贤, 黄胜海, 葛庆联, 高玉时, 韩威. 华东地区地方鸡品种mtDNA控制区遗传多样性. 生物多样性, 2017, 25(5): 540-548. doi:10.17520/biods.2017012.
JIA X X, TANG X J, FAN Y F, LU J X, HUANG S H, GE Q L, GAO Y S, HAN W. Genetic diversity of local chicken breeds in East China based on mitochondrial DNA D-loop region. Biodiversity Science, 2017, 25(5): 540-548. doi:10.17520/biods.2017012. (in Chinese)
[8] JIA X X, LU J X, TANG X J, FAN Y F, HUANG S H, GE Q L, GAO Y S. Genetic diversity of Jiangsu native chicken breeds assessed with the mitochondrial DNA D-loop region. British Poultry Science, 2018, 59(1): 34-39. doi:10.1080/00071668.2017.1395391.
[9] 黄勋和, 余哲琪, 翁茁先, 何丹林, 易振华, 李威娜, 陈洁波, 张细权, 杜炳旺, 钟福生. 广东省地方鸡线粒体遗传多样性与母系起源. 生物多样性, 2018, 26(3): 238-247. doi:10.17520/biods.2017259.
HUANG X H, YU Z Q, WENG Z X, HE D L, YI Z H, LI W N, CHEN J B, ZHANG X Q, DU B W, ZHONG F S. Mitochondrial genetic diversity and maternal origin of Guangdong indigenous chickens. Biodiversity Science, 2018, 26(3): 238-247. doi:10.17520/ biods.2017259. (in Chinese)
[10] 陈国宏. 中国禽类遗传资源. 上海: 上海科学技术出版社, 2004.
CHEN G H. Poul Try Genetic Resources in China. Shanghai: Shanghai Scientific & Technical Publishers, 2004. (in Chinese)
[11] FumihitoA, Miyake T, Sumi S, Takada M, Ohno S, Kondo N. One subspecies of the red jungle fowl () suffices as the matriarchic ancestor of all domestic breeds. Proceedings of the National Academy of Sciences, USA, 1994, 91: 12505-12509.
[12] 傅衍, 牛冬, 罗静, 阮晖, 何国庆, 张亚平. 中国家鸡的起源探讨. 遗传学报, 2001, 28(5): 411-417.
FU Y, NIU D, LUO J, RUAN H, HE G Q, ZHANG Y P. Studies of the origin of Chinese domestic fowls. Acta Genetica Sinica, 2001, 28(5): 411-417. (in Chinese)
[13] Kumar S, Stecher G, Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33: 1870-1874.
[14] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155(2): 945-959.
[15] RozasJ, Ferrer-Mata A, Sánchez-DelBarrio J C, Guirao-Rico S, Librado P, Ramos-Onsins S E, Sánchez- Gracia A. DnaSP 6: DNA sequence polymorphism analysis of large datasets. Molecular Biology Evolution, 2017, 34: 3299-3302.
[16] Excoffier L, Lischer H E. Arlequin suite ver. 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 2010, 10: 564-567.
[17] Peakall R, Smouse P E. GENALEX6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 2006, 6: 288-295.
[18] Miao Y W, Peng M S, Wu G S, Ouyang Y N, Yang Z Y, Yu N, Liang J P, Pianchou G, Beja-Pereira A, Mitra B, Palanichamy M G, Baig M, Chaudhuri T K, Shen Y Y, Kong Q P, Murphy R W, Yao Y G, Zhang Y P. Chicken domestication: An updated perspective based on mitochondrial genomes. Heredity, 2013, 110: 277-282.
[19] Peng M S, Fan L, Shi N N, Ning T, Yao Y G, Murphy R W, Wang W Z, Zhang Y P. DomeTree: a canonical toolkit for mitochondrial DNA analyses in domesticated animals. Molecular Ecology Resources, 2015, 15: 1238-1242.
[20] Bandelt H J, Forster P, Rohl A. Median-joining networks for inferring intraspecific phylogenies. Molecular Biology and Evolution, 1999, 16: 37-48.
[21] Liao Y Y, Mo G D, Sun J L, Wei F Y, Liao D J. Genetic diversity of Guangxi chicken breeds assessed with microsatellites and the mitochondrial DNA D-loop region. Molecular Biology Reports, 2016, 43: 415-425.
[22] Gao Y S, Jia X X, Tang X J, Fan Y F, Lu J X, Huang S H, Tang M J. The genetic diversity of chicken breeds from Jiangxi, assessed with BCDO2 and the complete mitochondrial DNA D-loop region. PLoS ONE, 2017, 12: e0173192.
[23] Huang XH, Wu YJ, Miao YW, Peng MS, Chen X, He DL, Suwannapoom C, Du BW, Li XY, Weng ZX, Jin SH, Song JJ, Wang MS, Chen JB, Li WN, Otecko NO, Geng ZY, Qu XY, Wu YP, Yang XR, Jin JQ, Han JL, Zhong FS, Zhang XQ, Zhang YP. Was chicken domesticated in northern China? New evidence from mitochondrial genomes. Science Bulletin, 2018, 63: 743-746.
[24] Herrera M, Thomson V A, Wadley J J, Piper P J, Sulandari S, Dharmayanthi A B, Kraitsek S, Gongora J, Austin J J. East African origins for Madagascan chickens as indicated by mitochondrial DNA. Royal Society Open Science, 2017(4): 160787.
[25] Dancause K N, Vilar M G, Steffy R, Lum J K. Characterizing genetic diversity of contemporary pacific chickens using mitochondrial DNA analyses. PloS ONE, 2011, 6(2): e16843.
[26] Langford S M, Kraitsek S, Baskerville B, Ho S Y W, Gongora J. Australian and Pacific contributions to the genetic diversity of Norfolk Island feral chickens. BMC Genetics, 2013, 14: 91.
[27] Thomson V A, Lebrasseur O, Austin J J, Hunt T L, Burney D A, Denham T, Rawlence N J, Wood J R, Gongora J, Flink G L, Linderholm A, Dobney K, Larson G, Cooper A. Using ancient DNA to study the origins and dispersal of ancestral Polynesian chickens across the Pacific. Proceedings of the National Academy of Sciences, USA, 111(13): 4826-4831.
[28] Liu Y P, Wu G S, Yao Y G, Miao Y W, Luikart G, Baig M, Beja-Pereira A, Ding Z L, Palanichamy M G, Zhang Y P. Multiple maternal origins of chickens: Out of the Asian jungles. Molecular Phylogenetics and Evolution, 2006, 38: 12-19.
[29] 瞿浩, 舒鼎铭, 杨纯芬. 广东优质鸡种质资源的现状及发展建议. 广东农业科学, 2004, 31(6): 4-7.
Qu H, Shu D M, Yang C F. The actuality and development proposal in Guangdong quality chicken germplasm resources. Guangdong Agricultural Sciences, 2004, 31(6): 4-7. (in Chinese)
[30] 张细权, 梁少东, 张德祥. 广东优质鸡新品种选育回顾. 中国家禽, 2006, 28(1): 9-11.
Zhang X Q, Liang S D, Zhang D X. Review on new breed selection of quality chicken in Guangdong Province. China Poultry, 2006, 28(1): 9-11. (in Chinese)
[31] Huang X H, Zhang J F, He D L, Zhang X Q, Zhong F S, Li W N, Zheng Q M, Chen J B, Du B W. Genetic diversity and population structure of indigenous chicken breeds in South China. Frontiers of Agricultural Science and Engineering, 2016, 3: 97-101.
[32] Zhang C Y, Lin D, Wang Y Z, PENG D Z, LI H F, FEI J, CHEN K W, YANG N, HU X X, ZHAO Y Q, LI N. Widespread introgression in Chinese indigenous chicken breeds from commercial broiler. Evolutionary applications, 2019, 12(3): 610-621.
[33] Huang X H, Otecko N O, Peng M S, Weng Z X, Li W N, Chen J B, Zhong M, Zhong F S, Jin S H, Geng Z Y, Luo W, He D L, Ma C, Han J L, Ommeh S C, Zhang Y P, Zhang X Q, Du B W. Genome-wide genetic structure and selection signatures for color in 10 traditional Chinese yellow-feathered chicken breeds. BMC Genomics, 2020, 21: 316.
[34] Qu L J, Li X Y, Xu G F, Chen K W, Yang H J, Zhang L C, Wu G Q, Hou Z C, Xu G Y, Yang N. Evaluation of genetic diversity in Chinese indigenous chicken breeds using microsatellite markers. Science in China Series C: Life Science, 2006, 49: 332-341.
Genetic Diversity of Indigenous Yellow-Feathered Chickens in Southern China Inferred from Mitochondrial DNA D-Loop Region

1Jiaying University/Guangdong Provincial Key Laboratory of Conservation and Precision Utilization of Characteristic Agricultural Resources in Mountainous Areas/Guangdong Science and Technology Innovation Centre of Wuhua Yellow Chicken, Meizhou 514015, Gugangdong;2College of Animal Sciences, South China Agricultural University, Guangzhou 510642
【Objective】Indigenous yellow-feathered chicken is an important poultry genetic resource in China. The genetic diversity of these chickens was evaluated to facilitate scientific protection policies and to improve their use.【Method】 A total of 1283 mitochondrial DNA fragments (mtDNA D-loop, 519 bp) were investigated including 694and 589 previously published units from 28 indigenous yellow-feathered chicken breeds from southern China. These data were subsequently used to calculate the genetic diversity, to construct median-joining networks of haplotypes, and to investigate geographical distribution patterns of haplotypes by using principal coordinate analysis, analysis of molecular variance, and neutral test. The origin of rare haplogroups was inferred from the geographical distribution pattern of mitochondrial DNA haplogroups of Asian and Pacific indigenous chickens.【Result】One hundred and one mutation sites were detected in 1 283 samples, of which 92 were polymorphic. One hundred and sixty-nine haplotypes, belonging to haplogroups A-E and G, were defined. The predominant haplogroups were A (15.43%), B (49.26%), C (18.55%), and E (16.37%), while D (0.31%) and G (0.08%) were the rare haplogroups within the total samples. Chickens from Henan Province covered all six haplogroups; Chickens from Guangdong Province had the largest number of haplotypes (64), whereas those from Zhejiang (19) and Hainan (12) Provinces had the lowest. Haplogroups A and B occurred in all 28 breeds. Haplogroup D existed only in Huainan yellow, Gushi, Ningdu yellow and Xiayan chickens, whereas haplogroup G only occurred in Gushi chickens. The distribution frequency of haplogroup D and the number of haplotypes indicated that haplogroup D of indigenous yellow-feathered chickens in southern China could originated from Southeast Asia. The evidence from geographical distribution and median-joining networks of haplogroup G indicated that haplogroup G of chickens from Henan Province and South Asia could originated from Southwest China. The indigenous yellow-feathered chickens in southern China had a total haplotype diversity of 0.903±0.005 and nucleotide diversity of 0.01269±n.d. Indigenous yellow-feathered chickens from Henan and Hunan Provinces had the highest level of haplotype diversity of 0.916±0.011 and 0.01358±0.00039, respectively, and the nucleotide diversity of 0.913±0.012 and 0.01345±0.00042, respectively. The lowest level of haplotype diversity was found in Hainan Province (0.736±0.076), and the lowest level of nucleotide diversity was found in Jiangxi Province (0.00981±0.00072). Huainan yellow, Gushi and Yunyang da chickens retained the highest level of haplotype diversity; Jianghan, Huainan yellow and Huanglang chickens had the highest level of nucleotide diversity, whereas the haplotype diversity and nucleotide diversity in Hongshan, Guangxi yellow and Xiaoshan chickens were the lowest. Wenchang chickens were genetically close to Hentian chickens, which was consistent with their breeding history. The population genetic structure of yellow-feathered chickens was less clear, and the molecular variance component of within-population was significantly higher than others. The neutral test indicated that the yellow-feathered chickens in southern Chinadid not sustain obvious population expansion at the breed level, with the exception of haplogroups A, B and E.【Conclusion】The results suggested that yellow-feathered chickens had a good conservation status with high level of genetic diversity, although Hongshan, Xiaoshan and Guangxi yellow chickens required further protection. Hybridization between chickens was common in yellow-feathered chickens, which had experienced population expansion. Indigenous chickens from Southeast Asia and Southwest China had substantial genetic contributions to indigenous yellow-feathered chickens in southern China.
yellow-feathered chicken; mitochondrial DNA D-loop; genetic variation; genetic divergence; demographic history

10.3864/j.issn.0578-1752.2022.22.016
2021-09-13;
2021-12-16
广东省自然科学基金(2014A030307018)、广东省公益研究与能力建设项目(2016A030303068)、广东省高等教育“冲补强”提升计划重点建设学科(农业资源与环境)建设项目(粤教科函[2018]181号)、梅州市应用型科技专项资金项目(2019B0201003、2019B0201006)
黄勋和,E-mail:hxh826@126.com。通信作者杜炳旺,E-mail:dudu903@163.com
(责任编辑 林鉴非)
