基于BSA-Seq技术的甜瓜抗霜霉病InDel标记开发
2022-01-18凌悦铭李寐华杨文莉伊鸿平张学军
凌悦铭,李寐华,杨 永,杨文莉,范 蓉,伊鸿平,张 红,张学军
(新疆农业科学院哈密瓜研究中心,乌鲁木齐 830091)
0 引 言
【研究意义】甜瓜(CucumismeloL., 2n= 2x= 24)是国内外重要的园艺作物和经济作物,据联合国粮农组织数据库(FAOSTAT)统计(http://www.fao.org/faostat/ zh/#data/QC),2015~2019年,我国的甜瓜种植面积从35.09×104hm2增加到38.37×104hm2,占全球甜瓜种植面积的36.91 %。甜瓜具有栽培周期短、市场需求量大、经济效益好、土地利用率和复种指数高等优点[1]。【前人研究进展】甜瓜霜霉病是一种由古巴假霜霉菌[Pseudoperonosporacubensis(Berk.&Curt.)Rostov.]引起的真菌性病害,主要危害甜瓜叶片,发病初期在叶片上形成水浸状小斑点,病斑扩展后,受叶脉限制呈黄褐色角斑,病斑背面伴有灰黑色霉层产生,最后叶片大面积发黄甚至枯焦。该病原菌孢子可随气流远距离传播,寄主范围非常广泛、容易迅速对杀菌剂产生抗药性、流行性强、防治难[2]。据不完全统计,世界上有17 %杀菌剂用于霜霉病的防治[3]。P.cubensis引起的作物经济损失在欧洲、中东和亚洲等许多地区都有报道[4]。甜瓜霜霉病流行年份可使甜瓜减产30 %~50 %,严重70 %~80 %,甚至绝产[5]。随着高通量测序技术的发展,下一代测序(next-generation sequencing, NGS)技术为挖掘功能标记或目标性状连锁标记及基因快速定位提供了一个新的有效手段。基于全基因组测序的集团分离分析法(Bulked segregant analysis, BSA)在不构建遗传图谱的情况下能高效快速地挖掘目的基因。BSA- Seq技术已被广泛应用在大田和蔬菜作物研究中,已经成功定位出水稻耐盐[6]、水稻稻瘟病[7]、黄瓜早花[8]、番茄果实重量[9]、大豆疫霉病[10]等基因。【本研究切入点】鉴于BSA- Seq技术能高效快速地挖掘目的基因,研究拟通过BSA- Seq技术定位甜瓜抗霜霉病基因,开发与抗病基因紧密连锁的分子标记或功能标记,以期在甜瓜苗期对育种材料进行分子辅助选择,提高筛选效率,加快育种进程,解决甜瓜霜霉病危害问题。【拟解决的关键问题】以野生高抗霜霉病资源DM-4和新疆感病自交系DM-2为亲本,构建F2抗病性分离群体,对抗、感亲本和2个极端表型子代混合池进行全基因组测序分析,利用SNP(单核苷酸多态性,Single nucleotide polymorphism)频率差异分析方法对抗霜霉病性状标记进行QTL(数量性状基因座,Quantitative trait locus)定位并关联候选区间,开发与目标性状紧密连锁的分子标记,应用于甜瓜抗病分子育种,也为精细定位甜瓜抗霜霉病基因及研究甜瓜抗霜霉病的分子机理奠定基础。
1 材料与方法
1.1 材 料
于2019~2021年在新疆农业科学院哈密瓜研究中心海南三亚育苗大棚进行。抗源DM-4(P1),引自美国农业部国家植物种质资源体系,现新疆农业科学院哈密瓜中心保存,高抗霜霉病,绿皮带墨绿斑、圆球型、软肉、网纹稀不全、品质差;感病自交系DM-2(P2)由新疆农业科学院哈密瓜研究中心提供,高感霜霉病、黄皮、脆肉、网纹粗密、风味好、口感佳。P1×P2杂交获得F1,F1自交获得1 000个F2单株。从中挑选50个抗病单株和50个感病单株构建极端抗、感基因池,用于霜霉病抗性基因定位。
古巴假霜霉菌[Pseudoperonosporacubensis(Berk.&Curt.)Rostov.]采自海南三亚地区早期自然发病的甜瓜叶片,单孢子分离后扩繁备用。
1.2 方 法
1.2.1 甜瓜霜霉病抗性的苗期人工接种鉴定
甜瓜霜霉病抗性鉴定材料包括F2群体单株及其双亲,其中双亲P1和P2各30株,F2群体单株1000株,于2019年10月鉴定。鉴定前将种子于68 ℃高温杀菌后再播入装有基质的营养钵中,基质由椰糠、有机肥和黄沙按体积8:1:1比例混合均匀而成,基质和营养钵都经熏蒸灭菌杀毒。
接种方法采用喷雾法[11]进行接种,接种浓度为5×103/mL个孢子,接种时期在甜瓜苗期第2片真叶完全展开时,16:00以后进行喷雾接种,用微型喷雾器将配制好的孢子悬浮液均匀喷于植株叶片正反面上,直到流水为止,接种后黑暗保湿24 h(相对湿度为80%~90 %),之后正常管理,夜间保湿,温度控制在25~30 ℃。接种后15 d调查发病情况。参照Criswell(2008)[12]和Epinat C[13,14]的抗性分级标准,并适当修正,将病情分为6个级别:0级:无任何症状;1级:病斑面积占整个叶片面积的1/10以下;2级:病斑面积占整个叶片面积的1/10~1/4;3级:病斑面积占整个叶片面积的1/4~2/4;4级:病斑面积占整个叶片面积的2/4~3/4;5级:病斑面积占整个叶片面积的3/4以上或者叶片干枯。病情指数(DI)计算方法:DI=∑(每个病级的植株数×级别数)/(总植株数×最高级别数)×100。抗性分级标准:高抗(HR):0<病情指数≤10;抗病(R):10<病情指数≤20;中抗(MR):20<病情指数≤40;感病(S):40<病情指数≤60;中感(MS):60<病情指数≤80;高感(HS):病情指数>80。
1.2.2 基因组DNA提取及测序分析
在F2群体中随机选取50株高抗单株和50株高感单株,分别取其幼嫩叶片0.5~1.0 g于液氮中研磨成粉末状,装入2.0 mL的离心管。用PlantZol试剂(北京全式金)提取2个亲本和F2群体单株叶片基因组DNA,并用核酸检测仪进行检测,统一稀释至15 ng/L后置于-80 ℃冰箱保存备用。将高抗单株和高感单株幼嫩叶各50份DNA分别等量混合,构建极端抗、感池。使用 Illumina HiSeq测序平台,对2个亲本和2个子代池分别进行全基因组重测序(委托北京诺禾致源生物信息科技有限公司)。参考基因组为甜瓜基因组(http://cucurbitgenomics.org/organism/18)。采用GATK3.3软件进行SNP及InDel的检测。
1.2.3 SNP检测及关联分析
使用bwa软件[15]将2个亲本重测序获得的有效测序数据reads与参考基因组比对。采用GATK3.3软件的Unified Genotyper模块进行多个样本SNP的检测,使用Variant Filtration进行过滤,过滤参数为-cluster Window Size 4,-filter Expression "QD < 4.0 || FS > 60.0 || MQ < 40.0 " ,-G_filter "GQ<20"。基于基因分型的结果,筛选亲本中纯和的SNP位点,以亲本DM-2为参考亲本,分析计算2个子代池在亲本间标记位点的SNP的频率(SNP-index)。其中,完全与其相同的SNP-index为0;完全与其不同的SNP-index为1。为减少测序错误和比对错误造成的影响,对计算出SNP-index后的亲本多态性位点进行过滤,过滤标准如下:(1)过滤掉两个子代中SNP-index 都小于0.3并且SNP深度都小于7的位点;(2)过滤掉一个子代SNP-index缺失的位点。计算(SNP-index),即两个子代SNP-index作差:(SNP- index)= SNP-index(极端性状B)- SNP-index(极端性状A)。进行1000次置换检验,选取95 %和99 %置信水平作为筛选的阈值。结合抗、感池SNP-indexes的信息,针对基因组位置绘制(SNP-index)图,分析获得候选区间。
1.2.4 InDel标记开发及有效性检测
采用GATK3.3软件的Unified Genotyper模块进行InDel的检测,使用Variant Filtration进行过滤,过滤参数为-filter Expression "QD < 4.0 || FS > 200.0"。在上述获得的候选区间内筛选插入或缺失片段大于4 bp的InDel位点,根据亲本的InDel信息,截取包含InDel位点上下游各100 bp的序列,并根据上下游序列设计引物,Tm为(58±3)℃,引物长度为(20±5)bp,PCR产物大小为200~350 bp,引物由上海生工生物科技有限责任公司合成。设计好的InDel引物在 2个亲本基因组DNA中进行多态性筛选,用具有多态性扩增的InDel引物在 F2群体上进行检测。
PCR反应总体系为:1 μL上下游引物(10 μmol/L)、1 μL DNA模板、5 μL 2×TaqMaster Mix,ddH2O补足至10 μL。PCR反应程序为:PCR 程序为94 ℃ 3 min;94 ℃ 30 s;57 ℃ 30 s;72 ℃ 30 s,共 30 个循环;72 ℃ 5 min。PCR 产物经8 %的聚丙烯酰胺凝胶电泳,银染法检测电泳结果。
2 结果与分析
2.1 甜瓜霜霉病的苗期抗性鉴定
研究表明,2019年10月,抗源DM-4病情指数为0,近似免疫;DM-2表现为高感,病情指数为90.00;对 F2群体病情级别绘制频数分布图,群体的病情级别分布图呈现正态分布。图1
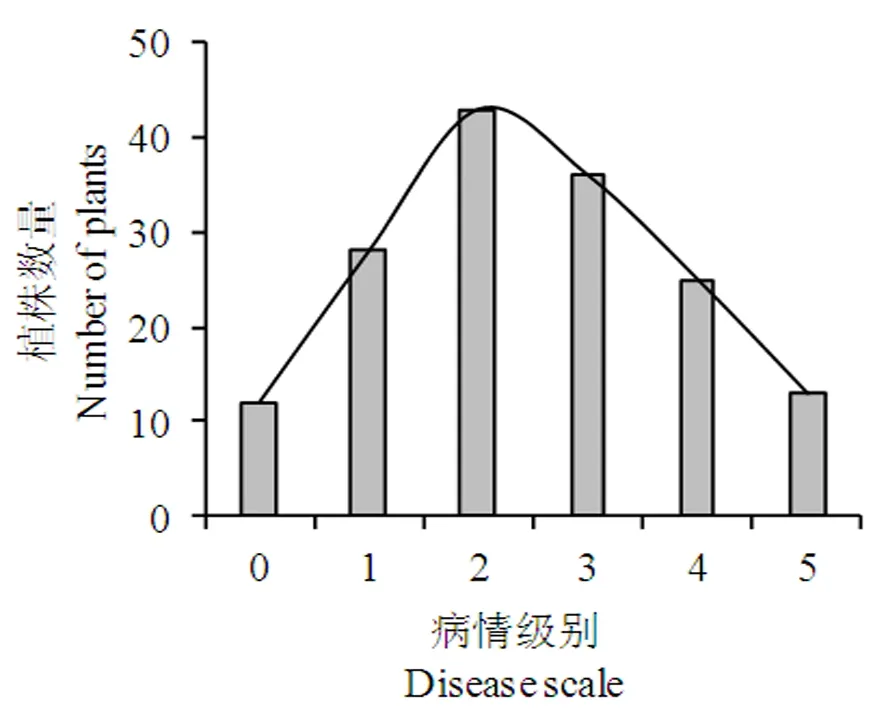
图1 病情级别在F2群体的频率分布Fig.1 Frequency distnbution of diseaseseverity in F2 population
2.2 测序数据
研究表明,对从F2选择50株高抗单株和50株高感单株分别构建高抗池和高感池,与2个亲本通过Illumina HiSeq重测序,过滤后得到的碱基数目为30.276 G,测序质量高(Q20≥97.74 %、Q30≥94.94 %),GC含量在37.12%~37.39 %,样本与参考基因组比对效率在92.28 %~93.4 %之间,平均覆盖深度为11.57~21.29X,1X覆盖度(至少有1个碱基的覆盖)为96.37%。所有样本的数据量足够,测序质量合格,GC分布正常,建库测序成功,与甜瓜参考基因组比对效率较高,可用于后续变异检测及目标性状的基因定位。表1,2
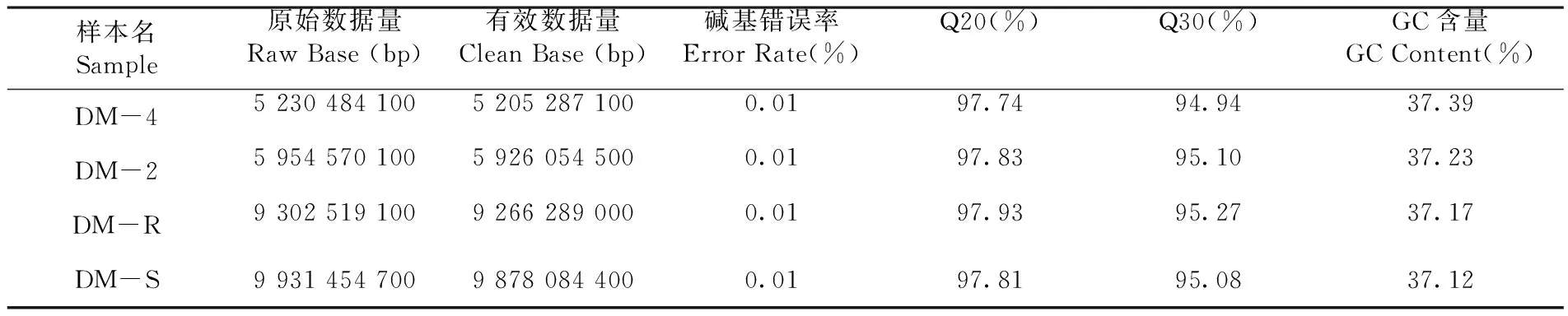
表1 测序数据质量情况汇总Table 1 The quality of sequencing data
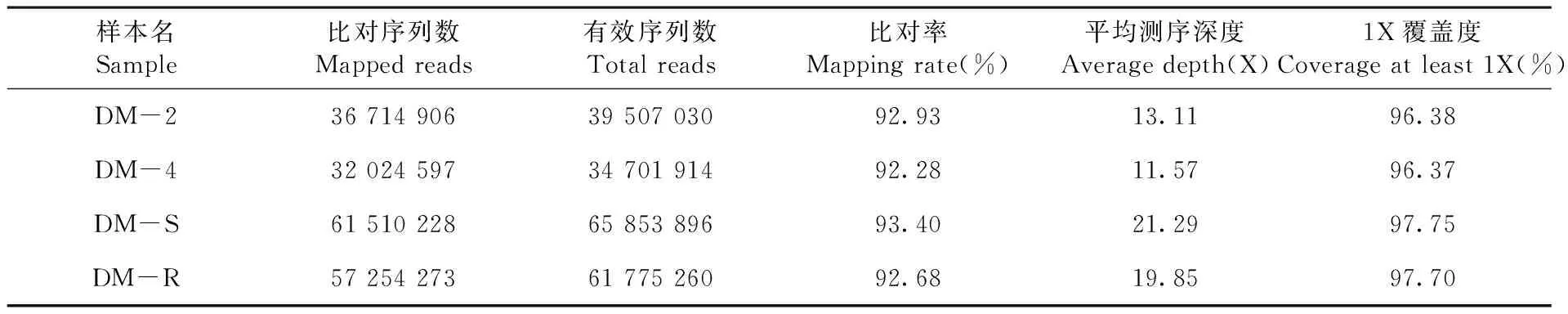
表2 测序深度及覆盖度统计Table 2 Sequencing depth and coverage
2.3 关联分析
研究表明,抗、感池之间存在117×104个不同SNPs。进一步过滤SNP,得到高质量可信SNP位点95 886个。采用SNP-index算法对这些高质量SNP进行关联,绘制2个子代池的( SNP-Index)的分布如图,甜瓜9号染色体21~24 Mb、10号染色体5~6 Mb、7~11 Mb、12~13 Mb和12号染色体21~23 Mb基因组位置的区域(SNP-Index)超过0.95和0.99置信度,是抗霜霉病基因的候选区间。其中,在9号染色体上关联到的区域(SNP-Index)值置信度达0.99,是甜瓜抗霜霉病主效QTL。图2

注:橙色曲线代表置信度为0.95的阈值线,绿色曲线代表置信度为0.99的阈值线
2.4 与甜瓜抗霜霉病基因连锁InDel标记的开发及筛选
研究表明,在9号染色体候选区域内共检测到2 882个InDel位点。以50 kb为步移区段,寻找附近碱基缺失4 bp以上的InDel位点,合成37对InDel引物。以高抗霜霉病资源DM-4和感病自交系DM-2的基因组DNA为模板,对37对引物的多态性进行PCR筛选。37对引物均能扩增出与预测大小一致的目标条带,其中9对引物具有多态性,多态率为32.14 %。图2,表3

注:M:Marker I;P1:以高抗霜霉病资源DM-4的DNA为模板进行PCR扩增;P2:以感病自交系DM-2的DNA为模板进行PCR扩增
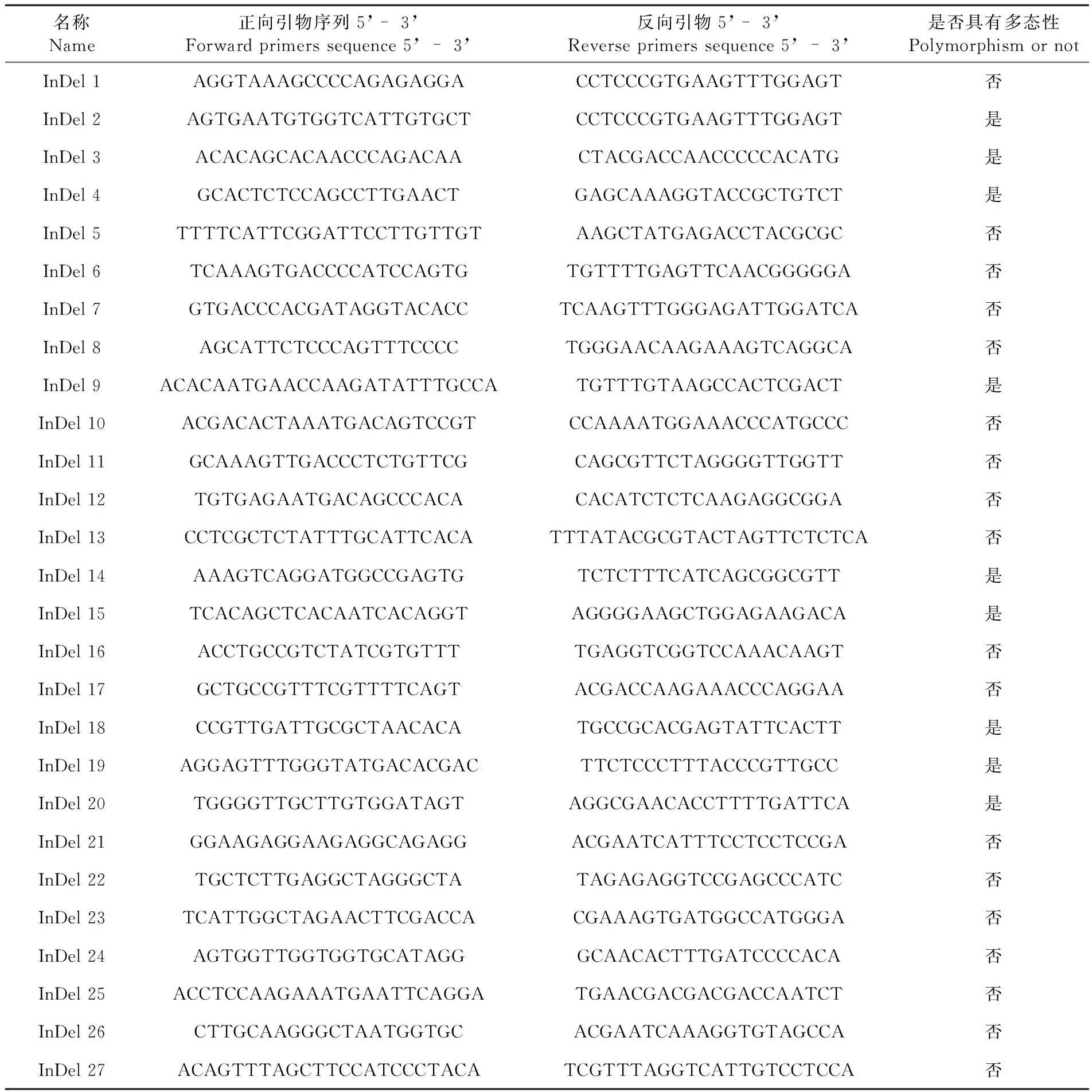
名称Name正向引物序列5’- 3’Forward primers sequence 5’ - 3’反向引物5’- 3’Reverse primers sequence 5’ - 3’是否具有多态性Polymorphism or notInDel 1AGGTAAAGCCCCAGAGAGGACCTCCCGTGAAGTTTGGAGT否InDel 2AGTGAATGTGGTCATTGTGCTCCTCCCGTGAAGTTTGGAGT是InDel 3ACACAGCACAACCCAGACAACTACGACCAACCCCCACATG是InDel 4GCACTCTCCAGCCTTGAACTGAGCAAAGGTACCGCTGTCT是InDel 5TTTTCATTCGGATTCCTTGTTGTAAGCTATGAGACCTACGCGC否InDel 6TCAAAGTGACCCCATCCAGTGTGTTTTGAGTTCAACGGGGGA否InDel 7GTGACCCACGATAGGTACACCTCAAGTTTGGGAGATTGGATCA否InDel 8AGCATTCTCCCAGTTTCCCCTGGGAACAAGAAAGTCAGGCA否InDel 9ACACAATGAACCAAGATATTTGCCATGTTTGTAAGCCACTCGACT是InDel 10ACGACACTAAATGACAGTCCGTCCAAAATGGAAACCCATGCCC否InDel 11GCAAAGTTGACCCTCTGTTCGCAGCGTTCTAGGGGTTGGTT否InDel 12TGTGAGAATGACAGCCCACACACATCTCTCAAGAGGCGGA否InDel 13CCTCGCTCTATTTGCATTCACATTTATACGCGTACTAGTTCTCTCA否InDel 14AAAGTCAGGATGGCCGAGTGTCTCTTTCATCAGCGGCGTT是InDel 15TCACAGCTCACAATCACAGGTAGGGGAAGCTGGAGAAGACA是InDel 16ACCTGCCGTCTATCGTGTTTTGAGGTCGGTCCAAACAAGT否InDel 17GCTGCCGTTTCGTTTTCAGTACGACCAAGAAACCCAGGAA否InDel 18CCGTTGATTGCGCTAACACATGCCGCACGAGTATTCACTT是InDel 19AGGAGTTTGGGTATGACACGACTTCTCCCTTTACCCGTTGCC是InDel 20TGGGGTTGCTTGTGGATAGTAGGCGAACACCTTTTGATTCA是InDel 21GGAAGAGGAAGAGGCAGAGGACGAATCATTTCCTCCTCCGA否InDel 22TGCTCTTGAGGCTAGGGCTATAGAGAGGTCCGAGCCCATC否InDel 23TCATTGGCTAGAACTTCGACCACGAAAGTGATGGCCATGGGA否InDel 24AGTGGTTGGTGGTGCATAGGGCAACACTTTGATCCCCACA否InDel 25ACCTCCAAGAAATGAATTCAGGATGAACGACGACGACCAATCT否InDel 26CTTGCAAGGGCTAATGGTGCACGAATCAAAGGTGTAGCCA否InDel 27ACAGTTTAGCTTCCATCCCTACATCGTTTAGGTCATTGTCCTCCA否
续表3 InDel引物序列信息Table 3 The information of InDel primers
2.5 InDel标记在F2分离群体中的鉴定
研究表明,利用抗病亲本DM-4、感病亲本DM-2以及F2群体中随机选取的5株极端抗病和5株极端感病单株DNA作为模板,聚丙烯酰胺电泳反复试验验证这9对引物的多态性。引物InDel 15和InDel 20在F2群体中随机选择的5个极端抗病单株和5个极端感病单株之间多态性高、条带清晰、扩增条带重演性好。进一步利用引物InDel 15和InDel 20在F2分离群体的100个单株中进行PCR鉴定,引物InDel 15在50个极端抗病单株中,有46个单株的基因型检测结果为抗病亲本带型,条带大小为251 bp;在50个极端感病单株中,有49个单株基因型鉴定结果为感病亲本带型,条带大小为349 bp。引物InDel 20在50个极端抗病单株中,49个单株的基因型检测结果为抗病亲本带型,条带大小为231 bp;在50个极端感病单株中,有49个单株基因型鉴定结果为感病亲本带型,条带大小为324 bp。该两个InDel标记条带能区分出F2群体中极端抗、感单株,并且和亲本带型高度一致,其中引物InDel 15准确率为95 %,InDel 20准确率为98 %。图4
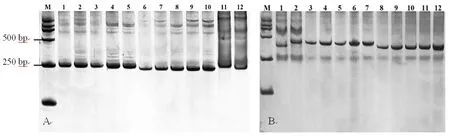
注:图2A为InDel 15引物扩增结果,其中11:抗霜霉病亲本DM-4,12:感霜霉病亲本DM-2,1~5:抗病单株,6~10:感病单株;图2B为InDel 20引物扩增结果,其中2:抗霜霉病亲本DM-4,1:感霜霉病亲本DM-2,3~7:抗病单株,8~12:感病单株;M均为:DL2000 DNA Marker
3 讨 论
世界上对甜瓜霜霉病抗性研究较早,与其他葫芦科作物相比,已经在甜瓜上报道了抗霜霉病材料[16]。Ivanoff[17]报道了4个高抗霜霉病的栽培品种。随后相继发现许多对霜霉病有较好抗性的甜瓜材料。研究选用的母本材料DM-4为野生高抗霜霉病资源,当受到霜霉病病原菌侵染时,侵染点附近会产生胼胝质,阻止菌丝的延伸,抑制病原菌的生长,这与文献[18]选用的高抗品种抗病表现一致。
目前已有学者进行了霜霉病抗性基因染色体定位,并开发出多个与霜霉病抗性相关的分子标记。杨柳燕等[19]以高抗品种DM3和高感品种DF4为材料,获得了与抗霜霉病基因连锁的SRAP标记me8em11,连锁距离为9.8 cM;贺玉花等[20]以抗病品种PI 414723为研究材料,发现了3个与抗霜霉病基因Pc-3连锁的SSR标记DE1887、DE1320和DE0854,遗传距离分别为26、26和13.69 cM。张学军等[21]结合SSR技术和QTL分析,检测到3个霜霉病抗性基因的QTL位点:qR1-1-1、qR2-2-1、qR1-3-1,分别位于chr-5、chr-9和chr-10染色体上。研究利用BSA- seq方法,检测到高抗野生资源DM-4的抗霜霉病位点定位在9号染色体上,这与张学军等[22]研究结果一致。同时,在10号染色体和12号染色体上也关联到候选区间,该区域(SNP-Index)值置信度为0.95。
InDel分子标记是不同个体间基因组同一位点的序列发生碱基的插入或缺失现象,具有在基因组内分布广、密度高、变异稳定、多态性强、检测容易等优点[23],在甜瓜果斑病[24]、蔓枯病[25]和白粉病[26]抗性鉴定中已有研究,但是利用InDel标记鉴定甜瓜霜霉病抗性的研究还没有相关报道。因此,开发甜瓜InDel标记对于准确鉴定甜瓜霜霉病抗性、快速培育抗病种质和品种具有重要意义。研究利用BSA-seq技术,在抗霜霉病候选区间内InDel位点设计37对InDel引物,9对引物具有多态性,其中引物InDel 15和InDel 20特异性好且条带清晰,检测F2群体单株时,扩增产物带型在抗病单株和感病单株之间的差异效果明显、稳定且准确率高,分别为95 %和98 %,可以用于甜瓜霜霉病抗性鉴定及分子标记辅助选择。同时,研究开发的与抗霜霉病基因连锁的InDel分子标记,缩小了抗霜霉病基因候选区间。今后将在此基础上进一步结合F2:3家系的基因型和表型进行精细定位,克隆抗霜霉病基因并探索在抗霜霉病功能基因上直接开发分子标记,这些共分离的功能标记可直接在苗期进行霜霉病抗性鉴定。
4 结 论
将甜瓜抗霜霉病QTL定位在9号染色体21~24 Mb,10号染色体5~6 Mb、7~11 Mb、12~13 Mb和12号染色体21~23 Mb候选区间。在9号染色体上关联到的区域(SNP-Index)值置信度达0.99,为主效QTL,并在此区域开发了InDel 15和InDel 20连锁标记,准确率分别为95%和98%。
