大厂茶紫芽品系P113不同季节花青素调控相关基因表达分析
2021-12-11刘霞李芳宋勤飞牛素贞吕立堂
刘霞,李芳,宋勤飞,牛素贞,吕立堂
大厂茶紫芽品系P113不同季节花青素调控相关基因表达分析
刘霞,李芳,宋勤飞,牛素贞*,吕立堂
贵州大学茶学院,贵州 贵阳 550025
为明确大厂茶紫芽品系P113在不同季节花青素积累的分子机理及合成途径上相关基因的表达特点,对春、夏、秋3个季节的P113一芽二叶进行转录组(RNA-Seq)和代谢组(UHPLC-MS/MS)分析。结果显示,检测到的10种花青苷衍生物含量随季节变化,其中4种随春、夏、秋季节变化呈上调表达,与其叶色表现一致;结构基因表达模式基本一致,均在夏季上调表达,在秋季的表达量与夏季无显著差异;酶基因获得的2个差异表达基因均在夏季上调表达;表达模式相似,均在夏季上调表达,且各有1个基因在秋季呈现下调表达;2个基因中有1个在夏季下调表达,1个随季节变化上调表达;的13个基因均在夏季上调表达,而在秋季呈现不同表达模式;修饰基因在夏季和秋季均出现两种表达模式;仅获得1个差异表达基因,随季节变化上调表达;3个基因中有1个在夏季下调表达,在秋季上调表达,另2个仅在夏季上调表达,在秋季的表达量与夏季无显著差异;获得的7个差异表达基因均在夏季上调表达,其中2个在秋季下调表达;调控基因随季节变化上调表达,对花青素合成有正向调控作用。研究表明,大厂茶紫芽品系P113在不同季节结构基因、修饰基因和调控基因的表达具有一定的时间特性,导致了花青素积累差异。
大厂茶;花青素;差异表达基因
茶树是世界三大主要饮料作物之一,具有重要的经济价值。随着人们生活水平的提高,功能性、特色茶树品种越来越受到关注。紫芽茶树品种作为一种富含花青素的特色茶树资源,其保健功能与高含量的花青素紧密相关,随着花青素的功能被逐渐阐明,选育富含花青素的特色紫芽茶树品种受到研究人员的广泛关注[1-3]。近年来在贵州发现了大量大茶树资源,表型及生化组分分析发现,这些资源的遗传多样性丰富,在品质、抗性、产量等相关性状上具有很好的优势。本课题组前期研究筛选出的紫芽品系P113,其芽叶性状与目前已知的紫芽品种存在显著差异,在分类上属于大厂茶(F. CZhang)。茶树花青素积累量与芽叶颜色深浅呈正相关[4],茶树叶片花青素的合成、积累和降解随叶片发育而变化,受时间、空间和内外因子综合影响。尽管有一些研究对茶树花青素代谢进行了初步的探讨,部分参与花青素合成的基因已被克隆验证,但茶树花青素代谢途径是一个复杂的网络结构,受到多个代谢途径的共同影响,由一系列的酶催化完成[5-7]。此外,不同紫芽资源的内含成分以及叶色调控机理都存在明显差异,因此很有必要对特异茶树资源芽叶的叶色调控机理进行探索,为后续的育种应用提供理论基础。目前高通量测序、基因芯片、代谢组分析和蛋白质组分析等技术已被应用到茶树花青素合成相关功能基因发掘领域[8],筛选到多个与茶树次生代谢、转录调控等生物学进程相关的差异基因,并克隆了编码类黄酮-甲基转移酶和类黄酮3'-羟化酶的基因[9-11]。
为进一步揭示花青素合成的遗传机制,本研究以大厂茶变异紫芽品系P113为材料,采用新一代高通量测序技术(RNA-Seq),研究其芽叶在不同季节发育过程中紫化程度和花青素生物合成相关基因的表达情况,筛选出茶树花青素合成途径相关基因,并进行功能注释,解析花青素生物合成及类黄酮生物代谢途径的转录机制,预测控制花青素合成的差异基因,并对差异基因进行GO与KEGG富集分析,为今后进行茶树叶片花青素合成相关基因的克隆和功能验证奠定基础。
1 材料与方法
1.1 材料
以紫芽品系P113为材料,选自普安县野生型茶树群体种,种植在普安县贵州大学实验基地内。其表型特征为乔木型,子房5室、无毛,柱头5裂,果实梅花形,顶芽无毛,花白色,直径5.8 cm,叶片革质、长14.2 cm、宽6.8 cm(图1)。材料常规水肥管理,于取材前半年进行修剪后封园,于3月中旬(春)、5月中旬(夏)、8月上旬(秋)分别采集长势良好、无病虫害的一芽二叶作为试验材料,各个时期材料取两组,一组微波制备干样用于代谢组检测,一组液氮速冻超低温冰箱保存用于转录组测序,试验设3个生物学重复。
1.2 花青素组分测定及总RNA提取
花青素测定参考李智等[12]方法。大厂茶总RNA提取参照Tri-Reagent试剂盒(武汉贝纳科技服务有限公司)说明书进行。从大厂茶芽叶中提取总RNA,采用琼脂糖凝胶电泳测试RNA质量,用Beads[带有Oligod(T)]对mRNA进行纯化,用Qubit RNA Assay Kit进行起始总RNA准确定量,采用Agilent 2100 BioAnalyzer检测RNA的完整性。取完整性好(28 S∶18 S约为2∶1)、A260∶A280为1.8~2.0、质量浓度≥200 mg·L-1的RNA样品,置于–80℃冰箱备用。
1.3 cDNA文库构建与RNA-Seq
选择质量合格的总RNA作为mRNA测序的建库起始样品,Illumina HiSeq TM2000测序平台对库检合格的文库进行测序获得raw reads,过滤低质量reads后得到clean reads。以舒茶早基因组作为参考基因组,用Trinity软件通过序列之间的重叠信息组装得到contigs,局部组装得到transcripts,用Tgicl和Phrap软件对transcripts进行同源聚类和拼接得到unigene。文库构建与测序皆由武汉贝纳科技服务有限公司完成。
1.4 基因差异表达分析
使用HTSeq软件统计样品的基因表达水平[13],样本合理性选择皮尔森相关系数的平方(2)检验[14],采用TMM对过滤后的数据进行标准化[15],采用DESeq对检测得到的基因进行差异表达分析[16],差异基因的筛选采用FPKM法,筛选阈值<0.005及|log2Fold change|>1,log2Fold change>0,认为是上调表达,反之为下调表达[17]。筛选出的差异表达基因结果用火山图进行直观表示。
2 结果与分析
2.1 花青素代谢物组分分析
紫芽品系P113在春、夏、秋3个季节的芽叶颜色存在明显的差异(图2)。通过对紫芽品系P113的花青素进行检测,得到10种花青苷衍生物(表1)。夏季,P113中的矢车菊素-3--咖啡酰槐糖苷-7--葡萄糖苷、矢车菊素-乙酰基葡萄糖、矢车菊素--丁香酸、矢车菊素-3--葡萄糖苷、飞燕草素-3--葡萄糖苷、矢车菊素-3,5--二葡萄糖苷、飞燕草素-3--芸香糖苷、天竺葵素-3--葡萄糖苷含量均高于春季;飞燕草素-3--(3'',6''-二丙二酰葡萄糖苷)和锦葵色素-3--葡萄糖苷含量低于春季。秋季,P113中矢车菊素-3--咖啡酰槐糖苷-7--葡萄糖苷、矢车菊素-乙酰基葡萄糖、飞燕草素-3--(3'',6''-二丙二酰葡萄糖苷)、锦葵色素-3--葡萄糖苷、飞燕草-3--葡萄糖苷、天竺葵素-3--葡萄糖苷等含量高于夏季,属于上调表达,其余花青苷衍生物含量均低于夏季。
2.2 转录组测序质量分析
紫芽品系P113春、夏、秋季叶片RNA-Seq分析结果显示(表2),去除带接头和质量低的reads后,获得clean reads范围为54 903 686~85 965 732个。碱基识别错误率均为0.01%,Q20(质量值≥20的碱基所占百分比)占整个reads长度的97.89%以上,Q30(质量值≥30的碱基所占百分比)占整个reads长度的94.03%以上,GC含量在43.16%~44.24%,测序质量较好,符合进一步分析要求。

图1 紫芽品系P113花、果、叶片形态特征

图2 紫芽品系P113不同季节的表型变化
2.3 差异表达基因相关性分析
皮尔森(Pearson)相关性系数是检验试验可靠性和样本选择是否合理的重要指标,相关系数越接近1,样品的相似度越高。对春、夏、秋3个季节基于差异表达基因的样品间皮尔森相关性分析显示(表3),春季和夏季、春季和秋季、夏季和秋季之间相关系数大于0.8,均为显著或极显著相关。3个季节样品生物学重复之间相关系数均在极显著范围。差异表达基因样品相关性显示,试验的重复性与可靠性均满足进一步分析的要求。
2.4 不同季节基因差异表达分析
通过对紫芽品系P113的春、夏、秋3个季节转录组差异表达分析,将相近两个季节转录组数据进行比对,筛选不同季节差异表达基因。结果显示,与春季相比,夏季有3 819个差异表达基因,其中2 524个上调,1 295个下调;与夏季相比,秋季有1 768个差异表达基因,其中958个上调,810个下调(图3)。利用KEGG数据库,将差异表达基因进行功能注释和富集分析,夏季的差异表达基因共注释得到294个代谢通路,主要涉及光合作用、细胞周期、黄酮生物合成、花青素生物合成、异黄酮生物合成及苯丙氨酸代谢和苯丙氨酸生物合成等;秋季的差异表达基因共注释得到272个代谢通路,主要涉及调节脂肪分解、cAMP信号通路、黄酮生物合成、花青素生物合成、异黄酮生物合成、苯丙氨酸代谢及苯丙氨酸生物合成等。通过筛选得到3个与花青素合成有关的代谢途径,分别为苯丙氨酸代谢、黄酮生物合成和花青素生物合成,共获得90个差异表达基因。对差异表达基因进行TPIA(http://teaplant.org)茶树基因组数据库比对,比对到参考基因组的有53个,被注释为14种酶基因,分别是黄酮生物合成途径中的10种酶基因,苯丙氨酸代谢途径中的4种酶基因,花青素合成途径中的1种,未比对到参考基因组的差异表达基因有37个。
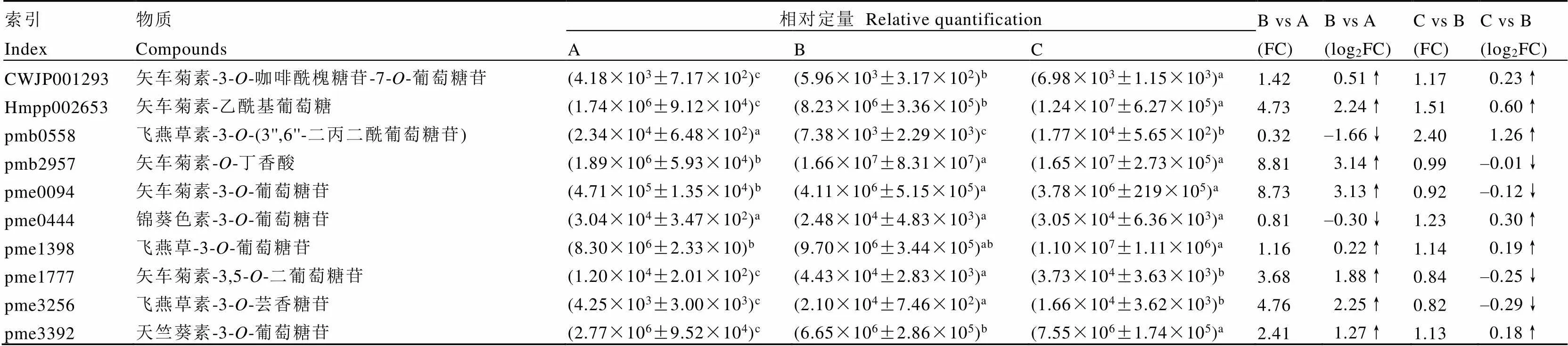
表1 不同季节P113的花青素组分含量分析
注:相对定量,通过每个单个峰的面积计算相对定量。A:春季淡紫色芽叶。B:夏季紫红色芽叶。C:秋季深紫色芽叶。同行不同字母表示差异显著(<0.05);B vs A:夏季与春季相比。C vs B:秋季与夏季相比。FC:差异倍数。↑表示上调,↓表示下调
Note: Relative quantification is calculated by the area of each discrete peak. A: lilac buds and leaves, spring. B: purple buds and leaves, summer. C: dark purple buds and leaves, autumn. The different lowercase letters in the same line indicate significant difference (<0.05). B vs A: summer compared with spring. C vs B: autumn compared with summer. FC: multiple of difference. ↑ indicates up regulation, ↓ indicates down regulation
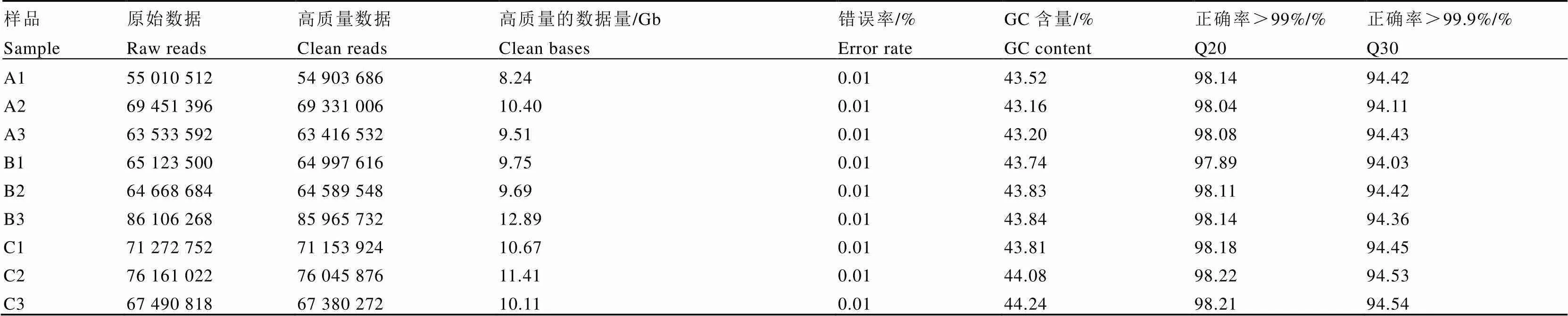
表2 紫芽品系P113转录组测序数据质量分析
注:A1、A2和A3:春季3个生物学重复。B1、B2和B3:夏季3个生物学重复。C1、C2和C3:秋季3个生物学重复。下同
Note: A1, A2 and A3: three biological repetitions in spring. B1, B2 and B3: three biological repeats in summer. C1, C2 and C3: three biological repeats in autumn. The same below
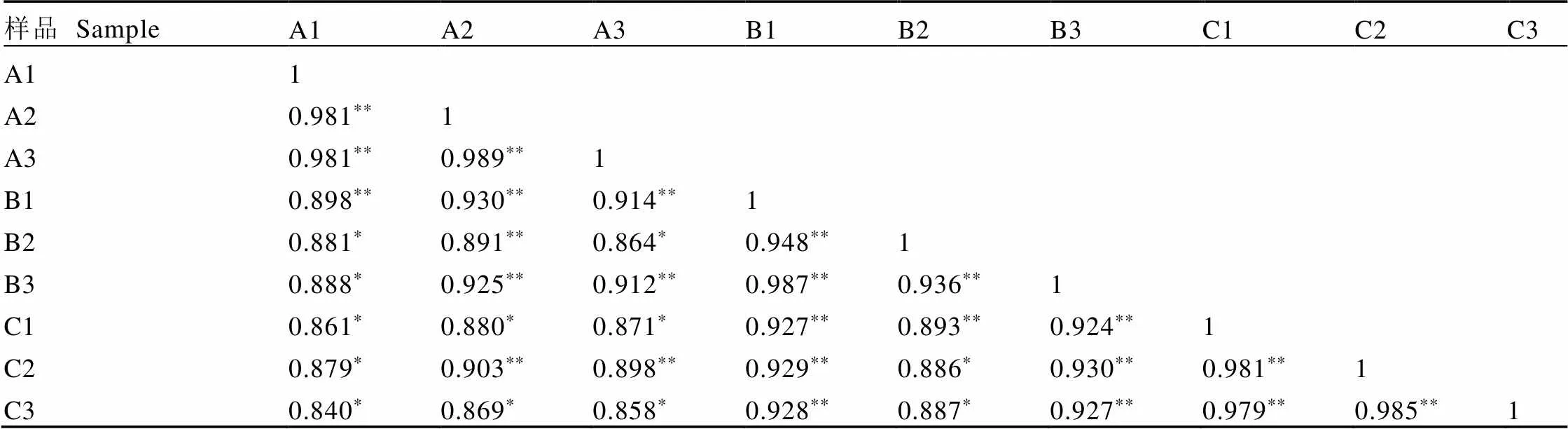
表3 样品间的皮尔森相关系数
注:*表示显著差异(<0.05),**表示极显著差异(<0.01)
Note: * indicates a significant difference (<0.05), ** indicates a very significant difference (<0.01)
2.5 花青素合成结构基因的表达分析
对紫芽品系P113不同季节花青素合成相关酶基因进行差异表达分析,结果表明(表4),共获得10个差异表达的基因,其表达模式基本一致,大多在夏季上调表达,在秋季与夏季无显著差异。酶基因获得2个差异表达的基因,其中TEA008357随着季节变化表达量显著升高,与花青素含量的积累情况基本一致;TEA027829的表达量夏季高于春季,秋季与夏季差异不显著。酶基因表达模式基本一致共获得9个差异表达的基因,其中6个差异表达基因在夏季上调表达,而在秋季与夏季的表达无显著差异;TEA016716、TEA010588、TEA000503基因则在夏季上调表达,在秋季下调表达。基因有2个差异表达,其中TEA010843在夏季下调表达,TEA034016基因随季节变化上调表达。基因共获得13个差异表达的基因,其中TEA002405、TEA013443、TEA014951、TEA020629、TEA023729基因的表达量在夏季上调表达,在秋季的表达量与夏季差异不显著;TEA010598、TEA024235、TEA025230、TEA033871基因在夏季上调表达,秋季下调表达;TEA010762、TEA018444、TEA023937、TEA023946基因与花青素积累情况相似,随着季节的变化呈增加趋势。
2.6 花青素合成修饰基因的表达分析
对紫芽品系P113不同季节芽叶中花青素合成修饰基因的表达分析表明(表5),共有8个差异表达基因,其中TEA015167、TEA027538基因随季节变化上调表达,夏季表达量高于春季,秋季表达量高于夏季;TEA006624、TEA015016在夏季下调表达,在秋季的表达量与夏季无显著差异;TEA015375基因在夏季上调表达,在秋季的表达量与夏季差异不显著;TEA025989基因在夏季上调表达,秋季时下调表达;TEA024761和TEA026458基因在夏季的表达量与春季无显著差异、而秋季时下调表达。只获得1个差异表达基因TEA022960,该基因随季节变化呈上调表达。共有3个差异表达的基因,TEA022238基因在夏季下调表达,在秋季上调表达;TEA025792和TEA025793基因在夏季上调表达,在秋季的表达量与夏季无显著差异。共有7个差异表达基因,其中TEA000392、TEA012735、TEA018304、TEA023870、TEA030958基因在夏季时表达量均高于春季,夏、秋季差异表达不显著;TEA022732、TEA029331基因夏季上调表达,秋季下调表达。
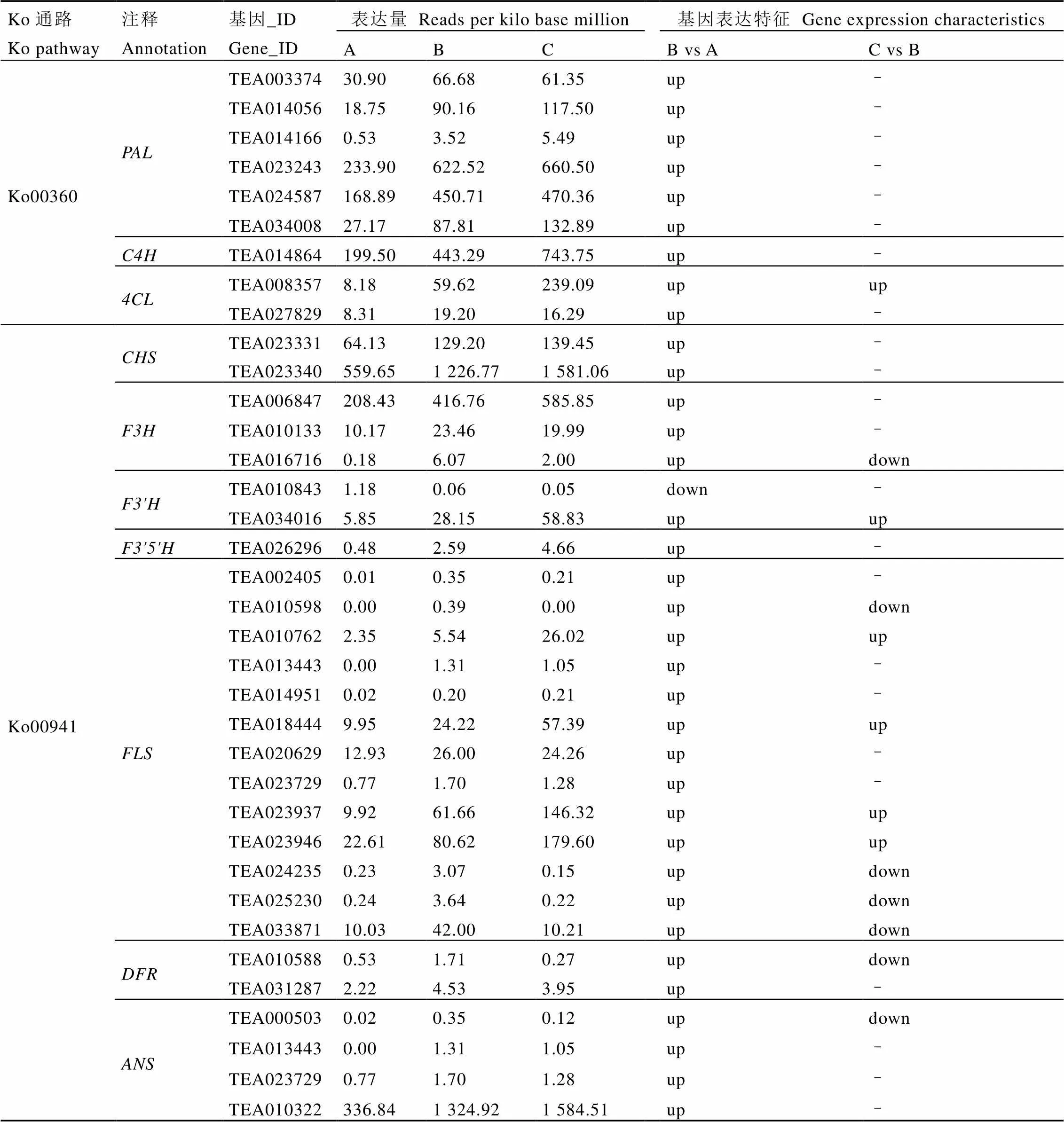
表4 不同季节花青素合成结构基因的表达
注:“up”为上调表达,“down”为下调表达,“–”为差异不显著。下同
Note: “up” means up-regulated expression, “down” means down-regulated expression, and “-“ means no significant. The same below
2.7 花青素合成调控基因的表达分析
对花青素合成相关调控因子、的表达分析表明(表6),在中获得7个差异基因,其中TEA016380、TEA017105基因随季节变化上调表达;TEA000421、TEA032260基因在夏季上调表达,秋季和夏季的表达量无显著差异;TEA000833、TEA019380、TEA032433基因在春季和夏季的表达无显著差异,秋季表达量相对夏季上调。蛋白中有10个差异基因,TEA028476、TEA026204基因随季节变化上调表达;TEA029605、TEA029615、TEA014193、TEA014430、TEA018997基因在春季和夏季的表达无显著差异,在秋季时上调表达;TEA033191、TEA014311、TEA011367基因在夏季上调表达,秋季的表达量与夏季无显著差异。
3 讨论
本研究利用UHPLC-MS/MS对大厂茶紫芽品系P113不同季节芽叶中花青素的代谢物组分进行检测,在紫芽品系P113芽叶中检测出10种花青苷衍生物,并对其进行分析。结果显示,大厂茶紫芽品系P113中10种花青苷衍生物含量随季节变化,在夏季时除了飞燕草素-3--(3'',6''-二丙二酰葡萄糖苷)和锦葵色素-3--葡萄糖苷相对含量减少外,其他8种花青苷组分含量均高于春季;在秋季时只有5种花青苷含量高于夏季。在10种花青苷衍生物中矢车菊素-3--咖啡酰槐糖苷-7--葡萄糖苷、矢车菊素-乙酰基葡萄糖、飞燕草-3--葡萄糖苷和天竺葵素-3--葡萄糖苷4种花青苷含量分别在夏季和秋季增加,在秋季时含量最高,这与蒋会兵等[18]和Shen等[19]的研究结果一致,这4种花青苷衍生物可能是影响大厂茶紫芽品系P113在不同季节紫化程度不同的原因。
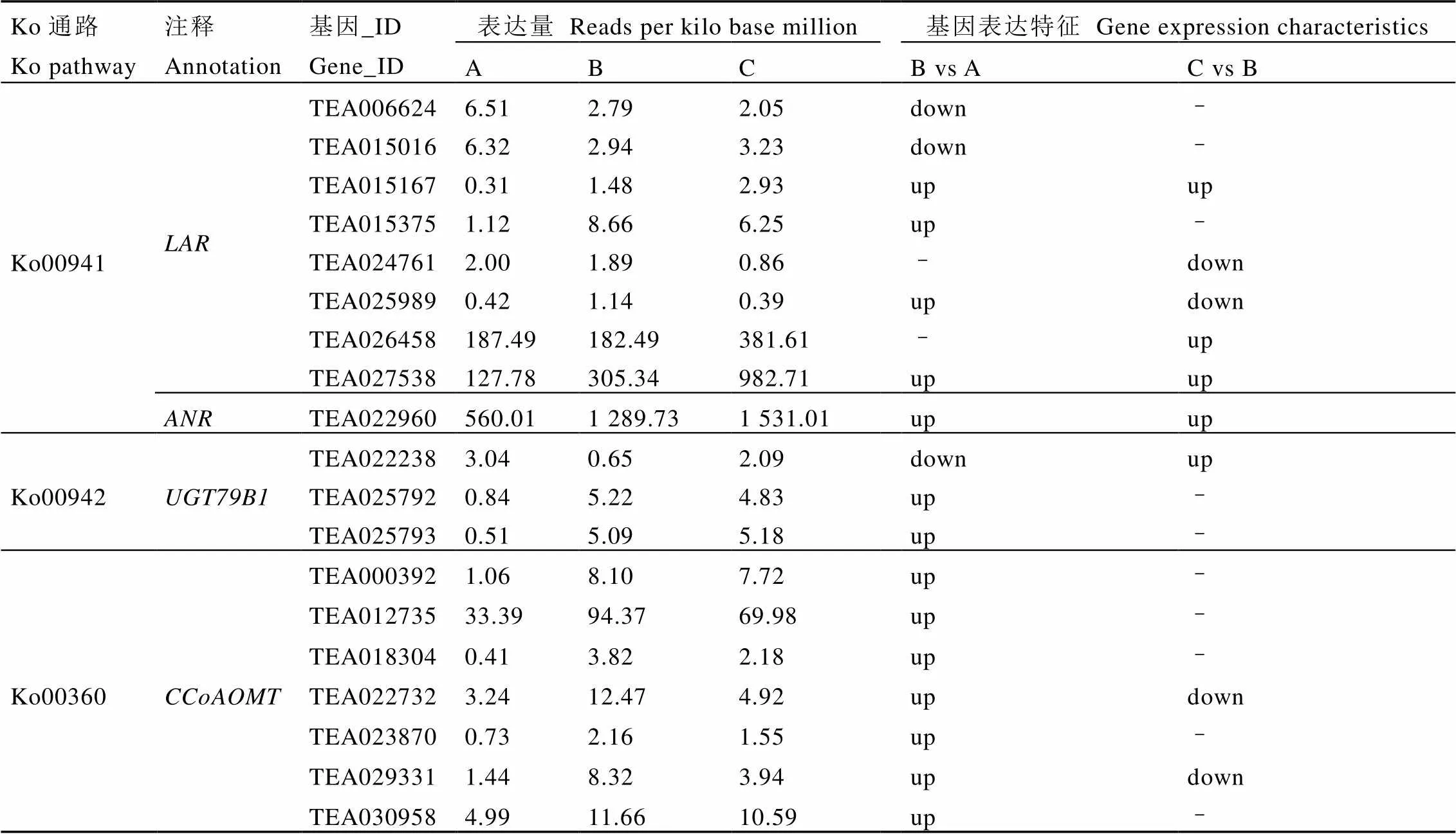
表5 不同季节花青素合成修饰基因的表达分析
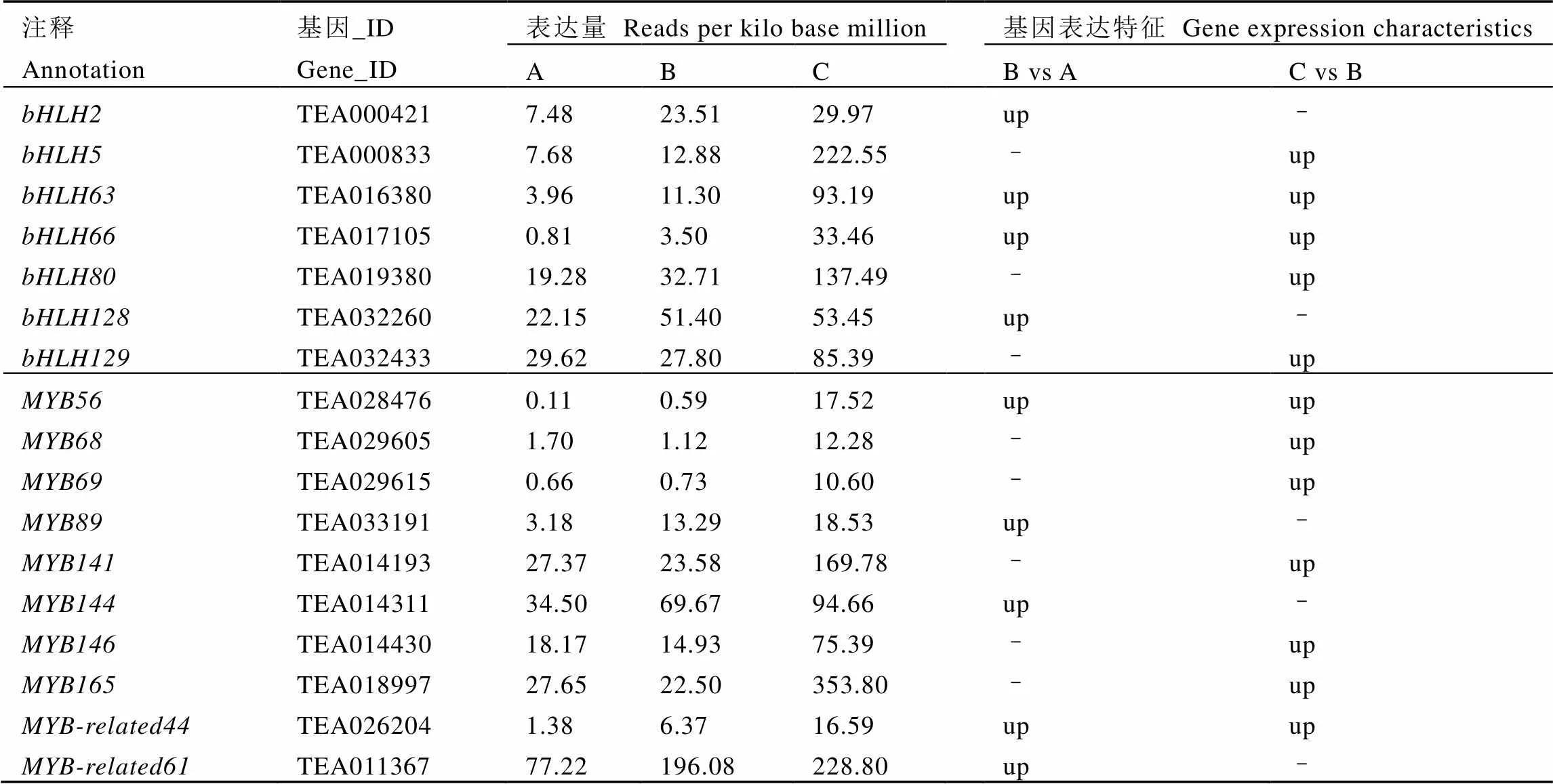
表6 大厂茶不同季节花青素合成调控基因的表达
苯丙氨酸解氨酶(PAL)是花青素生物合成过程中的关键酶之一,它催化-苯丙氨酸(-Phe)的脱氨基反应生成肉桂酸,为后续花青素的合成提供底物[20],桃金娘()果实中基因的表达与果实的颜色变化一致[21]。有研究表明,和基因的表达与花青素的积累呈正相关[22-24]。本研究结果显示,基因中获得10个差异基因,表达模式相似,在夏季上调表达,这与花青素积累情况基本一致,进一步验证了在花青素合成中的重要作用。基因表达情况相似,在夏季和秋季大部分上调表达。是控制天竺葵素合成的关键酶[25],本研究中天竺葵素-3--葡萄糖苷积累量在夏季和秋季均有所增加;在烟草()中的异源表达导致花青素积累增加,致使花色变深[26];受到抑制时会影响花青素的积累[27];基因的表达情况和菊花花瓣中花青素积累一致[28]。研究表明,与存在底物竞争关系[29],本研究中部分基因在秋季时出现表达量下调情况,这可能是二氢黄酮醇更多地向花青素代谢途径转化,通过底物竞争影响了花青素的合成。注释到中的差异基因存在两种不同的表达模式,这与其他植物中基因的表达与花青素积累呈正相关不同[30-31],其功能有待进一步研究。
花青素生物合成过程中修饰基因和是原花青素(PA)合成过程中的关键酶基因[32],可以将无色花色素转化为儿茶素(+)-C,可以催化花青素形成表儿茶素(–)-EC[33]。本研究中注释到中的差异基因出现两种不同的表达模式,在夏季和秋季均下调表达,这与在苹果中原花青素生物合成涉及的花青素还原酶基因表达相似[34],推测可能在紫芽品系P113中存在异位表达,其表达模式有待进一步研究。注释到的差异基因表达量在夏季和秋季均上调,与花青素积累情况相同,因此推测在原花青素生物合成过程中起着积极作用。糖基化有助于植物中花色苷的多样性和稳定性,属于花青素糖基化转移酶中的一种。在拟南芥的研究中敲除突变体中,花青素显著减少[35]。本研究中的基因表达量在夏季时上调,表明在花青素合成修饰过程中起重要修饰作用。作为木质素合成途径中的关键酶基因,与花青素的生物合成途径拥有相同的底物4-香豆辅酶A(4-coumaroyl-CoA)[36],本研究中基因在夏季均上调表达,部分在秋季时下调表达,表明在夏季时可能使底物更多的向木质素合成途径转化,秋季时可能更多的向花青素合成途径转化,促进花青素的积累。
已知花青素生物合成过程中受到转录因子调控,目前对花青素生物合成过程报道最多的调控因子主要是MYB-bHLH-WD40(MBW)复合体[37]。本研究中紫芽品系P113在春、夏、秋3个季节,表达特征与结构基因表达特征基本一致,表明调控因子可能通过正向调控这部分结构基因从而影响花青素积累[38]。转录因子在玫瑰花中过表达可以促进花青素生物合成下游基因的表达[39];在矮牵牛中基因过表达可以提高花青素生物合成过程中基因的表达量[40]。本研究中基因表达水平与花青素积累情况一致,表明可能在花青素生物合成过程中起到正向调控作用。植物花青素合成过程中转录因子的稳定表达对花青素积累起着重要作用。在矮牵牛中,转录因子可通过调控结构基因,从而调控花青素的积累[41];在马铃薯中,转录因子过量表达能调控基因表达,进而加深块茎表皮颜色[42]。而在本研究中转录调控因子未注释到显著差异表达的基因,其在紫芽品系P113中是否调控基因的表达有待进一步研究。
[1] 萧力争, 李勤, 谭正初, 等. 茶树紫色芽叶分级标准研究[J]. 云南农业大学学报(自然科学), 2008, 23(5): 668-672.
Xiao L Z, Li Q, Tan Z C, et al. Study on the classification standard of purple tea shoot [J]. Journal of Yunnan Agricultural University (Natural Science), 2008, 23(5): 668-672.
[2] 潘亚燕, 吴华玲, 李家贤, 等. 红紫芽茶利用研究进展[J]. 广东农业科学, 2015, 42(1): 8-12, 17.
Pan Y Y, Wu H L, Li J X, et al. Advances in research and utilization of purple tea [J]. Guangdong Agricultural Science, 2015, 42(1): 8-12, 17.
[3] 张向娜, 熊立瑰, 温贝贝, 等. 茶树叶色变异研究进展[J]. 植物生理学报, 2020, 56(4): 643-653.
Zhang X N, Xiong L G, Wen B B, et al. Advances in leaf color variation of tea plant () [J]. Plant Physiology Journal, 2020, 56(4): 643-653.
[4] 刘林峰, 向奕, 刘安, 等. 茶叶花青素的研究进展[J]. 茶叶通讯, 2018, 45(1): 3-8.
Liu L F, Xiang Y, Liu A, et al. Research progress of tea anthocyanin [J]. Journal of Tea Communication, 2018, 45(1): 3-8.
[5] 梁立军, 杨祎辰, 王二欢, 等. 植物花青素生物合成与调控研究进展[J]. 安徽农业学, 2018, 46(21): 18-24.
Liang L J, Yang Y C, Wang E H, et al. Research progress in plant anthocyanin biosynthesis and regulation [J]. Journal of Anhui Agricultural Sciences, 2018, 46(21): 18-24.
[6] 马成英, 吕海鹏, 林智, 等. 茶树类黄酮-甲基转移酶基因的克隆及原核表达分析[J]. 中国农业科学, 2013, 46(2): 325-333.
Ma C Y, Lv H P, Lin Z, et al.Cloning and prokaryotic expression of flavonoid-methyltransferase from[J]. Scientia Agricultura Sinica, 2013, 46(2): 325-333.
[7] Sun B, Zhu Z, Cao P, et al. Purple foliage coloration in tea (L.) arises from activation of the R2R3-MYB transcription factor CsAN1 [J]. Scientific Reports, 2016, 6: 32534. doi: 10.1038/srep32534.
[8] 唐秀华, 孙威江, 唐琴. 紫化茶树生理生化及其花青素调控机理研究进展[J]. 天然产物研究与开发, 2017, 29(6): 1077-1083, 999.
Tang X H, Sun W J, Tang Q. Advances in physiology biochemistry and regulation mechanism of anthocyanin in purple shoots of tea plant () [J]. Natural Product Research and Development, 2017, 29(6): 1077-1083, 999.
[9] 祝志欣, 鲁迎青. 花青素代谢途径与植物颜色变异[J]. 植物学报, 2016, 51(1): 107-119.
Zhu Z X, Lu Y Q. Anthocyanin metabolism pathway and plant color variation [J]. Chinese Bulletin of Botany, 2016, 51(1): 107-119.
[10] 张瑜, 徐志超, 季爱加, 等. bZIP转录因子调控植物次生代谢产物生物合成的研究进展[J]. 植物科学学报, 2017, 35(1): 128-137.
Zhang Y, Xu Z C, Ji A J, et al. Regulation of secondary metabolite biosynthesis by BZIP transcription factors in plants [J]. Plant Science Journal, 2017, 35(1): 128-137.
[11] 种康, 王台, 钱前, 等. 2014年中国植物科学若干领域重要研究进展[J]. 植物学报, 2015, 50(4): 412-459.
Zhong K, Wang T, Qian Q, et al. Important research progress in several fields of plant science in China in 2014 [J]. Bulletin of Botany, 2015, 50(4): 412-459.
[12] 李智, 王日为, 张丽霞, 等. 茶树紫色芽叶花青苷组分分析及结构推测[J]. 茶叶科学, 2014, 34(3): 279-287.
Li Z, Wang R W, Zhang L X, et al. Analysis and structural identification of anthocyanin components in purple buds and leaves of tea plant [J]. Journal of Tea Science, 2014, 34(3): 279-287.
[13] Trapnell C, Williams B A, Pertea G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation [J]. Nature Biotechnology, 2010, 28: 511-515.
[14] McKenna A, Hanna M, Banks E, et al. The genome analysis toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data [J].Genome Research, 2010, 20(9): 1297-1303.
[15] 陈林波, 夏丽飞, 田易萍, 等. 基于数字基因表达谱分析的茶树花不育基因挖掘[J]. 作物学报, 2017, 43(2): 210-217.
Chen L B, Xia L F, Tian Y P, et al. Exploring sterility gene from tea plant flower based on digital gene expression profiling [J]. Acta Agronomica Sinica, 2017, 43(2): 210-217.
[16] Anders S, Huber W. Differential expression analysis for sequence count data [J]. Nature Precedings, 2010, 11(10): R106. doi: 10.1038/npre.2010.4282.2.
[17] 庞丹丹, 张亚真, 徐礼羿, 等. 基于转录组测序的茶树儿茶素合成调控相关基因的挖掘[J]. 分子植物育种, 2019, 17(17): 5604-5612.
Pang D D, Zhang Y Z, Xu L Y, et al. Excavation of key genes involved in the regulation of tea tree catechin synthesis based on transcriptome sequencing [J]. Molecular Plant Breeding, 2019, 17(17): 5604-5612.
[18] 蒋会兵, 孙云南, 李梅, 等. 紫娟茶树叶片不同发育期花青素积累及合成相关基因的表达[J]. 茶叶科学, 2018, 38(2): 174-182.
Jiang H B, Sun Y N, Li M, et al. Anthocyanin accumulation and expression of synthesis-related genes in leaves of different developmental stages incv. Zijuan [J]. Journal of Tea Science, 2018, 38(2): 174-182.
[19] Shen J Z, Zou Z W, Zhang X Z, et al. Metabolic analyses reveal different mechanisms of leaf color change in two purple-leaf tea plant (L.) cultivars [J]. Horticulture Research, 2018, 5: 7. doi: 10.1038/s41438-017-0010-1.
[20] 郝向阳, 孙雪丽, 王天池, 等. 植物基因及其编码蛋白的特征与功能研究进展[J]. 热带作物学报, 2018, 39(7): 1452-1461.
Hao X Y, Sun X L, Wang T C, et al. Characteristics and Functions of Plantgenes and the encoded proteins [J]. Chinese Journal of Tropical Crops, 2018, 39(7): 1452-1461.
[21] Zhu B J, Wang Q, Wang J H, et al.andgene transcription and their correlation with anthocyanin accumulation in(Aiton.) Hassk. [J]. Turkish Journal of Biochemistry, 2018, 44(3): 289-298.
[22] 周天山, 王新超, 余有本, 等. 紫芽茶树类黄酮生物合成关键酶基因表达与总儿茶素、花青素含量相关性分析[J]. 作物学报, 2016, 42(4): 525-531.
Zhou T S, Wang X C, Yu Y B, et al. Correlation analysis between total catechins (or anthocyanins) and expression levels of genes involved in flavonoids biosynthesis in tea plant with puprple leaf [J]. Acta Agronomica Sinica, 2016, 42(4): 525-531.
[23] 曹冰冰, 王秋霜, 秦丹丹, 等. 红紫芽茶花青素合成关键酶活性与重要酚类物质相关性研究[J]. 茶叶科学, 2020, 40(6): 724-738.
Cao B B, Wang Q S, Qin D D, et al. Study on the correlation between the activities of key enzymes involving in anthocyanin synthesis and the contents of important polyphenols in purple tea [J]. Journal of Tea Science, 2020, 40(6): 724-738.
[24] Guo L P, Teixeira da Silva J A, Pan Q H, et al. Transcriptome analysis reveals candidate genes involved in anthocyanin biosynthesis in flowers of the pagoda tree (L.) [J]. Journal of Plant Growth Regulation, 2021. doi: 10.1007/s00344-020-10222-0.
[25] Yu L X, Li W J, Du Y, et al. Flower color mutants induced by carbon ion beam irradiation of geranium (×, Bailey) [J]. Nuclear Science and Techniques, 2016, 27(5): 37-44.
[26] Liu H L, Lou Q, Ma J R, et al. Cloning and functional characterization of dihydroflavonol 4-reductase gene involved in anthocyanidin biosynthesis of grape hyacinth [J]. International Journal of Molecular Sciences, 2019, 20(19): 4743. doi: 10.3390/ijms20194743.
[27] 戴思兰, 洪艳. 基于花青素苷合成和呈色机理的观赏植物花色改良分子育种[J]. 中国农业科学, 2016, 49(3): 529-542.
Dai S L, Hong Y. Molecular breeding for flower colors modification on ornamental plants based on the mechanism of anthocyanins biosynthesis and coloration [J]. Scientia Agricultura Sinica, 2016, 49(3): 529-542.
[28] 韩科厅, 赵莉, 唐杏姣, 等. 菊花花青素苷合成关键基因表达与花色表型的关系[J]. 园艺学报, 2012, 39(3): 516-524.
Han K T, Zhao L, Tang X J, et al. The relationship between the expression of key genes in anthocyanin biosynthesis and the color of chrysanthemum [J]. Acta Horticulturae Sinica, 2012, 39(3): 516-524.
[29] Park S, Kim D H, Lee J Y, et al. Comparative analysis of two flavonol synthases from different-colored onions provides insight into flavonoid biosynthesis [J]. Journal of Agricultural and Food Chemistry, 2017, 65(26): 5287-5298.
[30] 范月婷, 辛士超, Naychi Koko, 等. 橡胶树基因克隆及其功能分析[J]. 热带作物学报, 2020, 41(9): 1733-1740.
Fan Y T, Xin S C, Naychi K, et al. Cloning and functional verification of rubber treeGene [J]. Chinese Journal of Tropical Crops, 2020, 41(9): 1733-1740.
[31] 胡悦, 胡青霞, 谭彬, 等. 红皮石榴着色机制与花青苷合成研究进展[J]. 分子植物育种, 2019, 17(8): 2692-2700.
Hu Y, Hu Q X, Tan B, et al. Mechanism of red-skinned pomegranate fruit coloring and development of anthocyanin biosynthesis [J]. Molecular Plant Breeding, 2019, 17(8): 2692-2700.
[32] Yang Y N, Yao G F, Yue W Q, et al. Transcriptome profiling reveals differential gene expression in proanthocyanidin biosynthesis associated with red/green skin color mutant of pear (L.) [J]. Frontiers in Plant Science, 2015, 6: 795. doi: 10.3389/fpls.2015.00795.
[33] Shi L Y, Cao S F, Chen X, et al. Proanthocyanidin synthesis in Chinese bayberry (Sieb. et Zucc.) fruits [J]. Frontiers in Plant Science, 2018, 9: 212. doi: 10.3389/fpls.2018.00212.
[34] Liao L, Vimolmangkang S, Wei G C, et al. Molecular characterization of genes encoding leucoanthocyanidin reductase involved in proanthocyanidin biosynthesis in apple [J]. Frontiers in Plant Science, 2015, 6: 243. doi: 10.3389/fpls.2015.00243.
[35] Yonekura-Sakakibara K, Fukushima A, Nakabayashi R, et al. Two glycosyltransferases involved in anthocyanin modification delineated by transcriptome independent component analysis in[J]. The Plant Journal, 2012, 69(1): 154-167.
[36] Shaipulah N F M, Muhlemann J K, Woodworth B D, et al.down-regulation activates anthocyanin biosynthesis in petunia [J]. Plant Physiology, 2016, 170(2): 717-731.
[37] Xu W J, Dubos C, Lepiniec L. Transcriptional control of flavonoid biosynthesis by MYB-bHLH-WDR complexes [J]. Trends in Plant Science, 2015, 20(3): 176-185.
[38] Butelli E, Titta L, Giorgio M, et al. Enrichment of tomato fruit with health-promoting anthocyanins by expression of select transcription factors [J]. Nature Biotechnology, 2008, 26: 1301-1308.
[39] Zvi M M B, Shklarman E, Masci T, et al.transcription factor enhances production of phenylpropanoid and terpenoid scent compounds in rose flowers [J]. New Phytologist, 2012, 195(2): 335-345.
[40] Ai T N, Naing A H, Arun M, et al. Expression ofinenhances anthocyanin production in vegetative and floral tissues [J]. Scientia Horticulturae, 2017, 214: 58-65.
[41] Vetten N D, Quattrocchio F, Mol J, et al. The an11 locus controlling flower pigmentation in petunia encodes a novel WD-repeat protein conserved in yeast, plants, and animals [J]. Genes & Development, 1997, 11(11): 1422-1434.
[42] 杨波. 马铃薯块茎花青素差异累积及光诱导累积的研究[D]. 武汉: 华中农业大学, 2019.
Yang B. Research on differential accumulation and light-induced accumulation of anthocyanins in potato tubers [D]. Wuhan: Huazhong Agricultural University, 2019.
Analysis of Gene Expression Related to Anthocyanin Regulation of ‘P113’ Purple Strain ofF.C.Zhang in Different Seasons
LIU Xia, LI Fang, SONG Qinfei, NIU Suzhen*, LYU Litang
Tea College of Guizhou University, Guiyang 550025, China
In order to clarify the molecular mechanism of anthocyanin accumulation and the expression characteristics of related genes in the synthetic pathway of anthocyanin inF.C.Zhang purple bud line P113 in different seasons, one bud and two leaves of P113 in spring, summer and autumn were analyzed by RNA-Seq and UHPLC-MS/MS. The results show that the 10 anthocyanin contents changed with seasons, and 4 of them were up-regulated with the change of spring,summer and autumn, which were consistent with leaf color phenotypeof ‘P113’. The expression patterns of structural genes,,,,andwere basically the same, which were up-regulated in summer, but not significantlychanged in autumn. The two differentially expressedgenes were up-regulated in summer. The gene expression patterns of,andwere similar, and their expressions were up-regulated in summer and down-regulated in autumn. One of the twogenes was down-regulated in summer, and one was up-regulated with seasonal changes. The 13 genes ofwere all up-regulated in summer, but they showed different expression patterns in autumn. The modifier genehad two expression patterns in summer and autumn. There was only 1 differentially expressed gene inthat was up-regulated with seasonal changes. Among the threegenes, one was down-regulated in summer, up-regulated in autumn, andthe rest two genes were up-regulated in summer, but not significantly changed in autumn. Seven differentially expressedgenes were up-regulated in summer.Two of them down regulated in autumn, and the rest five genes were not significant changed. The regulatory genesandwere up-regulated with seasonal changes, and had a positive relationship with anthocyanin biosynthesis. The results show that the expressions of structural genes, modifier genes and regulatory genes of ‘P113’ (F.C.Zhang) had certain time characteristics in different seasons, which led to the differencesin anthocyanin accumulation.
F.C.Zhang, anthocyanins, differentially expressed genes
S571.1
A
1000-369X(2021)06-789-13
2021-02-22
2021-07-15
贵州省自然科学基金重点项目(黔科合基础[2019]1404号)、贵州省科技厅农业攻关项目(黔科合支撑[2017]2557、黔科合支撑[2017]2558)、国家自然科学基金(32060700)
刘霞,女,硕士研究生,主要从事茶树种质资源与遗传改良研究,liuxia199411@163.com。*通信作者:niusuzhen@163.com
(责任编辑:黄晨)
