戴瑞奶绵羊产奶性状的全基因组关联分析
2021-11-24米布农张立果乌日汉郭玉林王春伟徐全忠李光鹏苏小虎
米布农,张立果,乌日汉,郭玉林,王春伟,徐全忠,冯 爽,李光鹏,苏小虎*,张 立*
(1.内蒙古大学生命科学学院 省部共建草原家畜生殖调控与繁育国家重点实验室,呼和浩特 010020;2.乌兰察布市农牧局 乌兰察布市家畜畜牧工作站,乌兰察布 012000)
奶绵羊具有优良的产奶性能,且课题组前期的研究表明,绵羊奶的乳蛋白和乳脂显著优于山羊奶和牛奶[1]。戴瑞羊(Dairy Meade)是以新西兰本地柯泊华兹羊为母本、东佛里生羊为父本,经过20多年培育形成的新型乳用绵羊品种,于2016年被新西兰绵羊育种联合会认定为新西兰首个乳用绵羊品种,具有体型大、泌乳量高、繁殖性能好等优良生产性状[2]。戴瑞奶绵羊的泌乳期正常为180~230 d,初产平均产奶量为150~250 kg,经产平均产奶量为350~600 kg。我国历史上没有专用的奶绵羊品种。为填补这一空白,内蒙古大学于2016年引入了戴瑞奶绵羊胚胎,通过胚胎移植获得了国内纯种戴瑞奶绵羊。随后以本地小尾寒羊为母本,以戴瑞羊为父本开展级进杂交获得了一批新型奶绵羊繁育群体。
传统的育种方式主要是利用表型信息和系谱信息进行杂交选育,对遗传力低和测量复杂的性状,存在测定成本高、时间长、育种过程复杂的问题[3]。随着高通量测序技术、生物信息学及统计学方法的快速发展,全基因组关联分析(genome wide association study,GWAS)已成为禽畜重要性状主效基因挖掘与鉴定的主要手段[4-5]。目前,GWAS已在牛、羊等产奶性状的候选基因筛选方面取得了大量的研究成果。Liu等[6]使用90K Affymetrix Buffalo SNP阵列对意大利地中海水牛进行GWAS,发现与产奶性能相关的4个SNPs,2个基因组区域和5个候选基因。Deng等[7]对地中海水牛产奶量全基因组关联分析,发现了9个候选基因。Silva等[8]使用加权单步GWAS(weighted single-step GWAS,wssGWAS)对葡萄牙荷斯坦牛产奶性能全基因组关联分析,发现与产奶量相关的QTL有51个,乳脂含量相关的QTL有5个,乳蛋白含量相关的QTL有24个。一项使用90 K Axiom®水牛阵列对埃及水牛产奶性状进行全基因组关联分析的研究中,发现了47个显著SNPs和11个基因座[9]。García-Gámez等[10]使用Illumina OvineSNP50 BeadChip对西班牙Churra羊产奶性能进行全基因组关联分析,发现14个染色体水平显著的QTL和1个候选基因。Moioli等[11]使用Illumina SNP50K Bead chip对意大利Altamurana绵羊的产奶性能进行了基因组扫描和全基因组关联分析,发现2个候选基因。Li等[12]使用Illumina OvineSNP50对东弗里生和拉科讷杂交绵羊产奶性能全基因组关联分析,发现与180天产奶量相关的SNP有115个,180天乳脂含量相关的SNP有288个,180天乳蛋白含量相关的SNP有74个。Sutera等[13]使用Illumina OvineSNP50K BeadChip对意大利Valle del Belice绵羊产奶性能进行全基因组关联分析,发现与产奶性能相关的9个SNPs。
然而针对奶绵羊的研究较少,一定程度限制了奶绵羊育种的发展。本研究基于前期已形成的戴瑞羊品种群体,通过对其产奶性状测定和基因组重测序,利用GWAS方法挖掘其产奶性状相关的候选基因及关键位点,为奶绵羊新品种的培育提供理论技术支持,为我国绵羊奶产业发展提供参考。
1 材料与方法
1.1 试验动物来源与产奶性状
本试验所用的135只戴瑞奶绵羊繁育群体均来源于内蒙古自治区乌兰察布市蒙天然牧业科技发展有限公司。所选试验羊均为初产,15~18月龄。于相同的环境、饲料、摄食比例条件下饲喂直至泌乳期结束。每天06:00及17:00饲喂试验动物,试验期间试验动物可自由饮水。粗饲料是羊草,试验日粮饲料组成粗精比为6∶4,母羊泌乳精补料营养水平见表1。产奶性状为90天日均产奶量,150天日均产奶量和泌乳周期。所研究性状均为分级性状。根据群体泌乳量信息,以27%标准进行产奶性状等级划分。产奶量以前后连续5 d测定的平均值为准,90天日均产奶量性状根据低于0.5 kg和高于1 kg的标准划分为低产组和高产组,150天日均产奶量性状以0.5 kg划分低产组和高产组,泌乳周期性状根据低于125 d和高于180 d的标准划分为短周期组和长周期组。

表1 试验动物精补料营养水平
1.2 血液基因组DNA提取与检测
将采集的全血样本通过康为世纪CWE9600DNA提取试剂盒V2提取基因组DNA。琼脂糖凝胶电泳分析DNA降解程度以及是否有RNA污染;Nanodrop检测DNA的纯度(OD260 nm/OD280 nm比值);Qubit对DNA浓度进行精确定量。其中OD值在1.8~2.0之间,含量在1.5 μg以上的DNA样品被用来建库。
1.3 重测序基因分型和质量控制
为了获得核苷酸多态性信息,用illumina Novaseq6000平台对奶绵羊基因组进行重测序。有效测序数据通过BWA 0.7.17软件[14]比对到参考基因组,比对结果经SAMTOOLS 1.7软件[15]去除重复;并将其映射到Oar_rambouillet_v1.0参考序列;映射的读段通过SAMTOOLS分析获取变异位点的分型;使用Plink1.90 软件[16]进行质控,删除 SNP缺失大于10%的位点、最小等位基因频率小于1%的位点、哈迪-温伯格平衡检验P<10-6的位点。
1.4 样本群体结构分析
由于群体分层会使全基因组关联分析的结果出现假阳性[17],因此采用Plink1.90对SNP分型数据计算IBS(Identity-by-state)遗传距离矩阵和主成分分析,以研究奶绵羊群体的亲缘关系以及群体分布。通过Plink软件构建亲缘关系矩阵(G阵),之后采用R语言的gg plots包的heatmap.2函数绘制热图,采用解释方差最大的前三个主成分进行散点图绘制以分析群体结构。
1.5 全基因组关联分析及统计推断
采用GEMMA v0.98.1软件[18]进行混合线性模型[19]分析:
y=Xβ+Ζκγκ+ξ+e
式中,y为表型向量,Xβ为PCA固定效应,Ζκγκ为待检验标记效应,ξ~N(0,Kφ2)为多基因效应,e~N(0,1σ2)为残差效应。多基因效应中的K为标记推断的亲缘关系矩阵。最终,每个SNP位点对应一个关联值。采用Bonferroni校正的阈值为全基因组显著阈值设置为0.05/21 020 909,基因组水平潜在显著阈值设置为1/21 020 909。曼哈顿图以阈值线将关联信息与显著SNP进行了可视化。曼哈顿图和分位数图由GEMMA绘制。
1.6 候选基因注释
在ENSEMBL网站下载绵羊基因组序列版本Oar_rambouillet_v1.0(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/002/742/125/GCF_002742125.1_Oar_rambouillet_v1.0/GCF_002742125.1Oar_rambouillet_v1.0_genomic.fna.g),使用ANNOVAR软件[20]将显著SNP注释到其对应的基因上。
1.7 基因本体论和代谢途径分析
对于基因本体(GO)和代谢途径富集分析,使用cluster Profiler包[21]将注释到的候选基因根据GO和KEGG数据库进行基因功能富集分析。利用R语言的enrich plot包进行GO/KEGG的可视化画图。通过文献查阅基因生物学功能,综合分析确定影响奶绵羊产奶性状的候选基因。
2 结 果
2.1 表型数据分析
表2展示了产奶性状的平均值、标准差和变异系数。从表2中可以看出,90天日均产奶量的平均值是0.86 kg,150天日均产奶量的平均值是0.27 kg,泌乳周期的平均值是148 d,90天日均产奶量性状的变异系数是86%,150天日均产奶量性状的变异系数是140%,泌乳周期的变异系数是129%。
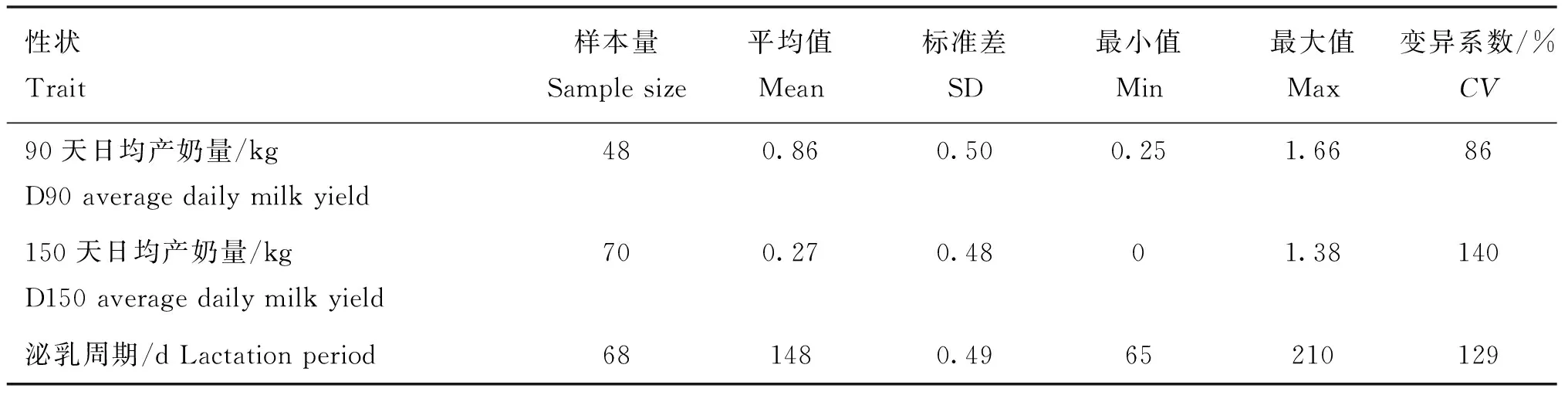
表2 产奶性状的表型描述性统计
2.2 基因分型和数据质量控制
通过基因分型共获得22 683 429个SNPs。在进行质量控制时,SNP缺失大于10%的位点,剔除696 899个;最小等位基因频率小于1%的位点,剔除0个;哈迪-温伯格平衡检验P<10-6的位点,剔除69 658个。经过质量控制后,有135个样本和21 020 909个 SNPs用于后续关联分析。基因分型率达到98.54%。
2.3 样本群体结构分析
图1为135只奶绵羊的亲缘关系矩阵热图。亲缘关系矩阵热图是用颜色的深浅不同表示每只羊之间的亲缘关系远近。颜色越浅,数值越小,亲缘关系越远。因此,从图1中可以看出,3个颜色深的地方代表3个家系。
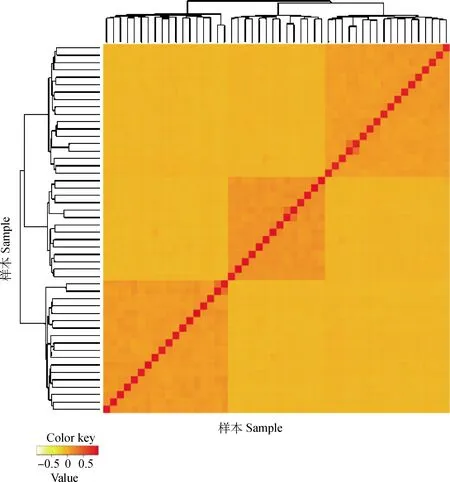
图1 亲缘关系矩阵热图
图2显示了该奶绵羊群体结构的分布,从图中可以看出试验群体分成了3个部分,说明该群体存在人工选育所引起的群体结构差异。因此在后续全基因组关联分析过程中需要考虑群体分层问题,并对其进行校正。
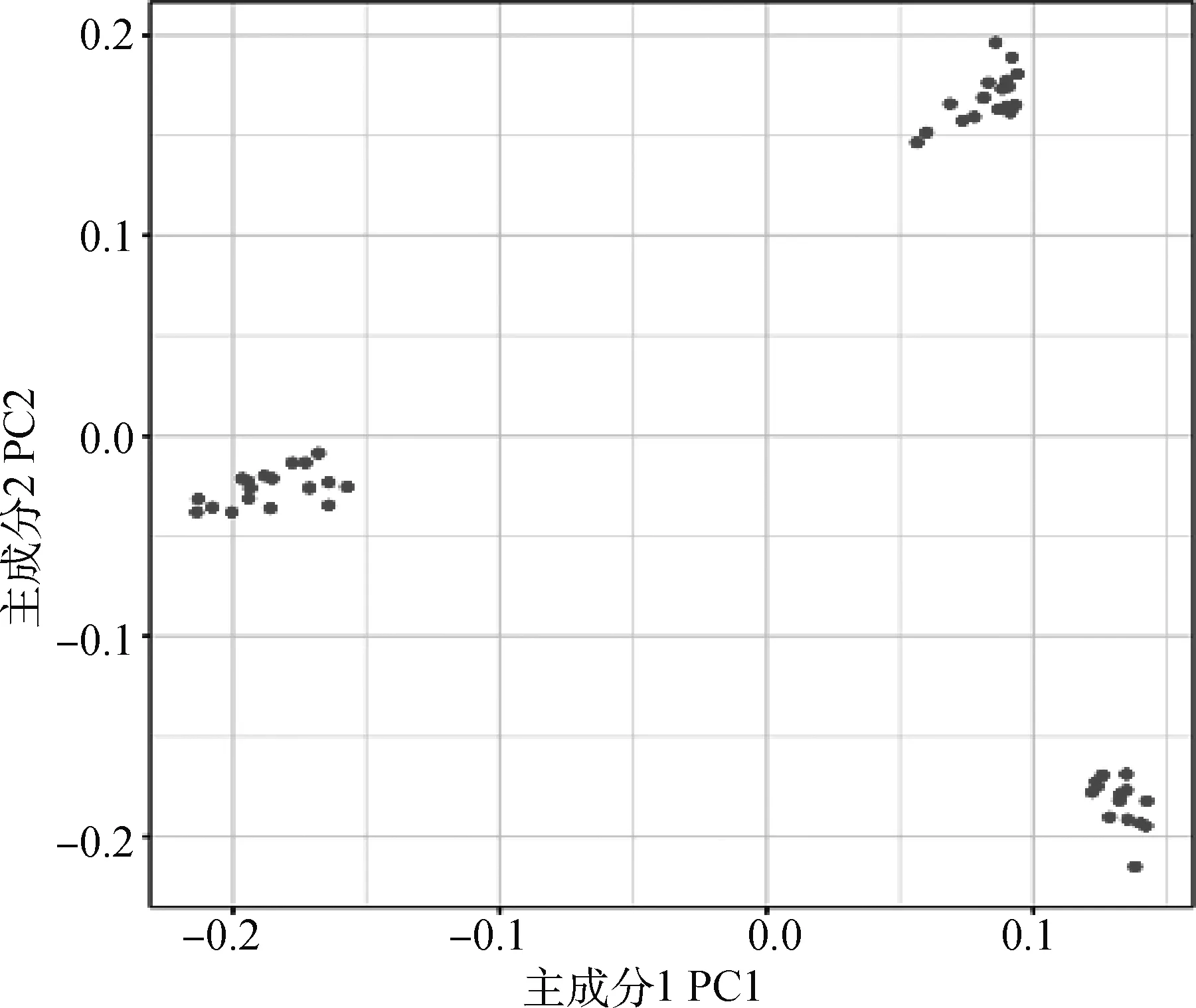
图2 PCA群体结构图
2.4 全基因组关联分析
图3为产奶性状的分位数-分位数(Quantile-Quantile)图,展示了90天日均产奶量、150天日均产奶量和泌乳周期的GWAS观测值和预期值。虚线表示在SNP与所研究性状不相关的原假设下SNP的分布。3个Q-Q图的观测值与预期P值的强烈偏差表明,与性状显著相关的SNP比预期的要多。

A.90天日均产奶量性状;B.150天日均产奶量性状;C.泌乳周期性状
图4为产奶性状的曼哈顿图,对分布在26条染色体上的21 020 909个SNPs进行了关联分析。Bonferroni校正后的显著水平阈值为2.38×10-9,基因组水平潜在显著阈值为4.76× 10-8。表3展示了与产奶性状显著相关的SNPs名称、物理位置以及邻近基因等。
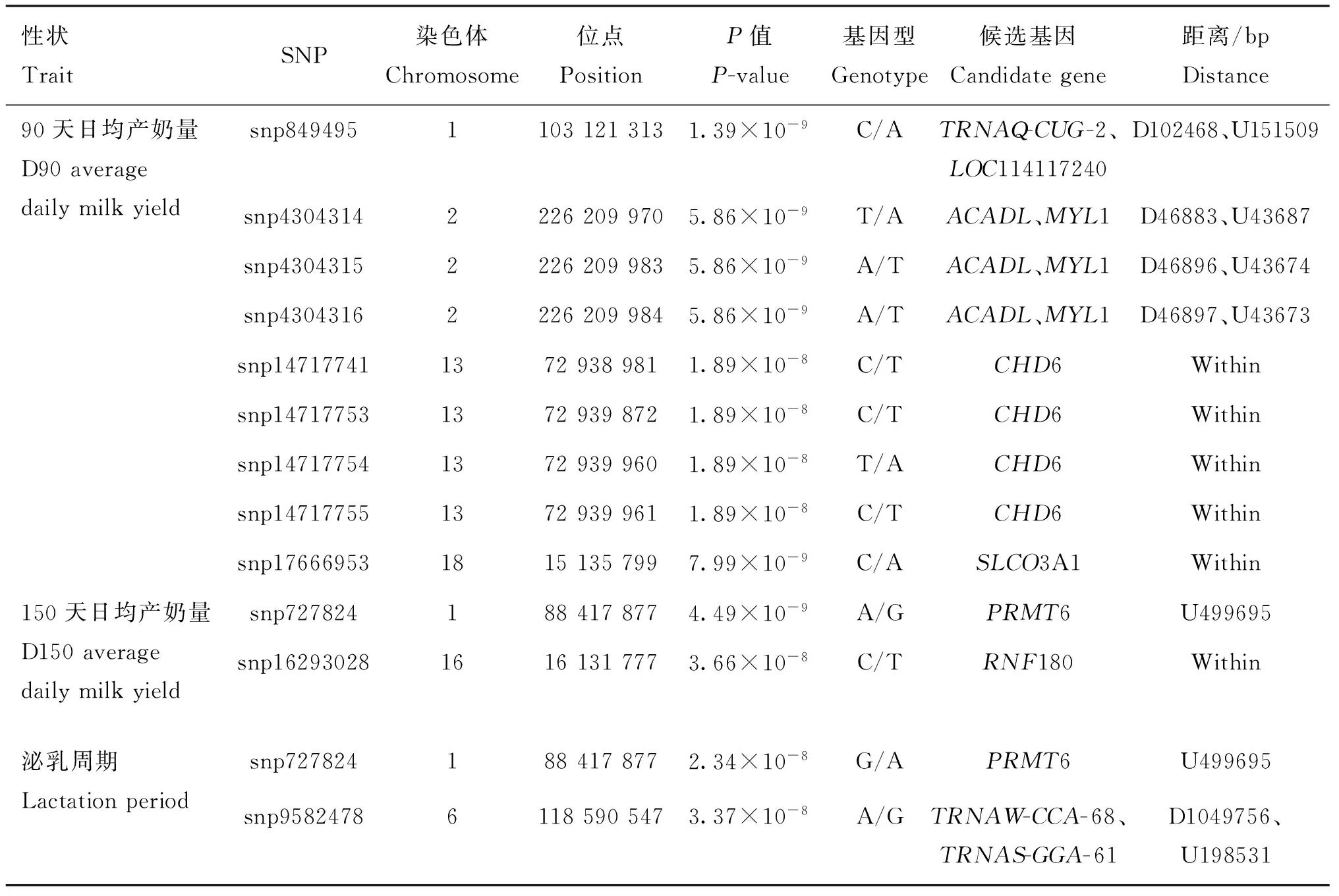
表3 与产奶性状关联的SNPs

黑色水平线代表全基因组水平显著阈值线,红色水平线为全基因组潜在显著关联阈值线。A.90天日均产奶量性状;B.150天日均产奶量性状;C.泌乳周期性状
与90天日均产奶量潜在显著关联的SNP有8个,分别位于2、13、18号染色体上。与90天日均产奶量显著关联的SNP有1个,位于1号染色体上。1号染色体上的1个显著SNP(849495)位于TRNAQ-CUG-2(未表征的基因)下游102 468 bp处和LOC114117240(未表征的基因)上游151 509 bp处;2号染色体3个潜在显著SNPs位于基因间区,邻近ACADL(酰基辅酶A脱氢酶长链)和MYL1(肌球蛋白轻链1)3 kb左右;13号染色体上4个潜在显著性SNPs位于CHD6(染色体域解旋酶DNA结合蛋白6)的内含子区域;18号染色体上1个潜在显著SNP(17666953)位于SLCO3A1(溶质载体有机阴离子转运蛋白家族成员3A1)的内含子区域。
与150天日均产奶量潜在显著关联的SNP共2个,分别位于1和16号染色体上。1号染色体上1个潜在显著SNP(727824)位于PRMT6(蛋白质精氨酸甲基转移酶6)上游499 695 bp处;16号染色体上1个潜在显著性SNP(16293028)位于RNF180(无名指蛋白180)的内含子区域。
与泌乳周期潜在显著关联的SNP共 2个,分别位于 1和6号染色体上。1号染色体上1个潜在显著SNP(727824)位于基因PRMT6(蛋白质精氨酸甲基转移酶6)上游499 695 bp处;6号染色体上1个 潜在显著SNP(9582478)位于TRNAW-CCA-68(未表征的基因)下游1 049 756 bp处和TRNAS-GGA-61(未表征的基因)上游198 531 bp处。
3 讨 论
在禽畜育种中,GWAS分析已成为识别表型变异的有力策略[22]。目前,尚无关于戴瑞奶绵羊全基因组关联分析对产奶性状影响的研究报道。本研究对奶绵羊群体产奶性状进行全基因组关联分析,共得到13个相关的SNPs。对于90天日均产奶量性状,共检测到9个显著关联的SNPs,1号染色体上有1个SNP达到基因组显著水平,达到潜在显著水平的8个SNPs分别位于2、13和18号染色体上;对于150天日均产奶量性状,有2个SNPs达到潜在显著水平,分别位于1和16号染色体上;对于泌乳周期性状,达到潜在显著水平的2个SNPs分别位于1和6号染色体上。
3.1 90天日均产奶量相关的候选基因分析
与90天日均产奶量关联的9个SNPs,snp849495邻近TRNAQ-CUG-2、LOC114117240基因,其功能尚未有报道;2号染色体46 800~43 690 bp区间内存在3个SNPs与90天日均产奶量潜在关联,邻近ACADL和MYL1基因。ACADL是长链脂肪酸β-氧化起始步骤的催化酶,在长链脂肪酸β-氧化中发挥着重要作用,与脂肪代谢有着密切的关系[23]。基因本体分析表明,ACADL主要在脂肪酸分解的生物过程(GO:0009062)、氧化还原酶活性的分子功能(GO:0016627)和线粒体的细胞成分(GO:0005759)中起作用,并参与脂肪酸降解(KEGG:ssc00071)、脂肪酸代谢(KEGG:ssc01212)、PPAR信号通路(KEGG:ssc03320)。Zhang等[24]的研究表明,ACADL催化长链脂肪酸(C13-C22)的β-氧化,并调节线粒体中脂肪酸的代谢途径,抑制ACADL会导致脂肪酸蓄积,从而激活过氧化物酶体增殖物激活受体(PPAR),PPARα激活剂通过拮抗NF-κB的转录活性而在几种细胞中显示出抗炎活性。有研究发现,16碳及以下脂肪酸合成增加会促使产奶量升高,而乳中16碳以上脂肪酸比例的增加使产奶量降低[25]。ACADL可能通过降解长链脂肪酸进而影响产奶量,因此,该基因可能是与90天日均产奶量相关的重要候选基因。MYL1基因是肌球蛋白轻链1,在骨骼肌不同发育时期发挥着重要作用[26]。对于MYL1基因与产奶相关的报道较少,是因为该品种也具有良好的产肉性能,所以才鉴定到MYL1基因。13号染色体上有4个SNPs位于CHD6内。CHD6是一种蛋白质编码基因。Lutz等[27]研究发现,CHD6和转录因子(p300,SRC1)结合形成PRIC复合物,该复合物可以促使PPAR激动剂和PPARα结合,PPARα参与脂质代谢和抗氧化过程。张雪莹[28]的研究表明,在促进脂肪酸分解代谢的组织中PPARα表达量相对较高。有文献报道过氧化物酶体增殖物激活受体γ(PPARγ)可能是乳脂合成调控核心[29]。PPARα和PPARγ属于同一家族。CHD6可能通过与PPARα作用参与脂质代谢调控。因此,CHD6可能也是与90天日均产奶量相关的候选基因。snp17666953位于SLCO3A1基因内。SLCO3A1属于溶质载体有机阴离子转运蛋白家族成员,是一种蛋白质编码基因。该基因与维生素、核苷、葡萄糖、胆汁盐和金属离子等的运输有关。SLCO3A1主要涉及的生物学过程有类花生酸转运(GO:0071715)和脂肪酸衍生物转运(GO:1901571)。与该基因相关的分子功能是有机阴离子跨膜转运蛋白活性(GO:0008514)。先前的研究中,Ibeagha-Awemu等[30]通过全基因组关联分析筛选到SLCO3A1是影响奶牛产奶性状和乳腺功能的新候选基因之一。Liu等[31]使用Illumina BovineSNP150 BeadChip对中国荷斯坦奶牛产奶性能进行全基因组关联分析,发现了10个数量性状基因座和8个候选基因,其中4个与脂肪和蛋白质显著相关的SNPs位于EPHA6、SLCO1A2、DGAT1和EP400内部。SLCO1A2 是溶质载体有机阴离子转运蛋白家族成员1A2,此基因和SLCO3A1基因都有转运有机阴离子的功能。故推测SLCO3A1可能是与90天日均产奶量相关的关键候选基因。
3.2 150天日均产奶量相关的候选基因分析
与150天日均产奶量存在潜在显著相关的2个SNPs,1号染色体上的snp727824,其下游499 695 bp处是PRMT6基因,是蛋白质精氨酸甲基转移酶6,编码的蛋白质属于精氨酸N-甲基转移酶家族。PRMTs主要参与转录、RNA剪接、DNA损伤修复、细胞信号转导、蛋白质运输等重要生物学过程[32-36]。该酶可催化甲基从S-腺苷甲硫氨酸(SAM)转移至组蛋白和其他蛋白质上的精氨酸残基。这种甲基化的失调在某些癌症的发展中至关重要。Chan等[37]研究发现,PRMT6在乳腺癌中异常表达。有研究发现同属于蛋白质精氨酸甲基转移酶家族的PRMT2基因表达的多少及部位与乳腺癌细胞的表达密切相关[38]。因此,该基因可能是影响150天日均产奶量的候选基因。snp16293028位于RNF180基因内。RNF180是E3泛素连接酶家族的重要成员,通过调节靶蛋白底物的泛素化而影响细胞的生长和发育,与各种生物过程如细胞生殖、分化、增殖、凋亡和肿瘤的发生等高度相关[39]。GO注释表明,RNF180主要在儿茶酚胺代谢的生物过程(GO:0006584)、与泛素结合酶结合的分子功能(GO:0031624)和内质网膜的细胞成分(GO:0031227)中起作用。有研究表明,转运膜蛋白的泛素化利于乳腺上皮细胞外脂肪酸的转入,使细胞利用更多的脂肪酸合成乳脂[40]。因此,RNF180可能是影响150天日均产奶量的重要候选基因。
3.3 泌乳周期相关的候选基因分析
snp9582478位于6号染色体,邻近TRNAW-CCA-68和TRNAS-GGA-61基因。TRNAW-CCA-68和TRNAS-GGA-61的基因功能尚未有报道。snp727824位于1号染色体,邻近PRMT6基因。
通过对以上候选基因的功能分析,ACADL、SLCO3A1可作为影响90天日均产奶量的候选基因,PRMT6可作为影响150天日均产奶量和泌乳周期的候选基因,后期仍需要进一步对挖掘到的候选基因进行功能验证。
4 结 论
本研究检测到13个与90天、150天日均产奶量和泌乳周期性状相关的SNPs,发现了10个候选基因,基因功能分析发现ACADL、SLCO3A1有可能是影响产奶性状的候选基因。本研究结果为深入揭示奶绵羊产奶性状的遗传效应和标记辅助选种提供理论技术支持。
