基于16S rDNA序列、MALDI-TOF-MS和VITEK的沙门氏菌和金黄色葡萄球菌的鉴定
2021-11-22孟令缘牛沁雅廉鲁昕黄巾凌崔生辉闫韶飞李凤琴杨保伟
孟令缘,牛沁雅,廉鲁昕,黄巾凌,崔生辉,闫韶飞,李凤琴,杨保伟*
(1 西北农林科技大学食品科学与工程学院 陕西杨凌 712100 2 中国食品药品检定研究院 北京 102629 3 国家食品安全风险评估中心 北京 100022)
沙门氏菌(Salmonella)和金黄色葡萄球菌(Staphylococcus aureus)是常见的食源性致病菌,也是导致公共卫生安全和健康问题的主要微生物种类。准确、高效的微生物鉴定技术是有效控制致病菌传播和预防相关疾病的前提。随着科学技术的高速发展,可供检测人员选择的微生物鉴定方法和技术越来越多。然而,不同方法间的差异常使得鉴定结果的可靠性和准确性受到质疑[1-2]。
目前,微生物的形态学、分子生物学、生理生化和蛋白质谱等特性通常作为检测、分类和鉴定的依据。形态学鉴定简单、快速,然而结果受检测人员主观因素影响较大,对检测人员操作水平要求较高,且不能作为最终评判依据[3-4]。分子生物学鉴定方法具有准确性高、结果稳定、数据库全面的特点,一直被广泛应用于临床、环境及食品微生物鉴定领域。其中,16S rDNA 序列长度适中,有保守区和可变区,是微生物鉴定和分子分型的最优标志物之一[5-6]。VITEK 全自动细菌鉴定系统和API等全自动微生物生化鉴定系统均以微生物的特征性生化反应作为鉴定依据,结果可靠,已被国内外多数政府部门和有资质的检验机构选择[7-9]。然而,全自动生化鉴定设备和配套耗材的经济成本较高,一般实验室难以负担。基质辅助激光解吸电离飞行时间质谱(Matrix-assisted-laser-desorptionionizer time of flight mass spectrometry,MALDITOF-MS)主要依据菌体蛋白质谱,与现有数据库进行比对得到鉴定结果,是20世纪80年代末问世的一项新技术,操作简单、鉴定迅速,为快速、准确的微生物鉴定带来新方向,被广泛应用于临床微生物鉴定。然而,其鉴定结果判断完全依赖于现有数据库,而我国缺少自有数据库的问题一直没有得到解决,导致鉴定结果不够准确[10-11]。截至目前,哪种方法为微生物鉴定的较佳选择尚未定论。
微生物鉴定的时效性、鉴定时间和水平被认为是鉴定方法选择的决定性因素。本研究选用16S rDNA 序列测定、MALDI-TOF-MS 和VITEK全自动细菌鉴定系统3 种方法鉴定沙门氏菌和金黄色葡萄球菌,以期确定一种时效性高的鉴定方法,为微生物参考物质研制奠定基础。
1 材料与方法
1.1 菌株
食源性沙门氏菌92 株,为西北农林科技大学食品科学与工程学院微生物食品安全研究室前期分离得到,菌株已通过PCR 和血清学鉴定。临床患者源沙门氏菌118 株,由上海市疾病预防与控制中心惠赠。食源性金黄色葡萄球菌18 株,由中国食品药品检定研究院惠赠。鉴定中使用大肠杆菌ATCC 25922 和金黄色葡萄球菌ATCC 29213作为质控对照。
1.2 试剂与仪器
Luria-Bertani(LB)琼脂培养基、Brain Heart Infusion(BHI)琼脂培养基,北京陆桥技术股份有限公司;VITEK COMPACT 2 GP 细菌鉴定卡、GN细菌鉴定卡,法国梅里埃公司;MALDI-TOF-MS鉴定用HCCA 基质,德国Bruker 公司;无水乙醇、乙腈、甲酸等,广东光华科技有限公司。
NU-425-400E 超净工作台,苏州净化设备有限公司;yCycler PCR 扩增仪、170-3672 琼脂糖凝胶电泳仪,Bio-Rad 公司;VITEK 2 COMPACT 60全自动细菌鉴定系统、手持式比浊仪,法国梅里埃公司;MALDI-TOF-MS,德国Bruker Auto-flexcontrol 公司;GNP-9080 生化培养箱,北京科伟实验仪器有限公司;LAC-5080S 冰箱,日本三洋公司。
1.3 试验方法
1.3.1 16S rDNA 扩增和测序 用无菌牙签挑取适量LB 琼脂培养基活化18~24 h 后的菌株培养物,均匀分散于装有100 μL 无菌ddH2O 的PCR管,振荡。PCR 仪中100 ℃加热10 min 后,13 200 r/min 离心10 min,取上清液,即为DNA 模板,备用。
PCR 扩增用引物为F:5'-AGAGTTTGATCC TGGCTCAGPCR-3' 和 R:5'-GGTTACCTTGTT ACGACTT-3'。扩增程序为:94 ℃10 min;94 ℃1 min,55 ℃1 min,72 ℃2 min,35 个循环;72 ℃10 min。PCR 产物经1%琼脂糖凝胶电泳检测后,低温条件送杨凌天润奥科生物科技有限公司测序。在NCBI 数据库比对并下载标准菌株同源序列,使用MEGA 7 软件邻接法(Neighbor-Joining)自举1 000 次构建进化树[12-13],使用iTOL 软件对进化树处理。
1.3.2 MALDI-TOF-MS 鉴定方法 样品预处理采用甲酸提取法,具体为:无菌接种环挑取已在BHI 琼脂平板上培养12 h 的单菌落,转移到装有300 μL 无菌超纯水的Eppendorf 管中,混匀。向Eppendorf 管中加入900 μL 无水乙醇,涡旋振荡1 min。13 200 r/min 离心2 min,去除上清液。相同条件再次离心。向装有菌泥的Eppendorf 管中加入50 μL 甲酸水溶液(V甲酸∶V水=7∶3),涡旋振荡,充分混匀。再向管内加入50 μL 乙腈,移液枪反复吹打混均,13 200 r/min 离心2 min。取1 μL 上清液滴加到清洁的靶板上,确认每个样本的靶位与所用样本跟踪方案一一对应。靶板在室温下晾干后,再将1 μL IVD HCCA 基质溶液均匀涂敷于每个样本表面,室温晾干。将靶板插入预热的MALDI-TOF-MS 仪运行程序。
1.3.3 VITEK 全自动细菌鉴定系统鉴定方法 用无菌牙签挑取适量已在BHI 琼脂培养基上活化12 h 的单菌落,均匀分散在装有3 mL VITEK 全自动细菌鉴定系统配套测试液的比浊管中,调整比浊管麦氏浊度为0.48~0.52。将VITEK 全自动细菌鉴定系统生化鉴定试剂板的虹吸管插入菌悬液后,整体移至VITEK 2 COMPACT 60 全自动细菌鉴定系统进行鉴定。沙门氏菌使用GN 卡,金黄色葡萄球菌使用GP 卡鉴定。
1.3.4 数据分析 使用R 语言软件包(v3.6.1)中的ggplot2 包对鉴定结果进行分析。
2 结果与分析
2.1 16S rDNA 序列测定
210 株(100.0%)沙门氏菌均可与NCBI 基因库中登录号为NR 074799.1 和NR 074910.1 的沙门氏菌聚为一簇,鉴定为沙门氏菌(图1)。

图1 210 株沙门氏菌16S rDNA 系统发育树Fig.1 Phylogenetic tree of 16S rDNA sequence of 210 Salmonella strains
NCBI 数据库比对结果显示,除1 株菌16S rDNA 序列与参比菌序列覆盖度小于95%外,其余序列的覆盖度(Cover number)均在98.0%~99.0%,鉴定结果可信。序列的相似性(Identity)均集中在99.0%~100.0%,且呈正态分布(图4a)。16S rDNA 序列分析结果表明鉴定菌株均为沙门氏菌,可将其鉴定到属水平。
18 株金黄色葡萄球菌中,15 株(83.3%)菌的16S rDNA 序列均可与基因库登录号为NR 113956.1 和NR 115606.1 的金黄色葡萄球菌聚在一簇,编号为SA002、SA004 和SA016 的菌株16S rDNA 序列与其它菌株亲缘性较远(图2),该3 株菌仅能被鉴定到葡萄球菌属水平,未能鉴定到种水平。18 株金黄色葡萄球菌16S rDNA 序列与参比菌株相应序列覆盖度集中在99.0%,相似性集中在99.0%,鉴定结果可信(图4a)。

图2 18 株金黄色葡萄球菌16S rDNA 序列系统发育树Fig.2 Phylogenetic tree of 16S rDNA sequence of 18 Staphylococcus aureus strains
2.2 MALDI-TOF-MS 鉴定
基于德国Bruker 公司MALDI-TOF-MS 数据库提供的评分标准,210 株(100.0%)沙门氏菌鉴定结果评分均大于2.0,为高度匹配,可被鉴定为沙门氏菌属,均未鉴定到种。其中,142 株(67.6%)沙门氏菌鉴定结果评分大于2.3,为极高度匹配(表1、图3)。取中间值2.376 作为标准值进行数据处理,沙门氏菌的匹配度集中在100.0%~105.0%(图4a)。
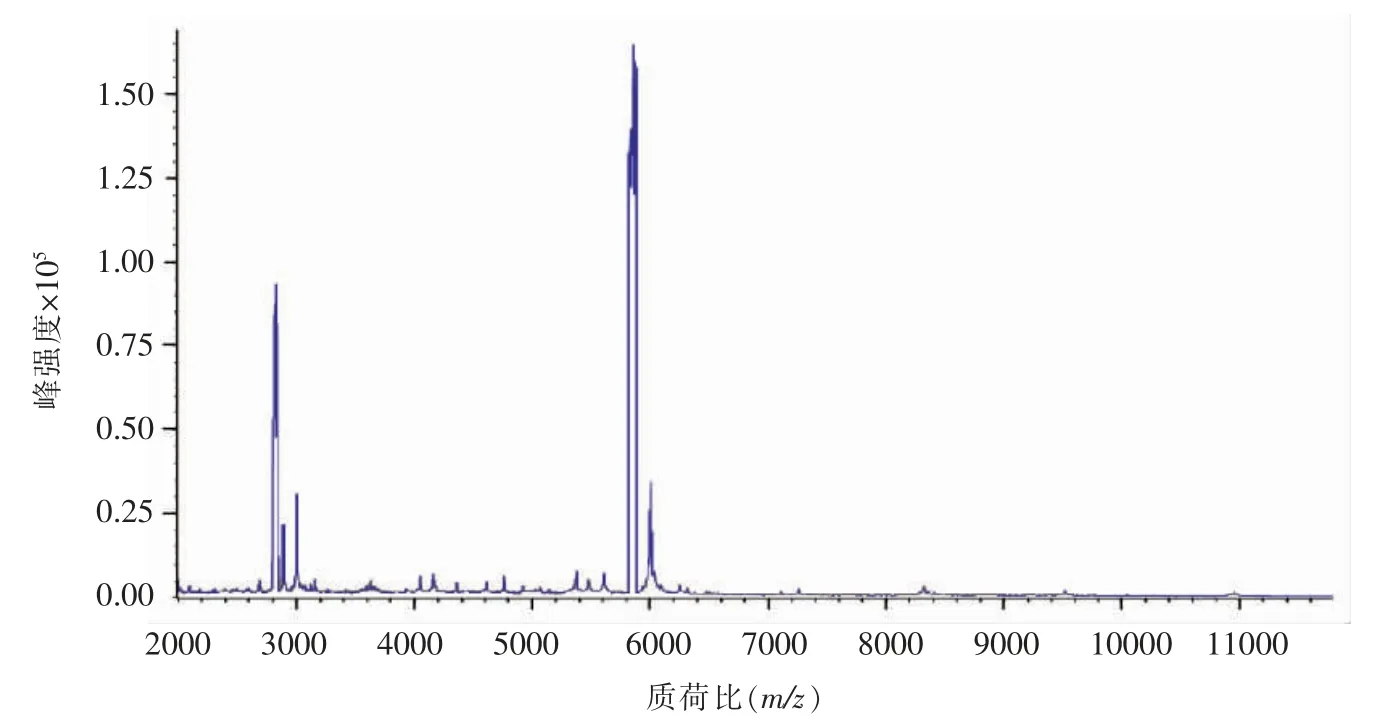
图3 1 株沙门氏菌MALDI-TOF-MS 鉴定色谱图Fig.3 MALDI-TOF-MS identification chromatogram of a Salmonella strain
18 株(100%)金黄色葡萄球菌MALDI-TOFMS 鉴定结果评分均大于2.0,为高度匹配,鉴定为金黄色葡萄球菌。其中,17 株(94.4%)菌鉴定评分均大于2.3,为极高度匹配(表1)。取中间值2.44作为标准值进行数据处理,匹配度集中在99.0%~100.0%(图4c)。

表1 210 株沙门氏菌和18 株金黄色葡萄球菌MADIL-TOF-MS 鉴定结果Table 1 Identification results of 210 Salmonella and 18 Staphylococcus aureus strains by MALDI-TOF-MS
2.3 VITEK 全自动细菌鉴定系统鉴定
210 株(100.0%)沙门氏菌均可被鉴定到沙门氏菌属水平,未鉴定到种。沙门氏菌VITEK 全自动细菌鉴定系统鉴定结果匹配度集中在97.0%~99.0%,其中,165 株(78.6%)菌鉴定结果匹配度大于等于98.0%,仅有2 个结果的匹配度小于95.0%(表2、图4a)。18 株(100.0%)金黄色葡萄球菌均被鉴定为金黄色葡萄球菌,其中16 株(88.9%)菌鉴定结果的匹配度大于等于98.0%,仅有1 个结果的匹配度小于95.0%。

表2 210 株沙门氏菌和18 株金黄色葡萄球菌VITEK 全自动细菌鉴定系统鉴定结果Table 2 Identification results of 210 Salmonella and 18 Staphylococcus strains using VITEK
2.4 数据分析
通过3 种方法对沙门氏菌检测结果间的相关性分析表明,各检测结果各自独立,没有影响,结果可信(图4b)。对金黄色葡萄菌检测结果相关性分析可知,质谱和16S rDNA 序列鉴定结果一致性间有0.46 的负相关,其余各检测结果独立,互相没有影响,结果可信(图4d)。

图4 3 种鉴定方法对210 株沙门氏菌及18 株金黄色葡萄球菌鉴定结果的相关性分析Fig.4 Correlation analysis of 3 identification methods for 210 Salmonella strains and 18 Staphylococcus aureus strains
3 讨论与结论
影响微生物检测和鉴定的主要因素有培养时间、培养基、菌株是否为纯培养物、生化反应是否明显、不同厂商提供的数据库差异、仪器差异和人员操作等[1]。就检测时间而言,基于细菌的保守基因序列,即16S rDNA 片段,扩增测序需要时间最长,约8~10 h,一次可检测约96 个样品;VITEK 全自动细菌鉴定系统鉴定不同种类细菌一般约需5~8 h,一次可检测约40 个样品;MALDI-TOF-MS约需2~4 h,针对本试验所用仪器靶板,一次最多可检测384 个样品。若基于检测时间和通量评判,MALDI-TOF-MS 效率最高,远远高于基因序列测定和VITEK 全自动细菌鉴定系统鉴定,因此MALDI-TOF-MS 也被认为是高通量鉴定最优最快速的方法。就鉴定结果的精确性而言,3 种方法均可以将沙门氏菌鉴定到属,准确性为100.0%,然而不能将其全部鉴定到种或具体的血清型。3种方法均可将金黄色葡萄球菌鉴定到属,准确性为100%,VITEK 全自动细菌鉴定系统和MALDITOF-MS 还可以进一步将其准确鉴定到种。何江等[14]比较了MALDI-TOF-MS 和16S rDNA 序列对19 株弧菌科细菌的鉴定结果,表明2 种方法的鉴定结果一致,MALDI-TOF-MS 可作为一种新的鉴定、检测技术,可缩短检测周期,然而有个别拟态弧菌和霍乱弧菌在16S rDNA 序列聚类树状图中的位置和亲缘关系差异较大。这与本研究通过16S rDNA 序列对金葡菌的鉴定结果比较相似,导致差异出现的原因可能与菌株16S rDNA 序列在系统发育树构建时所选择的参照菌株外群和聚类方法有很大关系,也可能与这些菌株来源比较广泛,其亲缘关系本身就比较远有关。Kang 等[15]研究发现,在对沙门氏菌血清型鉴定时,MALDI-TOFMS 比使用16S rDNA 序列分析更具有优势,然而种鉴定时没有明显差异;他们使用自建库作为蛋白质谱比对条件,而本研究以Bruker 公司提供的商业数据库作为对比,因此没有将沙门氏菌鉴定到血清型。刘娜等[16]也通过本研究使用的3 种方法鉴定了3 种常见的食源性致病菌,其中对金黄色葡萄球菌和沙门氏菌的质谱鉴定结果和基因测序结果与本研究基本相同,然而在生化鉴定时可以将少数特殊血清型的沙门氏菌鉴定到种。
采用分子生物学方法对菌株检测时,常使用PCR 扩增其特定的序列,然后对其测序分析。对于分离得到的某一种微生物,或者已经通过生化试验或其它方法初步确定种属的菌,可扩增其特异性基因片段,如沙门氏菌的保守基因invA[17-18]。然而,对于分离得到的多种微生物,或者菌种间亲缘关系较近,则可选择扩增其具有普遍存在性和序列保守性特点的16S rDNA 序列。近年来,随着高通量测序和大数据分析等技术的快速发展与应用,基于16S rDNA 的微生物鉴定技术已逐渐成为主流。由于16S rDNA 并不能将微生物鉴定到种及亚种以下水平,因此许多研究结合ITS 序列分析,弥补了16S rDNA 保守性强、变异程度弱的缺点[19]。有研究认为,16S rDNA 序列间相似性如果达到95%~97%以上即为同一属,相似性达到99%以上即为同一种[20-21],然而一些相似的种之间仍不能明确鉴定,如肠杆菌属(Enterobacter)和泛菌属(Pantoea)的16S rDNA 序列也存在99%相似性[22]。因此,也有学者基于16S rDNA 序列,建立了更快速、更灵敏的细菌鉴定方法[23-24]。当然,对沙门氏菌来说,还可以采用血清学方法进行鉴定,然而仍有部分沙门氏菌存在荚膜或缺乏鞭毛导致无法进行血清分型,因此也使用多位点序列分型(Multi locus sequence typing,MLST)和脉冲场凝胶电泳(Pulse field gel electrophoresis,PFGE)等分子分型技术辅助检测[25]。
MALDI-TOF-MS 通过检测微生物蛋白指纹图谱并将之与数据库中的标准菌株蛋白图谱对比进行鉴定。极高的准确性和较快的检测速度使得该方法被广泛应用于临床、环境和食品等研究领域的微生物检测[26]。然而,高额的设备和维护费使得一些普通实验室无法承担[11,27]。其次,基质的选择、培养条件、菌株处理方式、菌体量、数据库等因素也会影响试验结果[28-30]。本研究中5 株肠炎沙门氏菌的鉴定结果在第2 匹配项中评分低于2.0,为中度匹配;1 株爱丁堡沙门氏菌和1 株汤普逊沙门氏菌的鉴定结果在第2 匹配项中评分低于2.0且鉴定为克氏柠檬酸杆菌,这可能由于柠檬酸杆菌属和沙门氏菌属同属于肠杆菌科,其蛋白特性有相似之处,在Bruker 公司提供的数据库中沙门氏菌属数据不全,存在混淆的可能。本研究结果也证明了现有的商业菌种库谱图并不能将沙门氏菌属的微生物鉴定到亚种以下,无法确定其血清型,而一些学者通过自己建库则可以区分伤寒和非伤寒型沙门氏菌[31]。一些研究认为,鉴定评分≥2.0即鉴定到种,评分<2.0 且≥1.7 即鉴定到属水平,评分<1.7 即未鉴定出结果[11],按照这样的标准,本研究所有菌株均可通过MALDI-TOF-MS 鉴定到种水平。Seng 等[32]用此方法鉴定了1 660 株微生物,准确率高达95.4%,其中84.1%鉴定到种水平,11.3%鉴定到属水平,并且同步使用16S rDNA 基因检测,对比了2 种试验方法的效率,认为MALDI-TOF-MS 的综合成本约为传统基因检测的22%~23%。Liang 等[30]认为,与VITEK 全自动细菌鉴定系统鉴定方法相比,MALDI-TOF-MS 鉴定的经济成本约为其1/3,时间成本约为其1/7。质谱法检测使用的靶板一次性可以检测384 个样品,平均每个样品仅需30 s 左右,本研究采用的样品前处理方法每个样品约需3~5 min。当然,也可以采用直接点样的方法,更加节约时间,但对技术人员的操作要求较高。
VITEK 全自动细菌鉴定系统对微生物生长时间、是否为纯培养及菌种量都有较高的要求,该鉴定系统准确性较高,平均识别率为93.8%,因此,被众多机构推荐、批准作为参考标准[8,33]。本研究中所有菌株的鉴定结果准确无误,然而也有报道显示临床中VITEK 全自动细菌鉴定系统对不常见血清型的沙门氏菌判断错误[34]。对沙门氏菌的鉴定结果不能准确到血清型,然而其鉴定结果中的某些特性可以为血清型鉴定方向提供依据[7]。
综上所述,本研究采用的16S rDNA 序列测定、MALDI-TOF-MS 和VITEK 全自动细菌鉴定系统均可将210 株(100.0%)沙门氏菌鉴定为沙门氏菌属,MALDI-TOF-MS 和VITEK 全自动细菌鉴定系统可以将18 株(100.0%)金黄色葡萄球菌鉴定为金黄色葡萄球菌,16S rDNA 序列测定对18 株(100.0%)金黄色葡萄球菌准确鉴定到属水平,15株(83.3%)金黄色葡萄球菌准确鉴定到种水平。就鉴定结果对细菌种属的评判准确性而言,MALDITOF-MS 和VITEK 全自动细菌鉴定系统略高于16S rDNA 序列测定方法。相对而言,MALDITOF-MS 鉴定的时效性均远高于其它2 种方法,约占VITEK 全自动细菌鉴定系统鉴定时间的5%,16S rDNA 序列鉴定时间的10%。综合考虑,选择MALDI-TOF-MS 进行菌种鉴定最优,在实践中,具体如何选择试验方法需结合实验室设备配备和实验人员技术水平等综合考虑。
