基于改进免疫遗传算法的近红外光谱变量选择方法
2021-11-03陶焕明高美凤
陶焕明,高美凤
(江南大学 轻工过程先进控制教育部重点实验室,物联网工程学院,江苏 无锡 214122)
近红外光谱分析检测技术具有快速、无损以及低成本的优势,已经广泛运用于众多领域。如何快速有效地提取出有用光谱信息,即光谱变量选择,是近红外光谱技术研究的重点之一。目前,许多国内外学者提出了大量基于不同原理、策略的变量选择方法,如通过无信息变量消除法(UVE)对偏最小二乘(PLS)回归系数设定阈值限制来选择有效变量[1];区间偏最小二乘(iPLS)[2]、移动窗口偏最小二乘(MWPLS)[3]、向前和向后间隔偏最小二乘(FB-iPLS)[4]、区间随机蛙跳(iRF)[5]等则均为以光谱区间为筛选对象的方法。近年来,以智能算法及其改进算法为核心进行波长组合优化的算法研究较多,主要有遗传算法(GA)[6-8]、粒子群算法(PSO)[9]、模拟退火算法(SAA)[10]、蚁群算法(ACO)[11]等。GA算法具有群体搜索特性,广泛应用于多种领域,但易过早收敛。刘鑫等[12]将改进的GA 算法用于土壤养分预测,采用一种改进的实数编码差分变异算子,扩大了全局最优解搜索空间,加快了收敛速度,提高了预测精度。免疫遗传算法(IGA)在GA 算法的基础上引入了生物免疫机制,本质也是对GA 算法的一种改进[13-15]。相较于GA算法及其改进算法,IGA算法避免了容易过早收敛的缺点,维持了种群中个体的多样性,可有效防止算法陷入局部最优,但其精度性能仍存在提升空间。国内外对IGA 算法的研究也从未停止脚步[16-17]。
本文提出了一种改进免疫遗传算法(iIGA)用于近红外光谱变量选择。IGA 算法结合了适应度(亲和度)和抗体浓度形成聚合适应度进行后续的遗传操作来维持种群的多样性以及免疫平衡,抗体浓度越高,越受抑制,反之亦然。其中抗体浓度是指与抗体相似的个体总数和群体总数的比值,其中抗体之间相似度达到相应的阈值判定抗体相似。原算法中固定了抗体相似度阈值,可能会存在阈值过大或者过小而导致抗体浓度不具备差异性,使得引入的抗体浓度失去意义。本文提出自适应相似度阈值,所选取的阈值可使群体抗体浓度的标准差达到最大,完全体现出抗体浓度的差异性;同时采用精英保留策略,将亲和度高的若干个抗体存入到记忆细胞中,直接复制到下一代;另外在算法中引入贪心算法思想,将算法由局部最优扩展到全局最优。将每一次迭代中的亲和度最优基因随机反转一位基因位进行局部性探优,并且保证探优之后的基因亲和度值大于探优之前;然后以探优成功后的基因替换本次迭代中亲和度最差基因,使得算法朝着正确的方向进行局部性探优,最终达到全局最优,提升模型预测精度。
1 算法原理
1.1 IGA算法实现原理
GA算法中通过模拟种群个体对环境适应度大小的不同进行优胜劣汰,经过不断的迭代更新,使种群个体对环境的适应度不断提升,最终达到群体适应度的最佳表现。但在算法迭代后期,种群个体之间的差异性越来越小,导致算法陷入局部最优。因此,免疫遗传算法在个体对环境的适应度中引入抗体浓度,促使抗体浓度越高,适应度反遭抑制,维持了种群中个体的多样性,可有效防止算法陷入局部最优。
1.1.1 产生初始解 在种群中设置N个个体,作为算法模型的抗体角色。将需筛选的近红外光谱波段均分为L个波段,每个波段包括多个波长点,对应抗体基因上的基因位,按照0-1 形式对其进行随机编码,1表示选中该波段,0表示未选中。
1.1.2 计算亲和度 对抗体基因进行编码后,将选中的波段与对应的理化值进行PLS建模,亲和度即以建模得到的校正集均方根误差(RMSEC)和相关系数(R)为变量的函数值,本文设定亲和度Z为:

1.1.3 计算抗体浓度 抗体浓度用与抗体i相似的抗体个数ci与群体总个数N的比值di来表示,即:

抗体之间相似度达到一定阈值,即判定抗体相似。相似度是抗体与其他抗体之间的相似程度,其度量方法可基于信息熵、欧氏距离、基因值距离以及海明距离。其中海明距离主要用于计算离散编码,符合本文的0-1编码形式。因此本文采用海明距离作为抗体相似度的度量方法,具体公式如下:


式(5)表示抗体pi和pj的相似度,L为抗体基因编码长度;式(4)中分别表示第i个抗体基因上的第k位基因位和第j个抗体基因上的第k位基因位。
1.1.4 计算聚合适应度 将亲和度和抗体浓度融合成聚合适应度。在种群更新的过程中,抗体被选择的概率与聚合适应度成正比。因此,聚合适应度也反映了亲和度与抗体浓度在种群更新中的影响力。亲和度越高的个体,聚合适应度也越高;抗体浓度越高的个体应被抑制,以保证种群的多样性,因此聚合适应度越低。本文设定的聚合适应度PZ为:

式中λ、μ为某一常数,分别反映了亲和度和抗体浓度期望被选择到下一代的相对重要的参数。
1.1.5 遗传操作 通过轮盘赌选择法进行基因的复制。在轮盘赌中,种群中个体被选择的概率与对应的聚合适应度成正比,即聚合适应度越高的个体被选择的概率越大,反之,聚合适应度越低的个体被选择的概率越小。第i个个体被选择的概率Pi可表示为:

式中,PZi和PZj分别为第i个个体和第j个个体所对应的聚合适应度,n为种群大小。通过单点交叉对种群个体进行交叉处理。设定交叉概率为pc,单点交叉操作即交换两个个体的部分基因值。最后设定变异概率pm,对于0-1基因链码,随机挑选c个基因位,以变异概率pm对这些基因位上的值取反,即0变1,1变0。
1.1.6 更新迭代 迭代次数达到K时,选取种群中最优个体作为最终候选个体,其对应的波段作为最终筛选波段。
1.2 iIGA算法
1.2.1 自适应相似度阈值 原算法中固定了抗体相似度阈值,而每次更新迭代,抗体相似度都会发生变化,这可能会导致阈值过大或者过小而使抗体浓度不具备差异性,使得引入的抗体浓度失去意义。本文提出自适应相似度阈值,将相似度阈值以一定的间隔从群体相似度最小值遍历至群体相似度最大值,所选取的相似度阈值使得群体抗体浓度的标准差达到最大,可完全体现出抗体浓度的差异性。
1.2.2 精英保留策略 在种群中另外设置M个个体,作为算法模型中的记忆细胞角色,并对其基因进行0-1 随机编码,同时计算其亲和度。由于种群的选择具有不确定性,可能造成最优个体的丢失。因此将亲和度最高的M个个体,作为记忆细胞储存起来,直接以概率1 复制到下一代种群中。在每一次迭代中,将当前代数中亲和度最高的M个个体,存入到记忆细胞中以进行记忆细胞更新。
1.2.3 引入贪心算法思想 贪心算法在求最优解时,不必考虑整体最优,而是寻找在某种意义上仅是局部最优的解,用局部最优构造全局最优,一步一步逼近全局最优解。在算法的每一次更新迭代中,对亲和度最优个体基因进行局部性探优,即随机取反一个基因位并且保证探优之后的亲和度大于原先个体的亲和度,如果小于,则恢复被改变的基因位,再随机重取一个基因位进行探优,直到探优之后该个体的亲和度大于探优前个体的亲和度。如果重取次数达到上限值,则将原先最优个体直接替换最差个体。
1.3 iIGA算法实现步骤
Step1:初始化种群。在种群中设置N个个体,作为抗体角色。将光谱波段均分并进行0-1 随机编码。
Step2:计算亲和度。对抗体基因进行编码后,将选中的波段与对应的理化值进行PLS 建模,并利用式(1)计算亲和度。
Step3:初始化记忆细胞。在种群中设置M个个体,作为算法模型的记忆细胞角色,并对其基因进行0-1随机编码,同时计算亲和度。
Step4:计算抗体相似度。采用海明距离作为抗体相似度的度量方法,公式如式(3)~(5)所示。
Step5:自适应相似度阈值计算抗体浓度。采用自适应相似度阈值对抗体进行相似判定,将群体抗体浓度标准差达到最大时,即抗体浓度差异性最大时的阈值作为最终自适应相似度阈值。抗体浓度由式(2)计算。
Step6:计算聚合适应度。如式(6)所示,将亲和度和抗体浓度融合成聚合适应度。
Step7:引入贪心算法思想。对每一次迭代中亲和度最优的个体基因进行局部性探优,即随机取反一个基因位并保证探优之后的亲和度大于原先的个体。
Step8:更新记忆细胞。重新计算群体中个体的亲和度,选取最高的M个个体作为记忆细胞储存起来,直接以概率为1复制到下一代种群中。
Step9:进行遗传操作。通过轮盘赌选择法进行基因的复制。在轮盘赌中,个体被选择的概率如式(7)所示。然后通过单点交叉、变异对种群个体进行处理。
Step10:更新迭代。迭代次数达K时,选取种群中最优个体作为最终候选个体,其对应的波段作为最终筛选波段。
2 实验部分
2.1 实验数据
2.1.1 数据来源 本研究引用于eigenvector 网站上开源的一组标准玉米近红外光谱数据集。该数据集由80 个玉米样品组成,分别用m5spec、mp5spec、mp6spec 3 种不同的近红外光谱仪进行测试,包含了其淀粉、蛋白质、水分以及油脂含量。以间隔为2 nm在1 100~2 498 nm波长范围上收集(700 波长点)。本文选用mp5spec 仪器扫描得到的光谱数据,并选择其中的淀粉和蛋白质含量进行研究。
2.1.2 样本划分 图1 为原始光谱图,数据集由80 个玉米样品光谱组成。

图1 原始光谱图Fig.1 Original spectrogram
采用Kennard-Stone(KS)方法将80个样本分成校正集和预测集。划分结果为校正集样本52个,预测集样本28 个。校正集和预测集的淀粉和蛋白质含量值统计见表1。由表1 可知,校正集样本与预测集样本的平均值(Avg)和标准差(Std)相差不大,通过KS 方法划分的数据集,保证了校正集样本均匀分布。
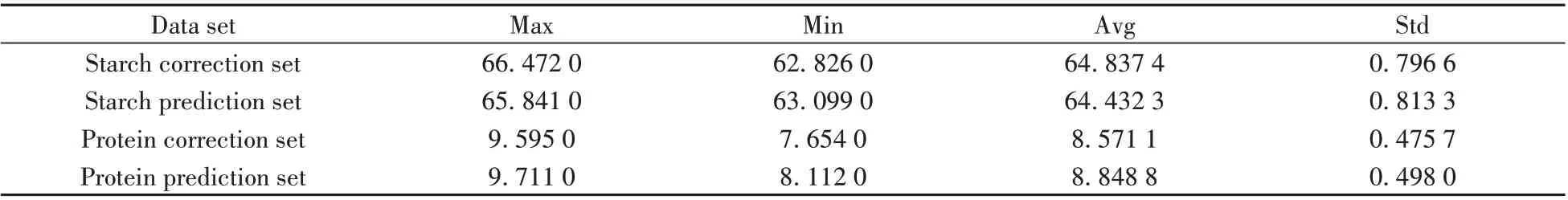
表1 校正集和预测集中淀粉和蛋白质含量值统计Table 1 Statistics of starch and protein content in correction set and prediction set(g/100g)
2.2 实验设备
本研究使用的是一台戴尔计算机,处理器是Intel(R)Core i5-9400,CPU 主频为2.90 GHz,Windows10、64位操作系统,显卡为GT710,所有计算均在MATLAB 2016a中进行。
2.3 实验方法
为了考察本文提出的iIGA 算法对建模预测的效果,验证该算法的有效性和优越性,分别用全谱波长、GA、IGA 以及iIGA 算法对数据集中玉米淀粉和蛋白质含量的建模效果进行了对比。由于原始光谱波长点数较多,如对波长点进行优选组合,运算效率将会非常低,因此将原光谱均分为若干个子区间,分别用上述算法进化迭代,获取最大适应度值所对应的优选子区间组合。根据基因优选的波段建立PLS 模型,计算出模型的相关系数(R)和预测集均方根误差(RMSEP),适应度函数F为:

将原光谱在1 100~2 498 nm 范围之间的700 个波长点数划分为175 个等距区间,即遗传编码长度为175,每一个基因位包含4个波长点数。设定群体个数为50个,交叉概率为0.85,变异概率为0.05,最大迭代数为100代。在种群进化过程中寻找最大迭代次数内进化过程的最优适应度个体。
对于iIGA 算法,通过实验选取的最佳记忆细胞数目为8 个。相似度阈值从抗体相似度最小值遍历至抗体相似度最大值,遍历跨度设置为0.001,当所选取的相似度阈值使得群体抗体浓度的标准差达到最大时,即为最终的相似度阈值。经过多次实验测试,设定聚合适应度最佳参数λ为0.7,μ为1.25。设定贪心寻优时的重取次数上限为20 次。由于上述算法存在随机性,因此将以上3种算法分别运行50次求平均。程序地址为https://github. com/thmguai/Journal_of_analytical_testing_thm.git。
3 结果与讨论
3.1 算法优化结果
3.1.1 自适应相似度阈值 相似度阈值从群体相似度最小值遍历至群体相似度最大值。如图2 所示,以玉米淀粉含量数据集为例,每取一个阈值,都会有与之一一对应的群体抗体浓度标准差(STD),当所选取的阈值使得标准差达到最大值时,表示群体抗体浓度差异性达到最大,可使IGA算法中提出的保留物种多样性思想(引入抗体浓度)最大化。

图2 群体抗体浓度标准差随抗体相似度阈值的变化Fig.2 Variation of antibody concentration STD with the threshold of antibody similarity
3.1.2 精英保留策略 将亲和度最高的8 个个体作为记忆细胞储存起来,直接以概率为1 复制到下一代种群中。如图3 所示,以玉米淀粉含量数据集为例,记忆细胞中储存的精英群体的亲和度均值远高于总群体,并且随着迭代次数逐渐提升,可防止某些最优个体的丢失,使种群朝着正确的方向进行更新,有效地避免了陷入局部最优。

图3 精英群体和总群体的抗体亲和度均值随迭代次数的变化图Fig.3 Variation of the mean antibody affinity of elite group and general population with the number of iterations
3.1.3 贪心算法思想 在算法的每一次迭代中,对抗体亲和度最优个体进行局部性探优,以局部最优逐渐构建出全局最优。如图4 所示,以玉米淀粉含量数据集为例,在对亲和度最优抗体进行局部寻优后,群体中最优个体较之前几乎都有了提升,突破了局部探优之前的最优领域,避免了过早陷入局部领域最优,使算法朝着通往全局最优的较合理的路径进行更新迭代。

图4 局部探优前后群体中最优抗体的亲和度随迭代次数的变化图Fig.4 Variation of the affinity of the optimal antibody with the number of iterations before and after local optimization
3.2 结果比较
iIGA 算法合理地优化了算法的性能,同时增强了算法全局寻优能力。图5 为用GA、IGA 以及iIGA算法筛选之后的变量分别与淀粉和蛋白质含量测试50次进行建模分析的预测均方根误差值。
由图5 可知,用iIGA 算法进行建模,预测玉米淀粉和蛋白质含量的精度较IGA 算法有着明显的提升,证明了该算法较原算法提升了性能,增强了全局寻优能力。表2和表3分别为各算法测试淀粉含量和蛋白质含量的各项性能数据。

图5 GA、IGA以及iIGA算法运行50次的淀粉预测RMSEP值(A)和蛋白质预测RMSEP值(B)Fig.5 Starch predicted RMSEP value(A)and protein predicted RMSEP value(B)after GA,IGA and iIGA run 50 times
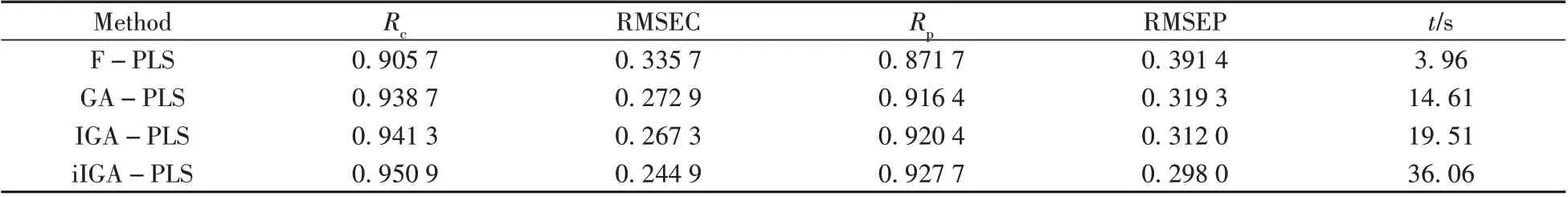
表2 不同变量筛选算法对玉米淀粉含量的预测结果Table 2 Prediction results of corn starch content by different variable screening methods

表3 不同变量筛选算法对玉米蛋白质含量的预测结果Table 3 Prediction results of corn protein content by different variable screening methods
从表2可得,在玉米淀粉含量的预测上,相比于全谱PLS(F-PLS),GA 算法模型的校正集均方根误差RMSEC从0.335 7降到0.272 9,校正集预测精度提升18.7%。模型的预测集均方根误差RMSEP从0.391 4 下降到0.319 3,预测集预测精度提升18.4%。而IGA 算法作为GA 的改进算法,预测精度有所提升。IGA 算法的RMSEC 为0.267 3,RMSEP 为0.312 0,预测精度分别较GA 算法提升了2.0%和2.3%。本文提出的iIGA 算法相较于IGA 算法,预测速度有所降低,但校正集相关系数Rc从0.941 3 提升至0.950 9,预测集相关系数Rp从0.920 4提升至0.927 7,RMSEC从0.267 3降到0.244 9,校正集预测精度提升8.4%;RMSEP 从0.312 0降到0.298 0,预测集预测精度提升4.5%。对IGA-PLS和iIGAPLS两组分别测试50次的RMSEP值进行显著性检验,F值为165.22,P值为9.5×10-23,P值小于0.05,因此两组数据存在显著性差别。由表2 数据分析可得,在淀粉含量光谱数据集中,iIGA 算法相较于原算法预测精度有了明显的提升。
从表3 可得,在玉米蛋白质含量的预测上,相比于全谱PLS,GA 算法模型的校正集均方根误差RMSEC 从0.147 4 降到0.116 0,校正集预测精度提升21.3%。模型的预测集均方根误差RMSEP 从0.178 9 下降到0.136 8,预测集预测精度提升23.5%。而IGA 算法的RMSEC 为0.112 4,RMSEP 为0.124 4,预测精度分别较GA 算法提升了3.1%和9.1%。本文提出的iIGA 算法相较于IGA 算法,预测速度有所降低,但校正集相关系数Rc从0.972 2 提升至0.976 4,预测集相关系数Rp从0.966 9 提升至0.974 2,RMSEC 从0.112 4降至0.103 7,校正集预测精度提升7.7%,RMSEP 从0.124 4降至0.110 3,预测集预测精度提升11.3%。同样对IGA-PLS 和iIGA-PLS 两组分别测试50 次的RMSEP 值进行显著性检验,F值为182.05,P值为4.5×10-24,P值小于0.05,因此两组数据存在显著性差别。由表3数据分析可得,iIGA算法应用在蛋白质含量光谱数据集中相较于原算法预测精度有了明显的提升。
4 结 论
提出了一种近红外光谱波长变量选择算法,即改进的免疫遗传算法,对原算法进行了3 个方面的改进优化,分别是自适应相似度阈值、引入精英保留策略和贪心算法思想。采用该算法进行波长选择后,建立了更加稳健、预测精度更高的PLS 模型。在对玉米光谱数据集中淀粉和蛋白质含量的预测建模中,模型的预测均方根误差RMSEP 分别为0.298 0和0.110 3,相比原算法,模型的预测精度分别提高了4.5%和11.3%,证实了iIGA 算法的优越性。在要求预测精度的场景下,本文提出的改进IGA波长变量选择算法是一个更佳的选择。
