DNA-纳米颗粒共聚体在最大匹配问题中的应用
2021-10-31麻晶晶
麻晶晶 许 进
①(山西财经大学统计学院 太原 030000)
②(北京大学信息科学技术学院 北京 100871)
1 引言
纳米技术的研究已经取得了很大的进步,尤其是由于DNA所具有的特殊的性质,DNA分子已经被用来构建不同的纳米组件和生物材料。DNA纳米技术不仅为纳米尺度的材料构建提供了新的方法和技术,而且在DNA计算、纳米尺度的生物机器和基因治疗等方面也具有特殊的用途。
利用DNA分子构建纳米尺度的组件是通过自组装的方式。在自然界中,自组装是一种非常常见的形成系统的方式。在生物系统中,生物有机体的细胞是由很多种生物分子通过自组装形成的,同时,生物有机体利用生物化学方法来控制它自身的分子和化学活动。分子计算将生物有机体的生物化学过程与电子微处理器控制电子机械元件的过程联系起来,提出了一种生物化学算法,这是一种全新的计算模型。
DNA自组装在生物有机体中也是非常常见的,例如两条具有互补的黏性末端的DNA单链能够杂交形成双链,这个过程是由一系列的非共价键实现的,例如氢键、范德华力、疏水性作用和静电力等等。因此分子计算可以通过特定实验室条件下的DNA分子自组装的生物化学过程来实现。
目前,利用DNA的自组装指导纳米颗粒形成3维结构的技术极大地促进了一些基本的科学问题的研究。Sharma等人[1]设计了一种利用DNA-纳米金颗粒共聚体的自组装过程来构建一种纳米金3维管状结构的方法,这种方法可以通过控制纳米金颗粒的大小构建不同的结构。Mastroianni等人[2]报道了一种构建3维纳米金结构的方法,他们利用修饰了DNA链的纳米金颗粒的自组装过程构建了一种金字塔结构,并且通过调整纳米金颗粒的大小构建了一种3维的手性结构。近年来关于DNA与纳米颗粒的研究也取得了很大的进展[3–9]。Dong等人[10]提出了用于求解图的连通度问题的DNA-纳米颗粒共聚体的自组装计算模型,列举了求解7个顶点的图的连通度问题的具体设计方法。Zhang等人[11]基于DNA-纳米颗粒共聚体的自组装,构建了不同种类的逻辑门,并且用实验实现了这些逻辑门的计算和检测。殷志祥等人[12]在DNA折纸基底上建立了一个与非门计算模型,通过DNA折纸基底上是否还保留纳米金颗粒来显示计算结果的真假。
本文提出一种DNA计算模型,利用DNA-纳米金颗粒共聚体的自组装来解决图论中的一个NP完全问题——最大匹配问题。根据模型我们设计了能够基于一个具体的图进行自组装的特殊的DNA-纳米金颗粒共聚体,然后利用一系列的实验方法来获得最终的解。这种生物化学算法可以极大地降低求解最大匹配问题的复杂度,这将为DNA自组装计算模型提供一种切实可行的方法。
2 DNA-纳米颗粒共聚体
纳米颗粒具有特殊的识别性质,生物分子具有信息存储能力,生物分子还能指导纳米颗粒形成空间立体结构,因此纳米颗粒与生物分子的共聚体在生物诊断和纳米技术领域具有很好的应用前景。构建纳米颗粒和生物分子的共聚体有很多种可行的方法,如使纳米颗粒与生物素、抗生物素蛋白、抗体、抗原、多肽、蛋白质等结合。在这些不同的分子中,DNA由于它内在的可编程能力吸引了越来越多的注意力。
当DNA分子的碱基对大约是12个时,它们在室温下是热力学稳定的。12个碱基对的长度大约是4 nm,基本上接近一个纳米颗粒的大小,并且这些碱基对已经足够用来指导纳米颗粒完成预先设计好的自组装过程。这种卓越的性能已经被用来形成纳米晶体的3维聚集[13],将纳米晶体连接在表面[14],构建纳米晶体的小集合[15–17]等。在这些类似的研究中,DNA-纳米颗粒共聚体的共同特点是如图1所示的一个纳米颗粒上连接了很多条DNA单链。

图1 DNA-纳米颗粒共聚体
对于诊断学来说,精确地控制每一个纳米颗粒上连接的寡核苷酸链的数目是至关重要的,因为有时仅仅检测是否存在某些序列是不够的,而定量地分析杂交的状况通常是十分必要的。Zanchet等人[18]展示了一种通过电泳来分离连接了图2所示的不同数目的寡核苷酸单链的纳米颗粒的方法。该方法通过电泳来分离不同的DNA-纳米金颗粒共聚体。纳米金颗粒在高离子强度的缓冲溶液中是非常稳定的,而这种缓冲溶液对于DNA的杂交反应来说是一种必要的条件。除此之外,在DNA链的末端修饰一个巯基,通过巯基将DNA连接到纳米金颗粒上也是简便易行的。通过调整DNA与金颗粒的比例,可以控制每个金颗粒上连接的DNA链的数目。

图2 连接了不同数目DNA单链的DNA-纳米金颗粒共聚体
凝胶电泳在生物学中是一项非常有用的技术,例如在分离不同大小的DNA分子时。在凝胶电泳中,带电的颗粒在电场下的凝胶中移动,颗粒的迁移率取决于颗粒的电荷和大小。图3展示了由于纳米颗粒上连接了不同数目的DNA链,纳米颗粒的迁移率也发生了很大的变化。

图3 琼脂糖凝胶电泳中连接了不同数目的DNA链的纳米颗粒的位置
尽管文献[18]的方法能够控制每一个纳米金颗粒上连接的DNA链的数目,但是每一个纳米金颗粒上连接的DNA链的种类都是相同的。2013年,Zhang等人[19]展示了一种新的方法,该方法能够选择性地控制每一个纳米金颗粒上连接的DNA链的数目和种类,即具有不同序列的DNA链能同时连接到一个纳米金颗粒上。
3 最大匹配问题
无向图G=(V,E)中具有顶点集V={v1,v2,···,vn}和边集E={e1,e2,···,em},其中m为边数,n为顶点数。图G的子图Gsub=(Vsub,Esub)满足Vsub∈V,Esub∈E。若图Gsub中任意两条边ek,ed在G中没有公共的顶点,则Esub为G的一个匹配。具有最多边的匹配称为图G的最大匹配。求解图的最大匹配是一个NP完全问题。本文以图4中的简单无向图G为例来说明基于DNA-纳米颗粒共聚体求解最大匹配问题的算法。
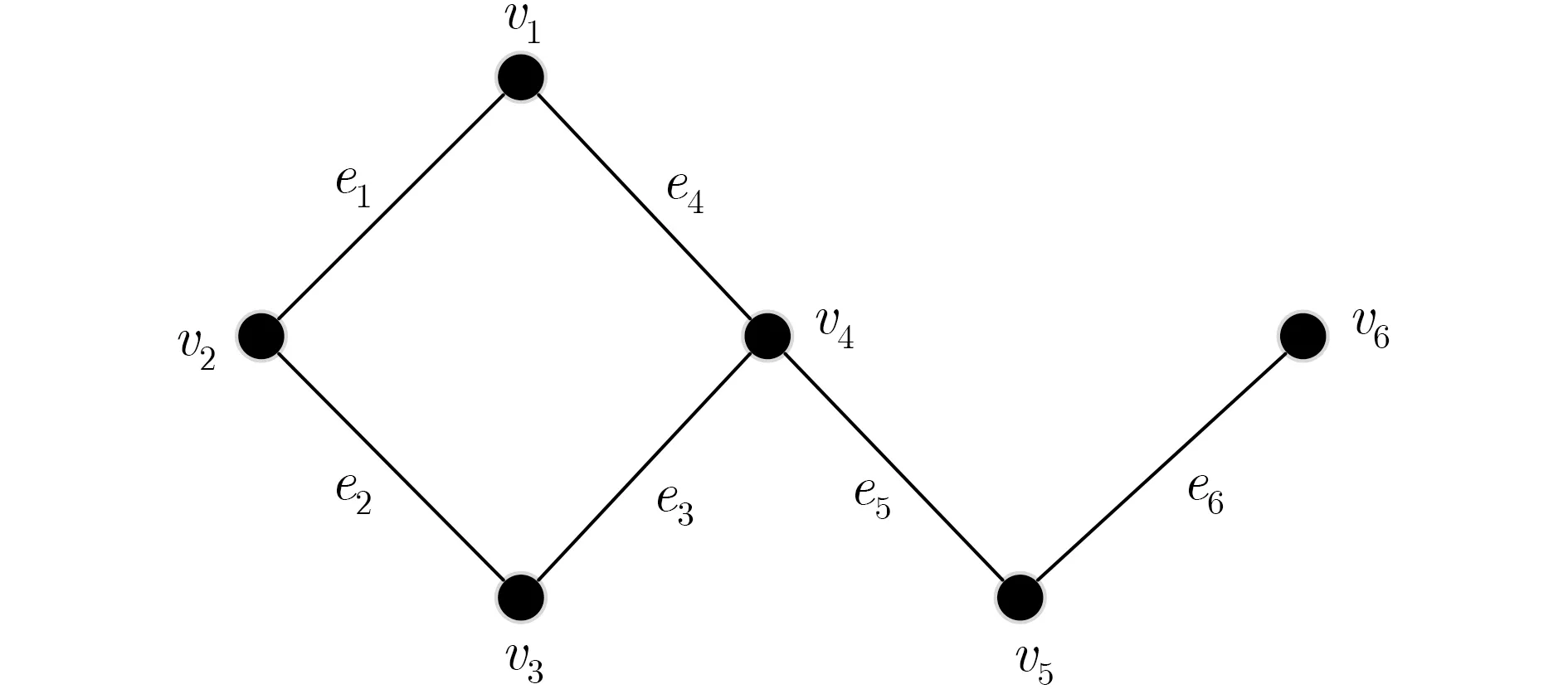
图4 简单无向图G
4 基于DNA-纳米颗粒共聚体求解最大匹配问题的算法
用DNA-纳米颗粒共聚体代表图G的每一个顶点,用DNA单链代表图G的每一条边。通过DNA-纳米颗粒共聚体与DNA单链分子的自组装形成代表图G信息的图结构。然后利用链置换反应来删除边。删除边后的子图Gsub是否为图G的最大匹配,本文通过琼脂糖凝胶电泳实验来检测。计算的过程包括以下4步:(1)编码DNA链和构建自组装过程中所需的DNA-纳米颗粒共聚体;(2)通过退火形成代表图G信息的自组装结构;(3)通过链置换反应删除边;(4)检测正确的解。
4.1 编码
图5展示了代表顶点的DNA-纳米颗粒共聚体结构和代表边的DNA链以及它们的杂交方式。每一个顶点都由一个DNA-纳米颗粒共聚体结构来表示。DNA-纳米颗粒共聚体结构根据图G中具体的顶点进行设计。如图5所示,以顶点v1和顶点v4为例来展示我们所设计的DNA-纳米颗粒共聚体结构。顶点v1的度数是2,因此图5中连接了两条DNA单链的DNA-纳米颗粒共聚体结构表示顶点v1。顶点v4的度数是3,因此图5中连接了3条DNA单链的DNA-纳米颗粒共聚体结构表示顶点v4。顶点v1和顶点v4之间以边e4相连,因此用图5中所示的一条DNA单链表示边e4。如图5所示,代表边e4的DNA单链能分别与代表顶点v1和顶点v4的DNA-纳米颗粒共聚体结构的相应序列杂交。此外,在设计代表边的DNA单链时,还要在单链中间设计一段序列(图5中代表e4的DNA单链上的粉色线段)用于之后需要进行的链置换反应。实验中所需的DNA链从提供引物合成的生物公司购买,构建自组装过程中所需的DNA-纳米颗粒共聚体采用我们在文献[19]中使用的实验方法。
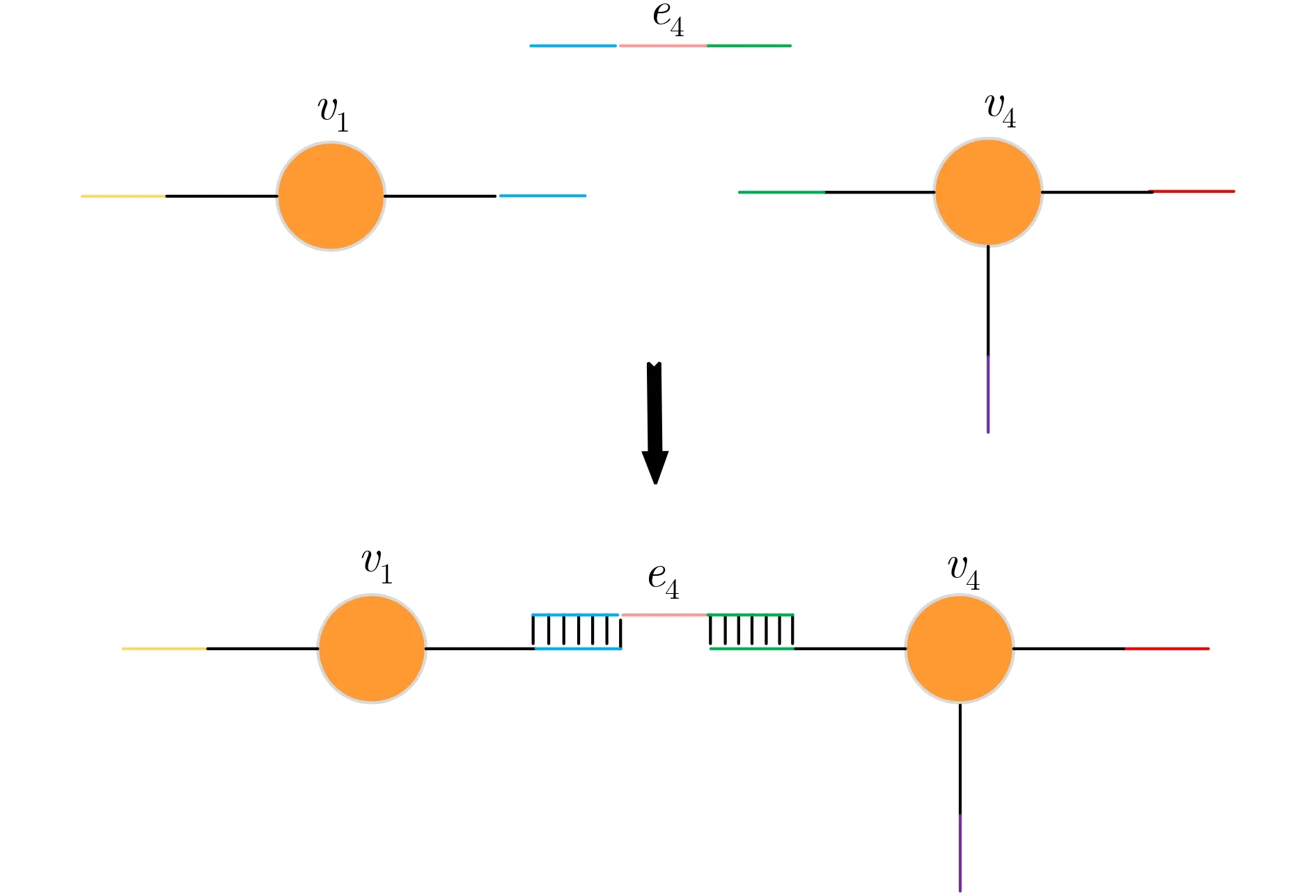
图5 代表顶点的DNA-纳米颗粒共聚体和代表边的DNA链及它们的杂交方式
4.2 退火
将代表顶点的所有DNA-纳米颗粒共聚体和代表边的所有DNA链在一定的缓冲溶液中混合,在适当的温度条件下DNA序列就可以特异地杂交,这个依据碱基互补配对的规则进行特异性杂交过程就是退火反应的过程,如图6所示,退火反应之后代表图G信息的自组装结构就形成了。

图6 退火后形成的代表图G信息的自组装结构
4.3 链置换反应删除边
求解最大匹配问题的一般算法是依次删除边,并测试删除边后的子图是否为最大匹配。以图G为例,删除边的组合数为26-8,因此需要根据这些组合分别删除边。图7展示了所用的删除边方法,即链置换反应。在上一步退火反应形成的自组装结构中,为了删除DTR所代表的一条边,需要加入其取代链dtr。dtr与DTR完全互补,因此能形成图7所示的双螺旋结构,并使原来连接在一起的两个顶点分开。链置换反应的实验条件与退火反应的实验条件一样,均是在合适的温度条件和缓冲液浓度条件下进行的。

图7 删除边的链置换反应
图8展示了删除边e1、边e3和边e6后的结构。用链置换的方法加入边e1、边e3和边e6的取代链后,原来退火反应形成的自组装结构就变为图8所示的结构。虚线椭圆圈住的结构为仍然连接在一起的顶点和边的结构。小椭圆圈住的为顶点v2,v3及边e2连接在一起的结构,即绿色实线框中所示的两个纳米金颗粒连接在一起的二聚体结构。大椭圆圈住的为顶点v1,v4,v5及边e4,e5连接在一起的结构,即蓝色实线框中所示的3个纳米金颗粒连接在一起的三聚体结构。此外,链置换反应删除顶点后还形成了代表顶点v6的结构,即红色实线框中的单个纳米金颗粒与DNA连接的共聚体。二聚体结构可以看作一条边。三聚体结构可以看作具有一个公共顶点的两条边。删除边e1、边e3和边e6后出现了三聚体结构,说明子图中存在具有公共顶点的两条边,也就是说删除边e1、边e3和边e6后的子图不是一个匹配。

图8 删除边e1、边e3和边e6后的结构
图9展示了删除边e2、边e4和边e5后的结构。用链置换的方法加入边e2、边e4和边e5的取代链后,原来退火反应形成的自组装结构就变为图9所示的结构。虚线椭圆圈住的结构为仍然连接在一起的顶点和边的结构。从图9可以看出,删除边e2、边e4和边e5后,原来退火反应形成的自组装结构变为全部是两个纳米金颗粒连接在一起的结构,即红色框中所示的两个纳米金颗粒连接在一起的二聚体结构。二聚体结构可以看作一条边,并且没有三聚体结构出现,说明删除边e2、边e4和边e5后子图中的边都没有公共顶点,即说明子图是一个匹配。
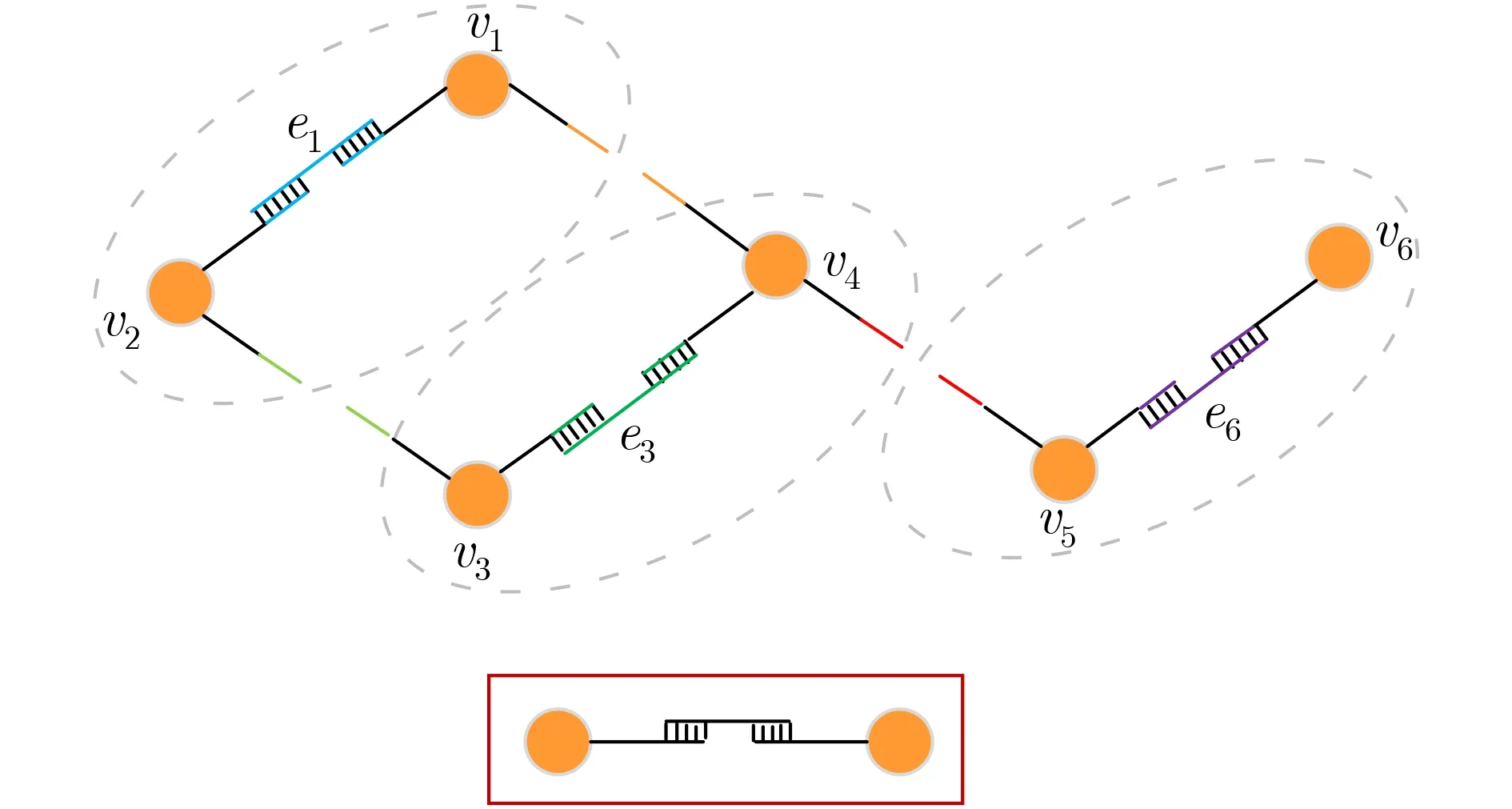
图9 删除边e2、边e4和边e5后的结构
4.4 琼脂糖凝胶电泳检测结果
经过上一步链置换反应删除边后就形成了各种子图,测试子图是否为图的匹配通过琼脂糖凝胶电泳实验来进行。具体的方法是预先合成图10所示的3种对照。蓝色实线框中的是3个纳米金颗粒通过两条DNA单链杂交而成的三聚体结构。绿色实线框中的是两个纳米金颗粒通过一条DNA单链杂交而成二聚体结构。红色实线框中的是一个纳米金颗粒与DNA的共聚体结构。在琼脂糖凝胶电泳中,如图10左侧粉色所示,由于分子量的不同,三聚体结构的电泳带位于最下方,二聚体结构电泳带位于中间,最上方的电泳带是单颗粒的DNA-纳米金颗粒共聚体。
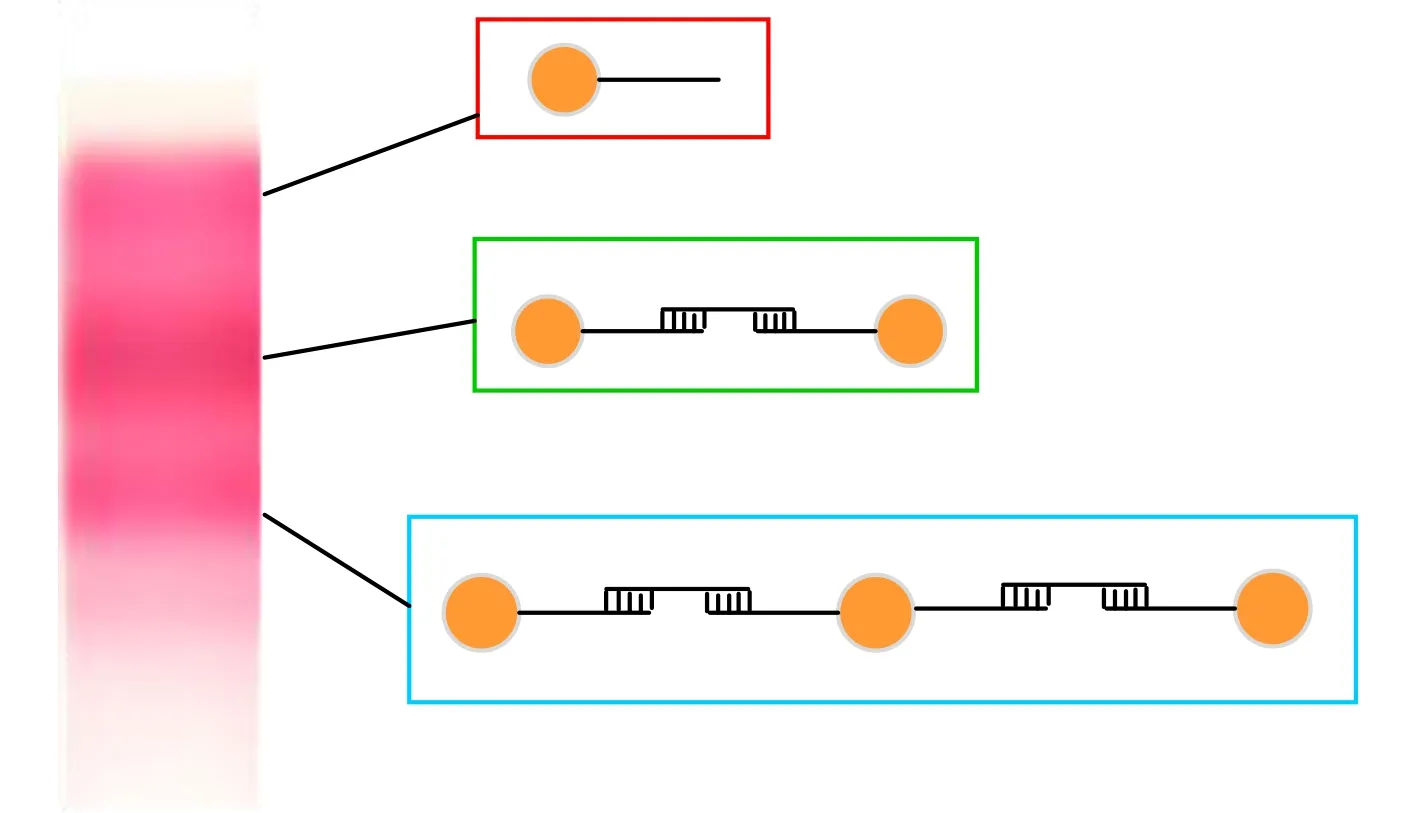
图10 琼脂糖凝胶电泳的对照
为了测试子图是否为图的匹配,就需要分别将子图进行琼脂糖凝胶电泳实验,与对照相比,如果一个子图的琼脂糖凝胶电泳结果出现了三聚体结构的电泳带就说明该子图不是图的匹配,因为三聚体结构表示具有一个公共顶点的两条边的结构。值得注意的是,如果一个子图的电泳结果出现的电泳带比三聚体的电泳带更靠下,说明该子图也不是图的匹配,因为更靠下的带可能是四聚体或更多聚体的情况,都代表子图的边具有公共顶点的情况。
如果一个子图的琼脂糖凝胶电泳结果出现了二聚体结构的电泳带和最上方的单颗粒DNA-纳米金颗粒共聚体的电泳带,并且没有出现三聚体结构或更高聚体结构的电泳带,就说明该子图是图的匹配,因为没有出现三聚体结构或更高聚体结构的电泳带就说明子图中的边都没有公共顶点。检查具有最多边数的匹配即为图的最大匹配。
5 结束语
本文用DNA-纳米颗粒共聚体代表图G的每一个顶点,用DNA单链代表图G的每一条边。通过DNA-纳米颗粒共聚体与DNA单链分子的自组装形成代表图G信息的自组装结构。然后利用链置换反应来删除边,并通过琼脂糖凝胶电泳实验来检测删除边后的子图是否为图的最大匹配。本文方法利用了DNA纳米技术的方法和技术,具有高度的并行性和可靠的结果检测能力,极大地降低了求解图的最大匹配问题的复杂度。
