低氧mtDNA3010A/G基因型变异诱导的lncRNA-mRNA共表达网络变化*
2021-10-22罗勇军
陈 郁, 龚 亮, 罗勇军
(陆军军医大学陆军卫勤训练基地军事医学地理学教研室, 重庆 400038)
当人体进入海拔2 500 m及其以上区域时,由于环境变化尤其是氧分压的下降,会对机体造成明显的生理学效应,因而称之为医学高原[1]。在移居高原的部分人群中,由于个体对高原环境刺激反应强烈,将发生包括急性高原反应、高原肺水肿(high altitude pulmonary edema, HAPE)、高原脑水肿(high altitude cerebral edema, HACE)等急性高原病[2]。目前,全世界有1.4亿人生活在海拔3 000 m以上地区[3],而2 500 m以上地区生活的人群更多。近年来,到高原的平原人群显著增多,如何维护其生命健康,保证其在高原正常生活是高原医学研究的热点和前沿。伴随着高通量技术尤其是大规模测序和芯片的应用,对人体在面临高原环境时的基因表达调控和作用网络的研究也越来越深入。其中,长链非编码RNA(long non-coding RNA, lncRNA)及其调控网络在低氧反应中的作用得到了越来越多的关注。
lncRNA是一类长度大于200个核苷酸的非编码RNA。研究发现,部分lncRNA具有编码肽类的能力[4]。随着研究的不断深入,发现lncRNA在转录、转录后修饰,以及RNA翻译等多个途径参与基因的表达调控,进而影响细胞的代谢、增殖和凋亡等[5]。按照来源和功能定位的不同,lncRNA也分为核基因来源和线粒体来源,两者也在发挥着不同的生物学效应[6]。研究发现,mtDNA3010位点的A基因型是汉族人群HAPE和急性高原反应的保护因素,而G基因型是危险因素[7, 8]。同时也发现,该位点的A基因型是高原移居汉族高原红细胞增多症的危险因素,而G基因型是保护因素[9]。mtDNA3010位点位于编码16s rRNA,难以从常规分子生物学途径解释其在缺氧过程中因变异引起的机制变化。而该位点A/G基因型在急性高原病和慢性高原病上相互矛盾的结果提示,mtDNA变异在缺氧反应过程中的作用更为复杂,需要从多维度探讨其可能的作用。
为进一步探讨mtDNA3010位点A/G基因型变异在缺氧反应中的作用,本研究在构建mtDNA3010位点A/G两种基因型融合细胞的基础上,采用表达谱芯片和qRT-PCR验证急性缺氧处理后lncRNA和mRNA表达谱的变化。通过生物信息学方法构建共表达网络,预测其潜在功能,为分析mtDNA变异在低氧反应中的作用提供理论依据。
1 材料与方法
1.1 细胞来源与处理
ρ0206细胞是由骨肉瘤细胞143BTK-,经溴化乙锭(EB)处理而形成一种特殊的无线粒体细胞,由浙江大学管敏鑫教授惠赠,作为供体用于构建mtDNA3010A和mtDNA3010G两种基因型融合细胞。
1.2 主要试剂
实验所需主要试剂如下:细胞总RNA提取试剂Trizol购于美国Invitrogen公司,RNA逆转录试剂盒、SYBR Green购于Takara公司;DMEM高糖培养基购于美国康宁公司,胎牛血清购于德国PAN公司;胰酶购于武汉博士德生物技术有限公司。
1.3 融合细胞缺氧处理
以ρ0206细胞为基础,本课题组前期成功构建了mtDNA3010A和mtDNA3010G两种基因型融合细胞,具体见参考文献[10]。细胞在37℃,5%CO2和常氧条件下培养,取对数期生长的两种融合细胞,计数后取20×104的细胞接种于培养皿中,经1%O2缺氧培养24 h后,建立细胞急性缺氧模型。缺氧结束后,弃培养上清,PBS清洗后加入Trizol,反复轻柔吹打以获取所有细胞。每种融合细胞各送样3份。
1.4 lncRNA表达谱和mRNA表达谱的筛选
采用北京博奥晶典生物技术有限公司提供的Human LncRNA Array v4(4x180K)芯片,包括了40916个lncRNA探针和34235个mRNA探针,涵盖了人体现有的绝大部分lncRNA和mRNA。总RNA的提取,标本质控以及后续芯片的处理、图像采集、数据读取在北京博奥晶典生物技术有限公司完成。
1.5 差异mRNA验证和功能预测
对两种融合细胞经缺氧处理后,采取Trizol法提取总RNA。根据mRNA芯片结果,以β-actin为内参,使用qRT-PCR法验证差异表达的mRNA,每组实验重复3次。采用Gene Ontology数据库和京都基因与基因组大百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)数据库对验证的差异基因进行功能注释,并进行一级、二级信号通路和功能预测。
1.6 lncRNA-mRNA共表达网络构建和靶基因预测
根据上述GO和KEGG的预测结果,包括其可能涉及到的基因、蛋白之间的相互作用关系,采用Cytoscape软件绘制lncRNA-mRNA共表达网络图,分析lncRNA和mRNA之间的信号转导网络。根据差异lncRNA预测其可能作用的mRNA,并通过GO和KEGG数据库预测靶基因的相关功能变化。
1.7 统计学处理
芯片数据分析由北京博奥晶典生物技术有限公司完成,其他数据分析采用SPSS20.0完成。芯片杂交扫描后的tiff格式图片数据采用Feature Extraction提数软件进行预处理分析,然后采用GeneSpring GX软件计算基因表达差异和统计学差异显著性P值。数据处理过程主要包括:芯片扫描tiff图用Feature Extraction软件处理得到原始数据文件 (.txt)。将原始数据文件(.txt)导入GeneSpring软件,写入分组等参数信息。进行每个样品的数据归一化和QC分析。采用Cluster3.0软件进行Cluster分析和图形化展示。根据分组信息,进行差异比较,得到差异基因。所得数据结果以mtDNA3010G基因型融合细胞为对照,以|FC|>1.2、P<0.05、FRD<0.05为标准,筛选差异表达的lncRNA和mRNA。在验证芯片结果时,按照2-△△Ct法计算分析mtDNA3010A和mtDNA3010G基因型融合细胞缺氧后基因表达差异。
2 结果
2.1 两种融合细胞缺氧后的lncRNA和mRNA表达谱差异
经1%O2缺氧培养24 h后,根据芯片检测结果,对不同样本中差异表达的lncRNA和mRNA进行聚类分析,系统热图分析结果见图1。其中,每一个纵列代表1个样本,AR和GR分别代表mtDNA3010A/G基因型融合细胞。红色代表表达上调,绿色代表表达下调。和mtDNA3010G基因型融合细胞相比:mtDNA3010A基因型融合细胞表达上调的lncRNA共有668个,上调倍数最大的为ENST00000553679.1倍数=2.89),表达下调的lncRNA共有1098个,下调倍数最大的为HIT000092395_03(倍数=2.80)(图2);表达上调的mRNA共有1151个,上调倍数最大的为成纤维细胞生长因子1(fibroblast growth factor 1, FGF1) (倍数=3.60),表达下调的mRNA共有539个,下调倍数最大的为G蛋白耦联受体151(G protein-coupled receptor 151, GPR151 )(倍数=2.07)(图2,表1,表2)。上述结果表明,即便线粒体仅有一个位点变异,在急性缺氧时也可能诱导显著的基因变化并被基因芯片检测到。
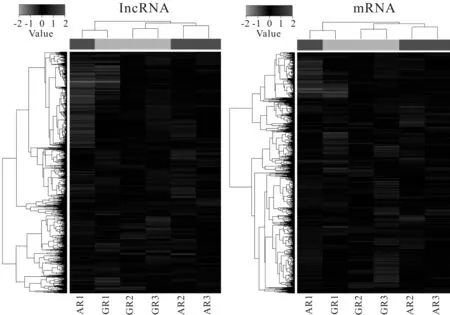
Fig. 1 Heat map of differential expressions of lncRNA and mRNA in mtDNA3010A/G fusion cells after acute hypoxia treatment
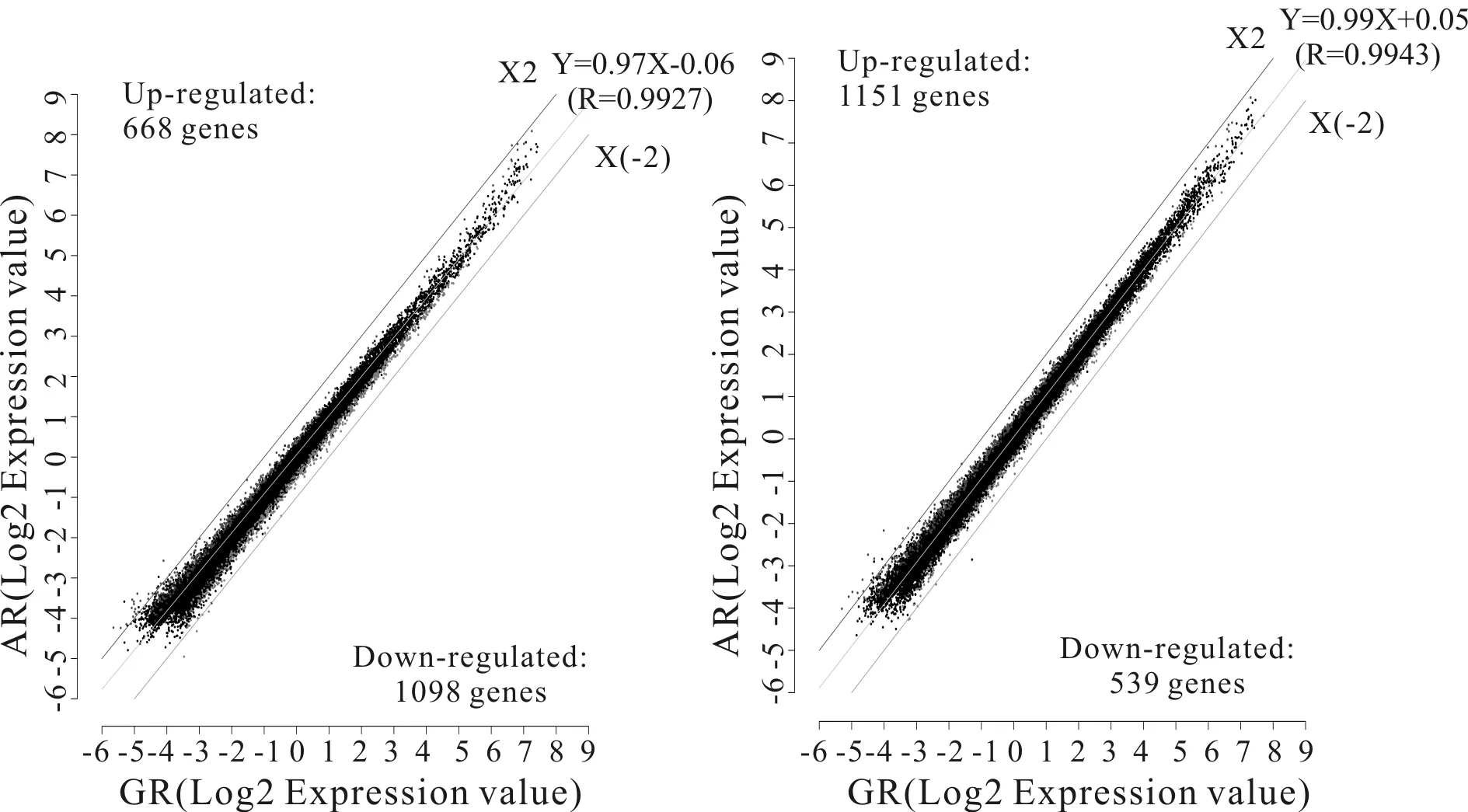
Fig. 2 Scatter plots of differential expressions of lncRNA and mRNA in mtDNA3010A/G fusion cells after acute hypoxia treatment
横坐标、纵坐标分别为mtDNA3010G基因型融合细胞和mtDNA3010A基因型融合细胞表达的lncRNA(图2左)、mRNA(图2右)经log2转化后的值。红色直线为基因上调表达阈值线,绿色直线为基因下调表达阈值线,中间灰色直线为总体表达量的拟合线,方程为该拟合线的方程,R为mtDNA3010A基因型和mtDNA3010G基因型的两组样本的相关系数。

Tab. 1 Top 10 lncRNAs showed significant differences in expression changes

Tab. 2 Top 10 lncRNAs showed significant differences in expression changes
2.2 差异mRNA验证和功能预测
根据mRNA芯片结果,筛选了部分基因进行验证,结果见表3。以FGF1为例,芯片结果为正 3.60,提示经急性缺氧处理后,与mtDNA3010A融合细胞相比,mtDNA3010G融合细胞的该基因表达升高3.60倍。然而经qRT-PCR验证后,发现该差异表达基因为假阳性。上述结果提示,表达谱芯片检测出的差异表达基因有许多假阳性的结果,必须通过qRT-PCR对结果验证。唯有验证成功的差异表达基因方能完成后续的生物信息学分析。
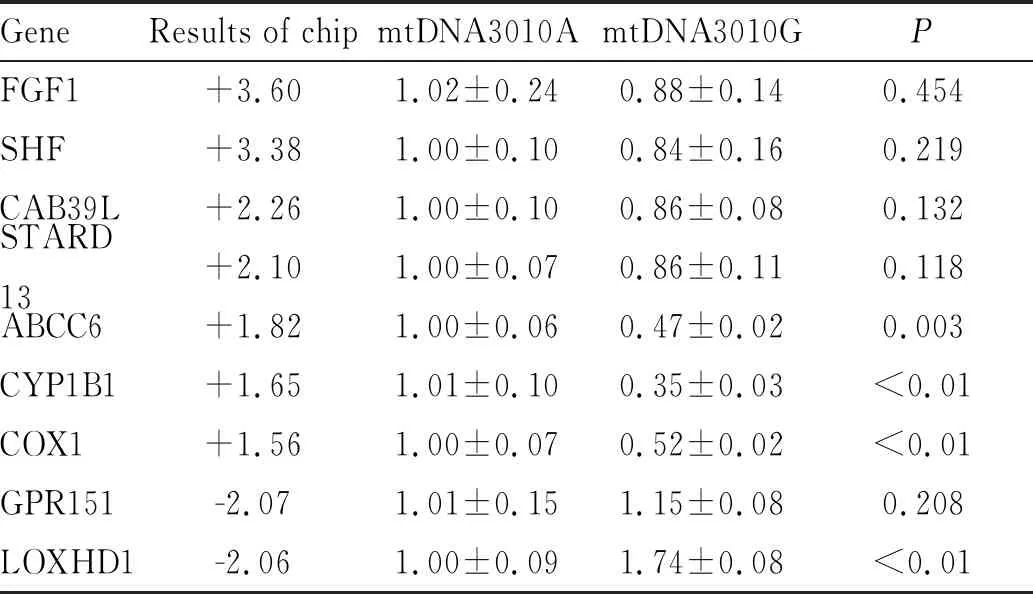
Tab. 3 Results of chips and verified by qRT-PCR
通过GO聚类分析,和mtDNA3010G基因型融合细胞相比,mtDNA3010A基因型融合细胞差异表达基因涉及的生物学过程包括纤毛合成、上皮纤毛运动、细胞微管运动、纤毛组装、糖脂类生物合成以及心血管系统发生;涉及的细胞组分包括DNA复制复合物、核小体、微管等细胞骨架、细胞器、蛋白-DNA复合体等;差异表达基因涉及的分子功能包括芳香胺乙酰转移酶活性,蛋白二聚体活性,磷脂酶A2活化因子活性,醌类辅助因子甲基转移酶活性,脂蛋白转运体活性以及C-甲基转移酶活性等。
KEGG通路分析提示,差异表达的基因涉及的信号通路包括到酒精中毒,系统性红斑狼疮,咖啡因代谢,化学物致癌,肿瘤转录失调节以及p53信号通路等(图3)。

Fig. 3 KEGG-PATHWAY enrichment analysis of differentially expressed genesin mtDNA3010A/G fusion cells after acute hypoxia treatment
2.3 lncRNA-mRNA共表达网络的构建
按照相关性系数大于0.90,并且P<0.05的标准,筛选出显著表达的lncRNA-mRNA前1000个共表达基因对,采用Cytoscape软件生成并分析新构建的lncRNA-mRNA共表达网络。正相关表明随着该lncRNA的表达增加,mRNA表达水平亦显著上升,负相关即显著下降,相关系数越大表明变化的幅度越显著。本研究中发现,在急性缺氧处理后,与mtDNA3010G基因型融合细胞相比,mtDNA3010A基因型融合细胞差异表达lncRNA关联程度最高的基因包括,锚蛋白3(Ankyrin 3, ANK3),B细胞淋巴瘤样因子13(BCL2 Like 13, BCL2L13)和发动蛋白结合蛋白(dynamin binding protein, DNMBP)等,其中最为显著的是ANK3基因,预测有1695个lncRNA可能与其有相互作用,提示上述基因也可能参与了mtDNA3010A基因型对急性高原反应和HAPE的保护作用。
2.4 lncRNA的靶基因预测
在构建lncRNA-mRNA共表达网络图的基础上,根据lncRNA在染色体上所属区域,预测其在10kb范围内可能有作用的mRNA(即顺式预测),同时利用blast工具比对lncRNA序列和mRNA的3`-UTR区域(即反式预测),并综合不同lncRNA预测的同一靶基因数目,共筛选了10个与差异lncRNA存在高度相互作用的靶基因,具体见表4。生物信息学分析提示mtDNA3010位点基因型变异后,缺氧能够诱导细胞出现一系列的动态调控网络变化,有可能参与了HAPE的发生过程。
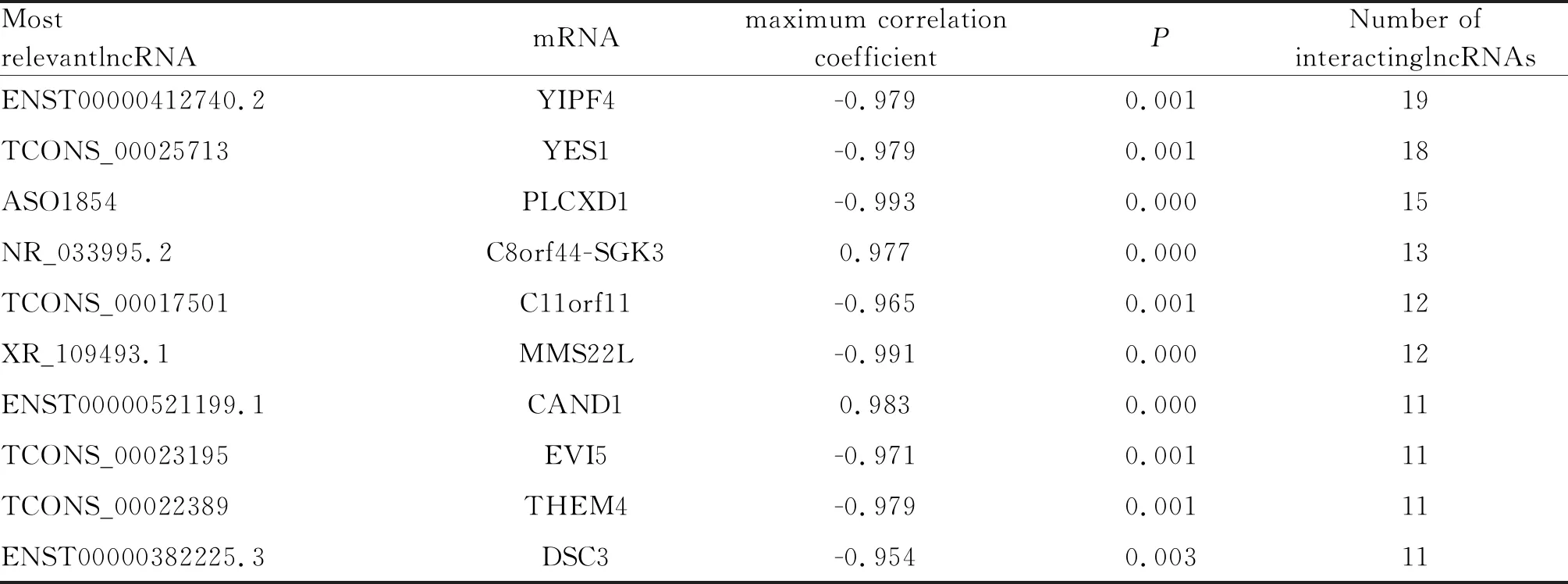
Tab. 4 Predicted target genes by differently expressed lncRNAs
2.5 预测靶基因的GO和KEGG分析
参照2.2的方法,对lncRNA预测的10个靶基因进行GO和KEGG分析。结果提示,mtDNA3010G基因型在缺氧后可能会影响细胞在能量代谢和炎症反应相关基因的表达,能量代谢异常和炎症反应过度可能是HAPE发生中的重要环节。GO结果表明,和mtDNA3010G基因型融合细胞相比,mtDNA3010A基因型融合细胞经急性缺氧处理后,差异lncRNA的靶基因涉及的生物学过程包括糖原分解代谢、细胞对白介素6(interleukin 6, IL-6)的反应、内皮细胞活化、碳水化合物的合成代谢等。在涉及到的细胞成分方面,主要包括糖原颗粒、蛋白磷酸酶1型复合体、Bcl3-Bcl10复合物、Bcl3-NFκB复合物和IL-6受体复合物等。在分子功能方面,主要为与IL-11结合、NADP转氢酶活性、黄嘌呤氧化酶活性、IL-11受体活性、氧化还原酶活性等密切相关。KEGG结果提示,靶基因的功能富集于烟酰胺代谢、酒精代谢、咖啡因代谢、细胞连接、肾素-血管紧张素系统、胰岛素抵抗和过氧化物酶活性等。
3 讨论
人体在经受高原环境刺激时,机体会出现多方面代偿变化。良好的代偿有助于机体习服高原环境,保持健康状态,而失代偿则会导致各种高原病的发生。大量的证据提示,在不同的人群中HAPE的发生与线粒体变异有关,表明线粒体变异及其诱导的功能变化是HAPE重要的发病环节[11]。本研究表明,mtDNA3010位点由A基因型突变为G基因型后,在缺氧作用下,会导致lncRNA-mRNA调控网络的显著变化,提示lncRNA和mRNA之间存在着复杂而又紧密的相互作用。本研究还提示,线粒体与核基因之间有联系,不仅仅是体现在mRNA表达差异,lncRNA同样介导了线粒体与细胞核之间的相互作用,进而影响细胞的代谢过程和机体功能的正常发挥。上述结果也表明,mtDNA3010位点由G基因型突变为A基因型时,在缺氧条件下会引起细胞基因表达改变,然而每个差异表达基因的功能并不能完全解释细胞在缺氧刺激时的功能变化,需要相互作用形成级联放大效应,最终导致疾病的发生。
lncRNA参与了动脉粥样硬化、肺动脉高压,尤其是缺氧性肺动脉高压的发生,而缺氧性肺动脉高压是HAPE发生的中心环节。研究发现,lncRNA LINC-PINT和LINC00599的多态性与汉族HAPE的发生风险有关[12]。LINC-PINTrs157928位点的CT基因型是中国汉族人群HAPE的保护因素,LINC00599rs2272026位点的CC基因型是32岁以下人群HAPE的保护因素,而在32岁以上则为危险因素。目前,lncRNA在HAPE发生中的作用研究报道较少,尚未涉及具体的分子机制。本次研究中虽然也发现了大量差异表达的lncRNA,其中ENST00000553679.1和ENST00000453008.2差异最为明显,但是有关这两种lncRNA的具体功能的研究尚鲜有报告,其机制还需要进一步探讨。
在本研究中,采用qRT-PCR验证芯片结果后发现,A基因型的ATP结合盒亚家族C成员6(ATP binding cassette subfamily C member 6, ABCC6)、细胞色素P450家族1亚家族B成员1(cytochrome P450 family 1 subfamily B member 1, CYP1B1)和细胞色素C氧化酶Ⅰ(cytochrome C oxidase I, COX1)表达显著上调,而脂氧合酶同源结构域1(lipoxygenase homology domains 1, LOXHD1)表达显著下调。ABCC6编码的是一类跨膜蛋白,能够转运细胞内外多种物质,并同时具备ATP酶活性,其突变与皮肤弹性假黄瘤有关。CYP1B1编码重要的代谢酶类,参与了包括脂肪酸,类固醇激素和维生素的代谢,是重要的炎症调控基因[13]。COX1是线粒体氧化呼吸链复合体Ⅳ的重要组成部分,在形成电化学梯度驱动中ATP合成发挥着关键作用。LOXHD1编码一种高度保守的蛋白,主要表达在内耳处的毛细胞,其突变与听力缺失有关。目前有关上述基因在缺氧反应及高原病发生中作用的研究报道。结合相关基因的功能预测,表达上调的ABCC6、CYP1B1和COX1可能会改善细胞的能量代谢,抑制炎症反应,促进能量生成,有助于机体应对高原环境的习服。表达下调的LOXHD1作用尚不清楚,根据其诱发听力障碍的同类型基因潜在功能提示,可能会影响钙泵的功能。钙是收缩血管的重要因素,而钙拮抗剂是降低血压的主要药物,而降低肺动脉压是治疗HAPE的重要方法。上述结果提示,本研究中发现的差异表达基因可能通过多种途径参与了HAPE的发生过程。
在多个不同人群的HAPE遗传易感性研究中,均发现HAPE的发生与mtDNA变异有关,进一步分析提示,这些变异在缺氧条件下诱发的能量代谢异常有可能是导致HAPE发生的重要机制。氧化磷酸化是细胞的主要供能方式,在缺氧条件下这一方式受到明显的抑制,难以提供足够的能量供细胞代谢,导致细胞的某些代谢过程发生异常,在缺氧刺激下不能发挥有效的应对,导致细胞功能失常,进而通过一系列级联反应导致了HAPE的发生,而mtDNA的变异可能加剧这一病理反应。此外,据报道,HAPE患者血清中的IL-6和肿瘤坏死因子(tumor necrosis factor-α, TNF-α)等炎症因子含量明显升高[14],提示炎症反应与HAPE的发生密切相关[15]。在本实验中,GO分析和KEGG富集均提示,差异lncRNA预测的靶基因,以及差异表达基因参与了炎症反应和细胞能量代谢,表明mtDNA3010位点由G基因型突变为A基因型时,也可导致与能量代谢和炎症反应相关因子的变化,为阐述HAPE的发生提供了新的理论依据。
综上所述,在急性缺氧条件下,mtDNA3010位点由A基因型突变为G基因型后,会导致相关lncRNA和mRNA的表达差异,并在生物学过程、细胞成分和分子功能,以及代谢途径等方面影响了细胞的炎症反应、能量代谢等核心进程,可能在诱导急性高原反应和HAPE的发生、发展中发挥了重要作用。上述差异lncRNA和mRNA有望成为HAPE的易感标志物,并为探索线粒体介导的细胞调控网络变化在高原病发生中的机制提供了重要的研究方向,也为从线粒体角度分析能量代谢和炎症反应在缺氧反应中的机制提供有益资料。
