32份茶树地方群体种资源的遗传多样性和群体结构分析
2021-10-16李长乐葛悦闫美琳李慧林青青王璞赵华王明乐王郁郭飞倪德江
李长乐,葛悦,闫美琳,李慧,林青青,王璞,赵华,王明乐,王郁,郭飞,倪德江
32份茶树地方群体种资源的遗传多样性和群体结构分析
李长乐,葛悦,闫美琳,李慧,林青青,王璞,赵华,王明乐,王郁,郭飞*,倪德江
华中农业大学园艺林学学院/园艺植物生物学教育部重点实验室,湖北 武汉 430070
选取均匀分布于茶树15个连锁群上的30对SSR引物,对来自12个省份的32份茶树群体种资源进行遗传多样性和群体结构分析,以期为茶树杂交育种亲本选择和演化路线推断提供参考。研究共获得149个等位基因,平均每个SSR标记检测到5.96个等位基因,引物多态性信息含量均值为0.660。32个茶树群体Shannon’s多样性指数范围为0.691~1.089,平均数为0.954;观测杂合度的范围为0.253~0.633,平均数为0.510;期望杂合度范围为0.430~0.653,平均数为0.590。供试茶树群体遗传分化系数()均值为0.205,遗传分化水平较高。基于Nei’s遗传距离的聚类和群体结构分析结果一致,供试种质可划分为四大类型,具有明显的地域分布规律。
群体品种;SSR分子标记;群体结构;遗传多样性;演化路线
茶树在我国分布广泛,经历了长期的自然选择及人工驯化和栽培过程,形成了丰富多样的种质资源。现有的茶树品种基本分为有性系品种和无性系品种两大类,有性系品种用种子繁殖,又称“群体品种”,群体品种包含的个体类型丰富,是天然的种质资源库。
近年来,随着茶产业的蓬勃发展,发掘和保护优异茶树种质资源的重要性日益凸显,关于茶树种质资源方面的文献越来越多,从表型性状、内含成分、适制茶类到细胞结构、基因序列的研究均有报道[1-4]。分子标记揭示的是基因组层面上DNA序列的差异,相对于形态学标记、生化标记等,受外界环境因素影响小。1980年至今,越来越多的分子标记技术成功用于茶树种质资源的遗传多样性分析、种质鉴定和分类研究[5-7]。在茶树中广泛应用的分子标记技术主要有限制性片段长度多态性标记(RFLP)、扩展片段长度多态性标记(AFLP)、单核苷酸多态性标记(SNP)、简单重复序列标记(SSR)等[8-10]。SSR是第二代分子标记,技术成熟,稳定可靠,获取途径便捷,且具有共显性、单个位点多态性高、重复性好、特异性强、数量丰富等特点,被广泛运用于茶树种质的遗传多样性评估、亲缘关系鉴定、群体结构划分、指纹图谱构建等[11-12]。黄丹娟[13]对我国茶树品种的遗传多样性水平进行分析,从128个SSR标记中筛选出2套核心引物,认为分析254个茶树品种遗传多样性至少需要用30对SSR引物。姜晓辉等[14]利用毛细管电泳荧光标记技术对广东茶树群体种质进行研究、聚类,综合分析了其遗传结构并推测了其可能的基因交流情况。杨军等[15]利用SSR分子标记对福建茶树种质资源展开研究,将供试样品划分类群并鉴定了亲缘关系。姚明哲等[16]对四川和重庆茶树群体种的遗传多样性和群体结构进行分析,发现川、渝茶树群体种在群体结构上有较明显的差异,推测出这两地种质可能具有不同的演化和传播途径。Zhao等[17]利用14个SSR分子标记分析了大理茶中25个居群内587个茶树个体的遗传多样性,发现各居群内部水平上野生居群遗传多样性的减少和遗传漂变的发生高于驯化居群。
国内外学者对茶树群体种资源的各方面做了大量研究,但对于全国范围内多省份茶树种质资源的综合研究相对较少,难以从整体范围去考量、发掘不同地区群体种资源的关联与差异性。茶树地方群体品种是茶树种质资源的重要组成部分,也是优质茶树选育的重要材料。本研究基于SSR分子标记对来自12个省份的32份茶树地方群体品种进行群体结构和遗传多样性研究,以期揭示不同地区茶树种质亲缘关系和地域分布规律,为优异茶树种质资源的发掘和利用奠定基础。
1 材料与方法
1.1 试验材料
供试材料为来自全国12个省份(包含32个地区)的茶树地方群体品种,课题组前期从各个产区实地考察、收集茶籽,统一播种、定植于华中农业大学茶学试验基地。分子标记样品为单株取样,以前期整理的供试地方群体品种典型形态学特征为参照,每个群体种选择6株代表性植株,共计192株单株,分别采集各单株幼嫩组织并储藏于–80℃冰箱备用。供试群体名称、来源、编号等信息见表1。
1.2 试验方法
1.2.1 DNA提取及SSR引物信息
茶树基因组DNA的提取使用CTAB法;提取所得DNA样品的浓度和质量使用超微量分光光度计和2%琼脂糖凝胶电泳检测;试验所用的30对引物均由北京擎科生物科技有限公司合成,引物序列参照文献[13]。
1.2.2 PCR反应体系和反应条件
PCR反应体系(10 μL):10 μmol·L-1的正反向引物各0.5 μL,2×Taq PCR MasterMix(Taq DNA Polymerase、MgCl2、dNTPs)5 μL,300 ng·μL-1的DNA模板1 μL,灭菌超纯水3 μL。PCR扩增程序为:94℃预变性3 min;94℃变性30 s,各引物退火温度退火30 s,72℃延伸30 s,35个循环;72℃延伸10 min,扩增完毕于4℃冰箱保存。
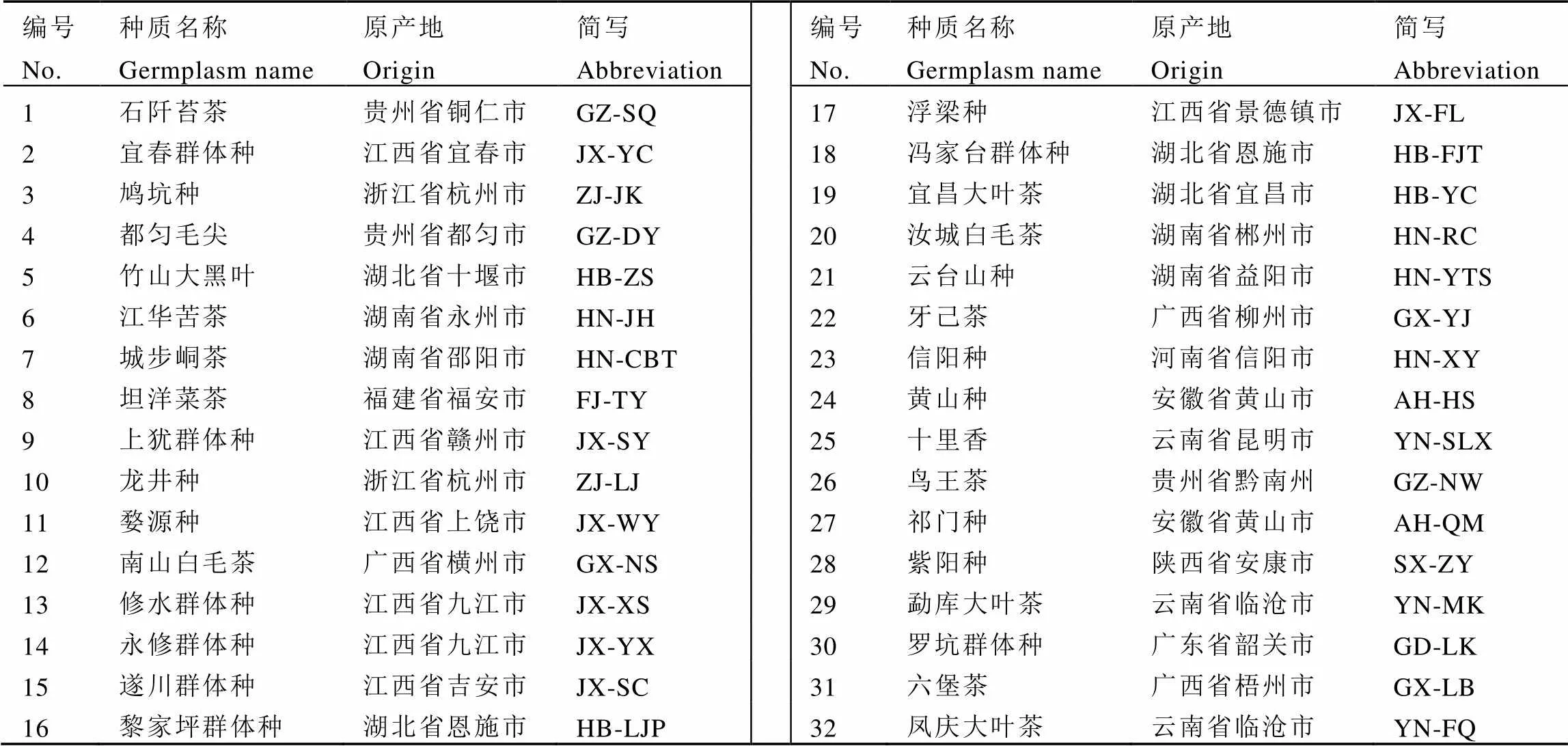
表1 供试材料的名称及原产地
1.2.3 毛细管电泳
毛细管电泳系统为QIAxcel Advanced全自动核酸分析仪,预制胶卡夹为QIAxcel DNA High Resolution Kit(1200),运行方法为OM800,吸取时间选择10 s。运行结束后,保存电泳结果的峰图和胶图进行数据统计分析。
1.3 数据统计与分析
1.3.1 数据统计
采用人工读带方法根据扩增条带分子量大小进行统计,有清晰目标条带记为“1”,无清晰目标条带的记为“0”,生成“0、1”数据矩阵。
1.3.2 数据分析
不同软件分析所需要数据格式的转换通过DataFormate实现;使用Popgen 32软件进行Hardy-Weinberg平衡检验、中性检验、各项遗传参数估算、遗传距离和遗传一致度计算;遗传多样性指数(值)计算公式为12,式中表示第个等位基因位点出现的频率[18];根据Nei’s遗传距离和邻接法(Neighbor-joining method),利用MEGA 6.0软件构建系统进化树。
应用Structure 2.3对供试茶树材料进行群体结构分析,假定群体数目(K值)为1~15,各位点相互独立,将蒙特卡罗算法开始时的不作数迭代次数(Length of burn-in period)设为50 000,将不作数迭代数的MCMC设为50 000,每个K值重复运行10次。以似然值最大原则,根据在线软件Structure Harvester计算结果确定最佳分组K值,进而得到最优群体结构。
2 结果与分析
2.1 引物的多态性信息量
用30对引物对供试的茶树样品进行扩增,其中有25对均匀分布于15条染色体上的SSR引物扩增效果较好,能够扩增出清晰的条带,且扩增产物经过毛细管电泳检测后能产生较好的多态性。引物TM209的部分毛细管电泳图谱如图1所示。
使用Popgene 32软件对供试茶树群体的25对SSR位点进行了Hardy-Weinberg平衡检验和中性测试分析(设置1 000次重复)。Hardy-Weinberg平衡检验可以评估供试群体与遗传平衡群体之间的偏离程度,是评估群体其他计算数据是否可靠的一种手段[19]。对茶树群体毛细管电泳数据进行Hardy-Weinberg平衡的卡方检验,800组数据中仅有136组显著偏Hardy-Weinberg平衡,且散落分布在不同群体的不同位点上,意味群体评估数据相对可靠。中性测试分析结果显示,25对SSR位点中有6对位点的统计量观测值()略偏离95%置信区间,其余19对SSR位点观测值均在95%置信区间范围内,表明这些基因座大多为中性基因,不受自然选择和其他因素的影响,可用于茶树群体遗传多样性分析(表2)。
利用25对SSR高多态性引物扩增结果对来自32份茶树群体的192个单株进行遗传多样性分析,共获得149个等位基因,其中最多为10个,最少为4个,平均为5.96个;有效等位基因数在1.786~5.532,平均为3.424个(表3)。值通常反映引物的多态性程度,当值>0.5时,为高度多态性;当0.25<值<0.5时,为中度多态性;当值<0.25时,表现为低度多态性[20]。供试样品的值变化范围较大(0.440~0.819),平均值为0.660,说明本研究中所选出的25对引物总体上存在较高的多态性,适用于茶树种质资源的鉴定。观察杂合度()和期望杂合度()的范围在0~1(0表示无多态性;1表示无限多个等位基因具有相同频率),供试群体的位于0.214~0.750,平均值为0.510;位于0.441~0.821,平均值为0.681。表明在整体水平上,供试茶树群体具有较高的遗传多样性。
2.2 茶树群体遗传多样性与遗传结构分析
2.2.1 不同地区茶树群体遗传多样性分析
如表4所示,32个茶树群体的观测等位基因数()范围为2.760~3.760,平均观测等位基因数为3.318;有效等位基因数()的范围为1.813~2.794,平均有效等位基因数为2.445;Shannon’s多样性指数()范围为0.691~1.089,平均数为0.954;观测杂合度()的范围为0.253~0.633,平均数为0.510;期望杂合度()范围为0.430~0.653,平均数为0.590。32个茶树群体中有30个群体的观测杂合度小于期望杂合度,说明茶树群体内存有一定程度的近交现象,导致纯合子过量。多态位点数为23~25个,说明各群体的遗传多样性水平较高。
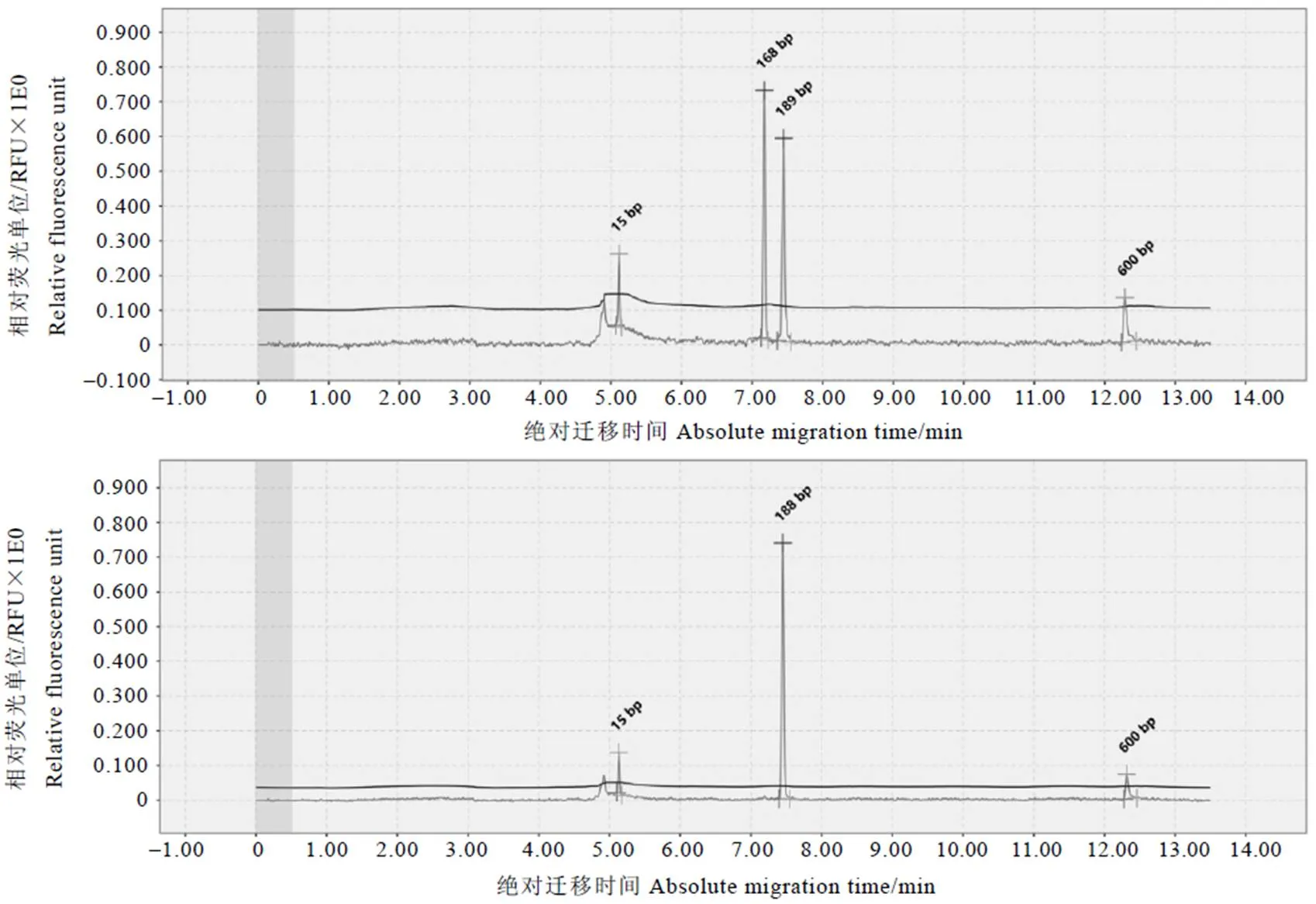
图1 引物TM209的部分毛细管电泳图谱
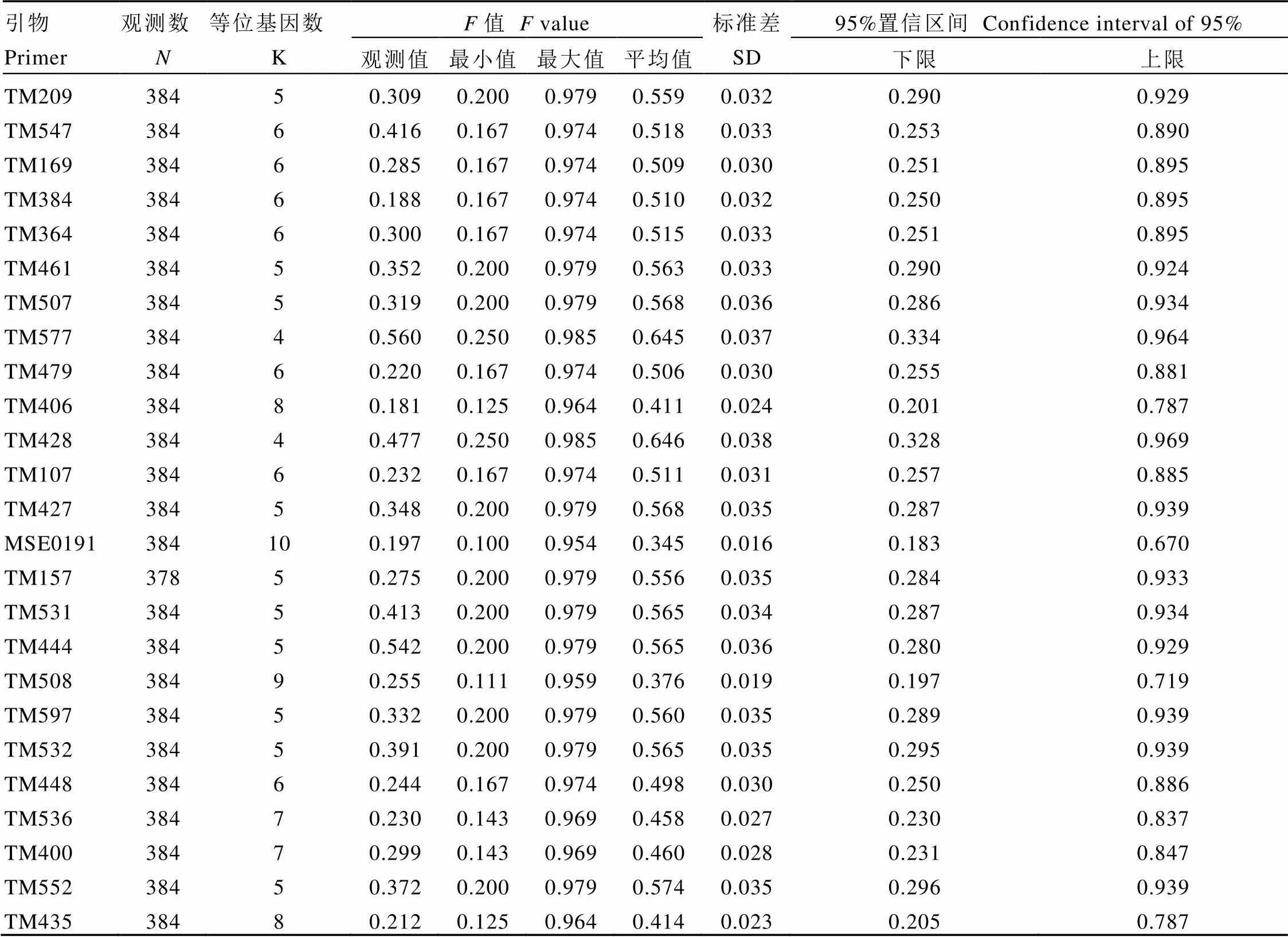
表2 25个位点中性检测信息
2.2.2 茶树群体遗传分化分析
对茶树群体遗传结构进行统计检验分析,结果如表5所示。25个SSR位点的基因流均值为0.969,约等于1,其中14个位点的基因流小于1,11个位点基因流大于1,说明茶树各群体内和群体间都存在一定程度的遗传分化,这可能与茶树作为经济作物受到人为干预较多以及茶树异花授粉、自交不亲和的特性有关。其总群体固定指数()的范围在0.022~0.660,平均值为0.250。群体内固定系数()平均值为0.056,接近于0,表明群体可能是Hardy-Weinberg随机交配群体[21]。群体间遗传分化系数()为0~<0.05时,群体间具有低水平遗传分化;位于0.05~<0.15时,群体间具有中等水平遗传分化;位于0.15~<0.25时,群体间具有较高水平遗传分化[22]。本研究供试茶树群体遗传分化系数()均值为0.205,说明群体间遗传分化水平较高。
2.2.3 系统发育分析及群体结构划分
基于供试样品两两之间的Nei’s遗传距离矩阵,应用邻接法对32份资源进行系统发育分析(图2)。其中来自同一省市不同地区的茶树地方群体品种显示出了较近的亲缘关系(簇a):黎家坪群体种和冯家台群体种(同属湖北省恩施市),龙井种和鸠坑种(同属于浙江省杭州市),黄山种和祁门种(同属安徽省黄山市)。整体分析结果表明,32个供试样品被划分为3个簇,簇a主要包括13份种质资源,分别为湖北(3份)、江西(2份)、浙江(2份)、贵州(1份)、安徽(2份)、福建(1份)、河南(1份)、陕西(1份)的种质资源;簇b包含12份种质资源,分别为江西(5份)、湖北(1份)、湖南(2份)、广西(1份)、贵州(2份)、云南(1份)的种质资源;簇c包括广西(2份)、广东(1份)、湖南(2份)、云南(2份)的种质资源。
应用Structure 2.3对供试材料进行群体结构分析,结果表明,当K=2时,ΔK的数值最大(图3)。按照似然值最大的要求分析来自32个地区的茶树群体种,可以将其分成2个遗传群体,红色表示亚群A,绿色表示亚群B(图4-A)。为了进一步研究茶树群体结构,本研究对K=4时的茶树群体结构也进行了分析(图4-B)。基于Structure 2.3软件分析得到的茶树群体分类结构与根据Nei’s遗传距离得到的邻接聚类分析结果一致。当K=2时,群体A包括整个簇c;群体B包含簇a和簇b。当K=4时,整体分类结构也和邻接聚类分析结果相符,在K=2时的分类结果上进一步细化,增加了亚群C(蓝色)和亚群D(黄色)。

表3 25个位点多样性信息
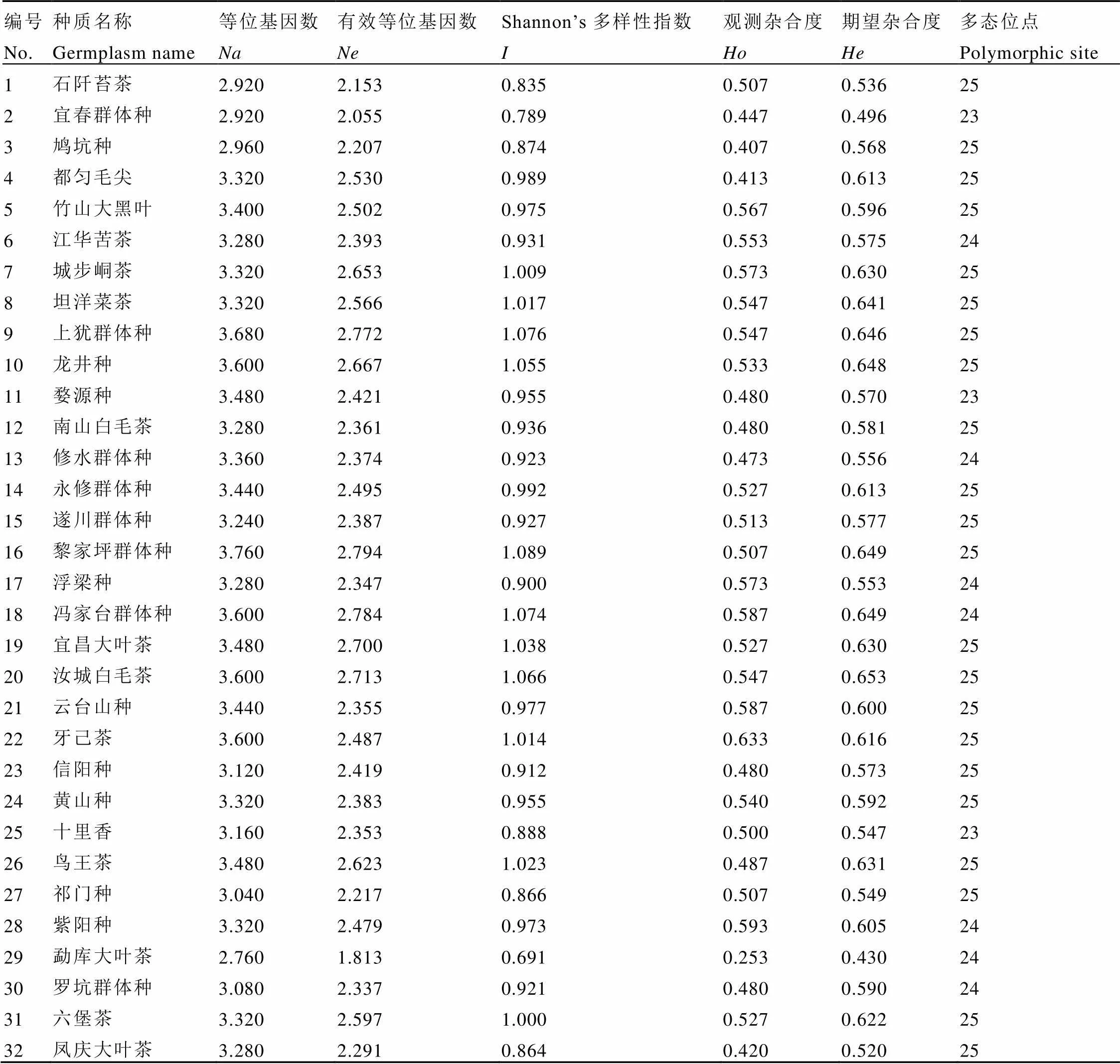
表4 群体遗传多样性信息表
综合系统发育树和群体结构分析的结果,供试群体可以划分为四大类。从原产地来看,这四大类茶树种质在地理分布上表现出一定规律:位于云南的群体种(凤庆大叶茶、勐库大叶茶)单独聚为一类;第二类在湖南南部和广西、广东区域集中分布;第三类主要分布在云南、贵州、广西、湖南、湖北、江西等地,这些地区位于供试茶区的中部;第四类主要分布在湖北、江西、福建、陕西、河南、安徽、浙江等地,这些地区位于供试茶区的东部和北部末端。
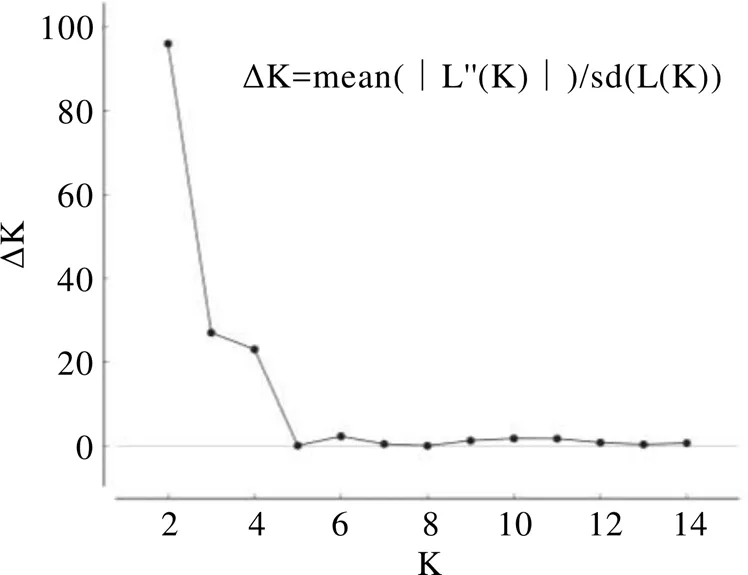
图3 ΔK随K值的变化趋势
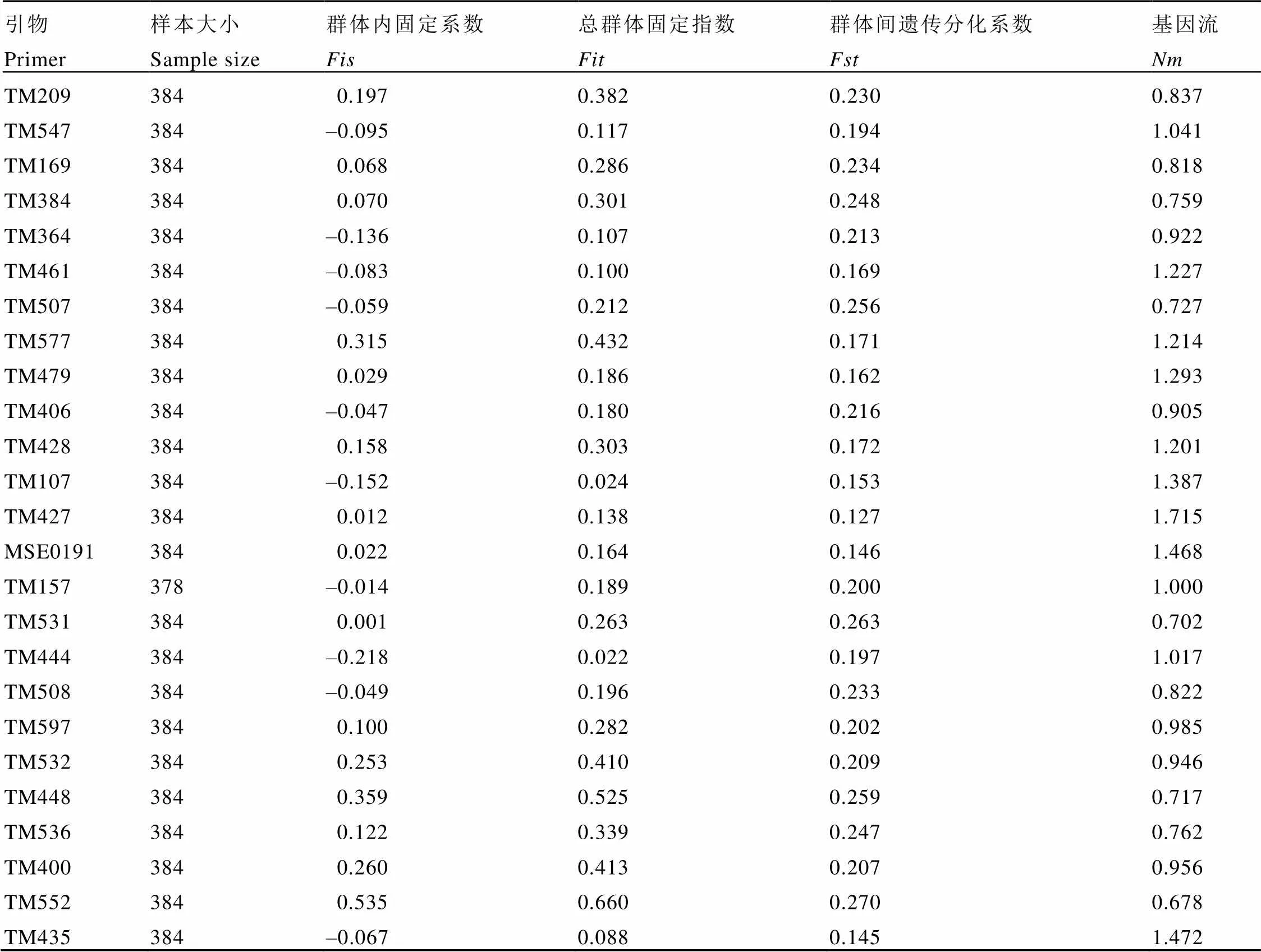
表5 F统计检验
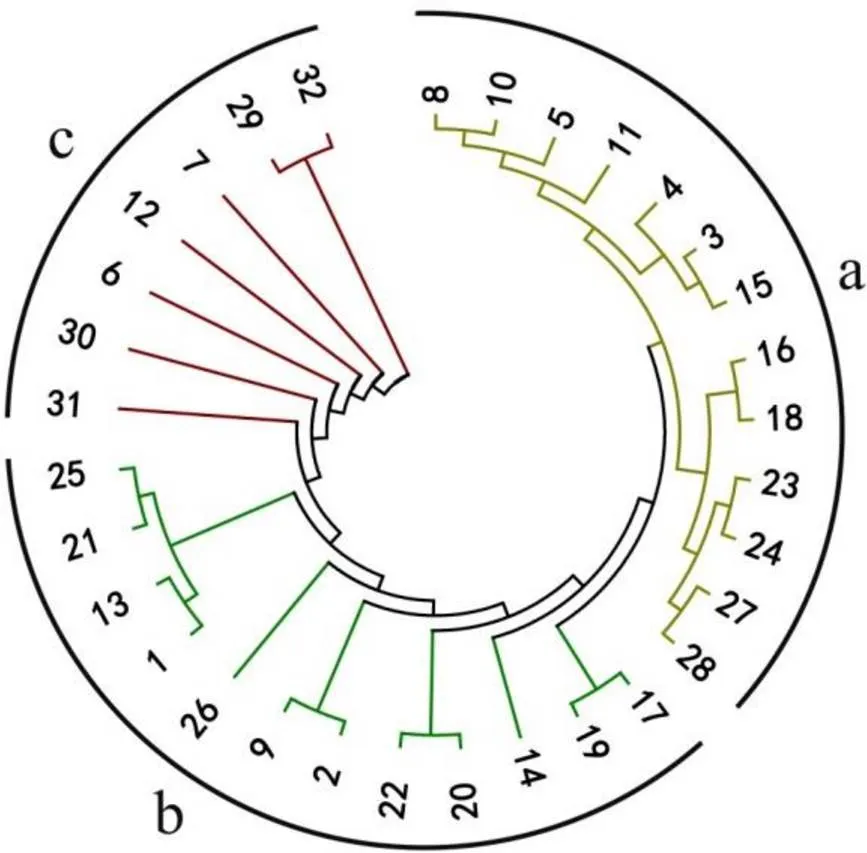
注:1:石阡苔茶;2:宜春群体种;3:鸠坑种;4:都匀毛尖;5:竹山大黑叶;6:江华苦茶;7:城步峒茶;8:坦洋菜茶;9:上犹群体种;10:龙井种;11:婺源种;12:南山白毛茶;13:修水群体种;14:永修群体种;15:遂川群体种;16:黎家坪群体种;17:浮梁种;18:冯家台群体种;19:宜昌大叶茶;20:汝城白毛茶;21:云台山种;22:牙己茶;23:信阳种;24:黄山种;25:十里香;26:鸟王茶;27:祁门种;28:紫阳种;29:勐库大叶茶;30:罗坑群体种;31:六堡茶;32:凤庆大叶茶。下同
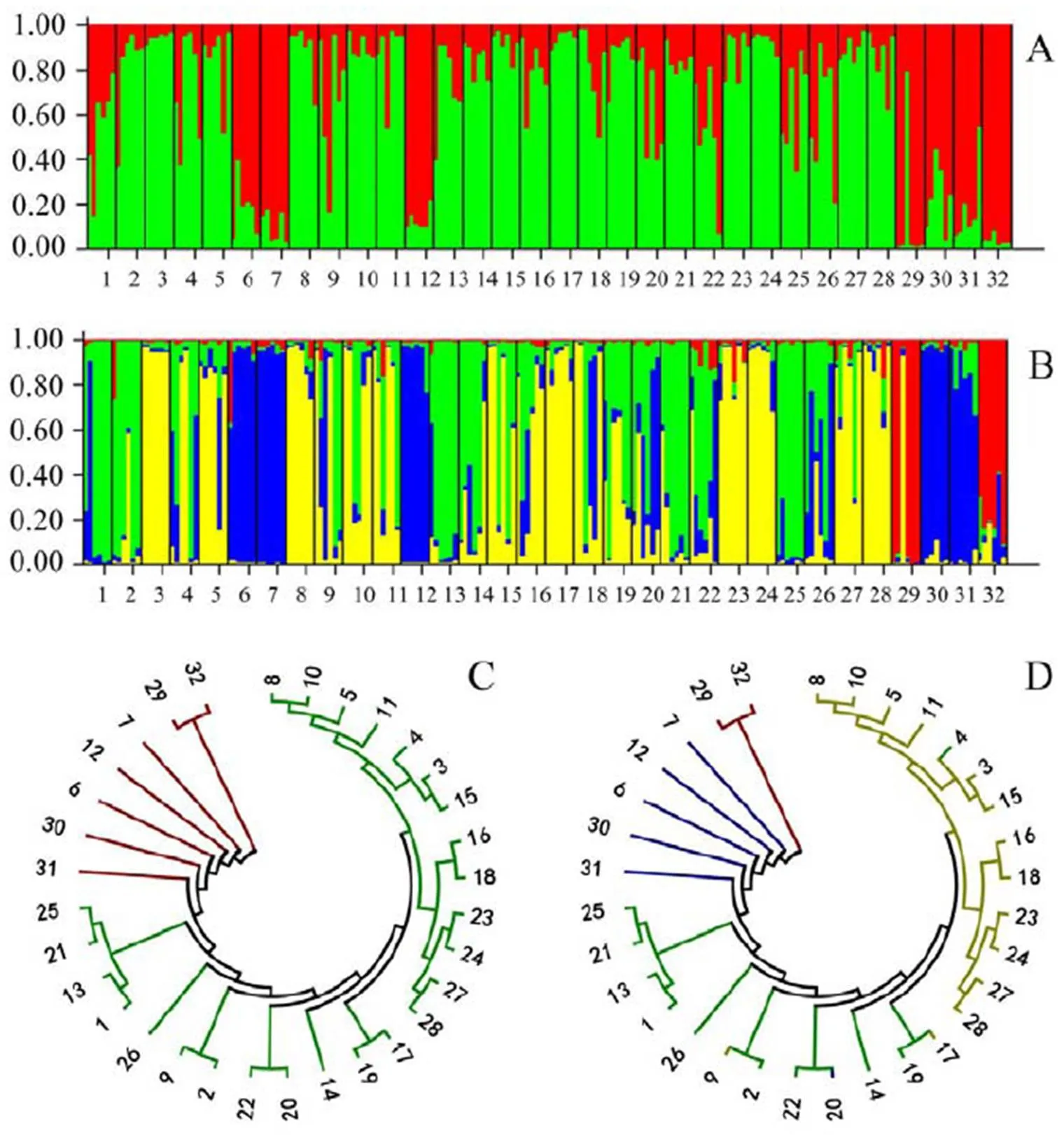
注:图A为K=2,红色表示群体A,绿色表示群体B。图B为K=4,红色表示群体A,绿色表示群体B,蓝色表示群体C,黄色表示群体D。图C为K=3时,Structure软件划分的A(红色)、B(绿色)两个亚群在系统发育树上的分布情况。图D为K=4时,Structure软件划分的A(红色)、B(绿色)、C(蓝色)、D(黄色)4个亚群在系统发育树上的分布情况
3 讨论
3.1 引物的多态性信息量
物种的遗传多样性分析是其种质资源评价和杂种优势利用的基础,遗传多样性评价指标易受到供试材料及标记方法的影响。本研究利用25对SSR引物对来自不同省份的192个单株进行遗传多样性分析,Hardy-Weinberg平衡检验和中性测试结果表明,所选的位点多为中性位点,各群体供试材料在遗传水平相对稳定、可靠,有利于茶树群体种质遗传多样性的研究。分析共获得149个等位基因,平均每个SSR标记检测到5.96个等位基因,引物多态性信息含量均值为0.660,表现为高多态性引物,有利于不同遗传背景供试材料的区分。与其他研究相比,本研究检测到的等位位点数偏高,这可能是由于本研究中研究材料涵盖面积广、类型丰富,且采用的毛细管电泳检测方法,数据较传统的聚丙烯酰胺凝胶电泳检测更为灵敏、精确,能区分3~5 bp的差异,故能检测到更多的等位位点。
3.2 供试样品的遗传多样性
乔婷婷[23]对不同省份茶树资源多样性进行统计,发现广西、广东和云南的资源多样性偏高,湖南、江苏、安徽和河南的多样性偏低。本研究中各省份的遗传多样性并没有明显的地域规律,这可能与所选取的供试材料及样本量不同有关。根据杂合度的概念,当杂合度大于0.5时,群体具有比较丰富的遗传多样性;且观测杂合度值和期望杂合度值越接近,群体的遗传多样性程度越高[24-25]。本研究供试茶树群体Shannon’s多样性指数为0.691~1.089,观测杂合度平均值为0.51,期望杂合度平均值为0.59,各群体的观测杂合度值和期望杂合度值非常接近,说明供试的32个群体都具有较高的遗传多样性,其中城步峒茶群体的观测杂合度及期望杂合度与刘振等[26]研究结果相似。遗传分化系数()平均值为0.205,表明供试茶树群体间遗传分化水平较高,侧面反映了研究选用的SSR位点对供试样品具有较好的区分效果。
3.3 供试样品的群体结构
黄丹娟[13]和乔婷婷[23]基于分子标记分别对不同省份优良茶树品种进行聚类分析,都发现福建、浙江、安徽和河南的种质资源聚在同一类群,与本研究分析得到的茶树群体聚类结果一致。本研究中,来自云南的凤庆大叶茶和勐库大叶茶并没有与十里香聚在一起,而是单独聚为一类,推测这可能与横断山脉独特的地貌有关。研究表明,山脉和河流可以改变植物的生境并影响物种的群体结构,横断山脉是中国最长、最宽和最典型的南北走向山系,也是植物区系东西、南北多样性融合和选择演化的关键地带[27-29],在地理分布上,凤庆大叶茶和勐库大叶茶原产地位于横断山脉西侧区域,而十里香原产地位于横断山脉东侧区域。
本研究中第二大类包含城步峒茶、江华苦茶、汝城白毛茶、六堡茶、南山白毛茶和罗坑群体种6份种质资源,聚集分布在3个彼此相邻的省份中,与凤庆大叶茶和勐库大叶茶亲缘关系较近;在K=2时,第四大类与第三大类聚为一类,第四大类主要分布于前人所推测茶树传播路线的末端区域,而第三大类在供试茶区西南到东北方向皆有分布,且从云南经贵州、湖南呈放射状在广西、江西、湖北等地分布。结合本研究系统发育分析结果,推测遗传背景不同的供试茶树分别通过以下2条途径传播:(1)一部分种质(第一大类和第二大类)从云南经广西向湖南、广东传播;(2)其他种质(第三大类和第四大类)从云南经贵州、湖南向湖北、江西传播,最终传播到福建、安徽、浙江、陕西、河南等地。
3.4 茶树地方群体品种的收集与保护
32份茶树地方群体种遗传多样性研究结果显示,每份茶树地方群体种内包含着丰富的种质资源,且不同群体种间遗传分化水平较高。系统发育及群体结构分析表明,供试茶区中,西南地区较东北地区种质遗传分化水平高,且传播中间地带(湖南、湖北、江西)包含茶树资源类型较为丰富,供试茶区东部末端及北部末端类型单一、遗传背景较为狭窄,与姚明哲等[30]的研究结果相似;对江南茶区茶树地方品种遗传多样性进行分析,湖南、湖北、江西的种质表现出了丰富的多样性[31];对来自安徽省内10个地区的茶树群体种进行遗传多样性分析,结果表明它们遗传相似性极高,遗传相似系数为0.946~0.987[32]。基因分型往往与表型性状相关联,余书平等[33]基于分子标记技术对开化县茶树群体种进行亲缘关系分析,发现了两个基于分子标记的基因分型结果一致、表型性状表现相似的特异种质。可以适当结合分子标记技术开展各地方种质遗传多样性研究,挖掘亲缘关系相对较远的核心种质,因地制宜的开展茶树地方群体种质的收集与保护工作。
感谢中国农业科学院茶叶研究所陈亮研究员课题组为本研究提供引物。
[1] 谢文钢, 李晓松, 李伟, 等. 四川地方中小叶茶树资源的表型遗传多样性[J]. 浙江农业学报, 2019, 31(9): 1405-1415.
Xie W G, Li X S, Li W, et al. Genetic diversity of “Small & Medium Leaf” tea resources in Sichuan Province based on phenotypic characteristics [J]. Acta Agriculturae Zhejiangensis, 2019, 31(9): 1405-1415.
[2] 黄晓敏. 闽南茶区不同乌龙茶品种叶片显微结构比较研究[D]. 福州: 福建农林大学, 2010.
Huang X M. Comparative research on leaf tissues for different oolong tea cultivars in southern tea region of Fujian Province [D]. Fuzhou: Fujian Agriculture and Forestry University, 2010.
[3] 许燕. 6个特色茶树品种(系)光合特性及叶绿体超微结构研究[D]. 成都: 四川农业大学, 2016.
Xu Y. Studies on photosynthetic traits and chloroplast ultrastructure of 6 tea characteristic varieties (lines) [D]. Chengdu: Sichuan Agriculture University, 2016.
[4] Martinez L E, Cavagnaro P F, Masuell R W, et al. SSR-based assessment of genetic diversity in south Americanvarieties [J]. Plant Science, 2006, 170(6): 1036-1044.
[5] Yao M Z, Chen L, Liang Y R. Genetic diversity among tea cultivars from China, Japan and Kenya revealed by ISSR markers and its implication for parental selection in tea breeding programmes [J]. Plant Breeding, 2008, 127(2): 166-172.
[6] Yao M Z, Ma C L, Qiao T T, et al. Diversity distribution and population structure of tea germplasms in China revealed by EST-SSR markers [J]. Tree Genetics & Genome, 2012, 8(1): 205-220.
[7] Zhao L P, Liu Z, Chen L, et al. Generation and characterization of 24 novel EST derived microsatellites from tea plant () and cross-species amplification in its closely related species and varieties [J]. Conservation Genetics, 2008, 9(5): 1327-1331.
[8] Bandyopadhyay T. Molecular marker technology in genetic improvement of tea [J]. International Journal of Plant Breeding and Genetics, 2011, 5(1): 23-33.
[9] Fang W P, Meinhardt L W, Tan H W, et al. Varietal identification of tea () using nanofluidic array of single nucleotide polymorphism (SNP) markers [J]. Horticulture Research, 2014, 1(1): 14035. doi: 10.1038/hortres.2014.35.
[10] Hu C Y, Tsai Y Z, Lin S F. Development of STS and CAPS markers for variety identification and genetic diversity analysis of tea germplasm in Taiwan [J]. Botanical Studies, 2014, 55(1): 1-15.
[11] 李婧慧, 金彦龙, 乔艳清, 等. 新疆150份陆地棉品种DNA指纹图谱构建与遗传多样性分析[J/OL]. 分子植物育种: 2020[2021-03-02]. http://kns.cnki.net/kcms/detail/46.1068.S.20201217.1805.004.html.
Li J H, Jin Y L, Qiao Y Q, et al. Construction of DNA fingerprint and genetic diversity of 150 upland cotton varieties in Xinjiang [J/OL]. Molecular Plant Breeding: 2020[2021-03-02]. http://kns.cnki.net/kcms/detail/46.1068.S.20201217.1805.004.html.
[12] 姜慧, 潘根, 常丽, 等. 亚麻分子标记辅助育种研究进展[J/OL]. 植物遗传资源学报: 2020[2021-03-02]. https://doi.org/10.13430/j.cnki.jpgr.20201124001.
Jiang H, Pan G, Chang L, et al. Advances in molecular marker assisted breeding of flax [J/OL]. Journal of Plant Genetic Resources: 2020[2021-03-02]. https://doi.org/10.13430/j.cnki.jpgr.20201124001.
[13] 黄丹娟. 我国茶树优良品种遗传多样性及指纹图谱构建[D]. 北京: 中国农业科学院, 2016.
Huang D J. Genetic diversity and fingerprinting of Chinese improved tea cultivars [D]. Beijing: Chinese Academy of Agricultural Sciences, 2016.
[14] 姜晓辉, 方开星, 陈栋, 等. 基于EST-SSR毛细管电泳荧光标记技术分析广东2个历史名茶群体遗传多样性[J]. 热带作物学报, 2018, 39(1): 46-54.
Jiang X H, Fang K X, Chen D, et al. Genetic diversity and relationship of two historical famousgroups in Guangdong by capillary electrophoresis detection with fluorescent EST-SSR marker [J]. Chinese Journal of Tropical Crops, 2018, 39(1): 46-54.
[15] 杨军, 王让剑, 孔祥瑞, 等. 94个茶树种质资源遗传多样性与亲缘关系分析[J]. 茶叶学报, 2018, 59(4): 186-192.
Yang J, Wang R J, Kong X R, et al. Genetic diversity and relationship of 94 tea germplasm [J]. Acta Tea Sinica, 2018, 59(4): 186-192.
[16] 姚明哲, 马春雷, 陈亮, 等. 川、渝地方茶树品种的遗传多样性和群体结构[J]. 茶叶科学, 2012, 32(5): 419-425.
Yao M Z, Ma C L, Chen L, et al. Genetic diversity and population structure of local tea landraces in Sichuan and Chongqing revealed by EST-SSR markers [J]. Journal of Tea Science, 2012, 32(5): 419-425.
[17] Zhao D, Yang J, Yang S, et al. Genetic diversity and domestication origin of tea plant(Theaceae) as revealed by microsatellite markers [J]. BMC Plant Biology, 2014, 14(1): 14. doi: 10.1186/1471-2229-14-14.
[18] Khawla G A, Jasmine K J, Rose L A. Chloroplast genome analysis of box-ironbark[J]. Molecular Phylogenetics and Evolution, 2019, 136: 76-86.
[19] 朱小虎. 新疆密叶杨群体遗传多样性及遗传结构研究[D]. 北京: 北京林业大学, 2019.
Zhu X H. Study on genetic diversity and genetic structure ofKom. population in Xinjiang [D]. Beijing: Beijing Forestry University, 2019.
[20] Hieter P A. Clustered arrangement of immunoglobulin lambda constant region genes in man [J]. Nature, 1981, 294: 536-540.
[21] 刘爽. 中国樟树群体遗传结构研究[D]. 南昌: 江西农业大学, 2019.
Liu S. Population genetic structure ofin China [D]. Nanchang: Jiangxi Agricultural University, 2019.
[22] 黄燕, 朱振东, 段灿星, 等. 灰葡萄孢蚕豆分离物的遗传多样性[J]. 中国农业科学, 2014, 47(12): 2335-2347.
Huang Y, Zhu Z D, Duan C X, et al. Genetic diversity ofisolates from broad bean [J]. Scientia Agricultura Sinica, 2014, 47(12): 2335-2347.
[23] 乔婷婷. 茶树资源遗传多样性及其表型性状关联EST-SSR位点的初步鉴定[D]. 北京: 中国农业科学院, 2010.
Qiao T T. Genetic diversity of tea ((L.) O. Kuntze) and association analysis of phenotypic traits with EST-SSR markers [D]. Beijing: Chinese Academy of Agricultural Sciences, 2010.
[24] 汤存伟, 余雄, 刘武军, 等. 新疆13个绵羊群体遗传多样性及遗传分化的研究[J]. 家畜生态学报, 2011, 32(1): 13-19.
Tang C W, Yu X, Liu W J, et al. Study on genetic diversity and genetic differentiation of 13 sheep populations in Xinjiang Uygur Autonomous Region [J]. Journal of Domestic Animal Ecology, 2011, 32(1): 13-19.
[25] Wright S. Evolution and the genetics of populations. 4: variability within and among natural populations [M]. Chicago: The University of Chicago Press, 1978.
[26] 刘振, 成杨, 杨培迪, 等. 基于nSSR和cpDNA序列的城步峒茶群体遗传多样性和结构研究[J]. 茶叶科学, 2020, 40(2): 250-258.
Liu Z, Cheng Y, Yang P D, et al. Genetic diversity and structure of Chengbudong tea population revealed by nSSR and cpDNA markers [J]. Journal of Tea Science, 2020, 40(2): 250-258.
[27] 王铠元, 孙克祥, 卢瑞甫. 滇西地区大地构造演化[J]. 青藏高原地质文集, 1983(4): 187-199.
Wang K Y, Sun K X, Lu R P. Tectonic evolution in western Yunnan [J]. Collected Works of Qinghai-Tibet Plateau Geology, 1983(4): 187-199.
[28] 明庆忠. 纵向岭谷北部三江并流区河谷地貌发育及其环境效应研究[D]. 兰州: 兰州大学, 2006.
Ming Q Z. The landform development and environmental effects in the north of Longitudinal Range-Gorge Region (LRGR) [D]. Lanzhou: Lanzhou University, 2006.
[29] Schultz M. Phylogeography’s past, present, and future: 10 years after Avise, 2000 [J]. Molecular Phylogenetics and Evolution, 2010, 54(1): 291-301.
[30] 姚明哲, 刘振, 陈亮, 等. 利用EST-SSR分析江北茶区茶树资源的遗传多样性和遗传结构[J]. 茶叶科学, 2009, 29(3): 243-250.
Yao M Z, Liu Z, Chen L, et al. Genetic diversity and structure of tea germplasm originated from region of north Yangtze River based on EST-SSR markers [J]. Journal of Tea Science, 2009, 29(3): 243-250.
[31] 姜燕华. 我国茶树地方品种遗传多样性及人为选择影响的研究[D]. 北京: 中国农业科学院, 2010.
Jiang Y H. Studies on the genetic diversity of traditional tea germplasm in China as well as the influence of artificial selection [D]. Beijing: Chinese Academy of Agricultural Sciences, 2010.
[32] 丁洲, 李烨昕, 袁艺, 等. 安徽茶区优良群体种的表型性状和遗传多样性分析[J]. 茶叶科学, 2018, 38(2): 155-161.
Ding Z, Li Y X, Yuan Y, et al. Phenotypic traits and genetic diversity of elite tea population in Anhui (China) [J]. Journal of Tea Science, 2018, 38(2): 155-161.
[33] 余书平, 徐礼羿, 吴荣梅, 等. 浙江开化县茶树种质资源的遗传多样性及亲缘关系分析[J]. 茶叶科学, 2020, 40(3): 341-351.
Yu S P, Xu L Y, Wu R M, et al. Genetic and phylogenetic analysis for resources offrom Kaihua County in Zhejiang Province [J]. Journal of Tea Science, 2020, 40(3): 341-351.
Analysis of Genetic Diversity and Population Structure of 32 Tea Landraces in China
LI Changle, GE Yue, YAN Meilin, LI Hui, LIN Qingqing, WANG Pu, ZHAO Hua, WANG Mingle, WANG Yu, GUO Fei*, NI Dejiang
Key Laboratory of Horticultural Plant Biology, Ministry of Education, College of Horticulture and Forestry Sciences, Huazhong Agricultural University, Wuhan 430070, China
Tea plant populations are natural populations of tea plants. They are cultivated in specific production areas and contain individuals with different economic and biological traits. There are great differences among individuals and there are many types. They are important materials for studying the evolutionary relationship and breeding of tea plants. In this study, 30 pairs of SSR primers distributed on 15 linkage groups were selected to analyze the genetic diversity and population structure of 32 tea plant populations from 12 provinces, in order to provide a reference for the selection of tea breeding parents and the inference of evolution route. In this study, a total of 149 alleles were obtained with an average of 5.96 for each SSR marker. The average polymorphism information content of primers was 0.660. Shannon's diversity index of 32 tea populations ranged from 0.691 to 1.089, with an average of 0.954. The observed heterozygosity ranged from 0.253 to 0.633, with an average of 0.510. The expected heterozygosity ranged from 0.430 to 0.653, with an average of 0.590. The average genetic differentiation coefficient () of tea plant population was 0.205, indicating a high level of genetic differentiation. The results of clustering based on Nei's genetic distance and population structure analysis were consistent. The germplasm to be tested was divided into 4 major types, with obvious regional distribution.
tea landraces, SSR molecular marker, population structure, genetic diversity, evolutionary route
S571.1;S324
A
1000-369X(2021)05-619-12
2021-03-02
2021-03-24
国家重点研发计划(2019YFD1001601)、园艺作物种质资源平台(2020DFE017)、中央高校基本科研业务费专项(2662020YLPY023)
李长乐,女,硕士研究生,主要从事茶树遗传育种研究。*通信作者:guofei@mail.hzau.edu.cn
(责任编辑:黄晨)
