小麦NPR1基因家族鉴定及其表达分析
2021-09-13李雅倩,王林,宋婧含,牛红莉,朱光,高春保,方正武,刘易科
李雅倩,王林,宋婧含,牛红莉,朱光,高春保,方正武,刘易科


摘要:【目的】對小麦NPR1基因家族成员进行鉴定及表达分析,为探究该家族基因的作用机制及小麦遗传改良提供理论参考。【方法】以拟南芥NPR1家族蛋白序列为参考序列,从小麦基因组中鉴定出小麦NPR1基因家族成员,利用生物信息学软件对其序列特征进行分析,并分别利用RNA-Seq原始数据和实时荧光定量PCR(qRT-PCR)分析小麦NPR1家族基因在不同组织及不同胁迫下的表达水平。通过STRING在线网站构建TaNPR1s蛋白的互作网络。【结果】共鉴定获得20个小麦NPR1基因家族成员,其编码蛋白的不稳定指数均大于40,为不稳定蛋白;平均总亲水性值(GRAVY)均为负值(除TaNPR5-D为正值外),为亲水蛋白;主要分布于细胞核内,在叶绿体、线粒体、内质网和细胞质等部位也有分布;二级结构均由α-螺旋、延伸链、β-转角和无规卷曲组成,以α-螺旋和无规则卷曲为主;对应的三级结构模型有10种。20个TaNPR1s蛋白均可与转录因子HBP-1b及未知蛋白A、B、C、D发生相互作用,这5种蛋白均含有bZIP结构域(含TGACG基序)和种子休眠特异基因结构域(DOG1)。TaNPR1s基因在不同组织中的表达模式不同,可分为在多个组织中表达、在特定的组织或发育阶段表达和在不同组织发育阶段均低表达或不表达,共三大类。随机挑选的8个TaNPR1s基因中,有3个基因在禾谷镰刀菌胁迫下表达量降低,但在白粉病菌胁迫下表达量升高;有2个基因在这两种菌胁迫下表达量均升高,有2个基因在两种菌胁迫下表达量均下降。TaNPR1s基因对6种非生物胁迫处理均有响应,但表达模式存在差异。【结论】小麦NPR1基因家族成员在不同组织生长发育过程和生物和非生物胁迫响应中发挥重要调控作用,且基因的可变剪接体也表现出不同组织表达特性,丰富了NPR1s蛋白功能。TaNPR1s蛋白可能通过与bZIP和DOG1结构域结合发挥其生物学功能。
关键词: 小麦;NPR1基因家族;鉴定;生物信息学分析;表达分析
中图分类号: S512.103.53 文献标志码: A 文章编号:2095-1191(2021)09-2339-11
Genome-wide identification and expression analysis of
wheat NPR1 gene family
LI Ya-qian1,2, WANG Lin1,2, SONG Jing-han1,2, NIU Hong-li1, ZHU Guang2,
GAO Chun-bao1,2, FANG Zheng-wu1*, LIU Yi-ke2*
(1College of Agronomy, Yangtze University/Hubei Collaborative Innovation Center for Grain Industry, Jingzhou, Hubei 434025, China; 2Food Crops Institute,Hubei Academy of Agricultural Sciences/Wheat Disease Biology
Research Station on Central China,Ministry of Agriculture and Rural Affairs/Hubei Engineering and
Technology Research Center of Wheat, Wuhan 430064, China)
Abstract:【Objective】The identification and expression analysis of NPR1 gene family members in wheat provided theoretical reference for exploring the mechanism of this family gene and wheat genetic improvement. 【Method】Wheat NPR1 gene family members from wheat genome were identified based on NPR1 family protein sequence from arabidopsis. The sequence characteristics were analyzed by bioinformatics software, and the expression levels of wheat NPR1 family genes under different tissues and different stresses were analyzed by RNA-Seq data and real-time fluorescence quantitative PCR(qRT-PCR). The online website STRING was used to predict and analyze the interaction relationship between related proteins of TaNPR1 family members. 【Result】A total of 20 members of wheat NPR1 gene family were identified, and the TaNPR1s encoded by them were unstable proteins with instability index greater than 40. The average total hydrophilic va-lue (GRAVY) was negative (except TaNPR5-D was positive), which was hydrophilic protein. It was mainly distributed in the nucleus, chloroplast, mitochondria, endoplasmic reticulum and cytoplasm. The secondary structure was composed of α-helix, extended strand, β-turn and random coil, and α-helix and random coil were the main components. There were 10 kinds of tertiary structure models. All 20 TaNPR1s proteins could interact with the transcription factor HBP-1b and unknown proteins A, B, C and D, the five proteins all contained bZIP domain(containing TGACG motif) and seed dormancy specific gene domain(DOG1). The expression pattern of TaNPR1s gene in different tissues was different, which could be divided into three categories:expressed in multiple tissues, expressed in specific tissues or developmental stages, and all low expression or no expression in different tissue development stages. Among the 8 randomly selected TaNPR1s genes, the expression levels of 3 genes decreased under Fusarium graminearum stress, but increased under powdery mildew stress. The expression levels of two genes increased under both strains, and the expression levels of two genes decreased under both strains. TaNPR1s gene responded to all six abiotic stress treatments, but their expression patterns were different. 【Conclusion】Members of wheat NPR1 gene family play an important role in regulating the growth and development of different tissues and the response to biological and abiotic stresses, and the variable spliceosomes of genes also show different tissue expression characteristics, enriching the function of NPR1s protein. TaNPR1s protein may play its bio-logical function by binding to bZIP and DOG1 domains.
Key words: wheat; NPR1 gene family; identification; bioinformatics analysis; expression analysis
Foundation item: Hubei Provincial Special Project of Central Government Guides Local Science and Technology Development(2020ZYYD011);National Wheat Industry Technology System Construction Project(CARS-03)
0 引言
【研究意义】小麦是世界广泛种植的主要农作物之一,也是人类的主要口粮来源,其生长发育过程中易遭到各种病原微生物的侵扰,从而影响其产量和品质(Wang et al.,2018;陈文烨等,2020)。植物在生物体进化过程中为了抵抗各种生物和非生物胁迫形成了各种免疫防御机制(Jones and Dangl,2006;陈文烨等,2020),其中,植物的系统性抗病机理存在两种发生类型:一种是致病菌所诱导的系统获得性抗性(Systemic acquired resistance,SAR);另一种为非致病性微生物所诱导的系统诱导性抗性(Induced systemic resistance,ISR)(Kogel and Langen,2005;郑世仲等,2020)。非表达病程相关蛋白(Non-expresser of pathogenesis related genes 1,NPR1)是植物防御信号中的一个重要调节因子,广泛参与植物体内的免疫防御反应(Mhamdi,2019),其在SAR反应过程中是水杨酸(Salicylic acid,SA)信号通路的重要组成部分,也是植物病程相关蛋白(Pathogenesis-rela-ted proteins,PRs)基因表達和SAR的激活子(Cao et al.,1997;Zhou et al.,2000;Backer et al.,2019);其在茉莉酸(Jasmonic acid,JA)介导的ISR反应过程中是JA信号途径的负调控因子(Pieterse et al.,1998;Spoel et al.,2003)。此外,NPR1是植物响应病原菌侵染的核心基因,参与调控植物体内SA与JA的平衡(韩永光等,2018)。在植物对冷环境适应过程中NPR1基因参与调节冷诱导基因表达(Olate et al.,2018)。因此,对小麦基因组中的NPR1基因家族成员进行鉴定及分析,对探究NPR1基因在小麦抗逆过程中的作用机制及培育小麦抗性品种具有重要意义。【前人研究进展】NPR1基因首次从拟南芥中被克隆,该基因启动子区域的W-box元件和WRKY转录因子结合进而调控NPR1基因的转录,其编码蛋白的C端含有锚蛋白重复序列(Ank)和NPR1-like C-terminal结构域,N端有一个BTB/POZ(Broad complex Tramtrack and Bric-a-brac/Pox virus and zinc finger)结构域(Cao et al.,1994;Cao et al.,1997;Aravind and Koonin,1999)。目前拟南芥基因组测序已完成,共鉴定出6个AtNPR1s基因(Initiative,2000),其中AtNPR1和AtNPR2是SA受体,在植物免疫中起转录共激活因子的作用(Castelló et al.,2018);AtNPR3和AtNPR4也是SA受体,在植物防御中起转录辅抑制因子的作用(Zhang et al.,2006);AtNPR5和AtNPR6基因又被称为AtBOP1和AtBOP2基因,因缺乏NPR1-like C-terminal结构域,在系统发育进化树中分支差异较大,二者主要在拟南芥生长发育过程和形态建成中发挥重要作用(Hepworth et al.,2005;Mikael et al.,2005)。目前大量研究证实,NPR1基因对植物的抗病性中发挥重要作用,如在苹果中过量表达MpNPR1基因能加强植株对火疫病、黑星病菌及胶锈菌的抗病性(Malnoy et al.,2007);将拟南芥AtNPR1基因转入草莓中可增强草莓对炭疽病、白粉病及叶斑病等的抗性(Silva et al.,2015);水稻中过表达OsNPR1基因能增强植株对叶枯病菌的抗性(Yuan et al.,2007)。抑制烟草NPR1基因表达可导致植株丧失烟草花叶病毒(TMV)抗性(Liu et al.,2002)。保守的条锈菌蛋白可与小麦NPR1蛋白发生相互作用,并降低病原体对致病相关基因(Pathogenesis-related genes,PR)的诱导(Wang et al.,2016)。NPR1基因在受到病原物侵染的条件下,才能激活下游防御基因表达,在未被诱导物或病原体激活时,PR基因的表达并未显著提高(蔡韡韡等,2019;Tripathi et al.,2019)。【本研究切入点】目前,NPR1基因家族已在番木瓜(Peraza-Echeverria et al.,2012)、苹果(焦鹏,2016)、香蕉(任陪娣等,2019)、小麦(Liu et al.,2019) 和油菜(Wang et al.,2020)等多种植物中鉴定分析。但针对小麦NPR1基因在生物和非生物胁迫下的表达模式分析较少且缺乏验证,同时对该家族基因的蛋白特性还缺乏系统研究。【拟解决的关键问题】以拟南芥NPR1家族蛋白序列为参考序列,从小麦基因组中鉴定出小麦NPR1基因家族成员,利用生物信息学软件对其序列特征进行分析,并分别利用RNA-Seq原始数据和实时荧光定量PCR(qRT-PCR)分析小麦NPR1家族基因在不同组织及不同胁迫下的表达水平;通过STRING在线网站构建TaNPR1s蛋白的互作网络,为探究该家族基因的作用机制及小麦遗传改良提供理论参考。
1 材料与方法
1. 1 试验材料
供试的小麦品种为鄂麦170,由湖北省农业科学院粮食作物研究所提供。RNA提取试剂TRIzol购自赛默飞世尔科技(中国)有限公司。反转录试剂盒(Prime ScriptTM RT reagent Kit with gDNA Eraser)和实时荧光定量PCR试剂盒(SYBR Premix Ex Taq)购自TaKaRa公司。主要设备仪器:人工气候箱(PQX智能型,宁波莱福科技有限公司)、凝胶成像系统(Bio-Rad,美国)和CFX Connect Real-Time System(Bio-Rad,美国)。
1. 2 样品处理及采集
参考Jiang等(2019)的方法对小麦种子消毒处理后进行无菌培养,生长出来的小麦幼苗在人工气候箱中培养至两叶一心时期。对小麦幼苗分别进行生物胁迫[禾谷镰刀菌(PH-1)、白粉病菌(Bgt strain E09)]和非生物胁迫[热(42 ℃)、冷(4 ℃)、脱落酸(ABA,100 μmol/L)、水杨酸(SA,100 μmol/L)、茉莉酸甲酯(MeJA,100 μmol/L)和氯化钠(NaCl,150 μmol/L)]处理24 h,对照组(CK)不作胁迫处理。收集幼苗根和叶部位组织,每3株混作一个样,液氮速冻处理后,-80 ℃保存备用,用于分析不同逆境胁迫下TaNPR1基因的表达特性。
1. 3 小麦NPR1基因家族成员鉴定
以拟南芥AtNPR1(AT1G64280.1)、AtNPR2(AT4G26120.1)、AtNPR3(AT5G45110.1和AT5G451 10.2)、AtNPR4(AT4G19660.1和AT4G19660.2)、AtNPR5(AT2G41370.1)和AtNPR6(AT3G57130.1)的蛋白序列为查询序列,在小麦基因组数据库中进行BLASTp比对(E值<1×10-10)(Altschul et al.,1997)。将检索到的序列提交到Pfam(http://pfam.xfam.org/)(Finn et al.,2006)和InterProScan(http://www.ebi.ac.uk/interpro/)(Mulder and Apweiler,2007)網站,筛选出同时含有BTB/POZ和Ankyrin结构域的蛋白,即为小麦NPR1基因家族成员。
1. 4 生物信息学分析及互作预测
小麦NPR1蛋白的理化性质通过ExPASy Ser-ver10(https://prosite.espasyp.org/)(Wilkins et al.,1999)进行预测。利用WoLF PSORT:Protein Subcellular Localization Prediction(https://www.genscript.com/wolf-psort.html)(Horton et al.,2007)在线预测亚细胞定位。利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测蛋白二级结构。使用SWISS-MODEL(https://swissmodel.expasy.org/interactive#alignment)(Schwede et al.,2003)对蛋白的三级结构进行预测。通过功能蛋白关联网络在线网站STRING(https://stringdb.org)(Szklarczyk et al.,2018)预测小麦NPR1基因家族成员相关蛋白的互作关系,可信度分数为0.70。
1. 5 转录组数据分析
从NCBI的Short Read Archive(SRA)数据库中下载关于不同组织生长发育相关的RNA-Seq原始数据(SBA编号:PRJEB25639),以及不同逆境胁迫处理的RNA-Seq原始数据(SBA编号:条锈菌PRJEB12497、禾谷镰刀菌PRJEB12358、干旱PRJNA306536和高温─干旱共处理PRJNA257938),并通过HISAT2将其映射到小麦基因组中。转录组数据采用TPM值(Transcripts Per Million)来表示基因的表达量。TPM值低于1的看作基因不表达或表达量很低。利用R语言“pheatmap”包,基于TPM值绘制基因表达谱,以Log2(TPM+1)值绘制NPR1基因差异表达热图。
1. 6 实时荧光定量PCR(qRT-PCR)表达分析
利用反转录试剂盒将RNA反转录合成cDNA。采用SYBR Premix Ex Taq进行qRT-PCR检测。反应体系(20.0 μL):cDNA 2.0 μL,正、反向引物各0.8 μL,50×ROX Reference Dye II 0.4 μL,TB Green Premix Ex Taq II 10.0 μL,灭菌水补足至20.0 μL。扩增程序:95 ℃ 3 min;95 ℃ 10 s,60 ℃ 30 s(同时收集信号值),共进行40个循环,溶解曲线分析为65 ℃。β-Actin作为相对定量的内参基因。利用Primer Premier 5.0设计内参基因和小麦NPR1基因家族成员的引物序列,如表1所示。采用2-ΔΔCt方法进行数据计算,每个样本技术重复3次,基因的相对表达量用平均值±标准差表示。对照的相对表达量为1。
2 结果与分析
2. 1 小麦NPR1基因家族成员鉴定及序列特征分析结果
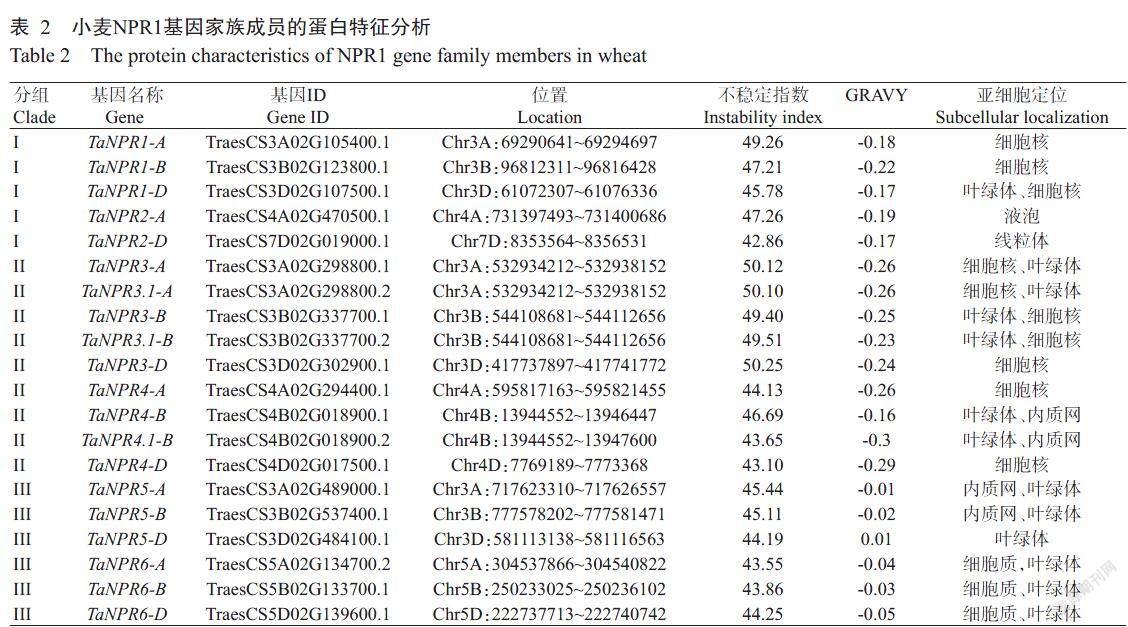
以8个拟南芥NPR1家族蛋白序列为参考序列,在小麦基因组数据库进行BLASTp比对分析,并结合Pfam和SMART网站去除不具有BTB/POZ和Ank保守结构域的序列,共鉴定获得20个小麦NPR1基因家族成员(其中包含3个可变剪接体:TaNPR3.1-A、TaNPR3.1-B、TaNPR4.1-B),并根据其与拟南芥NPR1基因家族的进化关系进行分组及命名(表2)。20个小麦NPR1家族蛋白(TaNPR1s)的不稳定指数均大于40,为不稳定蛋白;平均总亲水性值(GRAVY)均为负值(除TaNPR5-D为正值外),为亲水蛋白;主要分布于细胞核内,在叶绿体、线粒体、内质网和细胞质等部位也有分布。
2. 2 TaNPR1s蛋白结构预测结果
由表3可知,TaNPR1s蛋白二级结构由α-螺旋、延伸链、β-转角和无规卷曲4种元件组成,以α-螺旋和无规则卷曲为主,其中TaNPR1-A、TaNPR1-B、TaNPR1-D、TaNPR2-A、TaNPR2-D、TaNPR3-A、TaNPR3-B、TaNPR3-D、TaNPR4-A、TaNPR4-B和TaNPR4-D蛋白以α-螺旋比例最大,TaNPR5-A、TaNPR5-B、TaNPR5-D、TaNPR6-A、TaNPR6-B和TaNPR6-D蛋白中α-螺旋和无规卷曲所占比例较相近。此外,TaNPR5-A、TaNPR5-B、TaNPR5-D、TaNPR6-A、TaNPR6-B和TaNPR6-D蛋白结构不含NPR1-like C-terminal结构域,与该家族其他蛋白差异较大。
利用SWISS-MODEL构建小麦NPR1蛋白的三级结构模型,共有10种,如表3所示。TaNPR1-A、TaNPR1-B、TaNPR1-D、TaNPR2-A和TaNPR2-D蛋白的二级结构相似,但三级结构差异较大,除TaNPR1-B和TaNPR1-D对应的结构模型相同外,其余蛋白对应的结构模型均不相同;TaNPR3-A、TaNPR3.1-A、TaNPR4-A、TaNPR4.1-B和TaNPR4-D蛋白的二级结构相似,对应结构模型均为5y4d.1。5y4d.1三维结构在I和II组均有分布,其原因可能是与I和II组的遗传关系较近(Liu et al.,2019)。第III组内除TaNPR6-A和TaNPR6-B蛋白三级结构差异较大外,其余蛋白三级结构相似(图1)。TaNPR3-A和TaNPR3-B蛋白分别与其对应的可变剪接体TaNPR3.1-A和TaNPR3.1-B三级结构类似,而TaNPR4-B蛋白与其对应的可变剪接体TaNPR4.1-B三级结构差异相对较大。
2. 3 TaNPR1s蛋白互作网络构建
为了更好地理解基因功能,使用STRING在线数据库构建TaNPR1s蛋白的互作网络(图2)。所有TaNPR1s蛋白均可与转录因子HBP-1b及未知蛋白A、B、C、D发生相互作用。通过Pfam检测发现,这5种蛋白均含有bZIP结构域(含TGACG基序)和种子休眠特异基因结构域(DOG1)。TaNPR1可能通过与这两个结构域发生相互作用从而发挥其功能。
2. 4 TaNPR1s基因在不同组织中的表达分析结果
从NCBI下载与中国春小麦籽粒、穗、叶、茎和根5个组织在生长发育过程中的RNA-Seq原始数据(SBA编号:PRJEB25639),对TaNPR1s基因在小麦生长发育过程中表达水平进行分析。在20个TaNPR1s基因中,大部分基因在小麦生长发育过程中均有表达,TPM值为1.00~31.32(图3)。按照在不同组织的表达情况,TaNPR1s基因可分为三大类:第一类在多个组织中表达,包括TaNPR3.1-A、TaNPR3-B、TaNPR3-D、TaNPR4-A、TaNPR4-B、TaNPR4-D和TaNPR4.1-B共7个基因;第二类仅在特定的组织或发育阶段表达,包括TaNPR1-A、TaNPR1-B、TaNPR1-D、TaNPR2-A、TaNPR2-D、TaNPR5-A、TaNPR5-B、TaNPR5-D、TaNPR6-A、TaNPR6-B和TaNPR6-D共11个基因;第三类在不同组织发育阶段均低表达或不表达,包括TaNPR3-A和TaNPR3.1-B。以第二类中的TaNPR5-B基因为例,利用BAR在线软件绘制其基因表达的电子荧光象形图(图4),该图直观显示了TaNPR5-B基因在小麦早、中、晚3个生长发育阶段在不同组织的表达情况。由上述结果推测小麦NPR1基因家族成员在小麦不同组织生长发育过程中发挥重要作用。
2. 5 TaNPR1s基因在不同胁迫条件下的表达分析结果
从NCBI下载与生物胁迫(SBA编号:条锈菌PRJEB12497;禾谷镰刀菌PRJEB12358)和非生物胁迫(SBA编号:干旱PRJNA306536;高温─干旱共处理PRJNA257938)相关的RNA-Seq原始数据,对TaNPR1s基因在不同逆境胁迫下的表达模式进行分析,结果图5所示。TaNPR1s基因在不同逆境胁迫条件下表达模式不同。TaNPR1-A、TaNPR1-B、TaNPR1-D和TaNPR4.1-B基因在接种条锈菌后1 d时表达量上升,而后表达量下降;TaNPR3.1-A和TaNPR3-D基因在接种后3 d时表达量下降,而在5 d时表达量上升。禾谷镰刀菌侵染后,TaNPR1-A、TaNPR1-B、TaNPR1-D、TaNPR5-A、TaNPR5-B和TaNPR6-B基因的表达量总体上呈上升趋势,TaNPR4-B、TaNPR4.1-B和TaNPR5-D基因的表达量总体呈下降趋势。在干旱胁迫处理条件下,TaNPR1-A、TaNPR1-B、TaNPR1-D、TaNPR4.1-B和TaNPR4-D基因表达量下降。此外,TaNPR1-A、TaNPR1-B、TaNPR1-D、TaNPR4.1-B和TaNPR4-D基因在高温或高温—干旱共处理条件下表达量变化不显著,而TaNPR1-A、TaNPR1-B和TaNPR1-D基因在干旱处理1 h时表达量上升,随后下降,TaNPR4.1-B、TaNPR4-A和TaNPR4-D基因在干旱处理6 h时表达量下降。TaNPR3-B基因在干旱处理条件下表达量下降,在高温或高温—干旱共处理条件下表达量上升。TaNPR4-B干旱处理条件下表达量下降,在高温或高温—干旱共处理条件下表达量先上升后下降。综上所述,TaNPR1s基因参与逆境环境的响应及信号转导,且不同NPR1基因在不同胁迫下基因的表达量变化不同。
随机挑选8个TaNPR1s基因,利用qRT-PCR检测其在生物胁迫(禾谷镰刀菌和白粉病菌)下的表达情况,结果如图6-A和图6-B所示。与对照相比,TaNPR2-D、TaNPR3.1-A和TaNPR3-D基因在禾谷镰刀菌胁迫下的情况下相对表达量降低,而在白粉病菌胁迫下相对表达量升高;TaNPR3-B和TaNPR3.1-B基因在兩种菌胁迫下相对表达量均升高,TaNPR2-A和TaNPR4-A基因在两种菌的胁迫下相对表达量均下降。
利用qRT-PCR检测上述随机挑选的8个TaNPR1s基因在非生物胁迫(冷、热、NaCl、SA、MeJA和ABA)下的表达情况,结果如图6-C~图6-H所示。TaNPR1s基因对6种非生物胁迫处理均有响应,但表达模式存在明显差异。与对照相比,在冷胁迫下,除TaNPR3-B和TaNPR3.1-A基因的相对表达量上升外,其余基因相对表达量均下降;在热和SA胁迫下,所有基因的相对表达量均下降;在NaCl胁迫下,TaNPR2-A、TaNPR2-D和TaNPR3.1-B基因的相对表达量上升,其余基因相对表达量下降;在MeJA胁迫下,除TaNPR2-A基因外,其他基因的相对表达量下降;在ABA胁迫下,除TaNPR3-A基因外,其余基因的相对表达量下降。
3 讨论
NPR1是植物SAR及PRs基因表达的激活子,也參与SA与JA/乙烯信号途径的相互作用,是调节植物整体抗病性的重要作用因子,参与植物多种抗性代谢通路,在植物生长发育和抗病性研究中具有重要作用(Cao et al.,1997),因而植物NPR1基因的研究受到国内外学者的普遍关注。本研究在Liu等(2019)对于NPR1基因系统分类分析的基础上,对小麦NPR1家族蛋白的理化性质、结构预测、互作网络及在不同组织和不同逆境胁迫下的表达情况进行系统分析,初步揭示了小麦NPR1基因家族成员的结构和功能,为该基因家族在小麦遗传改良中的应用提供了理论参考。前人研究发现,NPR1蛋白的BTB结构域与TGA2的抑制区结合会导致TGA2抑制PRs基因表达的功能缺失,从而提升植物抗病性,可见,NPR1典型特征结构域对其功能发挥极为重要(Rochon et al.,2006;Boyle et al.,2009;李微巍,2014);NPR1蛋白的NPR1-like C-terminal结构域含有核定位信号,对定位于细胞核内的NPR1参与转录共激活发挥重要作用(Kinkema et al.,2000)。本研究通过构建蛋白互作网络发现,所有TaNPR1s蛋白均与转录因子HBP-1b及未知蛋白A、B、C、D发生相互作用,经Pfam检测发现这5种蛋白均含有bZIP结构域和DOG1结构域,这两类结构域均属于TGA转录因子。在细胞核内,NPR1蛋白能与TGA亚家族成员(含有TGACG基序,属于bZIP转录因子家族)结合,这些转录因子参与SAR反应,在SA的诱导下能激活下游防卫基因PR1的表达(Zhang et al.,1999;Fan and Dong,2002)。而DOG1结构域可以通过结合ABA信号传递的负调控因子,增加对ABA的敏感性从而引起种子休眠,在种子成熟和发育过程中发挥作用(宋松泉等,2020)。说明TaNPR1蛋白可能通过与bZIP或DOG1结构域结合,进而在生物和非生物胁迫过程中发挥功能。
本研究发现,部分TaNPR1s基因(TaNPR3.1-A、TaNPR3-B、TaNPR3-D、TaNPR4.1-B和TaNPR4-D)在不同组织中均有较高的相对表达量,即使在不同胁迫下,这些基因仍保持较高的相对表达量,表明这些基因在小麦不同生长阶段发挥重要的调控作用,可能直接或间接参与小麦的生长发育过程和形态建成。此外,本研究还发现,尽管TaNPR1s基因在不同生物胁迫和非生物胁迫下的相对表达量不同,但大多数TaNPR1s基因的相对表达量明显上调或下调,表明这些基因参与逆境环境的响应及信号转导(Ma and Bohnert,2007),且TaNPR3.1-A、TaNPR3-B和TaNPR4.1-B基因在不同发育时期均具有较高的相对表达量,但其对应的可变剪接体TaNPR3-A、TaNPR3.1-B和TaNPR4-B基因的相对表达量较低。可变剪切体可使同一个基因翻译成不同的蛋白,进而表现出不同的功能,可变剪接是基因在转录过程中普遍存在的现象,是导致蛋白质功能多样性的重要原因之一(Wang et al.,2015)。小麦的3个NPR1基因与其对应的可变剪切体在逆境胁迫下表达情况不同,说明可变剪切增加了小麦NPR1家族的转录本,丰富了其蛋白的遗传多样性。NPR1介导的逆境胁迫下植物信号通路非常复杂,今后应进一步比较TaNPR1s基因可变剪切体之间的差异,并探究其具体作用机制,对挖掘NPR1基因在响应逆境胁迫中的关键位点具有重大意义。
4 结论
小麦NPR1基因家族成员在不同组织生长发育过程和生物和非生物胁迫响应中发挥重要调控作用,且基因的可变剪接体也表现出不同组织表达特性,丰富了NPR1s蛋白功能。TaNPR1s蛋白可能通过与bZIP和DOG1结构域结合发挥其生物学功能。
参考文献:
蔡韡韡,曾志芳,魏春,杨华丽,佘文琴,陈桂信. 2019. ?NPR1同源基因全长cDNA的分离与表达分析[J]. 园艺学报,46(3):567-576. [Cai W W,Zeng Z F,Wei C,Yang H L,She W Q,Chen G X. 2019. Isolation and expression analysis of full-length cDNA of NPR1 homologous genes in Nai(Prunus salicina var. cordata)[J]. Acta Horticulturae Sinica,46(3):567-576.] doi:10.16420/j.issn.0513-353x.2019-0030.
陈文烨,杨帆,刘永伟,董福双,赵和,柴建芳,吕孟雨,周硕. 2020. 小麦TaNAC-B072基因的克隆和表达分析[J]. 江西农业学报,32(8):1-7. [Chen W Y,Yang F,Liu Y W,Dong F S,Zhao H,Chai J F,Lü M Y,Zhou S. 2020 Cloning and expression analysis of TaNAC-B072 in wheat[J]. Acta Agriculturae Jiangxi,32(8):1-7.] doi:10.19386/ j.cnki.jxnyxb.2020.08.01.
韩永光,马利刚,赵乐,冯卫生,郑晓珂. 2018. 植物抗性基因NPR1研究进展[J]. 安徽农业科学,46(26):18-20. [Han Y G,Ma L G,Zhao L,Feng W S,Zheng X K. 2018. Research progress on resistance gene NPR1 in plants[J]. Journal of Anhui Agricultural Sciences,46(26):18-20.] doi:10.13989/j.cnki.0517-6611.2018.26.006.
焦鹏. 2016. 苹果NPR1基因家族的全基因组鉴定与表达分析[D]. 杨凌:西北农林科技大学. [Jiao P. 2016. Genome-wide identification and expression analysis of NPR1 gene family in apple[D]. Yangling:Northwest A & F University.]
李微巍. 2014. 楊树NPR-like基因家族抗病相关基因的鉴定及功能研究[D]. 合肥:安徽农业大学. [Li W W. 2014. Identification of disease-resistance cancer-critical genes and functional research of NPR-Like family in poplar[D]. Hefei:Anhui Agricultural University.]
任陪娣,谢碧玉,陈翠,贾彩红,王静毅,张建斌. 2019. 香蕉 NPR1基因家族的鉴定及在枯萎病菌胁迫下表达分析[J]. 植物遗传资源学报,20(6):1621-1629. [Ren P D,Xie B Y,Chen C,Jia C H,Wang J Y,Zhang J B. 2019. Genome-wide survey of the NPR1 gene family in banana(Musa spp.) and expression pattern analysis upon infection with Foc TR4[J]. Journal of Plant Genetic Resources,20(6):1621-1629.] doi:10.13430/j.cnki.jpgr.20190416 002.
宋松泉,刘军,徐恒恒,刘旭,黄荟. 2020. 脱落酸代谢与信号传递及其调控种子休眠与萌发的分子机制[J]. 中国农业科学,53(5):857-873. [Song S Q,Liu J,Xu H H,Liu X,Huang H. 2020. ABA metabolism and signaling and their molecular mechanism regulating seed dormancy and germination[J]. Scientia Agricultura Sinica,53(5):857-873.] doi:10.3864/j.issn.0578-1752.2020.05.001.
郑世仲,王培育,张春柳,江胜滔,江金兰,颜沛沛,叶炜,赖钟雄. 2020. 文心兰NPR1基因的克隆与诱导抗性过程中的表达分析[J]. 福建农业学报,35(2):140-149. [Zheng S Z,Wang P Y,Zhang C L,Jiang S T,Jiang J L,Yan P P,Ye W,Lai Z X. 2020. NPR1 Cloning and expression du-ring development of induced systemic resistance in Oncidium hybridum[J]. Fujian Journal of Agricultural Scien-ces,35(2):140-149.] doi:10.19303/j.issn.1008-0384.2020. 02.004.
Altschul S F,Madden T L,Sch?ffer A A,Zhang J,Zhang Z,Miller W,Lipman D J. 1997. Gapped BLAST and PSI-BLAST:A new generation of protein database search programs[J]. Nucleic Acids Research,25(17):3389-3402. doi:10.1093/nar/25.17.3389.
Aravind L,Koonin E V. 1999. Fold prediction and evolutiona-ry analysis of the POZ domain:Structural and evolutio-nary relationship with the potassium channel tetramerization domain[J]. Journal of Molecular Biology,285(4):1353-1361. doi:10.1006/jmbi.1998.2394.
Backer R,Naidoo S,Berg N. 2019. The NONEXPRESSOR OF PATHOGENESIS-RELATED GENES 1(NPR1) and related family:mechanistic insights in plant disease resistance[J]. Frontiers in Plant Science,10:102. doi:10.3389/fpls. 2019.00102.
Boyle P,Su E L,Rochon A,Shearer H L,Murmu J,Chu J Y,Fobert P R,Després C. 2009. The BTB/POZ domain of the Arabidopsis disease resistance protein NPR1 interacts with the repression domain of TGA2 to negate its function[J]. The Plant Cell,21(11):3700-3713. doi:10.1105/tpc.109.069971.
Cao H,Bowling S A,Gordon A S,Dong X. 1994. Characteri-zation of an Arabidopsis mutant that is nonresponsive to inducers of systemic acquired resistance[J]. The Plant Cell,6(11):1583. doi:10.1105/tpc.6.11.1583.
Cao H,Glazebrook J,Clarke J D,Volko S,Dong X. 1997. The Arabidopsis NPR1 gene that controls systemic acquired resistance encodes a novel protein containing ankyrin repeats[J]. The Cell,88(1):57-63. doi:10.1016/S0092-8674(00)81858-9.
Castelló M J,Medina-puche L,Lamilla J,Tornero P. 2018. NPR1 paralogs of Arabidopsis and their role in salicylic acid perception[J]. PLoS One,13(12):e0209835. doi:10. 1371/journal.pone.0209835.
Fan W H,Dong X N. 2002. In vivo interaction between NPR1 and transcription factor TGA2 leads to salicylic acid-mediated gene activation in Arabidopsis[J]. The Plant Cell,14(6):1377-1389. doi:10.1105/tpc.001628.
Finn R D,Mistry J,Schuster-b?ckler B,Griffiths-jones S,Hollich V,Lassmann T,Moxon S,Marshall M,Khanna A,Durbin R,Eddy S R,Sonnhammer E L L,Bateman A. 2006. Pfam:Clans,web tools and services[J]. Nucleic Acids Research,34(S1):247-251. doi:10.1093/nar/gkj149.
Hepworth S R,Zhang Y,Mckim S,Li X,Haughn G W. 2005. BLADE-ON-PETIOLE-dependent signaling controls leaf and floral patterning in Arabidopsis[J]. The Plant Cell,17(5):1434-1448. doi:10.1105/tpc.104.030536.
Horton P,Park K J,Obayashi T,Fujita N,Harada H,Adams-collier C J,Nakai K. 2007. WoLF PSORT:Protein loca-lization predictor[J]. Nucleic Acids Research,35(S2):585-587. doi:10.1093/nar/gkm259.
Initiative T A G. 2000. Analysis of the genome sequence of the fowering plant Arabidopsis thaliana[J]. Nature,408(6814):796-815. doi:10.1038/35048692.
Jiang W Q,Yang L,He Y Q,Zhang H T,Li W,Chen H G,Ma D F,Yin J L. 2019. Genome-wide identification and transcriptional expression analysis of superoxide dismutase(SOD) family in wheat[J]. PeerJ,7(2):e8062. doi:10.7287/peerj.preprints.27402.
Jones J D G,Dangl J L. 2006. The plant immune system[J]. Nature,444(7117):323-329. doi:10.1038/nature05286.
Kinkema M,Fan W,Dong X. 2000. Nuclear localization of NPR1 is required for activation of PR gene expression[J]. The Plant Cell,12(12):2339-2350. doi:10.1105/tpc. 12.12.2339.
Kogel K,Langen G. 2005. Induced disease resistance and gene expression in cereals[J]. Cellular Microbiology,7(11):1555-1564. doi:10.1111/j.1462-5822.2005.00592.x.
Liu X,Liu Z G,Niu X H,Xu Q,Yang L. 2019. Genome-wide identification and analysis of the NPR1-Like gene family in bread wheat and its relatives[J]. International Journal of Molecular Sciences,20(23):5974. doi:10.3390/ ijms20235974.
Liu Y,Schiff M,Marathe R,Dinesh-kumar S P. 2002. Tobacco Rar1,EDS1 and NPR1/NIM1 like genes are required for N-mediated resistance to tobacco mosaic virus[J]. The Plant Journal,30(4):415-429. doi:10.1046/j.1365-313x. 2002.01297.x.
Ma S,Bohnert H J. 2007. Integration of Arabidopsis thaliana stress-related transcript profiles,promoter structures,and cell-specific expression[J]. Genome Biology,8(4):R49. doi:10.1186/gb-2007-8-4-r49.
Malnoy M,Jin Q,Borejsza-wysocka E E,He S Y,Aldwinckle H S. 2007. Overexpression of the apple MpNPR1 gene confers increased disease resistance in Malus x domestica[J]. Molecular Plant-Microbe Interactions,20(12):1568-1580. doi:10.1094/MPMI-20-12-1568.
Mhamdi A. 2019. NPR1 has everything under control[J]. Plant Physiology,181(1):6-7. doi:10.1104/pp.19.00890.
Mikael N,Mattias H,Ove N. 2005. The BLADE ON PETIOLE genes act redundantly to control the growth and development of lateral organs[J]. Development,132(9):2203-2213. doi:10.1242/dev.01815.
Mulder N,Apweiler R. 2007. InterPro and InterProScan:Tools for protein sequence classification and comparison[J]. Methods in Molecular Biology,396:59-70. doi:10. 1007/978-1-59745-515-2_5.
Olate E,Jiménez-gómez J M,Holuigue L,Salinas J. 2018. NPR1 mediates a novel regulatory pathway in cold acclimation by interacting with HSFA1 factors[J]. Nature Plants,4(10):811-23. doi:10.1038/s41477-018-0254-2.
Peraza-echeverria S,Santamaría J M,Fuentes G,Menéndez-cerón M,Vallejo-reyna M,Herrera-valencia V A. 2012. The NPR1 family of transcription cofactors in papaya:Insights into its structure,phylogeny and expression[J]. Genes & Genomics,34(4):379-390. doi:10.1007/s13258- 011-0218-7.
Pieterse C M J,Van Wees S C M,Van Pelt J A,Knoester M,Laan R,Gerrits H,Weisbeek P J,Van Loon L C. 1998. A novel signaling pathway controlling induced systemic resistance in Arabidopsis[J]. The Plant Cell,10(9):1571-1580. doi:10.1105/tpc.10.9.1571.
Rochon A,Boyle P,Wignes T,Fobert P R,Despres C. 2006. The coactivator function of Arabidopsis NPR1 requires the core of its BTB/POZ domain and the oxidation of C-terminal cysteines[J]. The Plant Cell,18(12):3670-3685. doi:10.1105/tpc.106.046953.
Schwede T,Kopp J,Guex N,Peitsch M C. 2003. SWISS-MODEL:An automated protein homology-modeling ser-ver[J]. Nucleic Acids Research,31(13):3381-3385. doi:10.1093/nar/gkg520.
Silva K J P,Brunings A,Peres N A,Mou Z,Folta K M. 2015. The Arabidopsis NPR1 gene confers broad-spectrum disease resistance in strawberry[J]. Transgenic Research,24(4):693-704. doi:10.1007/s11248-015-9869-5.
Spoel S H,Koornneef A,Claessens S M C,Korzelius J P,Van Pelt J A,Mueller M J,Buchala A J,Métraux J,Brown R,Kazan K,Van Loon L C,Dong X,Pieterse C. 2003. NPR1 modulates cross-talk between salicylate- and jasmonate-dependent defense pathways through a novel function in the cytosol[J]. The Plant Cell,15(3):760-770. doi:10.1105/tpc.009159.
Szklarczyk D,Gable A L,Lyon D,Junge A,Wyder S,Huerta-cepas J,Simonovic M,Doncheva N T,Morris J H,Bork P,Jensen L J,Mering C. 2018. STRING v11:Protein-protein association networks with increased coverage,supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Research,47(D1):D607-D613. doi:10.1093/nar/gky1131.
Tripathi D,Raikhy G,Kumar D. 2019. Chemical elicitors of systemic acquired resistance—Salicylic acid and its functional analogs[J]. Current Plant Biology,17:48-59. doi:10.1016/j.cpb.2019.03.002.
Wang X D,Bi W S,Gao J,Yu X M,Wang H Y,Liu D Q. 2018. Systemic acquired resistance,NPR1,and pathoge-nesis-related genes in wheat and barley[J]. Journal of Integrative Agriculture,17(11):60345-60347. doi:10.1016/S2095-3119(17)61852-5.
Wang X D,Yang B J,Li K,Kang Z S,Cantu D,Dubcovsky J. 2016. A conserved Puccinia striiformis protein intera-cts with wheat NPR1 and reduces induction of pathogenesis-related genes in response to pathogens[J]. Molecular Plant-Microbe Interactions,29(12):977-989. doi:10.1094/MPMI-10-16-0207-R.
Wang Y,Liu J,Huang B O,Xu Y M,Li J,Huang L F,Lin J,Zhang J,Min Q H,Yang W M,Wang X Z. 2015. Mechanism of alternative splicing and its regulation[J]. Biome-dical Reports,3(2):152-158. doi:10.3892/br.2014.407.
Wang Z,Ma L Y,Li X,Zhao F Y,Sarwar R,Cao J,Li Y L,Ding L N,Zhu K M,Yang Y H,Tan X L. 2020. Genome?wide identification of the NPR1 like gene family in Brassica napus and functional characterization of BnaNPR1 in resistance to Sclerotinia sclerotiorum[J]. Plant Cell Reports,39:709-722. doi:10.1007/s00299-020-02525-z.
Wilkins M R,Gasteiger E,Bairoch A,Sanchez J C,Williams K L,Appel R D. 1999. Protein identification and analysis tools in the ExPASy Server[J]. Methods in Molecular Biology,112(112):531-552. doi:10.1385/1-59259-584-7:531.
Yuan Y X,Zhong S H,Li Q,Zhu Z R,Lou Y G,Wang L Y,Wang J J,Wang M Y,Li Q L,Yang D L,He Z H. 2007. Functional analysis of rice NPR1-like genes reveals that OsNPR1/NH1 is the rice orthologue conferring disease resistance with enhanced herbivore susceptibility[J]. Plant Biotechnology Journal,5(2):313-324. doi:10.1111/j.1467- 7652.2007.00243.x.
Zhang Y L,Cheng Y T,Qu N,Zhao Q G,Bi D L,Li X. 2006. Negative regulation of defense responses in Arabidopsis by two NPR1 paralogs[J]. The Plant Journal,48(5):647-656. doi:10.1111/j.1365-313X.2006.02903.x.
Zhang Y L,Fan W H,Kinkema M,Li X,Dong X N. 1999. Interaction of NPR1 with basic leucine zipper protein transcription factors that bind sequences required for salicylic acid induction of the PR-1 gene[J]. Proceedings of the National Academy of Sciences,96(11):6523-6528. doi:10.1073/pnas.96.11.6523.
Zhou J,Trifa Y,Silva H,Pontier D,Lam E,Jyoti S,Klessig D F,Zhou J M,Shah J. 2000. NPR1 differentially interacts with members of the TGA/OBF family of transcription factors that bind an element of the PR-1 gene required for induction by salicylic acid[J]. Molecular Plant-Microbe Interactions,13(2):191-202. doi:10.1016/S1369-5266(00)80027-8.
(責任编辑 陈 燕)
