凡纳滨对虾转录组测序分析及肌肉生长发育相关基因的筛选
2021-09-13杨春玲,陈慧芳,彭敏,李强勇,曾地刚,刘青云,赵永贞,陈晓汉,陈秀荔
杨春玲,陈慧芳,彭敏,李强勇,曾地刚,刘青云,赵永贞,陈晓汉,陈秀荔


摘要:【目的】篩选出凡纳滨对虾生长发育的功能基因及代谢调控网络,揭示其生长发育的分子机制,为后续开展凡纳滨对虾分子生物学研究提供宝贵的基因数据来源。【方法】以快速生长群体和慢速生长群体的凡纳滨对虾肌肉组织为研究材料,通过Illumina HiSeqTM2500平台对构建的cDNA文库进行高通量测序分析,以StringTie进行拼接组装后利用DESeq2筛选差异表达基因,并基于KOG、GO、nr、COG、Swiss-Pro、KEGG和Pfam等数据库进行差异表达基因功能注释分析。【结果】经拼接组装共获得53458844条Clean reads,各样品Clean reads的Q30均在93.00%以上;Illumina测序获得的Clean reads与参考基因组的比对效率在87.75%~87.80%,说明转录组测序数据真实可靠。采用SnpEff进行SNP/InDel变异注释分析,结果显示,SNP位点中以A>G、G>A、C>T和T>C等4种类型的数量较多(14950~21562个),InDel位点则以SYNONYMOUS_CODING的数量最多(33630个)。使用StringTie对Mapped reads(比对到参考基因组的Reads)进行拼接,共发掘到4607个新基因,分别输入COG、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG和nr数据库中进行序列比对,最终发现共有1098个新基因被注释,以被nr数据库注释的新基因数量最多(1077个),而被COG数据库注释的新基因数量最少(416个)。基于Q-value<0.05且Fold Change>2的筛选条件,共获得1408个差异表达基因(661个为显著上调表达基因,747个为显著下调表达基因);1408个差异表达基因被注释到53个GO功能条目中,其中,22条被注释到生物学过程(Biological process),16条被注释到细胞组分(Cellular component),15条被注释到分子功能(Molecular function);KEGG信号通路富集分析发现3条重要的信号通路,分别是溶酶体通路(Lysosome)、氨基糖和核苷酸糖新陈代谢(Amino sugar and nucleotide sugar metabolism)及鞘脂类代谢(Sphingolipid metabolism)。在溶酶体通路中,CTSL基因、Nramp基因、MyoG基因和Myf5基因在凡纳滨对虾快速生长群体肌肉组织中呈上调表达,而Trypsin基因呈下调表达。【结论】通过转录组测序分析从快速生长群体和慢速生长群体的凡纳滨对虾肌肉组织中筛选出1408个差异表达基因(661个为显著上调表达基因,747个为显著下调表达基因),主要富集在溶酶体、氨基糖和核苷酸糖新陈代谢及鞘脂类代谢等通路上,在对虾肌肉生长发育过程中发挥重要作用。
关键词: 凡纳滨对虾;差异表达基因;肌肉;生长发育;转录组测序
中图分类号: S945.49 文献标志码: A 文章编号:2095-1191(2021)09-2319-10
Transcriptome sequencing and screening of genes related to muscle growth and development in Litopenaeus vannamei
YANG Chun-ling1, CHEN Hui-fang2, PENG Min1, LI Qiang-yong1, ZENG Di-gang1,
LIU Qing-yun1, ZHAO Yong-zhen1, CHEN Xiao-han1, CHEN Xiu-li1*
(1Guangxi Academy of Fishery Sciences/Guangxi Key Laboratory of Aquatic Genetic Breeding and Healthy Aquaculture, Nanning 530021, China; 2College of Animal Science and Technology, Guangxi University, Nanning 530004, China)
Abstract:【Objective】To screen out the functional genes and metabolic regulation network of growth and development in Litopenaeus vannamei, reveal the molecular mechanism of its growth and development, and provide a valuable source of genetic data for the subsequent molecular biology research of L. vannamei. 【Method】The muscle tissue of the fast-growing group and the slow-growing group of L. vannamei was used as the research material, high-throughput sequencing analysis was performed on the cDNA library through the Illumina HiSeqTM2500 platform, and the DESeq2 was used to screen differentially expressed genes after assembly using StringTie, and the functional annotation of differentially expressed genes was performed based on KOG, GO, nr, COG, Swiss-Pro, KEGG and Pfam databases. 【Result】A total of 53458844 Clean reads with Q30 more than 93.00% were obtained after splicing assembly. The comparison efficiency of Clean reads obtained by Illumina sequencing and the reference genome was 87.75% to 87.80%, indicating that the transcriptome sequencing data were true and reliable. SNPEff was used to annotate SNP and InDel variation, results showed that the number of 4 types of SNP loci, A>G, G>A, C>T, and T>C, was large(14950-21562), and the number of synonymous_coding points of InDel loci was the largest (33630). Using StringTie software to splice Mapped Read (Reads that were aligned to the reference genome), a total of 4607 new genes were discovered. The genes were input into COG, GO, KEGG, KOG, Pfam, Swiss-Prot, eggNOG and nr databases for sequence alignment. Finally, it was found that a total of 1098 new genes were annotated, with the largest number of new genes annotated by the nr database(1077), and the smallest number of new genes annotated by the COG database(416). Based on the screening conditions of Q-value<0.05 and Fold Change>2, a total of 1408 differential expression genes were detected, in which 661 genes were significantly up-regulated and 747 genes were significantly down-regulated. A total of 1408 differentially expressed genes were annotated to 53 GO function items, in which 22 items were annotated to biological process, 16 items were annotated to cellular component, 15 items were annotated to molecular function. KEGG signal pathway enrichment analysis revealed three important signal pathways, namely lysosome pathway, amino sugar and nucleotide sugar metabolism pathway and sphingolipid metabolism pathway. In the lysosomal pathway, CTSL gene, Nramp gene, MyoG gene and Myf5 gene were up-regulated in the muscle tissue of fast-growing L. vannamei, while Trypsin gene was down-regulated. 【Conclusion】Through transcriptome sequencing analysis, 1408 differentially expressed genes (661 are significantly up-regulated genes and 747 are significantly down-regulated genes) were screened from the muscle tissues of fast-growing and slow-growing L. vannamei, which are mainly enriched in lysosomes, sphingolipid metabolism, amino sugar and nucleotide sugar metabolism pathways. They play an important role in the growth and development of L. vannamei muscle.
Key words: Litopenaeus vannamei; differentially expressed gene; muscles; growth and development; transcriptome sequencing
Foundation item: Guangxi Key Research and Development Project(Guike AB19245032); Guangxi Innovation Team Building Project of National Modern Agricultural Industrial Technology System(nycytxgxcxtd-14-01); Special Project of Modern Agricultural Industrial Technology System Construction(CARS-48)
0 引言
【研究意义】凡纳滨对虾(Litopenaeus vannamei)又称南美白对虾,隶属于节肢動物门(Arthropoda)甲壳纲(Crustacea)十足目(Decapoda),具有抗逆性强、适盐范围广、肉质鲜美及经济价值高等优点,深受广大养殖户的喜爱(钱昭英,2014;彭敏等,2020)。自20世纪80年代末引进凡纳滨对虾以来,经过20多年的发展,我国已形成自辽宁至广西沿海的巨大养殖带,且近年来逐渐推广到湖南、湖北、江西和新疆等地区,养殖总面积达25.6万ha,年总产量达140万t,占全球对虾总产量的1/3以上,产值达2000多亿元,我国已发展成为凡纳滨对虾养殖第一大国(滕瑜等,2021)。生长性状是对虾养殖的最重要经济性状,直接影响其养殖经济效益,而遗传选育技术是提高动物机体生长性能最有效的措施之一。因此,揭示优良性状形成的分子机理,开展快速生长品系的分子标记辅助选育,是当前凡纳滨对虾遗传育种领域最重要的研究方向。【前人研究进展】在哺乳动物中,肌肉发育调控是研究机体生长发育规律最重要的内容之一,目前针对哺乳动物肌肉生长发育调控的分子机理已有深入研究(Buckingham et al.,2003;Carvajal et al.,2010)。肌细胞的分化与生长不仅受正、负决定因子的双向调控,还依赖于锌指蛋白及生长因子等组织特异因子的微调控(Buckingham et al.,2003;Carvajal and Rigby,2010),其中以bHLH(Basic-helix-loop-helix)为核心结构域的生肌调节因子(Myogenic regulatory factor,MRFs)在肌肉的发生发育过程中起正向调控作用,包括肌分化因子(MyoD)、生肌因子5(Myf5)、肌细胞生成素(MyoG)和生肌调节因子4(MRF4),且在肌肉的发生发育过程中具有时空表达特异性,其调控功能各异但又存在功能补偿(Pownall et al.,2002;Botzenhart et al.,2019)。肌肉生长抑制因子(Myostatin)则是肌肉生长发育过程中最重要的负调控因子(Otto and Patel,2010),在牛(Grobet et al.,1997;Kambadur et al.,1997)和小鼠(McPherron et al.,1997)中缺失或突变可导致其肌肉过度增长;近年来的研究还发现在牛、羊和狗等动物中Myogenin基因突变减弱其活性的同时对肌肉发育产生显著影响(Aiello et al.,2018)。在对虾方面,Lee等(2015)研究表明,dsRNA沉默凡纳滨对虾肌生长抑制素基因(LvMstn)表达后,凡纳滨对虾的生长速度变慢;Kong等(2020)通过分析中国对虾(Fenneropenaeus chinensis)FcMstn基因表达与生长性状的关系,发现FcMstn基因的表达与中国对虾生长性状呈负相关,且雌性个体的FcMstn基因平均表达量显著低于雄性个体,即FcMstn基因对中国对虾的肌肉生长呈负调控作用;Yan等(2020)也研究证实,FcMstn基因作为负调控因子参与肌肉生成过程,通过RNA干扰(RNAi)降低FcMstn基因表达水平的中国对虾显示出更快的生长速度。【本研究切入点】肌肉是对虾机体的主要组成部分,占其体重的60%以上,肌肉生长发育程度是决定对虾生长快慢、体形大小和生长性状优劣的主要因素。在家畜遗传改良工作中,已有研究通过筛查调控肌肉发育基因上的有益突变而选育出生长性能优良的新品种(Smith et al.,2000;Braunschweig,2010),但至今鲜见有关凡纳滨对虾肌肉发育调控机理的研究报道。【拟解决的关键问题】通过对凡纳滨对虾肌肉组织生长性状进行转录组测序分析,对比相关基因组序列,筛选出凡纳滨对虾生长发育的功能基因及代谢调控网络,进一步揭示其生长发育的分子机制,为后续开展凡纳滨对虾分子生物学研究提供宝贵的基因数据来源。
1 材料与方法
1. 1 试验材料
供试凡纳滨对虾由广西水产科学研究院南美白对虾遗传育种中心提供。通过对比凡纳滨对虾群体的体长和体重,分别筛选出快速生长群体30尾、慢速生长群体30尾,取其肌肉组织,液氮速冻后-80 ℃保存备用。
1. 2 试验方法
取适量凡纳滨对虾肌肉组织样本,按照RNA提取试剂盒及miRNA Isolation Kit试剂盒说明进行总RNA提取和分离,通过Qubit 2.0、NanoDrop及Aglient 2100等对总RNA的浓度、纯度和完整性进行检测(李隐侠等,2019;杨和川等,2020),取质量合格的RNA样品进行mRNA富集,并构建cDNA文库,然后采用Qubit 2.0和Agient 2100检测文库插入片段的长度及浓度,并以荧光定量PCR准确定量cDNA文库的有效浓度,最后在Illumina HiSeqTM 2500平台上完成高通量测序分析。
1. 3 数据处理分析
下机数据经质量评估和统计后,获得高质量的测序序列(Clean reads)。采用HISAT2对凡纳滨对虾基因组进行比对分析,将匹配的片段进行随机性检验和插入片段长度评估;利用StringTie对比对上的Clean reads进行组装以获得更完整的转录本,然后进行新基因发掘及表达量、可变剪接和基因结构分析;通过DESeq2分析不同肌肉样品组织中的基因差异表达,并基于KOG、GO、nr、COG、Swiss-Pro、KEGG和Pfam等数据库进行差异表达基因功能注释分析。
2 结果与分析
2. 1 转录组测序分析与组装结果
cDNA文库的Illumina测序数据经测序质量控制,共获得53458844条Clean reads(表1),然后与凡纳滨对虾基因组序列进行比对分析,并利用StringTie对比对上的Clean reads进行组装和定量分析,结果发现快速生长群体和慢速生长群体凡纳滨对虾肌肉样品的Clean reads与参考基因组的比对效率分别为87.75%和87.80%,具有较高的匹配率,基因組G+C含量在49.79%~50.23%,且Q30均高于93.00%,表明Illumina测序数据及参考基因组数据真实可靠,可用于后续研究。
2. 2 SNP/InDel分析注释结果
根据Illumina测序获得的Clean reads与凡纳滨对虾基因组序列的比对分析结果,采用GATK鉴定测序样品与参考基因组间的碱基错配情况,以识别潜在的SNP位点,并利用SnpEff分析注释SNP/InDel变异。统计每个样品SNP位点的数目、转换类型比例、颠换类型比例及杂合型比例,结果(图1)显示,A>G、G>A、C>T和T>C等4种类型的SNP位点数量较多(14950~21562个),而A>C、C>A、A>T、T>A、C>G、G>C、G>T和T>G等8种类型的SNP位点数量相对较少(2982~7326个)。同时统计每个样品InDel位点的数目和注释类型,结果(图2)发现,各种类型的InDel位点数量差异明显,以SYNONYMOUS_CODING的数量最多(33630个),其次是INTERGENIC、INTRON、DOWNSTREAM、UTR_3_PRIME和NON_ SYNONYMOUS_CODING等5种类型(8125~15934个),而INTRAGENIC、UPSTREAM、UTR_5_PRIME、SPLICE_SITE_ACCEPTOR、SPLICE_SITE_DONOR、
SPLICE_SITE_REGION、START_GAINED、START_ LOST、FRAME_SHIFT、CODON_INSERTION、CODON_DELETION、CODON_CHANGE_PLUS_CODON_INSERTION、CODON_CHANGE_PLUS_CODON_DELETION、SYNONYMOUS_CODING、NON_ SYNONYMOUS_CODING、SYNONYMOUS_STOP、 STOP_GAINED和STOP_LOST等类型的数量较少(4~3207个),其他类型的InDel位点数量为16011个。
2. 3 新基因功能注释分析结果
对匹配基因组的Clean reads进行拼接,并搜索参考基因组的注释信息,然后基于所选参考基因组序列,使用StringTie对Mapped reads(比对到参考基因组的Reads)进行拼接,并与原有的基因组注释信息进行注释分析,寻找未被注释的转录区,发掘新转录本和新基因。过滤掉编码肽链过短(少于50个氨基酸残基)或只包含单个外显子的序列,共发掘到4607个未被注释的转录区(新基因)。将发掘到的新基因分别输入COG、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG和nr数据库中进行序列比对,以获得新基因的注释信息。最终发现共有1098个新基因被注释,各数据库注释的新基因数量见表2。其中,以被nr数据库注释的新基因数量最多(1077个),而被COG数据库注释的新基因数量最少(416个)。
2. 4 差异表达基因分析结果
利用DESeq2对具有生物学重复样本进行样品间的基因差异表达分析,以获得表达基因集;同时以EBSeq对没有生物学重复样本进行基因差异表达分析。对凡纳滨对虾快速生长群体和慢速生长群体的转录本进行定量分析,设筛选差异条件为Q-value<0.05且Fold Change>2,共获得1408个差异表达基因,其中,661个为显著上调表达基因,747个为显著下调表达基因,二者差异不明显(图3)。
2. 4. 1 差异表达基因GO功能注释分析结果 通过对差异表达基因进行GO功能注释分析,结果显示1408个差异表达基因被注释到53个GO功能条目中,其中,22条被注释到生物学过程(Biological process),16条被注释到细胞组分(Cellular component),15条被注释到分子功能(Molecular function)。由图4可看出,在生物学过程分类中,差异表达基因主要集中在细胞过程(Cellular process)、代谢过程(Metabolic process)及单一生物过程(Single-organism process)等类别,说明凡纳滨对虾在生长过程中有大量参与调节代谢活动的基因存在;在细胞组分分类中,差异表达基因主要集中在细胞区域(Cell part)、细胞(Cell)和细胞膜(Membrane)等类别,说明细胞膜及其细胞组分在凡纳滨对虾肌肉生长发育过程中发挥重要作用;在分子功能分类中,差异表达基因主要集中在结合(Binding)和催化活性(Catalytic activity)等类别,说明各种酶的催化作用在凡纳滨对虾肌肉生长发育过程中发挥重要作用。
2. 4. 2 差异表达基因KEGG信号通路富集分析结果
在凡纳滨对虾肌肉组织转录组测序中,共有275个差异表达基因富集到49条KEGG信号通路上,可划分为环境信息处理(Environmental information processing)、新陈代谢(Metabolism)、遗传信息处理(Genetic information processing)、细胞过程(Cellular processes)、有机系统(Organismal systems)及人类疾病(Human diseases)等六大类(图5)。其中,以新陈代谢的通路最多,有27条KEGG信号通路,共富集有217个差异表达基因,占78.91%;有机系统和人类疾病的通路最少,均只有1条KEGG信号通路,分别富集有4个和6个差异表达基因。由图6可看出,在KEGG信号通路中富集程度排名前10的通路依次位溶酶体(Lysosome)、氨基糖和核苷酸糖新陈代谢(Amino sugar and nucleotide sugar metabolism)、鞘脂类代谢(Sphingolipid metabolism)、花生四烯酸代谢(Arachidonic acid metabolism)、细胞色素P450异生物质代谢(Matabolism of xenobiotics by cytochrome P450)、淀粉和蔗糖代谢(Starch and sucrose metabolism)、戊糖和葡萄糖醛酸相互转化(Pentose and glucuronate interconversions)、果糖和甘露糖代谢(Fructose and mannose metabolism)、甘氨酸、丝氨酸和苏氨酸代谢(Glycine,serine and threonine metabolism)及甘油脂代谢(Glycerolipid metabolism)。
2. 5 凡纳滨对虾生长相关基因差异表达分析结果
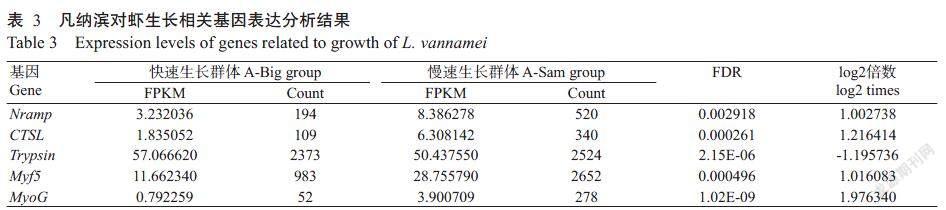
在筛选获得的差异表达基因中,天然抗性相关巨噬蛋白基因(Nramp)、组织蛋白酶L基因(CTSL)、胰蛋白酶基因(Trypsin)、MyoG基因及Myf5基因等已被证实在动物的生长发育过程中发挥重要作用(Pownall et al.,2002;Glenn et al.,2005;Otto and Patel,2010;Peracino et al.,2013)。这5个基因在快速生长群体和慢速生长群体凡纳滨对虾肌肉组织中的转录组测序结果(表3)显示,CTSL基因、Nramp基因、MyoG基因和Myf5基因在凡纳滨对虾快速生长群体肌肉组织中呈上调表达,而Trypsin基因在凡纳滨对虾快速生长群体肌肉组织中呈下调表达。
3 讨论
转录组测序分析是目前对动植物及微生物进行非模式生物研究的常用方法,能有效分析基因在特定条件下的表达情况,挖掘出生物网络调控及其分子机制(罗辉等,2015;李敏等,2019;肖韵铮等,2020)。至今,针对凡纳滨对虾的转录组测序已有较多研究报道。曾地刚等(2013)采用454高通量测序技术对凡纳滨对虾肝胰腺的转录组进行测序,结果获得500177条ESTs,经拼接后获得20225个Unigenes,其长度范围为50~8980 bp,平均507 bp。Lu等(2016)选取耐氨氮组和常规组肝的胰腺组织进行转录组测序分析,最终组装获得78636个Unigenes,并筛选得到136个显著差异表达基因。董丽君等(2019)以凡纳滨对虾低温胁迫组和常温组肝胰腺组织为材料进行转录组测序分析,结果获得50921个Unigenes,设置差异表达基因筛选阀值为Fold Change>2和Q-value<0.05,最终筛选得到243个低温胁迫相关基因,其中89个上调表达、154个下调表达。为了挖掘和利用凡纳滨对虾生长相关基因资源,本研究对生长差异显著的快速生长群体和慢速生长群体肌肉组织样本进行转录组测序和生物信息学分析,Illumina测序数据经测序质量控制后共获得53458844条Clean reads,各样品Clean reads的Q30均在93.00%以上,证明Illumina测序数据真实可靠,即转录组测序结果能满足后续研究的要求,为凡纳滨对虾生长差异基因的筛选打下基础。
凡纳滨对虾基因组数据已经能在互联网上检索获得,将测序获得的数据输入已知数据库中进行比对分析,发现Illumina测序获得的Clean reads与参考基因组的比对效率在87.75%~87.80%,说明比对结果有效,也进一步证实转录组测序数据和基因组数据真实可靠。根据比对结果,进行新基因发掘、基因结构优化分析及可变剪接预测分析,共发掘到4607个新基因;将这些新基因分别输入COG、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG和nr数据库中进行注释分析,最终发现共有1098个新基因被注释,其中注释到nr数據库1077个,eggNOG数据库948个,Pfam数据库917个,KOG数据库714个,Swiss-Prot数据库663个,KEGG数据库551个,GO数据库430个,COG数据库416个。KEGG信号通路富集分析结果显示,有385个差异表达基因富集成环境信息处理、新陈代谢、遗传信息处理、细胞过程、有机系统及人类疾病等六大类。此外,KEGG信号通路富集分析发现3条重要的信号通路,分别是溶酶体、氨基糖和核苷酸糖代谢及鞘脂类代谢。其中,溶酶体主要起消化作用,是细胞自溶、防御及其对某些物质利用等信号转导通路的重要共同交汇通路(Lamming and Bar-Peled,2019);氨基糖和核苷酸糖代谢属于能量代谢通路,影响生物的发育及其免疫等过程(张阳,2018)。本研究通过对比快速生长群体和慢速生长群体凡纳滨对虾肌肉组织中的差异表达基因,发现以溶酶体通路的富集程度最高,说明其在凡纳缤对虾肌肉生长发育过程中发挥重要作用。
本研究通過对快速生长群体和慢速生长群体的凡纳滨对虾肌肉组织进行转录组测序分析,最终筛选获得1408个差异表达基因,其中,661个为显著上调基因,747个为显著下调基因,为揭示凡纳滨对虾生长发育的分子机制及开展生长相关基因的功能研究打下了基础。在溶酶体通路中,发现CTSL基因和Nramp基因在凡纳滨对虾肌肉中的表达量存在显著差异。杨康等(2015)研究发现,在草鱼(Liza haematocheila)的生长过程及免疫反应中,Nramp基因在腮组织、血液、脾脏和肝脏中均呈上调表达。本研究也发现,Nramp基因在凡纳滨对虾肌肉中呈上调表达,可能是该基因参与了肌肉生长发育的免疫反应,且对虾类的生长与蛋白合成及消化密切相关。除了参与经典的肌肉发育信号通路基因(MyoG和Myf5)外,本研究还筛选出一些与蜕皮、肌肉生长和肌肉收缩等相关的基因,说明这些基因对肌肉的生长发育及蜕皮也发挥着至关重要的作用。凡纳滨对虾的生长发育是经过多次蜕皮而实现,蜕皮伴随着旧表皮的脱落、新表皮的形成及肌肉的收缩舒张等生理过程(闫允君等,2020)。朱景花等(2014)研究发现,CTSL基因SNP位点与凡纳滨对虾的生长速度密切相关。此外,Trypsin基因在凡纳滨对虾肌肉生长发育中显著下调表达,可能是由于蜕皮过程需通过影响胰蛋白酶活性变化来实现(JuanCarlos and JulioHumberto,2009),但具体作用机理还需进一步探究。
4 结论
通过转录组测序分析从快速生长群体和慢速生长群体的凡纳滨对虾肌肉组织中筛选出1408个差异表达基因(661个为显著上调表达基因,747个为显著下调表达基因),主要富集在溶酶体、氨基糖和核苷酸糖新陈代谢及鞘脂类代谢等通路上,在对虾肌肉生长发育过程中发挥重要作用。
参考文献:
董丽君,孟宪红,孔杰,罗坤,栾生,史晓丽. 2019. 基于转录组分析筛选凡纳滨对虾低温胁迫下的差异表达基因[J]. 中国水产科学,26(1):163-173. [Dong L J,Meng X H,Kong J,Luo K,Luan S,Shi X L. 2019. Screening of differentially expressed genes related to the cold tolerance in Litopenaeus vannamei based on high-throughput transcriptome sequencing[J]. Journal of Fishery Sciences of China,26(1):163-173.] doi:10.3724/SP.J.1118.2019. 18061.
李敏,郭聪,李玉娟,冯新民,王莹,张健,谈峰. 2019. 旱柳转录组测序及生物学分析[J]. 江苏农业学报,35(2):271-281. [Li M,Guo C,Li Y J,Feng X M,Wang Y,Zhang J,Tan F. 2019. Transcriptome sequencing and biological analysis of willow(Salix matsu-dana)[J]. Jiangsu Journal of Agricultural Sciences,35(2):271-281.] doi:10. 3969/j.issn.1000-4440.2019.02.005.
李隐侠,冯小品,张莉,张俊,钱勇,孟春花,王慧利,仲跻峰,曹少先. 2019. 热应激前后湖羊下丘脑差异表达新基因的筛选与注释[J]. 江苏农业学报,35(2):363-369. [Li Y X,Feng X P,Zhang L,Zhang J,Qian Y,Meng C H,Wang H L,Zhong J F,Cao S X. 2019. Screening and annotation of novel genes differentially expressed in hypothalamus before and after heat stress in Hu sheep[J]. Jiangsu Journal of Agricultural Sciences,35(2):363-369.]doi:10.3969/j.issn.1000-4440.2019.02.017.
罗辉,叶华,肖世俊,郑曙明,王晓清,王志勇. 2015. 转录组学技术在水产动物研究中的运用[J]. 水产学报,39(4):931-940. [Luo H,Ye H,Xiao S J,Zheng S M,Wang X Q,Wang Z Y. 2015. Application of transcriptomics technology to aquatic animal research[J]. Journal of Fishe-ries of China,39(4):931-940.] doi:10.11964/jfc.201411 09541.
彭敏,陈慧芳,李强勇,杨春玲,曾地刚,刘青云,赵永贞,陈晓汉,林勇,陈秀荔. 2020. 凡纳滨对虾连续3个世代选育群体的遗传多样性分析[J]. 南方农业学报,51(6):1442-1450. [Peng M,Chen H F,Li Q Y,Yang C L,Zeng D G,Liu Q Y,Zhao Y Z,Chen X H,Lin Y,Chen X L. 2020. Genetic diversity of three consecutive generations of Litopenaeus vannamei[J]. Journal of Southern Agriculture,51(6):1442-1450.] doi:10.3969/j.issn.2095-1191.2020.06.026.
錢昭英. 2014. 凡纳滨对虾生长性能相关候选基因的分离鉴定、表达规律及调控功能研究[D]. 杨凌:西北农林科技大学. [Qian Z Y. 2014. Isolation,identification,expression and function studies of growth performance candidate genes in Litopenaeus vannamei[D]. Yangling:Northwest A & F University.]
滕瑜,毕国栋,于爱美,王彩理. 2021. 凡纳滨对虾的加工利用和产业发展概述[J]. 食品安全质量检测学报,12(14):5727-5733. [Teng Y,Bi G D,Yu A M,Wang C L. 2021. Overview processing utilization and industrial development of Litopenaeus vannamei[J]. Journal of Food Safety and Quality,12(14):5727-5733.] doi:10.19812/j.cnki.jfsq11-5956/ts.2021.14.032.
肖韵铮,韩世明,秦昭,李春奇. 2020. 滇黄精转录组测序及类黄酮合成相关基因的分析[J]. 河南农业大学学报,54(6):931-940. [Xiao Y Z,Han S M,Qin Z,Li C Q. 2020. Analysis of transcriptome sequencing and related genes of flavonoids biosynthesis from Polygonatum kingianum[J]. Journal of Henan Agricultural University,54(6):931-940.] doi:10.16445/j.cnki.1000-2340.2020.06.004.
闫允君,卢霞,孟宪红,栾生,陈宝龙,孔杰. 2020. 基于转录组分析对中国对虾Myostatin基因调控的肌肉生长相关基因的筛选[J]. 渔业科学进展,42(4):55-63. [Yan Y J,Lu X,Meng X H,Luan S,Chen B L,Kong J. 2020.Screening of genes related to muscle growth under the Myostatin regulation by RNA-seq in Fenneropenaeus chinensis[J]. Progress in Fishery Sciences,42(4):55-63.] doi:10.19663/j.issn2095-9869.20200324002.
杨和川,苏文英,谭一罗,周振玲,秦裕营,李晓. 2020. 玉木耳转录组测序及褐变相关基因的挖掘[J]. 江西农业学报,32(5):7-12. [Yang H C,Su W Y,Tan Y L,Zhou Z L,Qin Y Y,Li X. 2020. Transcriptome sequencing of Auri-cularia cornea and investigating of genes involved in browning[J]. Acta Agriculturae Jiangxi,32(5):7-12.] doi: 10.19386/j.cnki.jxnyxb.2020.05.02.
杨康,王艺雅,刘铁柱,李同明,季相山,王慧. 2015. 草鱼天然抗性相关巨噬蛋白基因的表达特征与蛋白表达[J]. 中国水产科学,22(5):908-915. [Yang K,Wang Y Y,Liu T Z,Li T M,Ji X S,Wang H. 2015. Expression characteristics and protein expression of the natural resistance associated macrophage protein(Nramp) gene in grass carp (Ctenopharyngodon idellus)[J]. Journal of Fishery Scien-ces of China,22(5):908-915.] doi:10.3724/SP.J.1118. 2015.14448.
张阳. 2018. 口虾蛄不同地理群体线粒体DNA控制区和转录组变异研究[D]. 舟山:浙江海洋大学. [Zhang Y. 2018. Genetic variation analysis of different population mantis shrimp Oratosquilla oratoria based on mitochondrial DNA control region and transcriptome[D]. Zhoushan:Zhejiang Ocean University.] doi:10.7666/d.D01425045.
曾地刚,陈秀荔,谢达祥,赵永贞,杨春玲,马宁,李咏梅,陈晓汉. 2013. 基于高通量测序的凡纳滨对虾的转录组分析[J]. 基因组学与应用生物学,32(3):308-313. [Zeng D G,Chen X L,Xie D X,Zhao Y Z,Yang C L,Ma N,Li Y M,Chen X H. 2013. Deep sequencing-based transcriptome analysis of Litopenaeus vannamei[J]. Genomics and Applied Biology,32(3):308-313.] doi:10.3969/gab.032. 000308.
朱景花,李義军,王平,施泓,阮灵伟. 2014. 凡纳滨对虾CTSL基因与生长相关的SNP位点的特征[J]. 应用海洋学学报,33(1):53-59. [Zhu J H,Li Y J,Wang P,Shi H,Ruan L W. 2014. Characters of SNP associated with the growth of CTSL gene in Litopenaeus vannamei[J]. Journal of Applied Oceanography,33(1):53-59.] doi:10.3969/J.ISSN.2095-4972.2014.01.007.
Aiello D,Patel K,Lasagna E. 2018. The myostatin gene:An overview of mechanisms of action and its relevance to livestock animals[J]. Animal Genetics,49(6):505-519. doi:10.1111/age.12696.
Botzenhart U U,Gerlach R,Gredes T,Rentzsch I,Gedrange T,Kunert-Keil C. 2019. Expression rate of myogenic re-gulatory factors and muscle growth factor after botulinum toxin A injection in the right masseter muscle of dystrophin deficient(mdx) mice[J]. Advances in Clincal and Experimental Medicine,28(1):11-18. doi:10.17219/acem/ 76263.
Braunschweig M H. 2010. Mutations in the bovine ABCG2 and the ovine MSTN gene added to the few quantitative trait nucleotides identified in farm animals:A mini-review[J]. Journal of Applied Genetics,51(3):289-297. doi:10.1007/BF03208858.
Buckingham M,Bajard L,Chang T,Daubas P,Hadchouel J,Meilhac S,Montarras D,Rocancourt D,Rrlaix F. 2003. The formation of skeletal muscle:From somite to limb[J]. Journal of Anatomy,202(1):59-68. doi:10.1046/j. 1469-7580.2003.00139.x.
Carvajal J J,Rigby P W J. 2010. Regulation of gene expression in vertebrate skeletal muscle[J]. Experimental Cell Research,316(18):3014-3018. doi:10.1016/j.yexcr.2010. 07.005.
Glenn K L,Grapes L,Suwanasopee T,Harris D L,Li Y,Wilson K,Rothschild M F. 2005. SNP analysis of AMY2 and CTSL genes in Litopenaeus vannamei and Penaeus mono-don shrimp[J]. Animal Genetics,36(3):235-236. doi:10. 1111/j.1365-2052.2005.01274.x.
Grobet L,Martin L J,Poncelet D,Pirottin D,Brouwers B,Riquet J,Schoeberlein A,Dunner S,Ménissier F,Massabanda J,Fries R,Hanset R,Georges M. 1997. A deletion in the bovine myostatin gene causes the double-muscled phenotype in cattle[J]. Nature Genetics,17(1):71-74. doi:10. 1038/ng0997-71.
JuanCarlos S H,JulioHumberto C M. 2009. Activity of trypsin from Litopenaeus vannamei[J]. Aquaculture,290(3-4):190-195. doi:10.1016/j.aquaculture.2009.02.034.
Kambadur R,Sharma M,Smith T P,Bass J J. 1997. Mutations in myostatin (GDF8) in double-muscled Belgian Blue and Piedmontese cattle[J]. Genome Research,7(9):910-916. doi:10.1101/gr.7.9.910.
Kong J,Yan Y J,Lu X,Luan S,Meng X H,Dai P,Chen B L,Cao B X,Qiang G F,Luo K. 2020. Integrative phenoty-pic and gene expression data identify myostatin as a muscle growth inhibitor in Chinese shrimp Fenneropenaeus chinensis[J]. Scientific Reports,10(1):5985. doi:10.1038/ s41598-020-61382-8.
Lamming D W,Bar-Peled L. 2019. Lysosome:The metabolic signaling hub[J]. Traffic,20(1):27-38. doi:10.1111/tra. 12617.
Lee J H,Momani J,Kim Y M,Kang C K,Choi J H,Baek H J,Kim H W. 2014. Effective RNA-silencing strategy of Lv-MSTN/GDF11 gene and its effects on the growth in shrimp,Litopenaeus vannamei[J]. Comparative Bioche-mistry and Physiology. Part B. Biochemistry and Molecular Biology,179:9-16. doi:10.1016/j.cbpb.2014.09.005.
Lu X,Kong J,Luan S,Dai P,Meng X H,Cao B X,Luo K. 2016. Transcriptome analysis of the hepatopancreas in the pacific white shrimp(Litopenaeus vannamei) under acute ammonia stress[J]. PLoS One,11(10):e0164396. doi:10.1371/journal.pone.0164396.
McPherron A C,Lawler A M,Lee S J. 1997. Regulation of skeletal muscle mass in mice by a new TGF-beta superfamily member[J]. Nature,387(6628):83-90. doi:10. 1038/387083a0.
Otto A,Patel K. 2010. Signalling and the control of skeletal muscle size[J]. Experimental Cell Research,316(18):3059-3066. doi:10.1016/j.yexcr.2010.04.009.
Peracino B,Buracco S,Bozzaro S. 2013. The Nramp (Slc11) proteins regulate development,resistance to pathogenic bacteria and iron homeostasis in Dictyostelium discoideum[J]. Journal of Cell Science,126(1):301-311. doi:10.1242/ jcs.116210.
Pownall M E,Gustafsson M K,Emerson C P Jr. 2002. Myogenic regulatory factors and the specification of muscle progenitors in vertebrate embryos[J]. Annual Review of Cell Developmental Biology,18:747-783. doi:10.1146/annurev.cellbio. 18.012502.105758.
Smith J A,Lewis A M,Wiener P,Williams J L. 2000. Gene-tic variation in the bovine myostatin gene in UK beef cattle:Allele frequencies and haplotype analysis in the South Devon[J]. Animal Genetics,31(5):306-309. doi:10. 1046/j.1365-2052. 2000.00521.x.
Yan Y J,Lu X,Kong J,Meng X H,Luan S,Dai P,Chen B L,Cao B X,Luo K. 2020. Molecular characterization of myostatin and its inhibitory function on myogenesis and muscle growth in Chinese Shrimp,Fenneropenaeus chinensis[J]. Gene,758:144986. doi:10.1016/j.gene.2020. 144986.
(責任编辑 兰宗宝)
杨春玲(1979-),副研究员,主要从事水产动物分子遗传育种研究工作。先后主持广西重点研发计划项目“基于基因芯片的南美白对虾分子育种技术研究”及广西直属公益性科研院所基本科研业务费专项2项、广西水产遗传育种与健康养殖重点实验室自主研发项目2项;作为科研骨干参与完成国家级、省部级及地厅级科研项目20余项;参与育成国审水产新品种1个;参与制定国家农业行业标准1项、广西地方标准2项;获得广西科技进步二等奖1项;获得授权发明专利4项、计算机软件著作权10项;参编出版专著5部;在《CryoLetters》《Fish and Shellfish Immunology》《BMC Genomics》《水生态学杂志》《南方农业学报》《动物医学进展》等学术期刊上发表科技论文20余篇。
