GLIS1基因多态性与绵羊产羔数之间的关系研究
2021-08-28刘秋月胡文萍王翔宇张效生张金龙储明星
周 梅,刘秋月,狄 冉,胡文萍,王翔宇,张效生,张金龙,储明星∗
(1中国农业科学院北京畜牧兽医研究所,农业农村部动物遗传育种与繁殖重点实验室,北京100193;2安徽省农业科学院畜牧兽医研究所,猪分子数量遗传学安徽省农业科学院重点实验室,畜禽产品安全工程安徽省重点实验室,合肥230001;3天津市畜牧兽医研究所,天津300381)
产羔数是绵羊最重要的经济性状之一,据报道,它对绵羊繁殖力遗传进展的经济效益贡献率为74%—96%,因此,寻找与产羔数相关的主效基因一直是遗传育种研究者关注的重点[1-3]。
GLIS1来源于转录因子中最大的家族Kruppel样锌指蛋白,是一种在小鼠卵母细胞和受精卵一、二细胞期高表达的蛋白,在小鼠胚胎发育中的表达具有时空性[4]。GLIS1同时具有转录激活和转录抑制活性,能够对胚胎发育的特定阶段进行调控,对于小鼠的胚胎发育尤为重要,同时Gli蛋白的表达或活性又受其上下游BMP和Wnt蛋白家族的成员调控[5]。Takahashi等[6]报道,在体外培养条件下GLIS1基因在卵母细胞以及胚胎发育不同阶段的细胞中表达,且在牛胚胎发育过程中十分关键。王小海等[7]研究发现,GLIS1基因在小鼠的胚胎发育以及成纤维细胞的重编程过程中均发挥重要作用。胚胎发育成功与否对动物的的生殖力会产生直接影响,因此对影响胚胎发育过程的重要因子进行研究很有必要。
前期,本实验室对中国9个地方绵羊品种(分为单羔组和多羔组)共89只绵羊开展了全基因组重测序[8],筛选到绵羊产羔数候选基因GLIS1及相关多态位点。鉴于GLIS1在哺乳动物生殖中的作用还未见报道,本研究对该基因两个单核苷酸多态位点g.27775611 T>C和g.27857114 T>G在不同绵羊群体中的多态性进行检测,并与小尾寒羊产羔数进行关联分析,以探讨GLIS1基因与绵羊产羔数之间是否存在某种内在联系,以期为绵羊高效育种提供充分的理论基础。
1 材料与方法
1.1 样品采集
实验动物共包含760只绵羊,具体信息如表1所示。

表1 绵羊样本具体信息Table 1 Specific information of sheep sample
1.2 SNP位点检测
由北京君诺德生物技术有限公司采用Sequenom MassARRAY®SNP技术[9]对GLIS1基因两个多态性位点g.27775611 T>C和g.27857114 T>G在不同产羔数的绵羊品种中进行基因型检测。分型样品为DNA(样本量20μL,样品质量浓度为40—80 ng∕μL)。
1.3 数据分析
利用SPSS20统计软件对GLIS1两个SNP位点多态性与小尾寒羊各胎次产羔数分别进行差异显著性分析,采用比较均值和一般线性模型单因素方差分析中的最小显著差异(Least significant difference,LSD)法进行分析。使用的相关性分析模型与寸静宇[10]和文禹粱[11]论文中报道的一致。
2 结果与分析
2.1 GLIS1基因多态性分析
通过分型发现,GLIS1基因2个SNP位点在分型的绵羊品种中共存在3种基因型,g.27775611 T>C位点3种基因型分别是TT、CT和CC,g.27857114 T>G位点分别是TT、GT和GG(图1)。
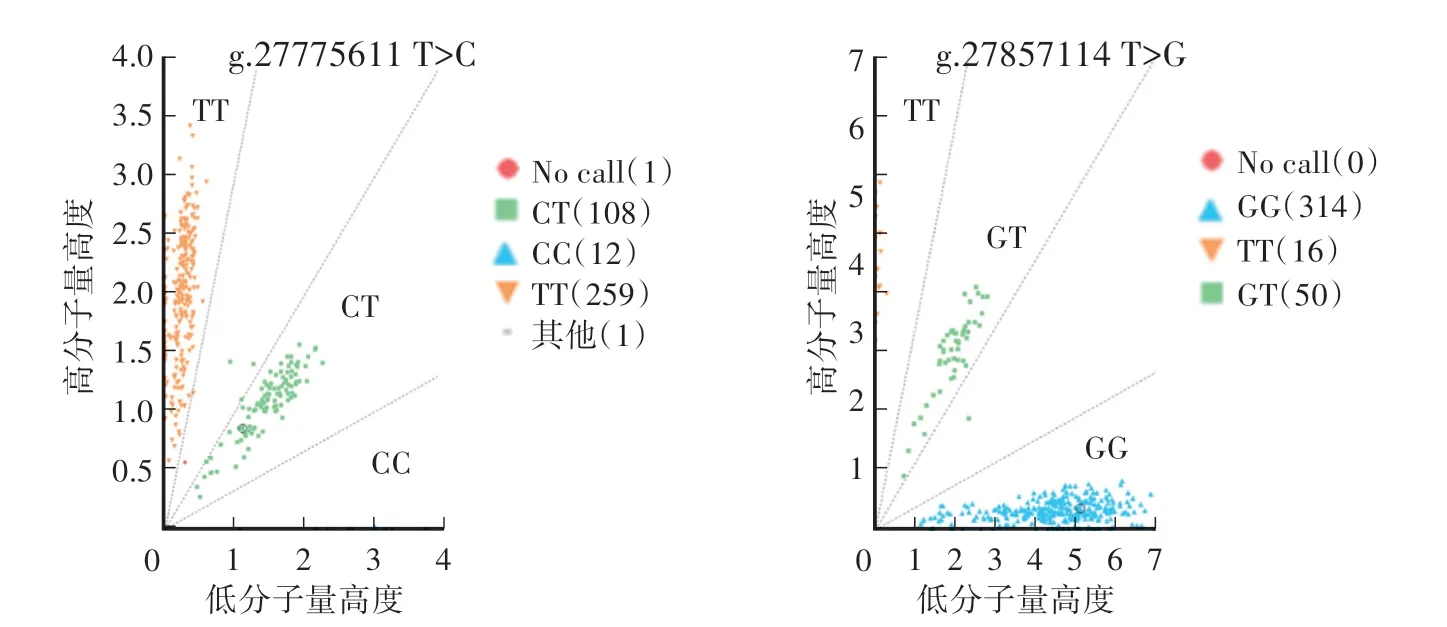
图1 GLIS1基因分型Fig.1 Genotyping of GLIS1 gene
根据分型结果,对2个SNP位点在单、多羔品种中的基因型频率和等位基因频率进行统计,并用卡方检验的方法验证单、多羔绵羊品种间的基因型频率和等位基因频率之间的差异。
由表2可知,在不同产羔能力的2个绵羊品种之间,GLIS1基因2个SNP位点的等位基因和基因型频率均达到极显著水平(P<0.01)。g.27775611 T>C和g.27857114 T>G位点在两个绵羊品种中的优势等位基因分别为T和G。

表2 GLIS1基因SNP位点在2个绵羊品种中等位基因和基因型频率分析Table 2 Analysis of allele and genotype frequencies of SNPs at GLIS1 gene in two sheep breeds
对2个SNP位点在6个绵羊品种中的基因型频率和等位基因频率分别进行统计,并计算该位点在各群体中的多肽信息含量(反映某个等位基因在群体中受选择的强度,其值大小与受选择强度成正比)、杂合度和有效等位基因数,并用卡方检验该位点在各群体中的平衡状态。从表3可知,GLIS1基因g.27775611 T>C位点在滩羊、萨福克羊和草原型藏羊这三个群体中呈中度多态(0.25≤PIC<0.5),在小尾寒羊、苏尼特羊和杜泊羊3个群体中均呈低度多态(PIC<0.25);g.27857114 T>G位点在萨福克羊和杜泊羊2个群体中呈中度多态(0.25≤PIC<0.5),在小尾寒羊、滩羊、苏尼特羊和草原型藏羊4个群体中均为低度多态(PIC<0.25)。χ2检验结果表明,除了杜泊羊的g.27775611 T>C位点以及草原型藏羊的g.27857114 T>G位点外,其余位点在各品种中均处于哈代温伯格平衡状态(P>0.05)。

表3 GLIS1基因SNP位点的群体遗传学分析Table 3 Population genetic analysis of SNPs at GLIS1 gene
2.2 GLIS1基因多态位点与小尾寒羊产羔数的关系
将2个SNP位点分别与小尾寒羊3个胎次的产羔数进行关联分析,结果见表4。GLIS1基因g.27775611 T>C位点突变显著影响小尾寒羊第二胎产羔数(P<0.05),但对第一胎和第三胎产羔数无明显影响(P>0.05),但总体而言,CC型产羔数最多,TT型其次,CT型最少;g.27857114 T>G位点与各胎产羔数之间均无显著关联(P>0.05)。
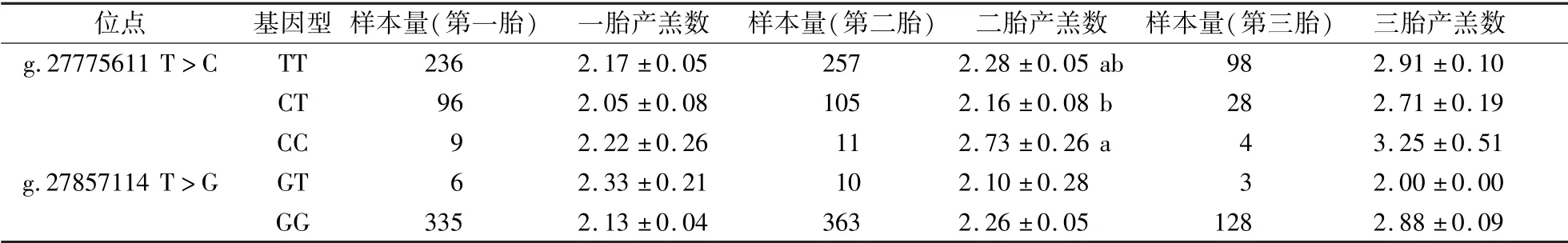
表4 GLIS1基因2个SNP位点与小尾寒羊产羔数关联分析Table 4 Analysis between the two SNP loci of GLIS1 gene and litter size in Small Tail Han Sheep
3 讨论与结论
绵羊在我国家畜中占据重要地位,是三大家畜之一,但绝大多数绵羊产羔数低的特点成为我国绵羊产业发展的主要限制因素[12]。因此,如何适当提高绵羊每胎的产羔数是养羊业亟待解决的科学难题。随着现代分子生物技术的迅速发展,高产羔数主效基因分子标记在绵羊生产中的应用可能是解决其产羔数低的有效方法。目前科学家已经定位了一些与绵羊产羔数相关的主效基因,其中最为著名的是BMPR1B[13-15]、BMP15[16-17]、GDF9[18-19]和B4GALNT2[20]。然而绵羊的产羔数性状是一个受微效多基因调控的复杂性状,仅仅靠已经发现的高产羔数主效基因分子标记很难在整体上提高绵羊的产羔数,继续挖掘与产羔数相关的基因仍十分必要。本实验室前期通过全基因组重测序发现GLIS1基因在单、多羔绵羊之间FST值达到了显著水平,虽然目前尚无GLIS1基因与动物繁殖性能之间关系的相关报道,但已有其在小鼠胚胎发育中的相关报道[5],而胚胎发育也是影响动物最终生殖力的重要阶段。因此,本研究首次对候选基因GLIS1多态性与绵羊产羔数之间的关系进行研究,旨在探讨其多态性与绵羊产羔数之间的关系,从而寻找与绵羊产羔数相关的候选基因及关键位点。
通过对GLIS1基因2个SNP位点g.27775611 T>C和g.27857114 T>G进行分型分析发现,在不同产羔能力的两个绵羊品种之间,GLIS1基因2个SNP位点的等位基因和基因型频率均达到极显著水平(P<0.01),表明这2个SNP位点很可能与绵羊的产羔数有关。群体遗传学分析表明,g.27775611 T>C位点在滩羊、萨福克羊和草原型藏羊中受到中等强度选择(0.25≤PIC<0.5),g.27857114 T>G位点在萨福克羊和杜泊羊中受到中等强度选择,2个位点在其他绵羊品种中受到选择的强度很低(PIC<0.25),该结果表明不同位点在不同的绵羊品种中会受到不同强度的选择,不过以上结果可能与某个绵羊品种遗传多样性较低有关,另外,本试验中有的品种样本量较少可能也对结果有一定程度的影响。哈代温伯格平衡分析结果表明,在长期的进化和选择下,两个位点在遗传适应性上可能具备一定的优势。杜泊羊的g.27775611 T>C位点和草原型藏羊的g.27857114 T>G位点均没有任何突变型,小尾寒羊和滩羊的g.27857114 T>G位点存在少数突变杂合个体,但均无突变纯合个体,可能是因为纯合突变型个体在胚胎发育过程中就死亡了,而突变杂合型的生存力可能相比野生型也削弱了很多,因此存在的个体很少。关联分析结果显示,GLIS1基因的g.27775611 T>C位点突变纯合子在各胎次中产羔数最高,突变杂合子在各胎次中产羔数最低且低于突变纯合子,且在第二胎中达到了差异显著水平,但由于突变纯合子在所选群体中个体较少,不具有很强的说服力,有待于增加样本量进一步研究。g.27857114 T>G位点没有野生型个体且突变杂合型个体很少,说明该位点突变纯合增加了个体的适应性,但该位点突变是否增加了产羔数,因没有野生纯合个体所以无法得知。突变纯合子在第二胎和第三胎中产羔数均最高,但与突变杂合子之间未达到差异显著水平,初步推测g.27857114 T>G位点虽然对绵羊产羔数有一定影响,但可能并不是影响绵羊产羔数的关键位点。
本研究初步得出以下结论:GLIS1基因错义突变位点g.27775611 T>C(苏氨酸→丙氨酸)与小尾寒羊产羔数显著相关,可能参与绵羊生殖调控;而错义突变位点g.27857114 T>G(苏氨酸→脯氨酸)与绵羊产羔数无显著关联。
