DNA自组装与信息存储
2021-08-16张晨昊王君珂
张晨昊, 王君珂, 晁 洁
(南京邮电大学 材料科学与工程学院, 江苏 南京 210003)
21世纪是信息量指数级增长的时代,目前全球范围内数字信息总量已经达到45泽字节(Zettabyte,ZB).如果继续存储一切即时访问数据,到2025年,全球内存需求将达到175 ZB[1-2],海量数据面临长期存储的问题亟待解决.当今使用最普遍的信息存储器件是闪存设备,但频繁地删除和重新存入数据会导致设备的快速老化,最终造成重要数据丢失.由于硅基设备的数据存储能力有限,且硅基材料本身对人体健康和环境保护有一定的危害性,科学家一直在寻找可代替的优质存储介质[3].
纵观该领域的研究现状,以合成DNA作为信息存储的新型介质将是解决问题的关键.与传统存储介质相比,DNA在数据保存时间及存储密度上更有优势.较之传统硬盘,它的存储容量是其10倍有余,存储密度更是其千倍.最新研究结果表明[4],仅4 g DNA就可以存储全球一年产生的数字信息.与仅有十几年使用寿命的磁带、光盘等存储设备相比,DNA在避光、干燥、适宜温度下可以保存数百年乃至数千年[5].然而,目前大多数基于合成DNA的存储技术通常以溶液中的DNA分子进行信息存储,这是一个缓慢而费力的过程,需要进行DNA扩增和高通量测序.相比之下,利用DNA自组装技术合成的纳米结构具有空间可寻址性、可编程性(对DNA形状进行逻辑编程,并利用可寻址的DNA链将生物分子、聚合物和无机纳米颗粒等修饰在其表面)等优点,目前这项技术已经能够提供与传统光刻技术相当甚至更高的空间分辨率[6-9].此外,一些设计巧妙的DNA组装体还能显示出模拟数字电路计算的能力[10-12].
鉴于以上这些纳米结构的特性,DNA自组装技术在信息存储领域凸显出其重要作用.本文首先简单介绍了DNA自组装技术的发展,然后从DNA origami和DNA tile 两个方面分析DNA自组装的合成原理,总结了基于DNA单链、DNA origami和DNA tile的信息运算和存储.最后讨论了DNA作为信息存储的介质目前面临的一些挑战.
1 DNA自组装技术
长期以来,将生物分子进行可寻址自组装一直是多学科领域的研究目标.早在20世纪80年代,Seeman等[13]就提出DNA自组装的概念,即将DNA单链作为基本结构单元,进而组装成具有几何特征的纳米结构.经过近40年的发展[14-18],各种各样的DNA自组装方法已被开发出来,如DNA origami、DNA tile、DNA砖和DNA线框自组装等,用于构建具有高精度和多维度的DNA纳米结构.特别是DNA origami和DNA tile的组装方法可以通过精确设计一系列互补的寡核苷酸,实现DNA基本单元自上而下的自组装.这类DNA纳米结构具有完全可寻址的特性,开创了DNA自组装的新时代,并为DNA计算和存储提供了有力的技术支撑[19].
1.1 DNA origami
DNA origami的发明是DNA自组装技术发展的里程碑之一[20-23].这项自组装技术最早是由Rothemund[24]在2006 年提出的,DNA origami是将支架链(长DNA单链)和数百个设计好的短DNA单链折叠在一起.其中,每个短DNA单链都和支架链有多个结合域,它们通过碱基互补配对结合在一起,以类似于编织的方式折叠成任意形状.与基于DNA tile的组装方案相比,DNA origami组装方案通常表现出更高的产率、稳定性和构建复杂几何形状的能力,研究人员使用这种技术已经制成了各种各样的一维(1-dimension,1D)、二维(2-dimension,2D)和三维(3-dimension,3D)DNA纳米结构,如星形线框结构、齿轮结构[25]、球形和3D瓶[26](图1a~图1c)等,这些纳米结构的尺寸被精确控制在10~100 nm之间.
DNA origami一般是基于DNA碱基互补配对原则,对单链DNA进行编程组装的纳米结构,然而,这种结构缺乏灵活性和可逆性.Gerling等[18]构建了基于核苷酸碱基堆积相互作用组装的三维DNA origami纳米结构(图1e),而且这些结构会随着盐浓度或温度的变化发生构型变化.最近,Zhou等[27]首次利用DNA origami实现了RNA长链的可控编程,并成功地调控了病毒蛋白装配的过程.该方案实现了人为调控病毒蛋白的各个复合组装步骤(图1f).
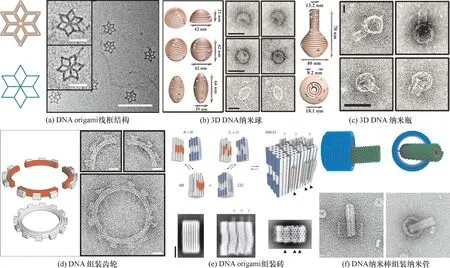
图1 DNA origami自组装结构示意图
目前,DNA origami自组装结构表面积通常为8 000~10 000 nm2,在该区域范围中大约包含200个可寻址点,因此,研究人员可以设计任意结构,并应用于多个领域.例如在DNA存储领域, DNA origami可被看作成信息存储盘,通过一系列生化反应实现数据信息的运算和存储[28-29].
1.2 DNA Tile
DNA自组装技术中另一个重要设计方案是DNA tile的组装,不同于DNA origami, DNA tile完全依赖于短DNA单链的组装,较短的链通常先组装成唯一或相同的单元块,然后进一步组装成高度有序的有限结构,每个DNA单元块的序列与占据的空间位置相关,并且是可单独寻址的[30].以这种方式产生的DNA结构的大小通常可以与DNA origami纳米结构相媲美,因此,将DNA tile结构用于多维结构组装引起了科学家的广泛兴趣[10,31].如图2a所示,Matthies等[32]通过使用对称的DNA tile结构成功组装了一系列周期性的DNA结构,例如菱形、管状和环形结构.Wang等[33]通过控制DNA tile的单元结构浓度进行复杂的二维和三维结构的自组装,包括正方形二维晶格、立方体和八面体等(图2b).
2017年,Manuguerra等[34]提出了DNA tile晶格和多面体结构的另一种装配方法,该方法使用小的DNA tile多面体作为点阵结构的连接点和支点,图2c中展示了立方体结构的单体单元和1D、2D和3D网格装配设计原理图.同年,毛承德课题组[35]设计了一种T形DNA tile单体结构,该结构可以将基于霍利迪(Holliday)连接体的DNA单体组装成大的DNA结构.基于这种组装原理,他们组装了一系列DNA多面体,如DNA四面体、八面体和二十面体(图2d)等.
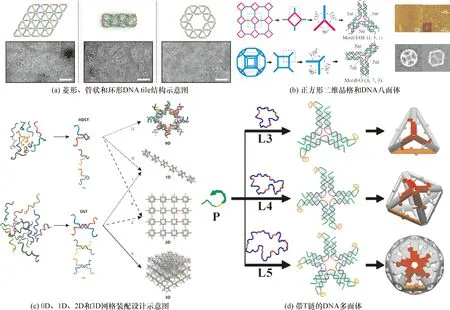
图2 线框DNA tile自组装的多面体
此外,DNA tile中不同结构单元的排列组合还可以进行信息运算,这些信息可以通过算法驱动的自组装本身固有的逻辑顺序进行信息处理[22,36].
2 DNA信息存储
1962年,科学家Wiener和Neiman提出:DNA作为存储介质在存储密度方面具有巨大的优势[37].但受当时DNA合成和测序技术的掣肘,直到1988年DNA存储概念才真正提出,Ceze等[4]开创性地将二进制中的1和0映射到DNA的4个碱基(AGCT)上,用DNA序列编码了一个古语言符号.2011年,哈佛大学知名遗传学家Church等[37]利用DNA单链合成技术存储了一本书,包括53 426个单词、11个图片和1个计算机程序,共5.27 兆字节 (Mebibyte,MB),该工作成为DNA数据存储方面的又一里程碑.随着研究的不断深入,Organick等[38]使用超过1 300万条寡核苷酸链,对35个不同类型的文件(数据量超过200 MB)进行编码和存储,并表明可以使用随机访问的方法单独恢复每个文件.他们还开发了一种算法,通过最大限度地利用所有序列读数的信息,大大降低了无错误解码所需的测序读数覆盖率.2018年,Nguyen等[39]基于DNA自组装的片上荧光切换系统,开发了一种可擦写且可随机访问的数据存储设备.Song等[40]展示了一种能够并行随机-随机存取的DNA多位非易失性存储系统.通过电场诱导的荧光标记互补探针的自组装来设计存储器的访问写和位移操作,并通过荧光成像读取数据位.
最近,包括微软、因美纳(Illumina)和西部数据(Western Digital)等在内的计算机和生物技术公司联盟宣布将组建DNA数据存储联盟,该联盟旨在构建DNA数据存储的整个生态系统,目前DNA的数据存储产业正蓬勃发展[9].总之,这些进展证明了可行的、大规模的、多样化的DNA数据存储和检索系统正不断完善和实现,因此,信息存储的未来就是DNA信息存储[41].
2.1 基于DNA单链自组装的存储
由于目前大多数DNA测序平台存在高成本、读写延迟和高错误率等问题,这使得DNA作为存储介质缺乏与现有存储设备竞争的能力.由于天然DNA链的碱基序列是固定的,研究人员可以修改拓扑结构来编码信息,为解决该问题提供了可能性.Tabatabaei等[42]介绍了一种大分子存储系统,与之前提出的所有基于DNA的数据存储方法不同,该系统将信息存储在DNA分子的糖磷酸盐骨架中,而不是DNA碱基的序列内容,即二进制信息的字符串被转换为位置编码.他们将文件信息打入大肠杆菌基因组DNA的聚合酶链式反应(PCR)产物中,并通过高通量测序和读数比对,准确地重建编码数据.同时,该存储系统可在正交DNA片段上并行插入二进制信息,并能实现单比特随机访问和逻辑运算.
一个DNA数据库通常不是完全静态的,它需要保持动态特性,并避免存储信息档案过程中出现冗余.因此,动态特性的实现将大大加强DNA存储的可行性和可操作性.Lin等[43]展示了一个由T7启动子和单双链杂交结构组成的简单体系结构,该方法利用DNA自组装的功能和优势完善了基于动态DNA的信息存储,其中单双链杂交结构提供了用于访问特定DNA链和存储信息的物理地址.通过扩展可编码的序列空间,该系统增加了理论上的存储密度和容量,并简化了设计正交文件地址集时的计算负担.同时,T7启动子在不破坏DNA的前提下可以从DNA单链中转录信息,从而实现了可重复的信息访问.
此外,DNA单链也能够以序列特异性的方式与无机纳米材料相互作用,这为基于DNA的数据存储提供了一种新的无杂交途径.Zhang等[44]提出了一种无DNA杂交的信息存储方案,他们通过在一维碳纳米管(Carbon nanotube,CNT)的表面组装DNA链,生成一种新型的管状核酸(Tubular nucleic acid,TNA)(图3).其中DNA以序列特异性的方式与CNT相互作用,表现出不同的结构构象.原子力显微镜(Atomic Force Microscope,AFM)成像显示TNA表现出独特的模式,具有特征性的高度和距离,这些特征高度和距离可以被用来对碳纳米管进行二维编码.此外,还可以利用具有不同手性的半导体碳纳米管扩展DNA序列以进行信息存储.
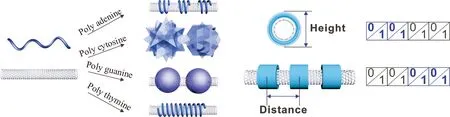
图3 管状核酸编码碳纳米管
目前基于DNA的数据记录架构都是将数据信息存储在合成DNA中,并通过NGS或第三代纳米孔测序技术检索所需信息.其中,固态纳米孔是读取分子三维形状的有力工具,可以将分子结构信息转化为电信号.Chen等[45]展示了一种用于鉴定DNA纳米结构的高分辨率集成纳米孔系统(图4),使用的DNA载体双链上的发夹长度仅有8个碱基对(base pair,bp)之差,该系统具有区分这种短DNA发夹长度的能力.使用该集成纳米孔系统,研究人员读取了多达112个DNA发夹,这些发夹的间隔为114 bp,连接在携带数字信息的DNA载体上,并有可能通过连接多个DNA载体双链进行扩展,用于数据存储.该数字数据存储方法展现了一种替代基于DNA碱基序列的信息存储方法,它的优势在于将DNA单链的简单自组装与纳米孔传感相结合,避免了复杂的合成装置和酶的使用,并且可以在微型规模上进行集成,从而易于编码和解码.总而言之,这些进展证明了基于DNA单链自组装的信息存储正不断完善和突破现有技术的桎梏,为信息存储的未来奠定了坚实的基础.
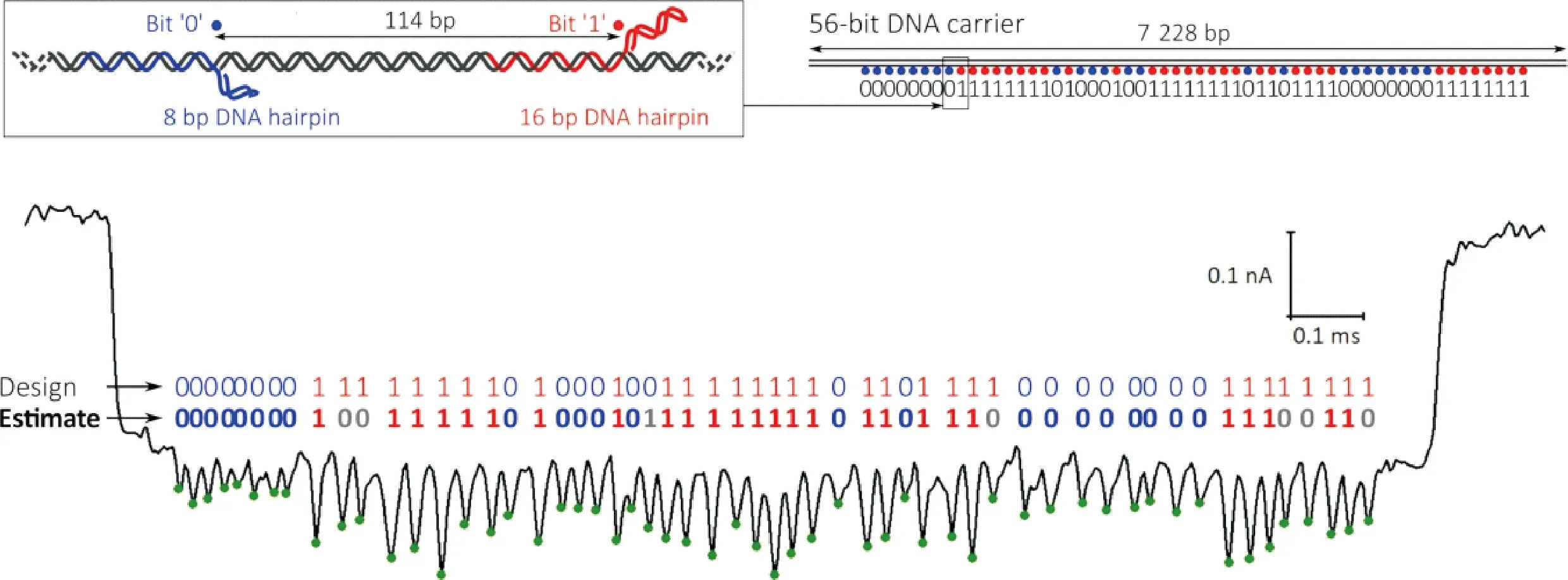
图4 基于DNA纳米结构和固态纳米孔的信息存储
2.2 基于DNA origami的信息存储
对于分子数据存储,常见的方法是根据编码规则设计DNA序列并合成,将数据存储在DNA序列中,具有密度高、耐久性好、维护成本低等优点.然而,基于合成DNA的数据存储模式是通过合成新的DNA链写入更多数据,该模式存在新数据写入繁冗,成本高等弊端.此外,DNA一旦合成,其固有的碱基序列很难修改,给数据操作和计算带来很大挑战.而DNA origami是基于沃森-克里克(Watson-Crick)碱基配对的灵活性功能纳米材料,能够解决上述问题.Rajendran等[46]展示了一种新颖的自组装技术,他们利用多重折纸结构,以特殊设计的形状在二维空间中放大折纸结构.在水平方向上该结构在π-π堆叠相互作用,形状互补和序列互补的基础上完成结构的拼接.研究人员一共设计了9个不同的拼图碎片,用DNA发夹结构装饰拼图碎片的表面,以显示字母表中的字母.如图5a所示,自组装的二维结构在纳米尺度显示完成了单词“DNA jigsaw”信息的存储,这种方案可以随意调配组装顺序,从而灵活地存储信息.

图5 基于DNA origami 的信息存储
数据的安全性和隐私性也是信息存储面临的难题.利用DNA分子的独特特性在物理密钥中进行加密,可以实现安全存储,因为只有使用正确的密钥才能检索到信息.Zhang等[47]开发了一种DNA折纸加密技术(DNA Origami Cryptography,DOC),该技术利用噬菌体病毒支架折叠成纳米级的盲文图案进行安全通信,可以创建大小超过700位的密钥.通过在携带部分信息的多个DNA折纸之间建立特定的链接,可以确保传输信息的完整性.DOC的多功能性还体现在传输各种数据格式上,包括文本、音符和图像(图5b).为了将该方法发展成为一种实用的数据加密技术,可以使用其他分子标记代替链霉菌素来编码传达信息的纳米图案,同时采用3D表征方法,实现高分辨检测的生物分子点阵图案.因此,随着分析技术的提高,DNA纳米图案中可以嵌入更多的信息点,并增加结构的信息存储容量.
同样,数据也可以存储在DNA变构的二维结构中,可以方便地写入和阅读数据,从而替代合成DNA的数据存储模式.具有纳米级精度的DNA origami已被用于模拟复杂的分子网络,可以通过模拟DNA origami复杂的动态行为进行主动模式操作.Fan等[48]提出了一种基于可重构DNA折纸多米诺骨牌阵列(DNA origami domino array,DODA)的动态模式操作系统,以执行复杂模式操作的邻近感应分子控制.可重构DODA的可激活平台通过一组“触发”DNA链,使DNA origami的构型发生横向改变.这种构型转化进一步使DNA单链之间的距离更加接近,产生临近感应,并进行DNA链置换级联反应,从而完成“写入”“擦除”和“移位”3种不同的模式操作,擦除操作如图5c所示.总体而言,可重构的DODA为安全信息存储,传输和恢复提供了出色的编码基础.同年,Fan等[49]提出,在系统中引入一组密钥DNA链时,可重构的DODA系统不仅可以演示分子信息编码,更能进一步用于高级信息安全的保障.首先,该系统将信息存储和编码在“前”构型的DODA中并进行加密,接着,添加一组解密DNA链后,引发DODA构型重排,转换成“后”构象,同时完成对信息的解码,结果可通过AFM扫描读出.例如,他们在DODA中成功呈现数字0~9的视觉图案和更复杂的交换代码等信息,安全性能高.此外,为了提高分子信息编码的复杂度,他们以两组DNA链为密钥,成功解码了“S-J-T-U”的防伪图案(图5d).这种方案提供的编码信息在经过特殊设计的解码过程后才能被读取,克服了信息安全编码技术中的关键难题.因此,DNA origami不仅仅是优秀的存储工具,其结构独特的可寻址性和可编程性还可以为信息的安全存储提供更精密的设计平台.
2.3 基于DNA tile的信息处理与存储
在DNA tile自组装技术中,DNA tile作为可编程自组装的基本构件通过Watson-Crick互补原则与排列,在一维、二维或三维晶格上与其它组件相结合.基本结构包括双交叉DNA tile、三交叉DNA tile和单链DNA tile(SST)[50].DNA tile通过自组装技术形成“唯一寻址”结构,其中每个DNA tile结构类型会精确地预设在固定位置,并有一组固定相邻的DNA tile组件.这些结构除了具有周期性和唯一寻址性外,不同结构单元的组合排列还可以存储信息,数据信息可以通过自组装本身固有的算法驱动进行处理.这种算法自组装可以对基础结构进行信息的编码,从而生成任意复杂的模式和结构,其本质上类似于基因组DNA遗传程序的编码.但到目前为止,DNA tile集很小而且很难编码用于特定的组装算法,这需要将算法自组装的DNA tile类型扩展到数百种,其中SST是最好的选择,这种DNA tile结构被称为“DNA算法画布”[11,30].Tikhomirov等[51]使用了一组独特的DNA链,并通过“分形”组装的方法创建了大小为0.5 μm2的二维阵列,并在放大的DNA tile结构上编写了复杂的2D图案,如蒙娜丽莎.因此,基于图案与DNA序列相互转换的软件工具和创建这些图案所需的实验设计,研究人员可以利用这种组装技术创建更大、更有用的DNA材料和器件,从而实现存储容量更大的DNA tile结构.
由于DNA tile在设计规则逻辑图案和直接可视化方面存在优势,因此,可以通过DNA tile结构设计实现单逻辑和多逻辑门,并构建各种生物逻辑电路.美国加州理工学院的Winfree课题组展示了一个DNA tile集的设计,该DNA tile集包含355个SST,可以重新编程并实现各种6位算法[52].他们使用该DNA tile集构建了21个电路,该逻辑系统设计从抽象的迭代布尔电路模型开始,该模型具有(n-1)个门,每个门有一个输入和一个输出,每个(n+1)门计算的逻辑函数由用户指定.该系统通过反复迭代电路层进行计算,最终达到固定点或一个周期.该模型包含了重复执行的布尔门的局部连接阵列,这是一个功能强大且通用的模型,可以模拟许多其他计算模型,例如图灵机、通用布尔电路和有界宽度的分支程序等.Tandon等[53]也展示了一种基于DNA tile的计算器(图6),该计算器可以通过实现正和负(输入和输出)的进位执行加法和减法运算.该系统具有很多优点,如相对简单的结合构件设计思路、高能量效率的数据计算和并行性强等.他们为了验证全加法器和全减法器的逻辑设计,将二进制加法和减法的DNA tile图集用AFM图像进行了显示.这种基于DNA的计算器可能对其他复杂的计算和逻辑运算(如解码器)很有帮助,拓宽了DNA电路在分子计算中的应用.这些研究结果表明,DNA分子工程和DNA分子科学正在进入算法时代,DNA自组装是可编程生物系统内可靠的算法组件,为信息存储提供了可靠的算法驱动.

图6 二进制减法DNA tile集的AFM图像示意图
3 DNA信息存储的挑战
基于DNA的信息存储拥有保存时间长、存储密度高等优点,可以预测,未来基于DNA的信息存储将会成为硅基信息存储的有力竞争者.然而,要实现这一目标,研究人员仍然面临许多巨大挑战.首先,与传统的存储技术相比,在DNA中编码并检索信息的完整流程需要耗费更多的时间和经济成本.因此,DNA信息存储在将来可能会面临来自光学、磁性或量子技术等领域的竞争和挑战.其次,以DNA作为介质存储数据对环境要求较高,极端条件下序列突变导致数据变化的可能性也不容忽视.最后,与传统和流行的存储系统,如闪存和硬盘相比,DNA数据存储系统很难做到快速擦除和重写数据,这也是DNA信息存储面临的一个巨大挑战,目前涉及该问题的研究还处于理论阶段.
3.1 合成成本的挑战
基于DNA的信息存储系统的主要缺点是在核苷酸序列上写入和读取数据所需的成本.合成DNA(写入/编码)的成本高于测序(读取/解码)的成本.Goldman等[54]在创建739 kb硬盘存储的实验过程中花费了12 660美元,其中98%用于合成,只有2%用于测序.基于DNA喷泉(Fountain)方案,Erlich等[55]花费7 000美元编码2.14 MB数据.因此,DNA喷泉每MB数据的写入成本约为3 500美元,另外1 000美元用于读取数据.最近,Yazdi等[56]花费2 540美元,先将10 894字节的数据压缩成3 633字节,然后用长度为16 880 bp的DNA进行编码.如果将这个成本与现代传统硬盘的成本进行比较,用DNA写入数据的成本将脱离实际.按照DNA Fountain方案,将现代1TB硬盘的数据存储在DNA中,成本将要高出约7×107倍.虽然DNA存储设备的成本必须与同类设备持平,才能对未来市场产生影响,但目前每MB数据的存储和读取成本很高,仍无法作为主流的存储介质.
3.2 长期存储的挑战
为了实现DNA介质在几个世纪甚至几千年的稳定保存,需要保护DNA免受湿度、辐射和温度等影响,通常的方法是将DNA完全密封在类似二氧化硅这样的基质中[57-59],但是这种存储方式不利于数据的随机读取和使用.
DNA链的长度也是影响数据存储时间的重要因素,虽然较长的DNA链可以减少用于文件地址和索引的单链成本,从而增加系统的信息密度.然而随着DNA链长度的增加,DNA的存储密度会逐渐降低,因此,较短的DNA链更具实用价值.研究表明较短的DNA链对环境要求更低[60].例如,与53 碱基(nucleartide,nt)长度的DNA链相比,113 nt DNA链的降解度在830 W/m2的阳光照射下升高了近2个数量级.与信息存储一样,评估冻融降解系数的一个重要考虑因素是DNA链的长度.研究发现,较小的DNA链更具抗冻融性.此外,DNA链在冷冻和解冻过程中会形成冰晶,这一过程产生的力将导致DNA链的断裂.证据表明[59],在类似的张力下,冷冻的DNA比非冷冻DNA更容易断裂.几项研究[61-63]已经发现,经过反复冷融化的DNA样品,通常会发生指数降解.例如,1次冻融后,缓冲液中的DNA降解了约10%,而20次冻融后DNA 降解约75%.
除了在冻融样品的过程中会发生DNA链的降解和缺失,也存在其他形式的DNA降解过程,这些过程包括脱嘌呤(从DNA主链上去除腺嘌呤或鸟嘌呤碱基)和氧化反应.在室温条件下,DNA链每年约有6%会发生脱嘌呤或氧化反应,这些反应速率随温度的升高而迅速增加.同时纯化操作也会导致测序过程中核苷酸缺失,而氧化反应则可能导致DNA链之间发生交联,从而抑制干燥DNA的再水化以及DNA的扩增和测序[64-67].以上研究结果表明,合适的存储条件是信息稳定存储的保障,寡核苷酸在进行长期存储过程中仍然面临很多挑战.
4 总 结
将大量的数据存储在双链DNA分子中,这已不再是科幻小说中的情景,DNA信息存储具有存储密度高和存储量大的显著优势,同时DNA信息存储的发展也将有利于DNA测序和合成技术的研究和开发.但是,基于DNA的数据存储仍然需要解决一系列的挑战,才能发挥其巨大潜力.目前DNA信息存储面临的首要挑战是合成大量DNA链的高昂成本,相信随着寡核苷酸合成技术的进步,在DNA上写入和读取信息的成本将会逐步下降.此外,使用DNA自组装结构进行通用信息存储需要大量的模板材料.如果没有成熟的DNA自组装工艺,在与传统信息存储器件竞争过程中,基于DNA自组装的信息存储将处于劣势.当然在实现DNA自组装结构进行信息存储的过程中,DNA纳米技术领域已经着眼于开发新的组装方法来降低生产成本,如利用细菌和病毒中的生物机制大规模生产DNA纳米结构.
相信随着DNA自组装技术和纳米尺度分析技术的发展,与利用合成DNA进行信息存储方式相比, DNA自组装结构在存储信息的灵活性和可编程性上将发挥巨大作用,并减少对DNA合成和测序技术的强烈依赖.
