肠道微生物群落研究进展及合成微生物群落面临的挑战
2021-07-30张蒙蒙肖敏凤
胡 政 周 茜 张蒙蒙 肖敏凤
1(中国科学院深圳先进技术研究院 合成生物学研究所 中国科学院定量工程生物学重点实验室 深圳 518055)
2(中国科学院大学 北京 100049)
3(深圳华大生命科学研究院 深圳 518083)
4(深圳市未知病原体应急检测重点实验室 深圳 518083)
1 引 言
人类肠道微生物群落由大量复杂的微生物组成,受到宿主的出生方式、生活方式和遗传等因素影响[1-4]。肠道微生物群落在机体免疫[5]、肠道内分泌[6]、药物作用和代谢[7]、宿主疾病(如炎症性肠炎[8-9]、肿瘤[10-13]、自闭症[14]、肥胖[1,15-16]和糖尿病[17-19])等方面都具有重要作用,是疾病治疗的理想靶点。然而,肠道菌群的种类及数量繁多且绝大多数肠道微生物都不能在体外培养,因此肠道微生物组在治疗中的研究及应用仍不成熟。尽管粪菌移植(Fecal Microbiota Transplantation,FMT)已经成为一种恢复正常肠道微生物群落结构的方法,但近年来的一些临床研究表明, FMT存在细菌播散感染的风险[20-21]。因此,需要采用更加可控的方式进行肠道微生物群落的调控。
目前已经通过基因组测序平台对肠道微生物群落进行了大量研究,但微生物表型及其在微生物群落中的作用尚不能完全通过测序数据进行准确预测。例如,2018 年的一项研究表明,仅基于基因组学和生长需求的文献信息,75% 肠道细菌种类代谢模型未能预测同一细菌种类在不同培养基中的生长情况[22]。近年来,培养组学的迅速发展使得可培养肠道微生物种类显著增加,然而这也仅仅占肠道微生物种类中极小的一部分[23]。解决这一问题的关键是引入工程化思维,结合基因组序列分析和培养组学的实验方法,严格控制实验系统中的变量,厘清微生物在肠道中的功能及其相互作用关系,从而为肠道微生物群落研究及应用扫清障碍。
合成微生物群落是一类微生物组成已知且复杂性降低的系统,可以通过实验干预和数学建模,实现对复杂的人类肠道微生物组的理解。宏基因组分析可促进对肠道菌群组成自上而下的观察[24],而自下而上的方法可以帮助理解这些复杂的微生物群落是如何形成、相互作用和影响宿主健康的[25-26]。采用合成生物学的方法能加深对肠道微生物群落功能的理解。本文综述了肠道微生物群落的研究方法、影响微生物群落稳定性的因素、合成肠道微生物群落领域面临的挑战,以期更好地利用合成微生物群落促进人类健康。
2 肠道微生物群落的研究方法
目前,合成微生物群落有自上而下和自下而上两种构建方法(图 1)。其中,自上而下的方法主要涉及宏基因组学研究,其重点在于了解肠道菌群组成,以及环境扰动下菌群组成变化;自下而上方法的重点在于理解不同微生物群落是如何形成、交流和影响其宿主健康的。
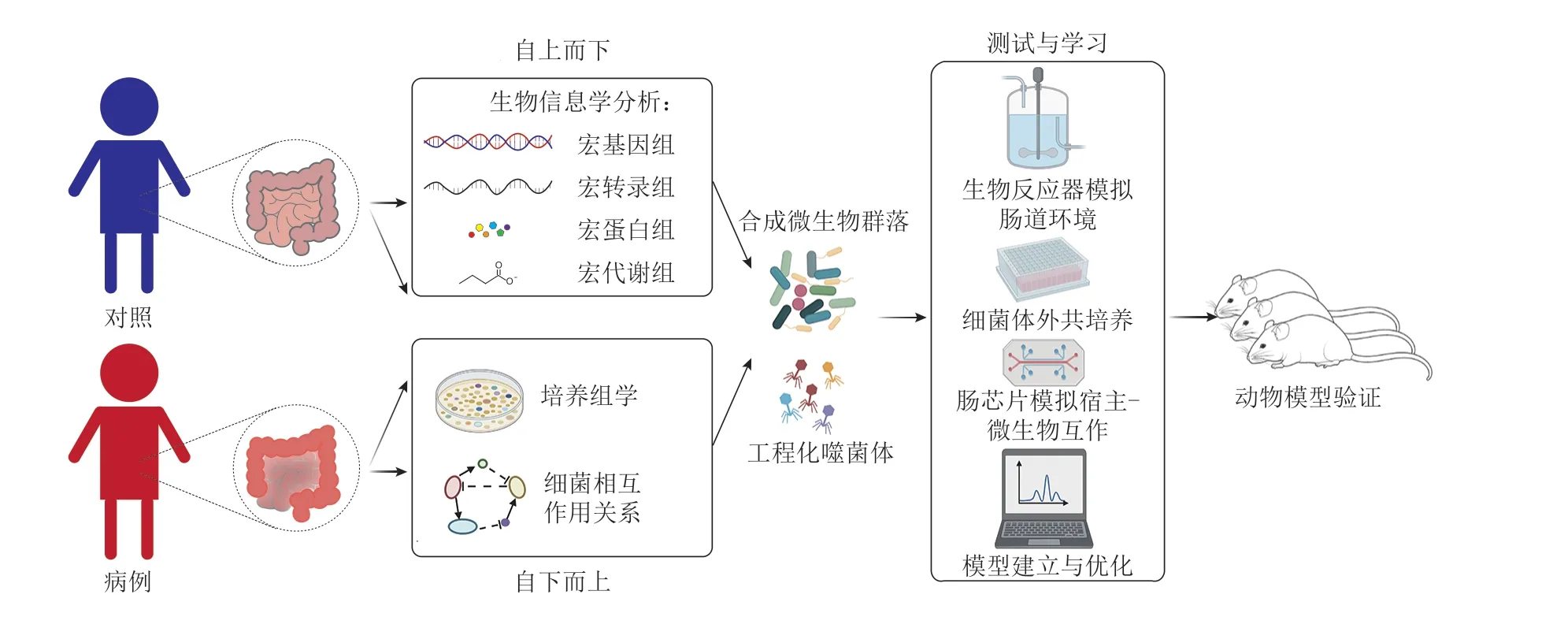
图1 肠道微生物群落的研究方法Fig. 1 Methods for the study of intestinal microbial community
2.1 自上而下——利用宏组学研究微生物群落组成及功能
传统微生物群落研究通常采用自上而下的方法,通过改变肠道或生物反应器的生理生化性质来获得具有特定功能的微生物群落。随着测序技术的飞速发展、测序费用降低而通量扩大,可以利用高通量测序的方法对肠道微生物群落进行研究。其中,16S rRNA 扩增子测序是最经典的肠道菌群鉴定方法[27-28]。目前已经有研究采用 16S rRNA绝对定量测序对微生物群落进行定量研究[28-29]。然而,这种方法仅限于“属”水平的分辨率,且不能揭示人类肠道微生物组的功能作用。目前应用最普遍的是采用宏基因组学方法对肠道环境中所有微生物的 DNA 进行测序。这种方式避免了传统培养方法的缺陷,显著提高了发现新微生物的能力,并能够对生境中微生物群落的组成、分布及其动态变化进行全面研究。大多数微生物数据都是通过粪便样本的宏基因组、宏转录组研究收集的,而针对合成微生物组的宏蛋白质组学的研究相对较少。在模拟人类合成肠道微生物群落时,宏蛋白质组学研究可以增加功能、分类和生物量维度。Liao 等[30]开发了一个 iMetaLab 平台并将其用于分析宏蛋白质组学数据,使用体外微生物培养进行宏蛋白质数据分析。宏基因组、宏转录组、宏蛋白质组和宏代谢组学等的出现给肠道微生物群落研究带来了新的变革,基于整合宏组学数据分析和合成微生物组模型已经成为新的趋势,使我们可以更全面地了解肠道微生物群落组成及其功能,更容易地控制微生物组培养过程中的时空变量,更好地优化合成微生物群落以治疗特定疾病,加快合成微生物群落的应用进程。
2.2 自下而上——利用分离的微生物构建合成微生物群落
采用合成生物学的思维自下而上地分离粪便样本中的微生物以重建微生物群落,是一种了解肠道微生物群落的有效方式。这种方式具备以下优势:(1)合成微生物群落中的细菌组成已知、可控且具有较好的重复性;(2)由于没有病毒和病原体,分离培养的微生物群落比粪便更稳定、更安全;(3)可以根据菌株对环境的敏感性来选择菌株,这进一步增大了合成微生物群落的成功率。通过将关键物种组合为稳定的微生物群落,并与患者的微生物群落进行比较,能有效帮助设计合成平衡的人体肠道微生物群落。Petrof 等[31]设计了一个由 33 种细菌组成的合成微生物群落,发现这种合成的微生物群落能够治疗耐抗生素的艰难梭菌结肠炎。
3 自下而上合成微生物群落的方法
3.1 利用营养互补关系合成微生物群落
肠道微生物与其他生物及其环境进行复杂的相互作用,其中大多数微生物是营养缺陷型,其生长依赖外部营养物质,包括氨基酸和维生素的交换[32]。因此,构建营养互补的微生物群落从而模拟微生物的互利共生关系是一种构建合成微生物群落的有效方式。一个典型的例子是 Mee 等[33]构建的一个由氨基酸营养缺陷型的大肠杆菌菌株组成的交叉喂养群落。他们从大肠杆菌 MG1655 菌株中获得了 14 个衍生营养缺陷型菌株,对每个菌株都敲除一个必需氨基酸的基因后,菌株两两共培养表现出显著的协同作用。400 代后,通过共享精氨酸、赖氨酸、蛋氨酸和苏氨酸而建立强相互作用,从而获得最快生长的配对营养缺陷型菌株占据了群落中的主导地位。类似地,在酿酒酵母和大肠杆菌等微生物中也构建了利用氨基酸营养缺陷型菌株的营养互补关系合成微生物群落[34-35]。
3.2 利用群体感应系统调控微生物群落
群体感应(Quorum Sensing,QS)是细菌通过分泌和感受自诱导剂响应环境变化的一种分子通讯的形式,利用信号分子增强微生物之间的群体感应行为来优化微生物群落结构与组成有助于增强群落结构的稳定性。但目前对群体感应如何影响微生物群落的组成和功能仍不十分明确。细菌之间可通过群体感应信号分子,如革兰氏阴性菌用于种内交流的高丝氨酸内酯(Acyl-HSL)、革兰氏阳性菌用于种内交流的寡肽类分子(AI-P)以及不同种属微生物之间可被多种细胞识别的 AI-2信号分子等进行交流[36]。在合成微生物群落中,可以通过改造群体感应系统来调控微生物群落组成。Stephens 等[37]设计了一种在两个大肠杆菌菌株中起作用的细菌群体感应系统,通过细胞间的信号传导来决定微生物群落内细菌亚群的生长速度。第一种菌株感应小分子信号 AI-2,产生自诱导因子 AI-1;第二种大肠杆菌菌株由前一种菌株产生的 AI-1 信号控制糖运输蛋白 HPr 的转录来调节菌株在共培养中的生长速度,从而通过 AI-1和 AI-2 之间的制衡调节群落的构成。Hong 等[38]基于细菌生物膜的共生关系,构建了两株交替形成不同菌膜的枯草芽孢杆菌。在E. coli中构建的LasR QS 回路不但可以特异性识别铜绿假单胞菌产生的信号分子,且能利用该信号分子调控自身基因的表达,通过表达和分泌抗菌肽 Microcin S和核酸酶 DNase I 降解成熟的生物膜基质并杀死包裹其中的细菌[39]。
3.3 利用噬菌体改造微生物群落
研究发现,人类肠道宏基因组测序读长中约5%~6% 来自噬菌体[40]。肠道微生物、噬菌体与宿主之间具有动态相互作用[41]。Ma 等[42]利用人体肠道微生物大数据鉴定了大量的肠道噬菌体,这些噬菌体携带的功能基因与细菌在肠道中的生存适应性相关。与广谱性抗生素相比,噬菌体感染宿主细菌具有较高的专一性,因此利用噬菌体携带小分子药物或 CRISPR 元件可能是精准地消除微生物群落中的特定菌株或编辑微生物群落中特定基因的有效方式[42-43]。Song 等[44]开发了一种识别活性噬菌体的工具,有助于构建工程化噬菌体并用于改造微生物群落。Hsu 等[45]使用限菌小鼠模型研究了噬菌体对肠道细菌群落的动态影响,发现噬菌体捕食不仅直接影响对噬菌体敏感的细菌,还通过细菌间的相互作用对其他细菌产生级联效应。此外,代谢组学分析显示,噬菌体捕食引起的菌群变化对肠道代谢组产生了直接的影响。这些工作表明,利用噬菌体特异性感染并杀死细菌的特性改造噬菌体来实现调节和改造肠道菌群不失为一种具有前景的微生物群落调控方法,在哺乳动物疾病治疗方面具有潜在的应用价值。
4 影响肠道微生物群落稳定性的因素
4.1 微生物之间的相互作用影响群落稳定性
微生物群落的稳定性十分重要,复杂群落内微生物的相互作用一直是菌群研究的瓶颈之一。微生物的种间互作关系可以是正向也可以是负向的,即协同或拮抗[46]。通常情况下,群落相比单一物种具有更好的稳定性和抗干扰能力。例如,Palleja 等[47]对健康成年人肠道微生物群落的研究发现,尽管抗生素处理初期出现了群落组成的改变,如肠杆菌和其他病原菌(粪肠球菌和核梭杆菌)的大量繁殖,以及双歧杆菌和丁酸生产量的减少,但健康人的肠道菌群对短期广谱抗生素干预有一定抵抗性,一段时间后群落组成逐渐恢复到接近原来的水平。然而,肠道中微生物相互作用如何影响群落的稳定性尚不十分清楚。自然条件下的多细胞体系常常形成错综复杂的相互作用关系网络[32,46]。传统观点认为,在稳态条件下,竞争单一限制性底物的物种不能共存,这样的竞争关系组成的微生物群落稳定性较低[48]。Coyte等[49]通过动力学分析表明,合作关系导致了物种与正反馈回路耦合,增强了微生物成员的依赖性,从而降低了群落的稳定性。而竞争有助于提高微生物群落的稳定性,即高物种丰度的微生物群落主要通过竞争关系抑制合作引起的正反馈回路,从而提高群落的稳定性。Allesina 和 Tang[50]研究表明,复杂生态系统的稳定性是由物种间相互作用的类型(捕食、竞争、互惠)和这些相互作用的强度决定的。应充分考虑不同菌株间的相互作用关系,以构建具有较高稳定性的合成微生物群落。
4.2 微生物群落对宿主肠道免疫耐受是其成功定殖的关键
据估计,人类微生物群落中含有多达 1014个细菌细胞,是体内存在的人类细胞数量的 10倍,肠腔成为人体和外来微生物直接接触的最大区域[51-52]。由于营养物质经上皮细胞吸收,所以肠道表面必须保持一定的通透性,但同时,肠道作为免疫系统的一道重要防线,健康的肠道必须具备阻挡病原体的能力。因此,微生物群落需要在肠道内成功生存并定殖才能发挥作用。微生物在肠道内成功定殖主要有以下两方面原因。
(1)肠道固有结构层面,宿主肠道物理结构(如肠绒毛)诱导肠道微生物区隔化。健康宿主肠道黏液屏障由高糖基化黏液蛋白 2(Mucin 2,MUC2)包围形成。肠道黏膜的存在使得肠道免疫系统能够对大量不断变化的无害微生物建立免疫耐受,同时保持免疫反应,以抵御致病菌或共生菌入侵无菌身体环境[53-54]。此外,MUC2 也通过抑制肠道树突状细胞向炎性状态转变,从而限制了肠道抗原的免疫原性[55-56]。黏膜单层上皮细胞将肠腔与下层组织物理分隔,限制了上皮细胞屏障的通透性。因此,从物理结构上保障了微生物群落在肠道内的生存能力。
(2)生物层面,肠道共生细菌的存在可以帮助抵御外来微生物的入侵。许多常见的肠道菌群物种,如乳酸菌和双歧杆菌可通过产生抗菌肽避免肠道感染外源微生物[57]。共生细菌也通过支持上皮屏障,协助伤口愈合,招募中性粒细胞和白细胞等方式调控免疫系统。在一些研究中,共生细菌 DNA 通过调节不同类型 T 细胞的水平,帮助宿主对潜在的病原体作出反应,甚至阻止肿瘤的发生[58]。微生物代谢物,如芳烃受体 AHR 的配体吲哚-3-醛,能刺激 ILC3,从而诱导 IL-22的产生。其中,IL-22 上调腺苷一磷酸(AMP)的表达,来提高肠道对病原菌的定殖抗性[59]。研究表明,无菌小鼠肠道固有层中产生 IgA 的浆细胞和 CD4+T 细胞水平急剧下降,黏液层变薄且组成异常,防御素表达受损。与微生物群落完整的小鼠相比,无菌小鼠肠道相关淋巴组织(Peyer’s Patches,肠系膜淋巴结)更小,包含的免疫细胞数量和种类更少。因此,无菌小鼠更容易受到细菌感染,某些疾病的症状也可能更严重[57,60-61]。利用这一点,通常采用抗生素处理减少肠道细菌,从而提高合成微生物群落在肠道内的定殖水平。
4.3 微生物群落的空间分布影响其环境适应能力
肠道微生物表现出不同的黏附特性和生长速率,这可能导致肠道微生物群落成员的空间分布差异[62-63]。基于微生物在黏液层或管腔中的定位而产生的差异复制可能进一步放大因不同黏附情况形成的差异[64-65]。空间结构对建立和维持肠道微生物群落成员与宿主之间关系的信号和代谢交换有较大的影响。然而,关于肠道微生物群落空间分布的信息有限。Mark Welch 等[66]从人类粪便中分离出 15 种微生物,组成合成生态系统用于研究肠道微生物群落空间分布。然而,微生物并未呈现出典型的位置特异性分布,只是在一些位置中的细菌组成比例有所差异,如肠内壁被黏液包裹的上皮细胞以及中央富含食物和黏液的肠腔中。这些位置的微生物种群与整体组成有所不同,即不同细菌的相对比例可能会发生波动,但整体上细菌分布并没有明显差异。随着测序技术的发展,MaPS-seq 得以实现肠道细菌生物地理图谱的绘制,揭示群落功能的相互作用。小鼠肠道不同区域内,微生物分布不均,但在小尺度(7 μm 左右)范围内部分细菌呈现一定的相关性[67]。高丰度拟杆菌门(Bacteroidales)类群的物种通常含有多种碳水化合物活性酶[68],且能通过协同代谢互养共生,促进细菌空间共生[69]。而共生群落在应对不同环境时表现出的种群空间协作行为,有利于提高其环境适应性[70]。Coyte 等[49]的模型表明,空间结构的引入在不减弱细菌相互作用的前提下提高了其群落稳定性,因此,肠道微生物可能会通过空间上的分区更好地发挥功能来适应环境。
5 优化合成肠道微生物群落面临的挑战
5.1 确定核心微生物及其代谢功能
目前,主要通过以下几个指标来评价合成肠道生态系统的有效性:核心肠道微生物的相对丰度、反映微生物丰富度和均匀度的微生物多样性指数、短链脂肪酸等主要肠道代谢产物的含量和比例等[51]。然而,人类肠道菌群的微生物种类及数量极为丰富,难以确定核心肠道微生物及其代谢功能。由于代谢相互作用的强弱与空间相对位置有关,在物种选择上有必要考虑物种空间位置及其代谢功能进行综合判断。群落的复杂性也是需要考虑的一个重要因素。其中,低复杂性的群落具有更高的可重复性和操作性,但有可能缺少自然群落中的关键物种或功能物种。高复杂性的群落在维持群落的多样性上更具优势,但这也意味着可操作性降低,需要对群落成分比例进行更精细的设定。此外,疾病类型、治疗方法、患者性别、年龄和饮食习惯等因素都会造成肠道菌群的改变[8,13,71-74],因此需要整合这些因素进行微生物群落组成分析,以建立个性化的合成肠道生态系统。
5.2 选择合适的培养系统分离肠道微生物
选择适合微生物群落中各种细菌生长的培养基是十分必要的。迄今为止,大多数肠道微生物群落都是在化学成分未知的复杂培养基中培养,而在特定或最小培养基中只有少数微生物能生长,这不利于肠道微生物的分离和培养。肠道中大多数微生物的分离需要采用厌氧培养。由于厌氧培养常用的培养基,如 GAM 厌氧培养基(Gifu Anaerobic Medium)、BHI 培养基(Brain-Heart Infusion Broth)、强化梭菌培养基、胰蛋白酶大豆培养基等具有特殊性,培养基的选择极大地限制了微生物群落的构建[75]。近年来,培养组学方法使得多种以前不能培养的微生物得以成功培养和鉴定,在此过程中培养系统的改进起着至关重要的作用[23,75-78]。Magnusdottir 等[79]结合了大数据和人工智能的方式构建了一个包含 773 个不同肠道微生物的计算机模型 AGORA(Assembly of Gut Organisms Through Reconstruction and Analysis)来模拟微生物代谢过程,分析各菌株对其他微生物及宿主代谢的影响,针对拟培养的特定的肠道微生物群落优化培养基组成。另外,Yang 等[80]采用基于质谱的靶向代谢组学方法研究了无机盐、胆盐、短链脂肪酸和黏蛋白等 4 种培养基成分对体外培养的肠道微生物群落的调控作用,结果表明,在体外培养过程中应补充相应成分以模拟肠道环境。
5.3 选择合适的研究模型
确定合成微生物群落的培养条件后,需要采用模拟肠道环境的不同体外模型对合成微生物群落的功能进行测试与评估。利用简单的肠道生物反应器系统(如微量孔板系统)能有效地研究益生菌和药物与肠道微生物群落的简单相互作用[81]。连续生物反应器系统(如多级结肠模型)能调控物质的流入和流出,能帮助理解营养物质、pH 及其他条件对肠道微生物群落的长期影响,有助于设计合成肠道微生物群落[82-83]。
早期研究中所用到的生物反应器与实际肠道环境有较大差异,因此,近年来一些研究采用生物反应器与模拟消化系统相结合来更好地模拟肠道生理生化环境,包括消化酶和胆盐的添加、pH 和停留时间的改变以及模拟人类肠道的蠕动运动等。然而,这类生物反应器系统均未考虑宿主上皮系统的影响。近年来,HuMiX 平台和肠芯片等体外肠道模型增加了宿主黏液层和人类细胞系,以更好地模拟宿主-微生物相互作用,因而得到了肠道微生物群落研究者的青睐[84-87]。然而,现有的肠道模型仍需要进一步发展。
尽管这些反映宿主效应的体外模型可以在一定程度上研究多种代谢活性肠道微生物群落的作用,但最终其代谢产物和微生物群落的组成可能与人类肠道微生物不同。因此,有必要在动物模型(如秀丽隐杆线虫和小鼠)中评价合成微生物群落的有效性。一些体内肠道微生物群落研究只使用了小鼠,但某些不可忽视的宿主特异性差异仍然存在于小鼠和人之间,包括免疫差异、转运时间及肠道结构等因素。现有的活体研究在直接预测合成微生物群落的作用方面也有局限性。因此,肠道微生物群落及宿主的相互关系需要通过合成肠道生态系统和活体动物模型相结合开展研究。
5.4 选择合适的数学模型
数学建模在理解肠道微生物群落动态和行为方面具有重要价值。通过宏组学可以获得肠道微生物群落的动态变化,结合数学模型又为理解群落动力学的机制和基本原理提供新的方向。由于肠道微生物群落组成极其复杂,因此,需要对群落结构进行简化,以进行数学模型开发和校正,校正后的模型可以进一步通过实验进行测试。不断改进的数学模型和实验验证可以更好地理解人类肠道微生物群落中的相互作用。
肠道微生物群落中常采用基于微分方程的动力学模型进行群落动态研究,如广义的 Lotka-Volterra 模型(GLV)。GLV 模型利用个体种群的内在增长率和微生物种群间相互作用调节的增长率来解释群落动态,由于模型假设的参数比其他模型要少,因此该模型便于结合实验进行验证[88-89]。已有研究表明,肠道细菌可能有助于控制调节性 T 细胞(Regulatory T Cells,Treg)的数量。特定的细菌组合可以刺激这些 Treg 细胞,并可能抑制炎症。通过调节肠道中不同种类的微生物可能为治疗溃疡性结肠炎等疾病提供一种新方法。然而,逐一测试肠道微生物群落的组成极具挑战性。因此,Stein 等[89]利用扩展的GLV 模型探究感染和炎症性疾病(包括艰难梭菌定殖和炎症性肠病)背景下微生物组的时序动力学并预测其稳定性。
随着高通量测序、多组学联合分析的发展,各种数学建模和实验的综合方法已被应用于不同分辨率、时间和空间尺度的肠道微生物群落,包括基因、个体和种群。构建基因组尺度代谢模型(Genome-Scale Metabolic Models,GEMs)描述微生物群落的代谢途径能更好地利用组学数据进行综合分析。GEMs 建模方法通常是基于高通量组学数据和已知的生化反应数据库,从而构建代谢网络。其中,代谢流平衡分析(Flux Balance Analysis,FBA)采用数学方法对代谢网络里的代谢流进行拟合分析,是目前微生物群落代谢网络重构及代谢模拟最常用的方法之一。FBA 分析主要用于描述稳态下(即群落代谢物的产生与消耗平衡)的系统特性,采用胞内代谢物达到平衡后的数据计算不同支路代谢流的分布情况,定量预测细胞内的反应通量、代谢物的消耗与产生速率以及细胞生长速率,从而获得特定条件下细胞代谢的全景。Cole 等[90]通过空间扩散模型耦合多个动态 FBA 模型的方式提出空间动态通量平衡分析(3-Dimensional Dynamic Flux-Balance Analysis,3DdFBA),模拟细菌与其他细菌的代谢产物交换,预测了大肠杆菌菌落的生长特征,并识别出醋酸盐和乳酸盐的交叉喂养情况。Chan等[91]在 FBA 模型的基础上给群落所有成员施加了恒定的平均增长率以确保群落的共存和稳定。在没有这种限制的情况下,生长速度更快的微生物最终将取代群落中的其他微生物。
近年来,机器学习和深度学习算法在肠道菌群数据挖掘方面取得了较大的进展:基于组学数据的建模可以预测多菌株群落的交叉喂养和竞争机制,根据微生物代谢产物(如丁酸盐和功能性胞外多糖等)设计最佳微生物群落[79,92]。有研究团队利用机器学习从多组学数据中识别噬菌体并挖掘噬菌体与细菌宿主的关系,为利用噬菌体改造微生物群落奠定了基础[44,93-97]。然而,尽管利用机器学习算法进行肠道菌群数据分析具有很好的应用前景,但不可否认的是其仍存在以下不足:(1)缺乏对机器学习算法的设计及特异性优化;(2)机器学习通常需要大量的训练数据,若训练数据集的规模不够大则容易导致构建的模型不具有普适性;(3)肠道菌群的数据影响因素较多,导致数据可能不具有足够的特征性,难以获得性能良好的训练模型[98]。机器学习和深度学习算法作为肠道微生物群落数据分析及合成微生物群落构建的工具仍需要进一步优化(图 2)。
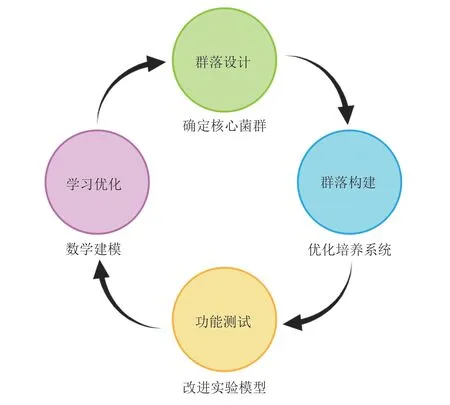
图2 合成肠道微生物群落面临的挑战Fig. 2 Challenges in synthesizing the gut microbiome
综上,选择合适的数学模型有助于更好地分析肠道微生物群落数据、理解微生物群落中的相互作用并优化肠道微生物群落的组成。
6 总结与展望
自上而下的宏组学研究方法及自下而上的合成生物学技术手段在肠道微生物群落研究中均发挥着独特的作用,具有较大的应用潜力,但仍有较多问题亟待解决。例如,需要优化宏组学大数据分析方法,获得更准确的微生物代谢及相互作用等信息;需要确定在不同类型肠道疾病治疗中,什么情况下合成微生物群落较单菌更具有优势;微生物群落的物种数量是否存在最优解;如何优化细胞通信基因回路、改进微生物与宿主代谢互作网络提高合成微生物群落各组分在肠道中承担复杂任务、发挥特定功能的能力[99];如何通过揭示控制肠道微生物自身及其与宿主的相互作用规律帮助我们理性设计肠道微生物群落,从而帮助改善人体健康;随着测序技术的进一步发展,如何整合繁杂的宏组学数据进一步优化微生物群落用于疾病监测与治疗。未来应深化宏组学、合成生物学与其他学科的交叉,充分利用宏组学的大数据特征及合成生物学的定量、工程化等优势,进一步开展对天然微生物群落相互作用机制的研究,最终设计一套普适性强、稳定性高、精确可控的合成微生物群落构建方法,以应用于人类健康事业。
