利用转录组学分析三峡肽素生物合成的基因簇
2021-07-23白晓轩杨宇纯薛艳红刘士平
刘 超,李 坤,白晓轩,杨宇纯,薛艳红,刘士平
(三峡大学生物与制药学院,中国轻工业功能酵母重点实验室,湖北宜昌 443002)
近年来,寡肽类抗生素在医药、食品、农业和饲料添加剂等领域正在发挥越来越大的作用[1−4],如环孢霉素等。与传统的抗生素相比,寡肽结构多样、广谱高效、分子量小、稳定性好、溶解度高且不易引起耐药性,受到了科学家的广泛关注[5−8]。
寡肽类天然产物在生物体中有两种合成途径,即核糖体途径和非核糖体途径[9]。前者是指通过依赖核糖体的翻译途径来合成,通过该途径合成的寡肽是一种初生代谢产物,在其基因组中一般会存在编码目标寡肽的氨基酸重复序列[10−12],其结构组分一般是20 种天然的氨基酸,如羊毛肽、硫肽和半乳糖肽[13];而后者是由一种不依赖于翻译途径的非核糖体肽合成酶(non-ribosomal peptide synthetase,NRPS)所催化,通过该途径合成的寡肽是一种次级代谢产物,其结构中大都含有许多非天然的氨基酸[14−16],如环肽素等[17]。为了方便研究寡肽合成的NRPS基因簇,科学家们根据非核糖体肽的结构和NRPS的序列及催化特点,开发出一系列预测软件[18],如antiSMASH、fungiSMASH、NRPS predictor、Norine等,为解析寡肽生物合成途径提供了有利条件[19−21]。特别是随着转录组测序技术(RNA-Seq)的应用[22−23],人们可以方便地从表达水平上确定候选的合成基因簇[24−25],如刘伟等[26]在2018 年利用转录组测序技术,筛选出了银杏类黄酮生物合成途径的关键基因,并分析了各基因的表达特征,确定了银杏黄酮醇类物质合成的限速酶基因。
在前期工作中,从三峡地区的疏花水柏枝(Myricaria laxiflora)的内生草酸青霉SG-4(CCTCC M2015270)中,发现了一种新线性五肽“三峡肽素”,结构是:O-Me-BABA-N-Me-L-Thr-D-Thr-N-Me-LVal-L-Ser,其中BABA是β-氨基丁酸,能抑制柑橘致腐菌,对人体安全,是一种潜在的保鲜剂[27−28]。为了揭示三峡肽素在草酸青霉中的合成机制,本研究在完成不同草酸青霉菌株的转录组分析的基础上,测定了SG-4 在不同培养条件下的转录组序列,为剖析三峡肽素的合成机理提供了实验依据。
1 材料与方法
1.1 材料与仪器
土豆、葡萄糖 科密欧化学试剂有限公司;琼脂 金燕海洋生物股份有限公司;色谱级乙腈 美国天地有限公司;真菌RNA提取试剂盒 美国Omega Bio-Tek公 司;Goldenstar™ RT6 cDNA Synthesis Kit Ver.2、T3 Super PCR Mix北京擎科新业生物技术有限公司。
BS-224s电子天平 德国赛多利斯;SW-CJ-2FD型双人实验操作台 苏州净化设备厂;XPX-9052 MBE数显培养箱 上海博讯实业有限公司医疗设备厂;ZQZY-85BN全自动控温摇床 上海知楚仪器有限公司;Master-s15 和泰纯水仪 上海和泰仪器有限公司;MyCyclerTM Thermal PCR仪 美国BIO-RAD公司;GDS-8000 凝胶成像系统 美国UVP公司;Allegra 64R台式高速冷冻离心机 美国BECKMAN公司;DYCP-31DN水平电泳仪 北京市六一仪器厂;YM30F不锈钢智能型立式电热蒸汽消毒器 上海三申医疗器械有限公司;SPD-16 岛津高效液相色谱仪紫外-可见光检测器 岛津仪器苏州有限公司;C8液相柱,YMC-Triart C8(250 mm×4.6 mm)日本YMC公司。
1.2 实验方法
1.2.1 实验菌株及培养条件 草酸青霉(P.oxalicum)SG-4 为来源于三峡河岸带植物疏花水柏枝(M.laxiflora)的内生真菌,菌株保藏于4 ℃的PDA固体培养基斜面。
PDA固体培养基:土豆200 g,葡萄糖20 g,琼脂15~20 g,水1000 mL,pH自然,121 ℃灭菌20 min。
草酸青霉SG-4 经活化培养以后,采用无菌操作取 1 环孢子分别接种于装有PDA液体培养基的三角摇瓶中,培养条件为28 ℃、120 r/min。
1.2.2 菌体量测量和三峡肽素含量检测 将SG-4接种至200 mL PDA液体培养基中,培养条件按1.2.1 中进行,培养2~9 d,每天获取菌丝和发酵液,菌丝烘干后称量,记录2~7 d的菌体量;发酵液使用等体积无水乙醇混匀沉淀24 h,将上清液旋蒸冻干获得粗提物,使用纯水将粗提物配制成5 mg/mL的溶液,按照杨宇纯等[27]检测方法,记录2~9 d的三峡肽素出峰的峰面积。
具体检测方法如下:利用C8色谱柱及高效液相色谱设备进行分析。流速1 mL/min,柱箱温度为35 ℃,流动相A为乙腈,流动相B为纯水。梯度洗脱:0~20 min 90%~0% B。进样量为80 μL,检测波长为210 nm,记录5~6 min出峰的峰面积。
1.2.3 SG-4 在不同培养条件下三峡肽素含量检测将SG-4 分别接种至200 mL和300 mL的PDA液体培养基中,其中200 mL分别培养4 d和7 d,300 mL培养7 d,培养条件及三峡肽素检测按照1.2.1 和1.2.2 中进行。
1.2.4 RNA提取及转录组测序 离心获取200 mL培养4、7 d及300 mL培养7 d的菌丝体,液氮研磨后采用OMEGA试剂盒提取总RNA,提取方法按试剂盒操作方法进行,使用Agilent 2100 和NanoDrop对RNA浓度、OD值(OD260nm/280nm和 OD260nm/230nm)、28S/18S、RIN/RQN值进行检测,根据结果对RNA进行质量评估。将质量检测合格的RNA样品使用BGISEQ-500 进行高通量转录组测序,操作简述如下:先用带有OligodT的磁珠富集有polyA尾巴的mRNA,再用DNA探针杂交rRNA,利用RNase H选择性消化DNA/RNA杂交链,再用DNase I消化掉DNA探针,纯化后即得到所需RNA。然后将RNA片段化,用随机的N6 引物进行反转录,进一步形成双链DNA。将双链DNA末端补平并进行5’端磷酸化,3'端形成突出一个“A”的粘末端,再连接一个3'端有凸出“T”的鼓泡状的接头。然后连接产物通过特异的引物进行PCR扩增和上机测序。
1.2.5 转录组数据分析 转录组数据使用过滤软件SOAPnuke进行统计,使用Trimmomatic进行过滤,得到clean reads;使用Bowtie2 软件将clean reads比对到参考基因序列上。根据比对结果,利用RSEM软件进行定量分析,并以FPKM计算基因的表达水平。差异表达分析重点在于找出样本之间差异表达的基因,并对这些基因进行深入挖掘分析。在分析中,差异表达基因默认定义为 FDR≤0.001 且倍数差异在1 倍以上的基因。筛选获得的差异表达基因,根据GO和KEGG注释结果进行分类,同时使用R软件中的phyper函数进行富集分析。同时将检测到的基因进行NR和NT注释,明确基因所在物种以及基因功能。
1.2.6 RT-PCR验证 使用Oligo 7 软件设计引物,由生工生物工程(上海)股份有限公司合成,使用反转录试剂盒Goldenstar™ RT6 cDNA Synthesis Kit Ver.2 将所提RNA反转录成cDNA,添加引物进行RT-PCR验证,所用的引物序列见表1。

表1 实验中所用的引物序列Table 1 Primer sequences in the experiment
1.3 数据处理
数据处理软件使用Graph Pad Prism 8。
2 结果与分析
2.1 草酸青霉SG-4 不同培养条件下生产能力比较
为了明确三峡肽素的合成特点,分析了SG-4 菌体生长规律和合成三峡肽素的时期,结果显示,菌体的生长在第6 d质量达到最大(图1A),而三峡肽素在第4 d开始累积,第8 d达到最大值(图1B),而在300 mL培养下,SG-4 不产三峡肽素(图2A,箭头标记处),暗示三峡肽素生物合成的基因受培养时间和装夜量的影响,这为研究三峡肽素合成机制提供了线索。由于基因表达与物质合成呈相关性[28],因此,三峡肽素在不同培养条件下的含量差异与其控制合成的基因表达量相对应,所以选取SG-4 在200 mL分别培养4、7 d以及300 mL培养7 d的菌丝提取RNA进行高通量转录组测序,通过对差异基因分析,挖掘三峡肽素合成基因簇。
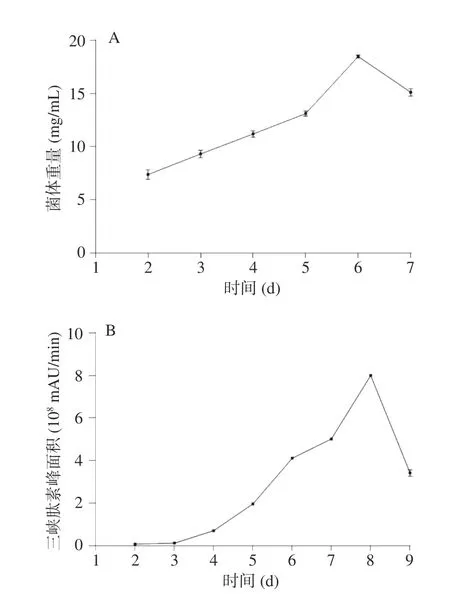
图1 SG-4 的生长(A)和三峡肽素的生产曲线(B)Fig.1 Growth curve (A) of SG-4 and production curve of sanxiapeptide (B)
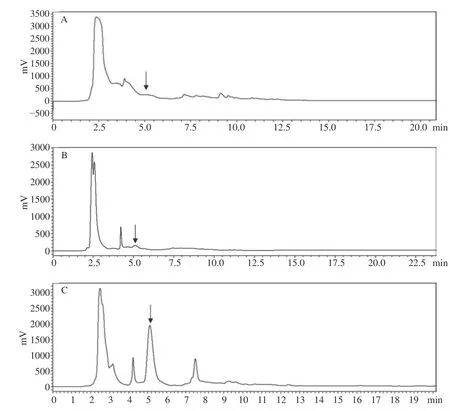
图2 三峡肽素在不同培养条件下液相检测Fig.2 Determination of sanxiapeptide in different culture conditions by liquid chromatography
2.2 转录组测序质量分析
将测序的原始数据(raw reads)中的低质量、接头污染以及未知碱基含量过高的数据过滤后,平均每个菌株产生了4278 万条净序列(clean reads),平均容量达6.42 Gb,高质量数据高达97.19%。使用Bowtie 2 软件将获得的clean reads比对到参考基因序列上,平均比对率达到93.88%(表2),上述结果表明,此次测序的质量有保证,结果可靠;同时使用Trinity对clean reads进行组装,然后使用Tgicl对转录本进行聚类去冗余得到Unigene,获得的All-Unigene为 22731 条,将 Unigene与 NR (RefSeq non-redundant proteins) 数据库进行BLAST比对,发现本样品SG-4 转录组测序得到的Unigene与草酸青霉114-2 的Unigene相似数目最多,高达85.08%(表3),其它占比则非常少。
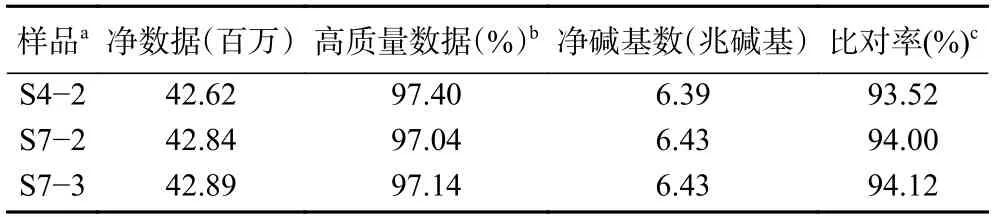
表2 转录组测序概况Table 2 A survey of transcriptome sequencing

表3 NR物种注释Table 3 NR Species notes
2.3 差异表达基因分析
由于三峡肽素含量差异会体现在基因表达差异上,因此按照差异的倍数(|log2(FoldChange)|)和错误发现率(FDR)来筛选差异表达基因[29],分析中将同时满足|log2(FoldChange)|≥1 且 FDR ≤0.001 的转录本确定为差异表达基因[30−31]。三个转录组中的表达发生显著变化的基因数见表4。

表4 差异表达的基因数Table 4 Number of genes differentially expressed
上述结果表明,高产(S7-2)相较于低产(S4-2)和不产(S7-3),其差异表达的基因占比相近,且明显集中在上调基因中,这表明在它们之间,五肽调控存在一定的共性,同时与三峡肽素含量差异也呈现一致性;而高产和低产相较于不产,其差异基因均达到了27%以上,特别是低产和不产之间,差异基因显著存在于下调基因,这表明在它们之间,五肽合成调控存在差异性。由于控制三峡肽素合成的基因簇相同,因此S7-2 与S4-2、S7-3 差异基因会趋于一致,且呈现上调趋势,所以在后期的比较中,重点关注在S4-2 对S7-2 ∩ S7-2 对S7-3 中的3312 个基因,特别是上调基因(图3)。
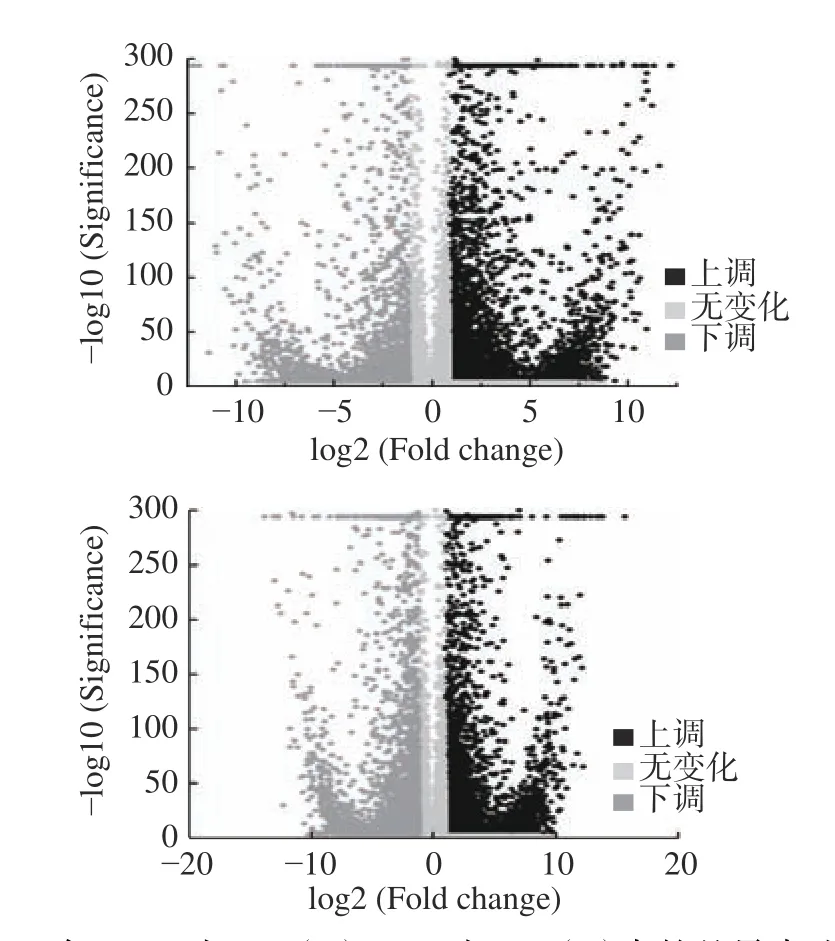
图3 在S4-2 对S7-2(A)S4-2 对 S7-3(B)中的差异表达基因Fig.3 Differentially expressed genes in S4-2 vs S7-2 (A)and S4-2 vs S7-3 (B)
2.4 差异表达基因的功能注释
对于差异表达的基因分别采用GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Genesand Genomes)分类富集分析了其可能的功能及所参与的细胞代谢过程。GO分类表明上述3312 个基因,主要参与细胞进程、代谢过程、细胞膜、催化活性以及连接相关等生物学过程中(图4A)。而KEGG分类表明差异集中在运输和分解代谢、翻译、碳水化合物代谢以及氨基酸代谢等过程中(图4B)。GO富集表明差异基因主要在细胞膜成分、碳水化合物代谢以及单加氧酶活性等过程中(图4C),KEGG富集表明差异基因主要集中在抗生素合成、糖酵解和糖异生过程以及氨基酸合成等途径(图4D)。

图4 差异基因表达分析Fig.4 Differential gene expression analyses
2.5 NRPS基因家族表达分析
在此之前,本课题组通过对不同草酸青霉菌株(114-2、SG-4、SJ-3、114-2 来源于山东大学微生物技术国家重点实验室,SG-4 和SJ-3 菌株均系三峡河岸带植物疏花水柏枝的内生真菌;相同培养条件下,114-2 不产三峡肽素,而SJ-3 产量高于SG-4)进行了转录组分析[32],筛选出7 个可能的NRPS基因簇(表5),结合实验数据,预测三峡肽素合成基因簇为PDE_01071(Non-ribosomal peptide synthetase,NRPS基因簇);因此,对此次转录组进行NR和NT注释,注释结果与预测基因簇PDE_01071 进行比对,结果如下表,其中Unigene5452_All包含PDE_01066 和PDE_01067 两个基因的部分CDS序列;同时,也对上述3312 个差异基因进行了比对,发现其中有21 个差异基因与PDE_01071 基因簇一致,且在S7-2 中均为上调基因(表6);而且RT-PCR结果与测序表达结果完全一致(图5A),为了保证结果的准确性,也对另外6 个可能的NRPS基因簇进行了表达验证(图5B),RT-PCR结果表明6 个基因表达结果和转录组数据不一致,因此将这6 个基因排除,该结果进一步说明PDE_01071基因簇可能参与草酸青霉中三峡肽素的合成。

图5 RT-PCR验证表达结果Fig.5 RT-PCR to verify the expression
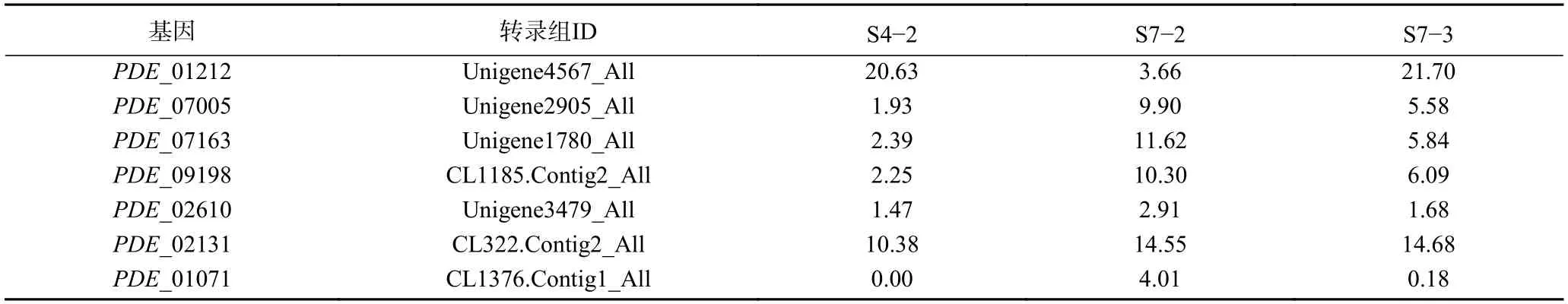
表5 7 个可能的NRPS基因在不同培养条件下的表达量Table 5 Expression levels of the 7 possible NRPS genes under different culture conditions
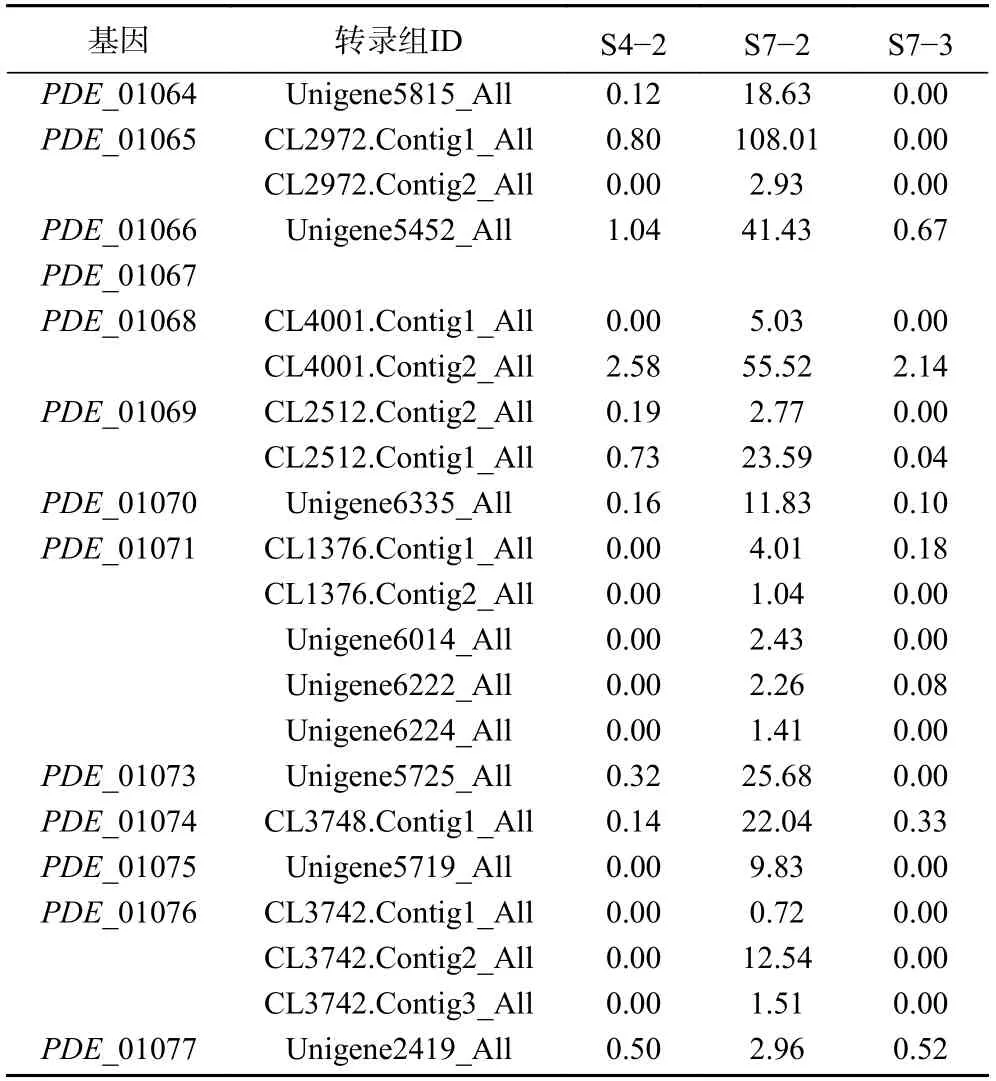
表6 差异基因与基因簇PDE_01071 比对Table 6 Comparison of differential genes with gene cluster PDE_01071
3 结论
通过分析SG-4 菌体生长规律和合成三峡肽素的时期,表明菌体的生长在第6 d质量达到最大,而三峡肽素在第4 d开始累积,第8 d达到最大值,因此三峡肽素合成特点符合次生代谢途径,同时通过对草酸青霉基因组序列进行检索,并未找到Ser、Thr、Val的重复编码序列,所以三峡肽素合成途径符合NRPS途径;由于三峡肽素含量与其控制合成的基因表达相关,所以草酸青霉SG-4 在不同培养条件下的含量差异与基因表达差异会趋于一致,因此本研究基于BGISEQ-500 测序平台,分析比较了草酸青霉SG-4 在不同培养条件下无参转录组测序数据,对差异表达基因进行了分析,将可能的合成基因锁定在3312 个差异基因中;由于此前完成了不同草酸青霉(114-2、SG-4、SJ-3)转录组测序及分析,分析结果认为NRPS基因簇PDE_01071 可能参与三峡肽素的合成,因此将3312 个基因同此基因簇进行NR及NT比对,比对结果表明:3312 个差异基因中有21 个基因与基因簇PDE_01071 基因一致,而这21 个基因均为上调基因,且集中在高产样本S7-2 中,这更进一步验证了此前的猜想,而所有这些尝试为明确其生物合成机制奠定了基础。后面的工作中,需对基因簇成员进行针对性敲除,在明确酶活性和基因功能的基础上,对其合成机理进行解析。
