不同生育期转基因抗虫棉根际土壤细菌群落特征
2021-07-17王甜甜陈彦君李俊生
王甜甜, 闫 冰, 陈彦君, 关 潇, 李俊生*
1.中国人民大学环境学院, 北京 100872
2.江西省科学院能源研究所, 江西 南昌 330096
3.海南大学热带农林学院, 海南 海口 570228
4.中国环境科学研究院, 国家环境保护区域生态过程与功能评估重点实验室, 北京 100012
转基因抗虫棉在棉花虫害预防中发挥重要作用,其大规模的商业化种植极大地提高了投入产出比,带来了巨大的经济效益;与此同时,Bt蛋白在土壤中的释放和持续残留,也对自然和农田产生了潜在生态环境风险[1-3]. 转Bt基因抗虫作物根际分泌的Bt蛋白在土壤中的吸附和积累可能影响根际土壤微环境[4-5],对土壤生态系统产生一定的影响[6]. 土壤微生物作为生物地球化学循环的重要驱动者[7-8],对土壤肥力、植物生长与健康、有机物质的周转以及生态系统功能的维持等方面发挥重要作用,在农业生态系统中扮演着重要角色. 土壤微生物群落通常被认为是反映土壤生态系统变化的早期敏感指标[9],然而转Bt基因抗虫棉对土壤微生物群落的潜在生态影响仍然知之甚少.
已有研究[10]发现,转Bt基因作物对土壤微生物群落结构产生影响. 转Bt基因抗虫棉可能会刺激细菌和真菌生长,导致土壤微生物数量升高,改变土壤微生物群落组成,进而增加土壤微生物群落多样性[11]. 然而,有研究[12-13]认为转基因作物并未造成土壤微生物群落的显著变化. Rui等[14]发现,转Bt基因抗虫棉根际土壤可培养功能细菌数量仅在棉花生长期与亲本对照之间呈显著差异,且纯化Bt蛋白添加试验发现,除固氮菌外,其余功能细菌数量未受到Bt蛋白添加的显著影响,即Bt蛋白不是导致根际土壤细菌数量减少的直接因素. 基于16S rRNA高通量测序技术研究转Bt基因作物根际细菌群落的研究[15]表明,一些稀有菌门在根际微生物群落中占重要位置,其可调节植物营养的吸收和土壤养分的循环. 然而,作物不同生育期对养分吸收不同,不同生育期转基因作物基因漂移发生的频率存在差异,因此不同生育期转基因抗虫棉是否对土壤微生物群落产生影响仍存在不确定性,且其群落变化过程有待进一步研究.
因此,该研究选取转Bt基因抗虫棉作为研究对象,基于高通量测序技术分析了不同生育期土壤细菌群落的组成、结构及多样性差异,以揭示转基因抗虫棉不同生育期对土壤微生物群落的影响,并探讨Bt蛋白残留对土壤微生物群落的驱动过程,以期为转基因抗虫棉种植的生态风险评估提供基础数据.
1 材料与方法
1.1 试验地区域概况
该研究在河北省邢台市转基因棉花种植示范基地(115.45°E、37.03°N)进行,所在区域属暖温带亚湿润大陆性季风气候,四季分明,年内温差大. 夏季高温多雨,冬季寒冷干燥,春、秋季短促,全年无霜期203 d,年均气温13.1 ℃,降水量476 mm. 降水季节分配不均匀,全年60%的降水集中在6—8月,土壤质地为砂质壤土. 试验地无转基因抗虫棉及其对照的连续种植史.
1.2 试验设计与土壤样品采集
该试验设置转基因组和亲本组,每组设置4个平行样地,每个样地面积为10 m×15 m,每个样地之间设置20 cm宽的隔离带,用于防止样地间相互干扰. 试验分别于播种前期、苗期、吐絮期进行根际土壤样品采集,取样时采用“抖根法”采集根际微生物样品,采用五点法采样混匀装入无菌自封袋中用冰盒带回实验室于-80 ℃冰箱中保存待用.
1.3 研究方法
1.3.1土壤Bt蛋白残留测定
基于美国Envirologix公司的ELISA试剂盒,测定土壤Bt蛋白残留量,具体操作步骤根据操作说明完成.
1.3.2DNA提取、PCR扩增与高通量测序
土壤总DNA提取采用FastDNA®SPIN Kit for soil(MP Biomedicals, Santa Ana, CA, USA)试剂盒进行,具体操作步骤根据操作说明进行;提取得到的样品总DNA利用琼脂糖凝胶电泳进行定性检测,经SMA4000超微量分光光度计(Merinton, USA)测定其浓度、纯度后,再将提取的总DNA分成3份放置于-80 ℃冰箱保存待用.
采用高通量测序研究土壤细菌群落组成;针对细菌16S rRNA基因V4区,使用通用引物515F (5′-GT GCCAGCMGCCGCGGTAA-3′)和806R (5′-GGACTAC HVGGGTWTCTAAT-3′)进行PCR扩增;PCR反应体系为10×ExTaq buffer 4 μL、dNTPs 2 μL、BSA 0.2 μL、2.5 U Takara Ex Taq DNA聚合酶0.25 μL、引物各1 μL、DNA模板1 μL,灭菌超纯水补足体系至25 μL. PCR反应条件为预变性温度94 ℃ (2 min);变性温度94 ℃ (30 s),退火温度53 ℃ (30 s),延伸温度72 ℃ (45 s),25次循环后72 ℃终止延伸10 min. PCR扩增产物经2%的琼脂糖凝胶检测,使用E.Z.N.A.TM公司的Gel Extraction Kit凝胶回收试剂盒进行纯化,并用QuantiFluorTM-ST (Promega,USA)进行定量分析后,构建Miseq文库,利用Illumina Miseq测序平台完成测序. 该研究测序工作委托北京诺禾致源生物信息科技有限公司完成.
1.4 数据处理与统计分析
高通量原始测序数据的处理用Mothur v1.39.5 软件参照Schlos等[16]标准操作流程进行. 对原始测序数据分离、正反向序列拼接、分配样品名称后进行序列质控,即去除测序过程中产生的低质量序列;然后分析序列并且基于序列相似度(cutoff=97%)分成不同的可操作分类单元(Operational taxonomic units, OTU);采用RDP Classifier v2.2 (http://sourceforge.net/projects/rdp-classifier)对OTU代表序列进行分类学注释,默认置信度为0.7,比对数据库为Greengene 13.5 (http://greengenes.secondgenome.com). 通过Mothur v1.30.2 软件计算alpha多样性值(Shannon-Wiener指数、Simpson指数、Sobs指数、PD指数). 使用FastTree v2.1.3 软件根据最大似然法构建进化树,然后利用FastUniFrac分析得到样本间距离矩阵.
对不同分组数据门水平相对丰度、多样性指数进行两两Student-t检验. 用主坐标分析(Principal coordinates analysis, PCoA)(Bray-Curtis距离)比较根际土壤样品间群落组成的beta多样性差异,并用非参数多元方差分析(ADONIS)进行差异检验. 采用Pearson相关分析探讨土壤微生物门水平相对丰度及多样性指数与Bt蛋白残留的相关性. 该研究所有统计分析及制图均基于R 3.5.1软件不同程序包实现.
2 结果与分析
24个根际土壤样品的高通量测序序列经过质控过滤后,共有 2 002 553 条细菌16S rRNA基因高质量序列. 其中,最少的样品有 68 208 条序列,最多的样品有 97 053 条序列,样品平均序列条数为 83 440. 根据样品中最小序列数,细菌每个样品随机挑选 68 208 条序列进行样品测序深度的均一化处理. 从根际土壤中共获得 8 429 个OTU,细菌序列的平均样品覆盖率分别为98.5%,表明测序取样深度几乎涵盖了大部分的根际土壤细菌.
2.1 不同生育期根际土壤微生物群落组成
所有细菌16S rRNA基因序列比对被分类成47个门、106个纲和1 228个属,有1.03%的序列在门水平不能进行分类. 不同样品细菌门水平(相对丰度>0.01)组成一致,分别为变形菌门(Proteobacteria,相对丰度为 0.433 8~0.499 3)、拟杆菌门(Bacteroidetes,相对丰度为 0.106 0~ 0.136 6)、放线菌门(Actinobacteria,相对丰度为 0.074 9~ 0.146 4)、芽单胞菌门(Gemmatimonadetes,相对丰度为 0.054 2~ 0.072 3)、酸杆菌门(Acidobacteria,相对丰度为 0.053 8~0.089 9)、疣薇菌门(Verrucomicrobia,相对丰度为 0.032 4~0.046 9)、厚壁菌门(Firmicutes,相对丰度为 0.021 6~0.049 5)、蓝细菌门(Cyanobacteria,相对丰度为 0.012 0~0.032 0)、绿弯菌门(Chloroflexi,相对丰度为 0.012 5~0.026 9)、浮霉状菌门(Planctomycetes,相对丰度为 0.013 0~0.018 2),以及未鉴定出的菌门unclassified_k_norank_d_Bacteria和Patescibacteria (见图1).
比较不同生育期转基因抗虫棉及亲本根际土壤的细菌群落组成(见图2)发现,转基因抗虫棉根际土壤酸杆菌门相对丰度在苗期、吐絮期均高于亲本同期(P<0.05),芽单胞菌门在苗期的相对丰度显著低于亲本(P<0.05),放线菌门在吐絮期的相对丰度低于亲本在苗期(P<0.05),疣微菌门在吐絮期的相对丰度高于亲本在播种前期 (P<0.05),绿弯菌门在苗期的相对丰度显著高于亲本同期(P<0.05),蓝细菌门在苗期的相对丰度高于亲本在播种前期. 比较转基因抗虫棉及亲本根际土壤细菌群落组成随生育期的变化发现,亲本根际土壤中变形杆菌门的相对丰度在吐絮期高于苗期(P<0.05),芽单胞菌门的相对丰度在苗期低于播种前期(P<0.05),酸杆菌门的相对丰度在吐絮期低于播种前期(P<0.05),蓝细菌门的相对丰度在苗期低于播种前期(P<0.05);转基因抗虫棉根际土壤中蓝细菌门的相对丰度在吐絮期低于播种前期(P<0.05),而变形杆菌门、拟杆菌门、芽单胞菌门等菌门的相对丰度随生育期变化不显著. 转基因抗虫棉根际土壤中拟杆菌门、厚壁菌门等菌门的相对丰度在各时期与亲本差异不明显.
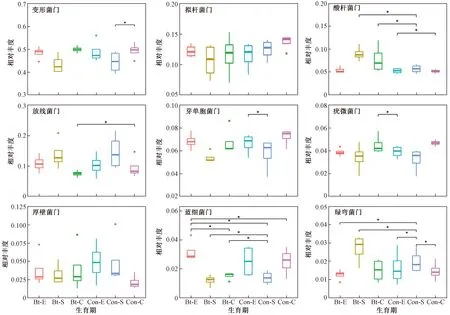
注: *表示P<0.05.
在属水平,共有40.03%的总细菌序列可以进行正确分类,丰度最高的6个属分别为拟杆菌门的鞘脂单胞菌属(Sphingomonas,占6.27%)、变形菌门的根瘤菌属(Rhodanobacter,占4.80%)、放线菌门的Gaiellales属(Gaiellales,占3.18%)、厚壁菌门的芽孢杆菌属(Gemmatimonadaceae,占2.43%)、拟杆菌门的噬几丁质属(Chitinophagaceae,占2.40%)和变形杆菌门的黄单胞菌属(Xanthomonadaceae,占2.33%)(见图3).
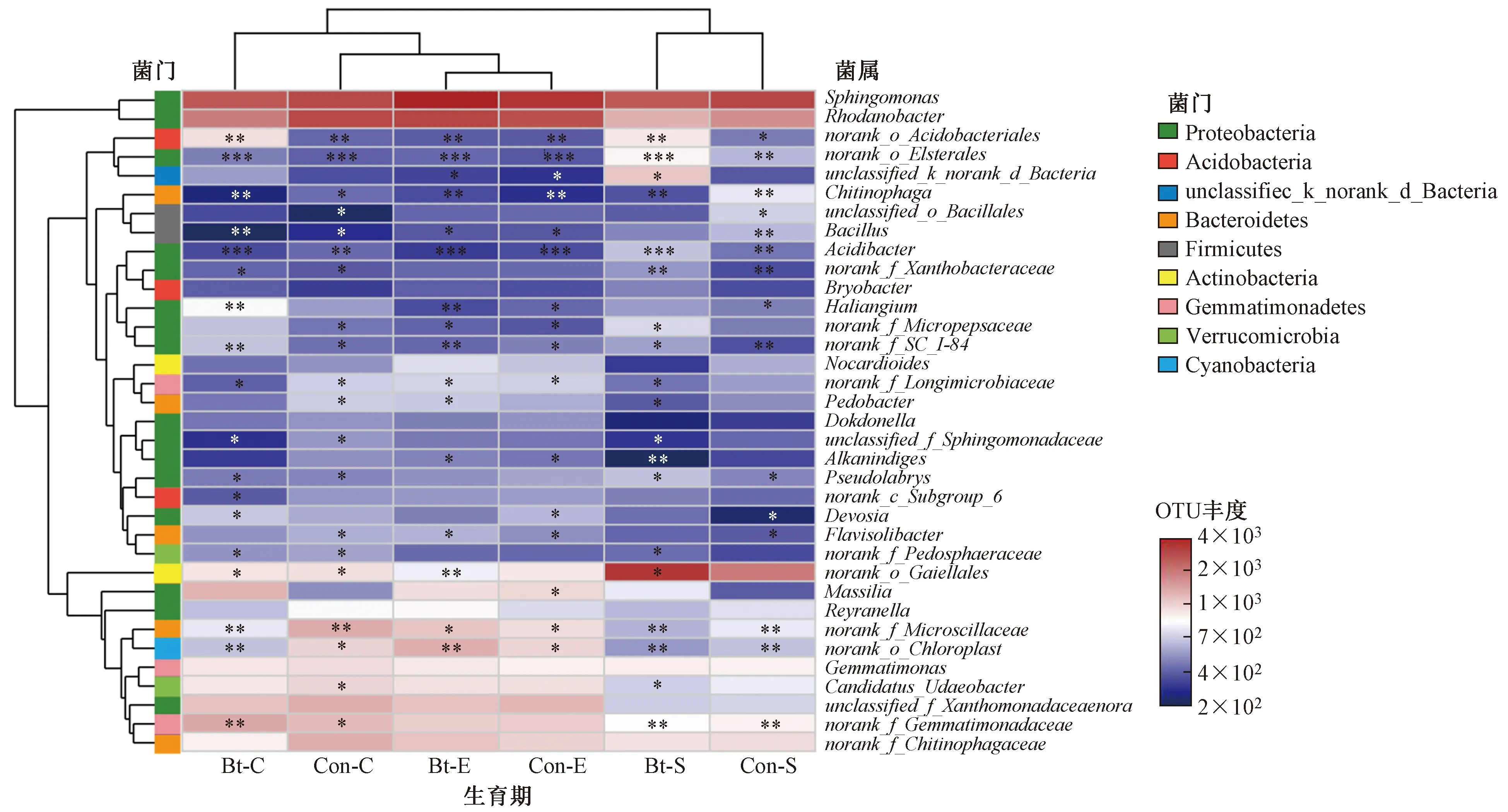
注: *表示P<0.05; ** 表示P<0.01.
根据所有样品在属水平的物种注释,选取相对丰度排名前35位的属绘制成热图. 由图3可见:吐絮期,转基因抗虫棉根际土壤中norank_f_Microscillaceae、norank_f_Longimicrobiaceae等属含量均低于亲本;苗期,转基因抗虫棉根际土壤中norank_o_Acidobacteriales等属显著高于亲本,Chitinophaga属显著低于亲本;播种前期,转基因抗虫棉及亲本根际土壤细菌相对丰度前35位的属差异不显著.
2.2 不同生育期根际土壤细菌群落多样性
转基因抗虫棉与亲本在播种前期、苗期、吐絮期根际土壤细菌群落的alpha多样性(Shannon-Wiener指数、Simpson指数、Sobs指数、PD指数)均无显著差异(P>0.05)(见表1);同时,转基因抗虫棉根际土壤细菌群落alpha多样性在不同生育期也无显著差异(P>0.05)(数据未列出). 这说明相对于亲本而言,转基因抗虫棉对根际土壤细菌群落多样性无显著影响,并且不同生育期对根际土壤细菌群落的多样性也无显著影响.
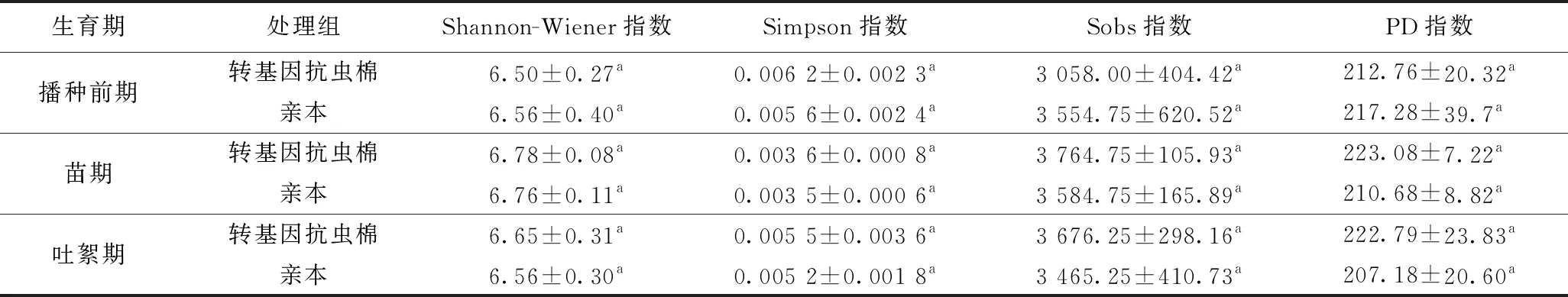
表1 转基因抗虫棉及亲本在不同生育期根际土壤细菌群落的alpha多样性
利用主坐标分析(PCoA)方法对不同生育期转基因抗虫棉根际土壤细菌群落差异进行分析(见图4),结果显示不同生育期根际土壤样品中细菌群落存在明显的空间分异规律(ADONIS,P<0.05). 播种前期,转基因抗虫棉和亲本的土壤细菌群落无显著差异(ADONIS,P=0.411),说明两组样地间土壤细菌群落相似;而苗期和吐絮期,转基因抗虫棉和亲本的土壤细菌群落呈显著差异(ADONIS,P<0.05),表明转基因棉花的种植对土壤细菌群落产生显著影响.
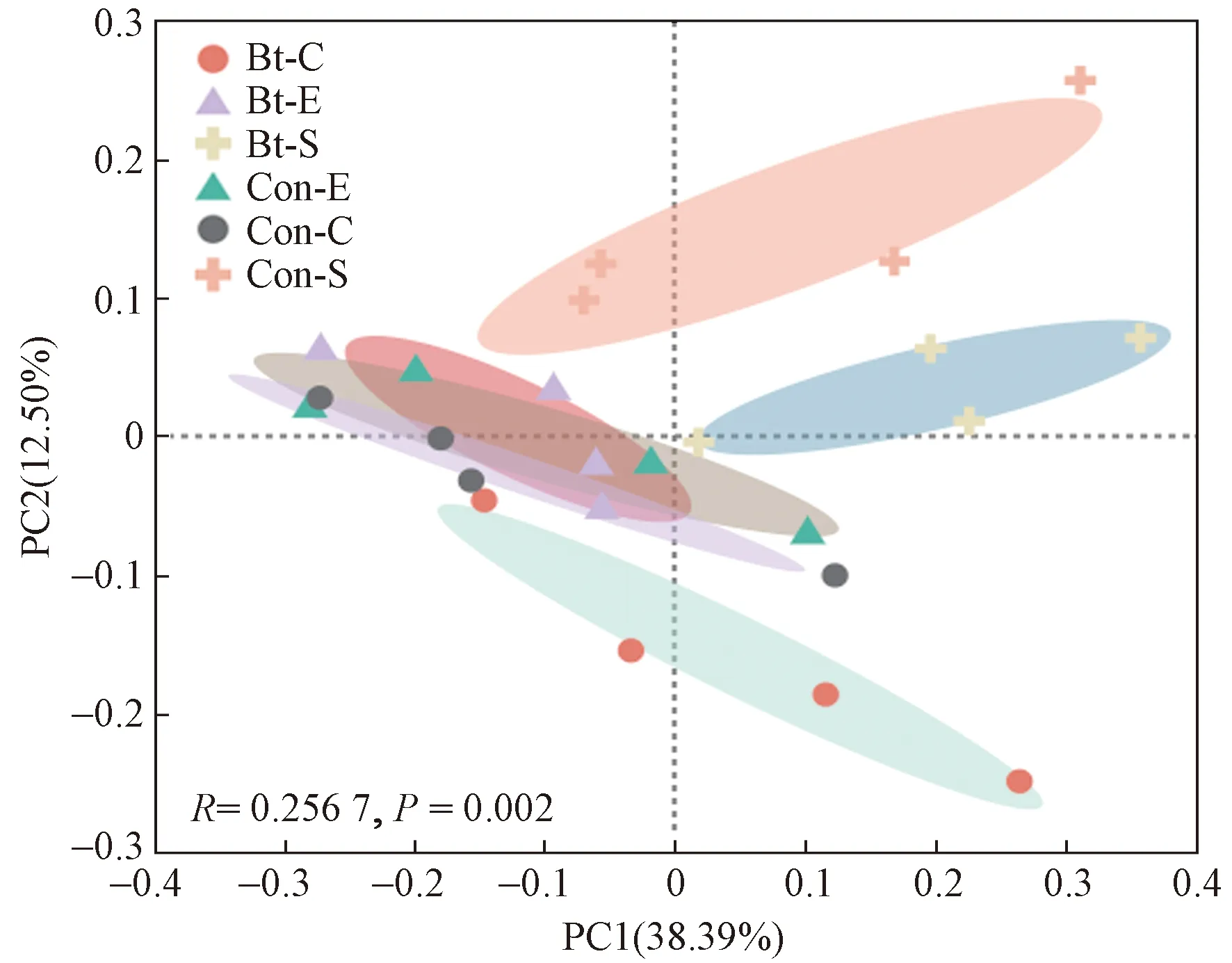
图4 不同生育期转基因抗虫棉及亲本根际土壤细菌PCoA图
2.3 根际土壤Bt蛋白残留及其与菌门相对丰度之间的关系
为了进一步明确转基因抗虫棉种植对土壤细菌群落的影响,分析了转基因抗虫棉及其亲本在不同生育期根际土壤Bt蛋白残留量及其与细菌门水平相对丰度的关系(见图5、6). 亲本根际土壤Bt蛋白本底值在不同生育期无显著差异(P>0.05),转基因根际土壤Bt蛋白残留量在不同生育期呈显著差异(P<0.01),且Bt蛋白残留量在不同生育期呈播种前期<吐絮期<苗期的特征(见图5). 不同生育期对比发现,播种前期转基因抗虫棉和亲本Bt蛋白残留量无显著差异(P>0.05),而苗期和吐絮期转基因抗虫棉和亲本Bt蛋白残留量均呈显著差异(P<0.05).
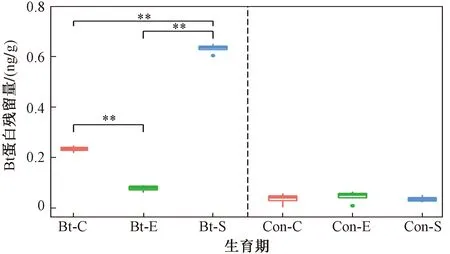
注: ** 表示P<0.01.
对根际土壤Bt蛋白检出量与门水平相对丰度之间进行相关分析(见图6)发现,根际土壤Bt蛋白残留量与Acidobacteria、Armatimonadetes、Chloroflexi、GAL15、WPS-2的相对丰度均呈显著正相关(R分别为0.706、0.569、0.515、0.44、0.413,P均小于0.05),而与BRC1、Cyanobacteria、Gemmatimonadetes的相对丰度均呈显著负相关(R分别为-0.423、-0.461、-0.406,P均小于0.05);同时,相关分析(数据未列出)也发现,根际土壤Bt蛋白残留量与差异菌属gnorank_f_Microscillaceae、norank_f_Longimicrobiaceae的相对丰度均呈显著性负相关(R分别为-0.531、-0.479,P均小于0.05),而与norank_o_Acidobacteriales的相对丰度呈正相关(R=0.637,P=0.001).

注: 图中灰色区域代表95%置信区间.
3 讨论
不同生育期转基因抗虫棉和亲本的根际土壤优势细菌门相同,均为变形菌门、拟杆菌门、放线菌门、芽单胞菌门、酸杆菌门、疣薇菌门、厚壁菌门、蓝细菌门、绿弯菌门和浮霉状菌门,对不同植物根际土壤微生物研究[17-19]也发现上述菌门是优势菌门,表明植物根际土壤微生物优势菌门组成具有高度相似性,同时对棉花根际土壤细菌群落的研究[20]也发现类似结果. 因此,转基因抗虫棉对根际土壤细菌门水平群落组成影响较小,这与Zhang等[21]研究结果一致,其发现转基因棉花和亲本的生长过程不会或很少引起根际土壤细菌优势类群的变化. Dohrmann等[15]通过16S rRNA高通量测序技术研究转基因抗虫棉根际土壤细菌群落发现,一些稀有菌门是根际微生物群落中重要的组成部分. Qiao等[20]研究发现:苗期Rhizoctonia、Alternaria受到促进从而相对丰度升高,Fusarium受到抑制从而相对丰度降低;蕾期Thanatephorus、Verticillium、Gibberella、Fusarium受到促进从而相对丰度升高,Rhizoctonia受到抑制从而相对丰度降低. 然而,该研究对不同生育期根际土壤细菌相对丰度分析发现,棉花生育期对细菌门水平和属水平相对丰度产生显著影响,并且转基因和亲本间在苗期和吐絮期菌群的相对丰度存在显著差异,这可能是由于转基因棉花在不同生育期根际分泌物含量或组成不同所致,但仍需进一步研究验证.
笔者研究发现,转基因抗虫棉及亲本在各生育期,其根际土壤细菌群落alpha多样性无显著差异. 张永鑫[22]通过对转Bt基因抗虫玉米功能多样性指数的检测发现,在多个时期其土壤微生物群落的功能多样性指数高于对照,认为种植转Bt基因抗虫玉米会增加土壤微生物多样性. Zhang等[23]研究发现,转基因抗虫棉秸秆还田30 d后,转基因抗虫棉田土壤微生物群落的Shannon-Wiener和McIntosh指数均显著高于非转基因棉田,且微生物对土壤碳源的利用能力也显著增加,说明转基因棉花土壤微生物功能多样性显著高于亲本. Yang等[24]研究也发现,转Bt基因抗虫棉的种植使细菌和真菌多样性增加. 然而,Zhang等[21]研究发现,转基因棉花不同生育期会引起根际土壤微生物群落的大幅波动,但转基因和亲本之间无显著差异;陈彦君等[25]也发现,转基因抗虫玉米种植对根际土壤细菌群落alpha多样性无持续性影响. Zhou等[26]基于1个种植季节的研究也发现,转基因作物种植对根际土壤微生物群落多样性无显著影响. 综上,转基因作物种植对土壤微生物多样性的影响仍存在不确定性,影响机制也不清楚,仍需要进一步深入研究.
笔者研究发现,生育期对根际土壤微生物群落beta多样性产生影响,并且苗期和吐絮期转基因与亲本根际土壤微生物群落也存在明显差异,与Qiao等[20]研究结果一致. 已有研究[27-29]发现,根际微生物受到植物发育阶段的显著影响. Boudoin等[30]研究发现,植物生长过程中根系分泌物的数量和质量会发生变化,从而导致不同发育阶段根际微生物群落组成的变化. 因此,推测转基因植物与亲本根际土壤微生物群落的时空分异可能是不同生育期植物根系分泌物变化所致,而苗期和吐絮期转基因抗虫棉根际土壤Bt蛋白含量的差异也是重要原因之一. 值得注意的是,对转基因大豆根际细菌群落的研究[31]发现,对照和亲本Beta多样性在开花期无显著差异,但在营养期和灌浆期均存在显著差异;而转基因玉米根际土壤细菌群落的Beta多样性与对照相比,在抽穗期和开花期均没有显著差异[32].
与亲本相比,转基因抗虫棉种植使根际土壤Bt蛋白残留量显著增加,且转基因抗虫棉不同生育期根际土壤Bt蛋白残留量呈显著累积现象,与陈彦君等[33]研究结果一致,说明转基因作物种植导致土壤Bt蛋白残留产生累积效应[34-35]. Stotzky[36]认为,完整的Cry1Ab蛋白与土壤颗粒结合可保持其杀虫特性,并在土壤中持续至少234 d. 笔者研究中相关分析表明,根际土壤Bt蛋白含量与细菌8个门的相对丰度均呈显著相关,说明转基因抗虫棉对根际土壤细菌群落的影响可能是Bt蛋白残留所致. 然而,Miethling-Graff等[37]基于长期(3个生长季)试验发现,种植转Bt基因玉米土壤中未发生Bt蛋白的积累,并且种植转基因抗虫棉的土壤中也未检测到Bt蛋白残留累积[38]. 基于土壤微生物功能和碳循环研究[39]发现,长期种植转Bt基因水稻可能不会对土壤生态环境产生显著影响.
4 结论
a) 转基因抗虫棉及其不同生育期对根际土壤细菌优势菌门组成的影响不显著,但对细菌菌门的相对丰度产生显著影响(P<0.05). 不同生育期,转基因抗虫棉及其亲本根际土壤变形菌门、酸杆菌门、芽单胞菌门、蓝细菌门、绿弯菌门的相对丰度存在显著差异(P<0.05).
b) 转基因抗虫棉及亲本在不同生育期对根际土壤细菌群落alpha多样性无显著影响(P>0.05),但PCoA分析发现,苗期和吐絮期转基因抗虫棉和亲本的土壤细菌群落均出现显著空间分异(ADONIS,P<0.05).
c) 转基因抗虫棉导致根际土壤Bt蛋白含量显著增加(P<0.05),且不同生育期存在显著差异(P<0.05),呈播种前期<吐絮期<苗期的特征.
d) 转基因棉花根际土壤Bt蛋白残留量导致土壤细菌群落门水平相对丰度发生变化,其与酸杆菌门、装甲菌门、绿弯菌门的相对丰度均呈显著正相关(P<0.05),而与蓝细菌门、芽单胞菌门的相对丰度均呈显著负相关(P<0.05).
