肝组织特异性基因IGFALS、CYP3A4、SLC22A1和CYP2E1可能与肝癌预后不良相关
2021-07-09张岩岩陈晓彤陈俊辉郑玉宝
张岩岩,曹 静,陈晓彤,陈俊辉,郑玉宝
中山大学附属第三医院感染科,广州 510630
肝癌是全球最致命的癌症之一,70%~90%的原发性肝癌为肝细胞癌,发病率逐年增加,男性患病率高于女性[1-2]。1997至2016年,中国肝癌的患病率从28.22/10万增至60.04/10万,发病率从27.33/10万增至41.40/10万,死亡率从27.40/10万增至31.49/10万,伤残调整生命年(disability-adjusted life years,DALYs)从10 311 308年增至11 539 102年[3]。肝癌的患病率和死亡率仍在持续上升,其不良预后也给国家和家庭带来沉重负担。对于肝癌目前仍缺乏有效治疗手段,除放射线、消融、手术切除、移植和经动脉化学栓塞(transcatheter arterial chemoembolization,TACE)等物理和化学治疗方法外,就只有一些少量的效果甚微的昂贵药物[4]。研究显示,肝癌的危险因素有乙型肝炎病毒(hepatitis B virus,HBV)、丙型肝炎病毒(hepatitis C virus,HCV)、酒精脂肪肝疾病、酒精相关性肝硬化、吸烟、肥胖、糖尿病、铁超载和各种饮食暴露,但肝癌发生发展的具体机制尚未阐明,仍待进一步探索[5-6]。
微阵列和RNA测序技术的发展促进了研究人员对RNA和DNA的进一步研究,已成为生物医学研究的一个重要组成部分[7-8]。目前,美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)的GEO数据库中储存了3万多个系列和100万个基因表达数据样本[9],为深入研究肿瘤基因以及寻找肿瘤生物标志物和预后指标提供了新方法。本研究从GEO数据库中鉴定了差异表达基因(differentially expressed genes,DEGs),并对其功能和关键基因在肝癌发生发展及预后中的作用进行了系统分析。
资料和方法
DEGs筛选从NCBI的GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)选择GSE57957、GSE121248、GSE36376和GSE14520 4个数据集,采用GEO数据库官网提供的Analyze with GEO2R在线工具进行整合和差异分析,以P<0.05和|log2FC|>1为标准筛选出4个数据集中的DEGs。使用VENN图软件分别显示4个数据集的上调和下调基因元素集合重叠区域,将前20个上调基因和前20个下调基因以热图形式呈现。
基因本体富集和京都基因与基因组百科全书通路分析采用DAVID6.8(https://david.ncifcrf.gov)在线工具进行基因本体(gene ontology,GO)分析和京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)分析,P<0.05被认为具有统计学意义。GO注释内容包括生物过程(biological process,BP)、细胞成分(cellular components,CC)和分子功能(molecular function,MF)3大类。
蛋白质相互作用网络构建及分析采用相互作用基因搜索工具(search tool for the retrieval of interacting genes,STRING)(http://string-db.org)建立蛋白质相互作用网络(protein-protein interaction network,PPIN),然后使用公共生物信息学软件平台Cytoscape3.6.1软件(http://cytoscape.org/)中的插件Cytohubba分析可视化分子相互关联密切程度,分子复合物检测插件(molecular complex detection,MCODE)从PPIN中选择枢纽基因的重要模块(其中Degree Cutoff=2,Node Score Cutoff=0.2,K-core=2和Max.Depth=100)。
GEPIA和HPA筛选关键基因以P<0.05,|log2FC|>2为标准从DEGs中筛选出差异表达显著的上下调基因。为了分析这些基因的总体存活率,采用基因表达谱交互式分析(gene expression profiling interactive analysis,GEPIA)(http://gepia.cancer-pku.cn)进行生存和统计学分析,以log rankP<0.05为标准鉴定出主要表达在肝脏组织中且预后明显较差的关键基因。同时,将基于抗体和抗原之间结合的高度特异性的方法与转录组学数据相结合,使用人类蛋白质图谱(human protein atlas,HPA)(https://www.proteinatlas.org)观察比较在正常组织和肝癌组织中这些关键基因蛋白质的表达水平。
统计学处理GEO2R采用limma包筛选差异表达基因,GEPIA采用one-way ANOVA方差分析方法进一步验证,P<0.05为差异有统计学意义。
结 果
DEGs鉴定GSE57957、GSE121248、GSE36376和GSE14520 4个数据集共包含1063个样本,其中573个肝癌组织和490个邻近的癌旁组织。通过GEO中的GEO2R整合分析鉴定,以P<0.05和|log2FC|>1为筛选标准筛选出DEGs,并采用RRA绘制火山图,呈现每个数据集中上下调基因(图1)。GSE57957、GSE121248、GSE36376和GSE14520数据集中分别筛选出109、306、423和486个显著上调基因及308、590、266和557个显著下调基因,采用VENN图软件从4个数据集中得到45个共同上调基因和132个共同下调基因(图2),前20个上调基因和前20个下调基因见图3。
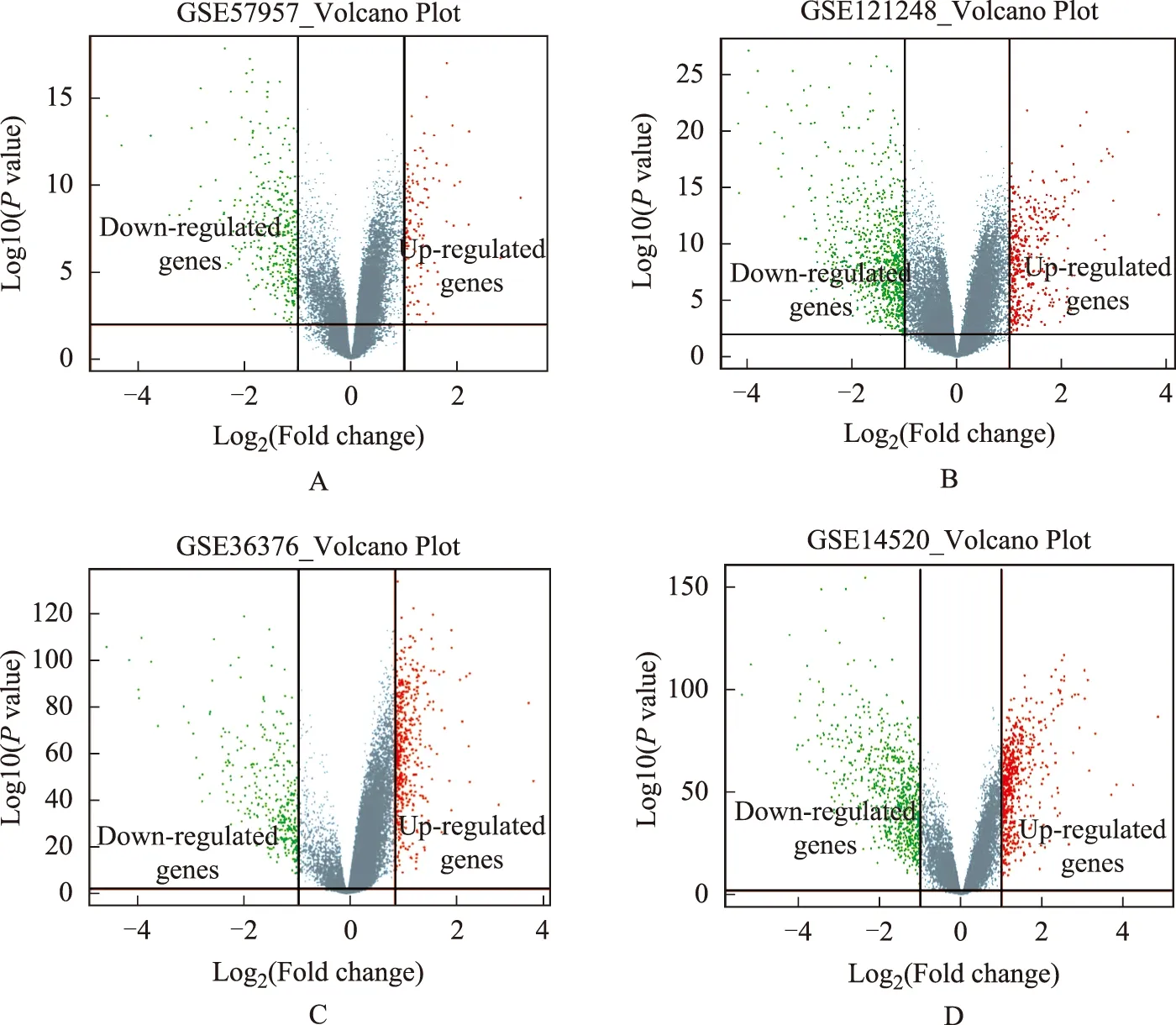
A.GSE57957;B.GSE121248;C.GSE36376;D.GSE14520图1 健康对照组与肝癌组差异表达基因火山图Fig 1 Volcano plots of differentially expressed genes in the healthy control group and liver cancer group
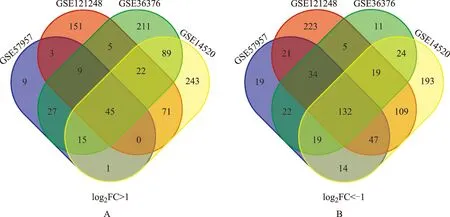
A.4个数据集中的45个共同上调基因(log2FC>1);B.4个数据集中的132个共同下调基因(log2FC<-1)A.45 differentially expressed genes were up-regulated in the four datasets(log2FC>1);B.132 differentially expressed genes were down-regulated in four datasets(log2FC<-1)图2 4个数据集上调基因交集和下调基因交集Fig 2 Intersections of significantly up-regulated and down-regulated genes in the four datasets

横坐标代表四个数据集,纵坐标代表基因名称,红色代表log2FC>1,绿色代表log2FC<-1,值代表log2FC值The abscissa represents the four data sets,and the ordinate represents the gene name;the red represents log2FC>1,the green represents log2FC<-1,and the value represents the log2FC value图3 前20个上调基因和前20个下调基因的log2FC热图Fig 3 Log2FC heatmap of the top 20 up-regulated genes and the top 20 down-regulated genes
DEGs的GO功能富集及KEGG通路分析采用DAVID6.8在线数据库分别对筛选出的45个共同上调基因和132个共同下调基因进行GO和KEGG分析,结果显示:上调DEGs主要富集注释:BP(氧化还原作用、有丝分裂核分裂、视黄醇代谢和胶原分解过程),CC(细胞核、胞浆、细胞外空间和外泌体),MF(蛋白结合、氧化还原酶活性、微管结合和细胞外基质结构组成);KEGG途径(M受体相互作用、细胞分裂、二次循环和消化吸收通路)。下调DEGs主要富集注释:BP(氧化还原作用、环氧合酶P450途径、细胞对锌离子的反应、生长负性调节、异型生物质和药物的代谢过程),CC(细胞外区域、细胞外泌体、细胞器膜、血液微粒和细胞外空间),MF(金属离子结合、血红素结合、氧化还原酶活性、铁离子结合和同源蛋白结合);KEGG途径(代谢通路、凝血级联反应、致癌通路和细胞色素P450对异种生物的诱导作用)(图4)。
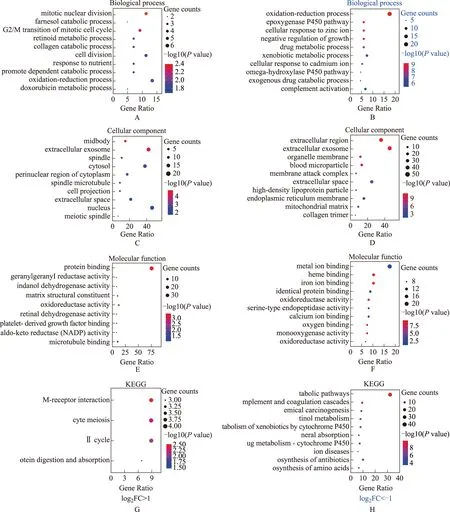
KEGG:京都基因与基因组百科全书KEGG:Kyoto Encyclopedia of Genes and GenomesA.生物过程(log2FC>1);B.生物过程(log2FC<-1);C.细胞成分(log2FC>1);D.细胞成分(log2FC<-1);E.分子功能(log2FC>1);F.分子功能(log2FC<-1);G.KEGG(log2FC>1);H.KEGG(log2FC<-1)A.biological process(log2FC>1);B.biological process(log2FC<-1);C.cellular component(log2FC>1);D.cellular component(log2FC<-1);E.molecular function(log2FC>1);F.molecular function(log2FC<-1);G.KEGG(log2FC>1);H.KEGG(log2FC<-1)图4 显著上调基因和下调基因的GO富集与KEGG通路分析Fig 4 GO enrichment and KEGG pathway analysis of significantly up-regulated and down-regulated genes
PPIN网络构建和分析STRING建立了177显著表达基因的PPIN,其中152个DEGs被过滤到PPIN复合体中,该复合体包含153个节点和607个边(average node degree:6.9,avg.local clustering coefficient:0.46,PPI enrichmentP-value:<1.0e-16)。采用Cytoscape3.6.1的插件Cytohubba分析可视化Hub基因的相互关联密切程度,结果显示,MCODE聚类发现13个模块,score值较大的Cluster1和Cluster2分别包括16个基因和13个基因,其中Cluster1基因分别为NCAPG、PRC1、KIAA0101、TOP2A、PTTG1、MCM4、RFC4、CDC20、MELK、AURKA、CCNB2、NUSAP1、CDKN3、ASPM、RACGAP1、HMMR(图5)。

A.蛋白质-蛋白质相互作用网络;B.Cytoscape3.6.1的插件Cytohubba分析可视化分子之间相互关联的密切程度,颜色越深,则关联越多;C和D:通过Cytoscape软件进行的模块分析:聚类模块1和聚类模块2(Degree cutoff=2,Node score cutoff=0.2,K-core=2,Max Depth=100)A.protein-protein interaction network;B.the Cytoscape3.6.1 plug-in CytoHubba visualizes the degree of molecular associations,and darker color represents stronger association;C and D:cluster 1 and cluster 2(Degree Cutoff=2,Node Score Cutoff=0.2,K-core=2,Max.Depth=100)identified by the Cytoscape plug-in MCODE图5 蛋白质相互作用网络及紧密关联程度分析Fig 5 Protein-protein interaction network and the association degree
筛选肝癌预后相关关键基因以P<0.05及|log2FC|>2进一步筛选出差异表达特别显著的32个上下调基因,通过GEPIA生存分析发现其中12个基因与预后显著相关,包括:TOP2A、CDC20、SPINK1、AKR1B10、CLEC1B、IGFALS、HGFAC、CYP3A4、SLC22A1、TAT、CYP2E1。进一步分析显示,12个基因中,IGFALS、HGFAC、CYP3A4、SLC22A1、TAT和CYP2E1等6个基因在肝癌中低表达,在肝脏组织中的表达明显高于其他器官或者几乎不表达于其他器官(图6)。
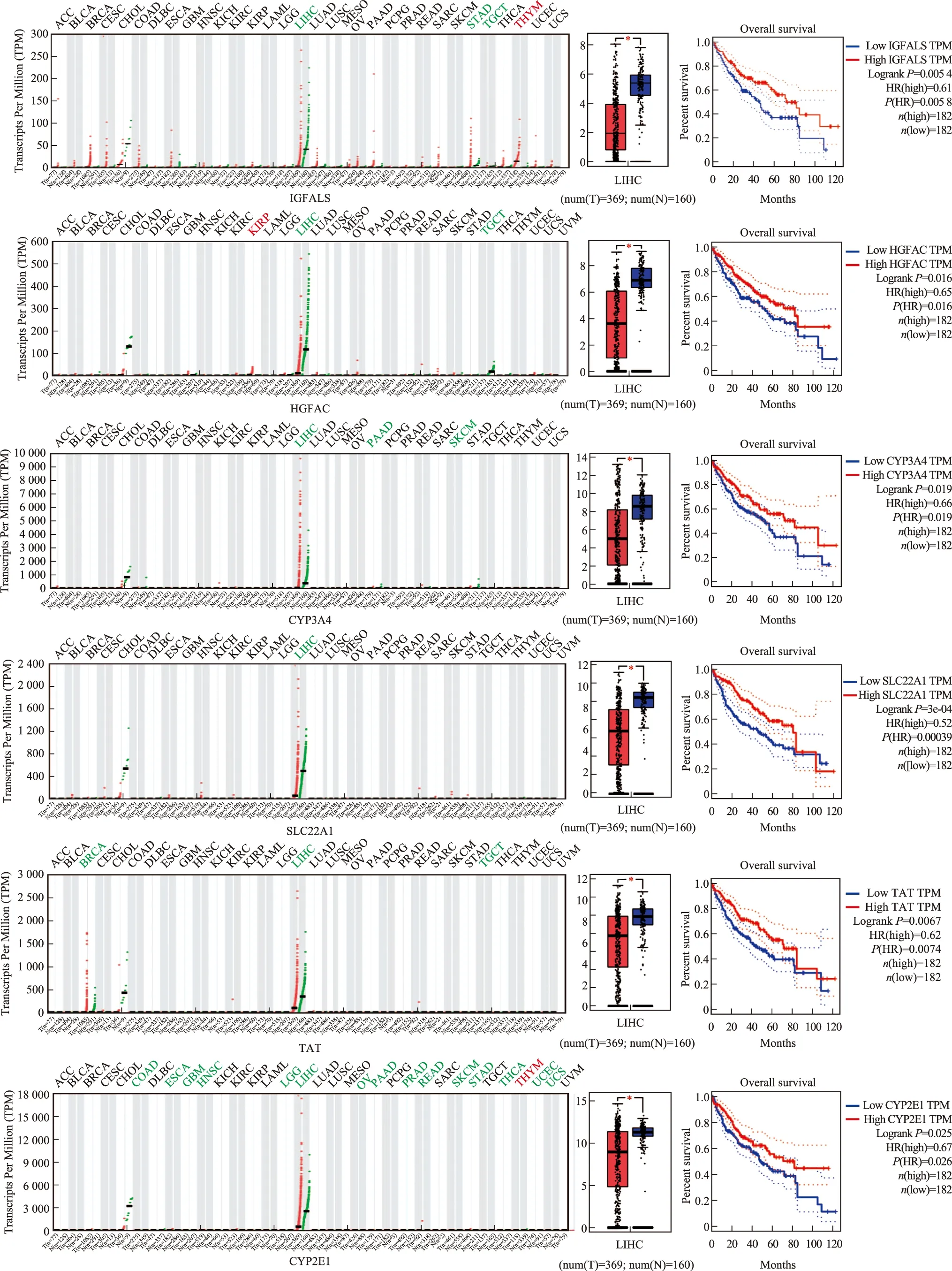
图6 关键基因(IGFALS、HGFAC、CYP3A4、SLC22A1、TAT、CYP2E1)在GEPIA的TCGA和GTEx中表达及预后分析Fig 6 Expression of key genes(IGFALS,HGFAC,CYP3A4,SLC22A1,TAT,CYP2E1)in TCGA and GTEx of GEPIA and prognosis analysis
通过HPA的免疫组织化学(immunohisto-chemistry,IHC)数据库检索发现,与正常组织相比,肝癌组织中的IGFALS、CYP3A4、SLC22A1、CYP2E1均显示下调;HGFAC在正常肝脏与肝癌之间表达的差异不明显;TAT在该数据库中尚未提供相关数据证明(图7)。

图7 关键基因(IGFALS、HGFAC、CYP3A4、SLC22A1、CYP2E1)在正常肝脏组织和肝癌组织中蛋白质的表达Fig 7 Expression of key genes(IGFALS,HGFAC,CYP3A4,SLC22A1,CYP2E1)in normal liver tissue and liver cancer tissue
讨 论
肝癌是全球第6最常见癌症,也是癌症相关死亡的第2常见原因[10],其中,半数以上的新病例和死亡病例发生在中国[11]。肝癌的进展速度快,缺乏有效的靶向药物,患者存活率较低[12]。同时,个体差异、各类危险因素、遗传易感性及微环境差异等极大限制了肝癌的早期发现及治疗进展[13]。因此,研究与肝癌发生发展相关的生物标志物并找到肝癌预后指标,可为未来早发现早治疗从而降低中国肝癌疾病负担提供有效策略。
本研究采用生物信息学方法筛选出在GSE57957、GSE121248、GSE36376和GSE145204数据集中都显著上调的基因45个,下调基因132个,并对其GO和KEGG功能进行富集分析。结果发现,IGFALS、CYP3A4、SLC22A1和CYP2E1基因在肝脏中特异性表达,其在肝癌中的低表达可能与肝癌的不良预后相关。
IGFALS是一种蛋白质编码基因,可与胰岛素生长因子-I(insulin-like growth factor-I,IGF-I)和胰岛素样生长因子结合蛋白质3(insulin-like growth factor binding proteins-3,IGFBP-3)形成三元复合物[14],人胰岛素样生长因子酸不稳定亚基(the insulin-like grouth factor binding protein acid labile subunit,IGFALS)突变可导致三元复合物形成障碍,从而影响乳腺癌和其他癌症的发生和发展[15]。同时,全基因组甲基化分析证明,IGFALS是人类肝癌发生的抑制基因。当IGFALS高表达时会显著降低HuH7细胞的总体活力和克隆形成[16]。转录组序列分析进一步确定了IGFALS是肝癌中优先下调的关键遗传决定因素基因,IGFALS的下调是肝癌发生的遗传生物标记[17]。
CYP3A4是编码酶的细胞色素P450超家族成员,位于内质网,其表达是由糖皮质激素和某些药理作用剂诱导,相关通路包括药物代谢-细胞色素P450和萘代谢。研究发现,CYP3A4可能是通过调节性激素代谢产物水平或者代谢激活一些外源性致癌物,从而在乳腺癌和前列腺癌的发生中发挥重要作用[18];孕烷X受体可通过CYP3A4依赖性途径调节利托那韦的肝毒性[19];CYP3A4基因下调的患者比正常表达的患者更容易发生肿瘤分化[20]。因此,CYP3A4被认为可能是一种与肝细胞癌预后不良有关的新型抑癌基因。
CYP2E1是细胞色素P450超家族的另一个成员,与CYP2E1相关的疾病包括酒精性肝硬化和脂肪肝。其相关通路包括药物代谢-细胞色素P450和萘代谢。CYP2E1可以增强大量饮酒后引起的肠道渗漏、脂肪性肝炎和细胞凋亡[21]。研究显示,CYP2E1 RsaI/PstI和DraI多态性可能影响肺癌的易感性[22],筛选CYP2E1致癌基因多态性的遗传和测定易感对象该基因表型及开发其抑制剂,对预防其介导的癌症十分重要[23]。但也有研究表明,CYP2E1基因多态性可能是亚洲人对膀胱癌的保护因素[24]。因此,CYP2E1在肝癌中的调控作用机制仍待进一步探索。
SLC22A1是一种蛋白质编码基因,与其相关的疾病有白血病、慢性髓样和染色体17Q23.1-Q23.2缺失综合征。与该基因有关的GO注释包括蛋白质均二聚活性和跨膜转运蛋白活性。SLC22A1作为金属运输溶质载体可能对癌症的发生和进展以及抗癌药和肿瘤显像剂的输送做出重大贡献[25],并且其下调与肿瘤进展和患者生存率降低有关[26-27]。编码有机阳离子转运蛋白1(organic cation transporter 1,OCT1)的下调可能影响肝细胞癌和胆管癌对索拉非尼(一种阳离子药物)的反应[28]。综上,推测SLC22A1可能通过调节OCT1表达从而在肝癌发生发展中发挥重要作用。
由于本研究是基于公共数据库的生物信息学分析,这些肝癌相关的信号通路和关键基因还需要分子生物学方法和实验数据来进一步验证。
