大菱鲆(Scophthalmus maximus L.)tlr3基因的鉴定及其在病原感染前后的表达
2021-07-07王羽李新雨王敏傅强宋琳
王羽,李新雨,王敏,傅强,宋琳,
(1. 青岛农业大学海洋科学与工程学院,山东青岛 266109; 2. 青岛农业大学生命科学学院)
先天免疫系统是宿主对病原体感染的第一道防御线,特别是对生存在富含大量病原菌的水生环境中的硬骨鱼类而言[1],病原体附着和入侵的主要组织为皮肤、鳃和肠等黏膜组织,这些构成了先天免疫的黏膜免疫系统,对水生动物防止病原菌感染尤为重要。先前研究认为,先天免疫系统识别病原菌及特异性受体是依赖一群被称为病原体相关分子模式(pathogen-associated molecular patterns,PAMPs)的分子,如鞭毛蛋白、脂多糖(LPS)、脂蛋白、肽聚糖(PGN)、脂磷壁酸(LTA)酵母聚糖、CpG-DNA、微生物核酸等,它们分布于细胞表面、胞内区室或血液、组织液中,被种系编码模式识别受体(pathogen recognition receptor,PRR)识别[2]。尽管包括多种抗菌肽、凝集素,各种病原体识别受体家族成员,如Toll样受体、NOD样受体(nod-like receptor,NLR)和RIG-I样受体(RIG-like receptor,RLR)的黏膜免疫因子作为刺激物和感受器,已经在先天免疫系统中得到广泛研究,但人们对鱼类黏膜免疫的了解仍然有局限性,尤其在疾病控制和预防检测等方面。在各种病原体识别受体中,Toll样受体是首个且为最有效地被表征和鉴定的检测感染的先天免疫受体,称为病原体相关分子模式识别受体,可直接或间接通过抗原呈递细胞(antigen presenting cell, APC)检测外源性致病微生物的含量,如脂多糖、肽聚糖、核酸、鞭毛蛋白等。因此,Toll样受体在调配适当的适应性免疫反应中起着重要作用[3-4]。
目前,人们已经在哺乳动物中发现了大约13种Toll样受体(人类的为TLR1—TLR10,小鼠的为TLR1—TLR9、TLR11—TLR13),在其他动物中也发现了几种Toll样受体,如在低等脊椎动物中鉴定出的TLR14、TLR23[5]、TLR15[6]、TLR18—TLR20[7]、TLR21—TLR22[8]、TLR24[9]。脊椎动物TLR可分为6个主要家族:TLR1(TLR1、TLR2、TLR6、TLR10、TLR14),TLR3,TLR4,TLR5,TLR7(TLR7、TLR8、TLR9)和TLR11(TLR11、TLR12、TLR13、TLR21、TLR22、TLR23)[5,10]。有研究表明,已经在十几种鱼中鉴定出至少20种TLR基因(tlr1—tlr4、tlr5M、tlr5S、tlr7—tlr9、tlr13、tlr14、tlr18—tlr26)[11]。
TLR属于I型跨膜蛋白,根据不同的一级结构和识别配体,Toll样受体被分成两个主要亚族,分别为识别微生物脂质、糖、蛋白质组的细胞表面亚族和识别病毒或细菌来源的核苷酸衍生物核酸感应亚族[12-14]。通常,TLR包含几个结构域:胞外N-末端富含亮氨酸重复序列(LRR_NT)、可变数目的富含亮氨酸重复序列(LRR)、胞内C-末端LRR(LRR_CT)、跨膜结构域和Toll/IL-1受体同源结构域(TIR)。LRR结构域的序列和数目的变化赋予TLR特异性。细胞质TIR结构域不仅在同一物种的不同TLR之间,而且在不同动物物种之间高度保守[15-16],并且参与信号传导以及TLR的定位[17-18]。与胞内TIR结构域相反,胞外LRR结构域参与病原体识别[15]。迄今为止,TLR信号通路已被充分研究,包括髓样分化因子(myeloid differentiation primary response protein 88, MyD88)依赖性和MyD88非依赖性[含TIR结构域的接头蛋白诱导的IFN-β,也称TICAM-1(TIR domain-containing adaptor inducing IFN-beta,TRIF)依赖性]途径[19]。
最近,因为与其他脊椎动物TLR基因序列有相似性,具有特殊功能的tlr3在多种硬骨鱼中被识别,如斑马鱼(Daniorerio)[7]、青鳉(Oryziaslatipes)[20]、半滑舌鳎(Cynoglossussemilaevis)[21]、牙鲆(Paralichthysolivaceus)[22]和斑点叉尾鮰(Ictaluruspunctatus)[11]。TLR3能识别来自病毒的双链RNA(dsRNA)[23]。当硬骨鱼被注射刺激物dsRNA同系物聚肌苷酸胞苷酸polyI:C、RNA病毒、革兰氏阴性菌Edwardsiellatarda和Edwardsiellaictaluri之后,tlr3是上调表达的;当被注射刺激物草鱼呼肠孤病毒GCRV、革兰氏阴性菌Yersiniaruckeri和E.ictaluri之后,tlr3是下调表达的[24-26]。在斑马鱼中注射革兰氏阳性菌Mycobacteriummarinum之后,tlr3表达量没有明显变化[7]。另外,功能性研究表明,用polyI:C刺激河豚体外细胞,河豚Tlr3可诱导IFN-β表达[27]。
大菱鲆是中国水产养殖业中一个重要海水养殖品种,很容易受到各种病原体的侵袭,包括爱德华氏菌、海豚链球菌,以及各种弧菌,如鳗弧菌和溶藻弧菌。在大菱鲆养殖场中,这些病原体感染会导致疾病的爆发,可造成巨大的经济损失。目前已有许多研究致力于识别大菱鲆的免疫功能,表征其在先天免疫应答中的机制,阐明其在抵御病原菌附着过程中的宿主免疫应答反应机理,许多免疫蛋白已在大菱鲆中被识别和分析,如Tlr2[28]、肽聚糖识别蛋白(Pgpr2)[29]、Myd88[30]、干扰素调节因子5(Irf-5)[31]、热激蛋白70(Hsp70)[32]和G型溶菌酶[33]。
许多研究已经阐明感染过程中的基因活性,但控制大菱鲆抵抗病原体的具体机制仍然不明确,而对大菱鲆TLR成员的综合调查研究有助于了解识别感染的适应性免疫应答。tlr3的直系同源基因在几种鱼中已经被识别,但是大菱鲆tlr3基因依然没有被鉴定。因此,本文通过检索RNA-seq转录组数据库捕获tlr3基因序列[34],探究tlr3在健康组织、经革兰氏阴性菌鳗弧菌或革兰氏阳性菌海豚链球菌感染后的表达模式。
1 材料和方法
1.1 序列鉴定和分析
在作者所在团队构建的大菱鲆转录组数据中[34],以来自哺乳动物和鱼类的TLR3序列作为查询序列查找Toll样受体基因的全长序列,期望值(E值)截点为1e-10,用常规测序鉴定该识别序列,使用数据库NCBI ORF Finder[35]来预测所检索序列的可读框(open reading frame,ORF)。在非冗余蛋白质序列数据库下,用BLASTP工具[36]进一步验证从ORF中预测到的氨基酸序列。用简单模块化结构研究工具SMART[37]鉴定该序列的保守区和信号肽,使用SignalP 4.1网络服务器[38],并设置参数:物种选择真核“eukaryotes”,默认D值截点(default D-cutoff value),跨膜信号肽类型为“SignalP-TM”,进一步分析信号肽序列。利用TMHMM Server v2.0服务器[39]预测氨基酸序列tlr3的结构域,使用ExPASy服务器[40]分析大菱鲆Tlr3的理论等电点pI、相对分子质量和N-糖基化位点。最后使用MatGAT程序计算不同物种中Tlr3氨基酸的相似性和同一性的百分比[41]。
1.2 系统发育分析
挑选出大菱鲆,以及包括人(Homosapiens)、鼠(Musmusculus)、鸡(Gallusgallus)、蛙(Xenopuslaevis)、斑马鱼(Daniorerio)、青鳉(Oryziaslatipes)、半滑舌鳎(Cynoglossussemilaevis)和斑点叉尾鮰在内的其他物种Tlr3的氨基酸和DNA序列来构建进化树[42],用ClustalW2程序进行多重蛋白质序列比对,用分子进化遗传学分析软件MEGA6中的邻接法进行系统发育与分子进化分析[43],用泊松定理选项进行数据分析,并用完全删除选项移除差异。设置1 000次重复,用以评估进化树的拓扑稳定性。
1.3 细菌感染和样品收集
从大菱鲆孵化场(中国山东海阳市)获得大菱鲆苗种,苗种平均体质量15.6 g,平均体长5.5 cm。试验前,将所有的鱼放在实验室溢流法养殖系统中至少暂养1周,使其适应环境。暂养水温控制在(27±0.5)℃,大菱鲆随机分成4组暂养在4个60 L水槽中,每个水槽暂养30尾,恒定通风,并保持12 h—12 h的光照—黑暗周期节奏。在感染试验期间不饲喂。选择革兰氏阴性菌鳗弧菌(Vibrioanguillarum)和革兰氏阳性菌海豚链球菌(Streptococcusiniae)作为感染菌种,用以展现宿主在防御病原菌感染时TLR基因的免疫作用。两种菌的细菌分离株均由青岛农业大学鱼类免疫实验室提供。在预感染之后,按标准流程从单一有症状的鱼上重新分离出病原菌,并在培养之前进行生化鉴定,将该病原菌接种到卢里亚-贝尔塔尼培养基(Luria-Bertani medium,LB培养基),在振荡培养箱中(180 r/min,28 ℃)过夜培养。
试验过程中,将鱼浸泡在细菌溶液(约27 ℃)中2 h,然后放回池中,并进行行为观察和样品收集。对照组单独浸泡在灭菌培养基中,并进行相同处理。使用200 mg/L的三卡因甲基磺酸盐(MS-222)将鱼麻醉,缓冲液为碳酸氢钠溶液。在振荡培养箱(180 r/min,28 ℃)中过夜孵育后,将处理组分别浸泡在终浓度为5×107CFU/mL的鳗弧菌和终浓度为5×106CFU/mL的海豚链球菌水体中2 h。在鳗弧菌感染后2 h、6 h、12 h、24 h,在海豚链球菌感染后2 h、4 h、8 h、12 h的时间点,从15条鱼(养鱼池随机分成3个,从每个养鱼池中取5条鱼)中收集黏膜组织(肠、皮肤和鳃)。将收集的样品在液氮中速冻,然后保存于-80 ℃的超低温冰箱中,以备后续的RNA提取试验。
1.4 总RNA提取
RNA提取之前,在液氮中用研钵与研杵将样品磨成粉末。按Trizol®Reagent试剂盒(Invitrogen,Carlsbad,CA,USA)说明书提取总RNA,并按说明书用不含核糖核酸酶(ribonuclease,RNase)的DNA酶(Promega)处理。用微量分光光度计Nanodrop 2000(Thermo Electron North America LLC, FL)测出每个样品RNA的质量与浓度。所有提取样品在260 nm和280 nm的吸收度比值A260/A280均大于1.8,并稀释样品至250 ng/μL,最后在琼脂糖凝胶上显现RNA样品的完整性。
1.5 实时荧光定量PCR分析
用实时荧光定量PCR反应(qPCR)测定tlr3的mRNA转录本在对照和处理组的大菱鲆不同组织中的表达情况。根据大菱鲆TLRs基因转录本,用Primer3软件设计基因特定引物。以大菱鲆的18SrRNA基因作为参照基因,用以标准化同一样品中基因表达水平,并列于表1中。根据产品说明书,利用PrimeScript RT试剂盒(Perfect Real Time,TaKaRa Bio Inc.)合成单链互补DNA(cDNA),每10 μL反转录反应体系需要500 ng RNA。使用如下的标准循环程序在CFX96实时PCR检测系统(Bio-Rad Laboratories,Hercules,CA)上进行反应:热循环曲线,变性,95 ℃ 30 s,95 ℃ 5 s进行40个循环,58 ℃ 5 s;解离曲线,65 ℃ 5 s;以0.1 ℃/s的速度增温到95 ℃来检测扩增的特定性。该反应共含有200 ng/μL cDNA 1.0 μL,5 μmol/L的正向和反向引物各1.0 μL,5.0 μL SYBR Green supermix试剂,2.0 μL无核酸酶的水。对代表性的聚合酶链反应(polymerase chain reaction,PCR)产物进行测序以确认基因扩增的特异性和正确性。

表1 用于实时荧光定量PCR分析的引物
对大菱鲆健康组织的基因表达模式,用肌肉组织的阈值循环数(Ct值)作为对照,其他组织作为处理组。将对照组和处理组的mRNA表达水平标准化到同一样本的18SrRNA基因的表达水平。试验重复3次,进行基因表达分析,用文献[44]的软件Relative Expression Software Tool (REST)分析结果来捕获P<0.05的显著性表达。
分析每个时间点、所有板上(包括不含核酸酶的水的对照组)来自健康和感染组织的3个RNA样品的三重RNA样本基因表达。用REST程序分析结果来捕获P<0.05的显著性表达[39]。
2 试验结果
2.1 大菱鲆tlr3基因的鉴定
用从其他鱼类物种中获得的TLR序列作为查询序列搜索大菱鲆转录组数据库之后,Tlr3的氨基酸序列可被鉴定并测序。序列号为KX216854的tlr3与先前鉴定出的大菱鲆tlr3[45]相同,其转录物具有3 592 bp的核苷酸,含有编码926个氨基酸的2 781 bp ORF。此外,多腺苷酸化信号序列ATAAAA在tlr3基因的下游。推测Tlr3蛋白质相对分子质量为103.97 kD,理论pI=8.89,磷酸化位点具有11个丝氨酸和2个苏氨酸,推测N-糖基化位点具有9个天冬酰胺。推定的大菱鲆Tlr3蛋白含有包含N-末端区域的前29个氨基酸残基的信号肽序列D值(两个序列间每个位点氨基酸替代总数)截点为0.450,网络Networks为SignalP-noTM,表明少于4个位置被预测为处于跨膜状态。预测Tlr3氨基酸有几个重要结构特征,包括13个LRR结构域、LRR_CT结构域和145个氨基酸的TIR结构域(图1)。大菱鲆Tlr3与牙鲆的同源性最高,达到82%,其次是半滑舌鳎的,可达74.1%,与其他鱼类的同源性在52%~60%之间,与人的同一性为52.6%(表2)。大菱鲆tlr3基因与牙鲆基因最相似,具有88.2%的同一性和78.2%的相似性。与ORF整体相比,TIR结构域之间的氨基酸序列同一性和相似性高4%~10%,在大菱鲆和牙鲆之间具有最高同一性(92.4%)和相似性(82.8%)(表2)。
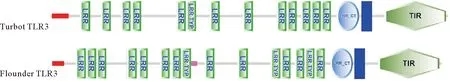
LRR,富含亮氨酸重复序列;LRR_CT,富含亮氨酸重复序列C-末端结构域;TIR,Toll/IL-1受体同源结构域;LRR_NT,富含亮氨酸重复序列N-末端结构域;LRR TYP,富含亮氨酸的重复序列典型的(最密集的)亚科;,跨膜结构域;,低组分复杂性段;,信号肽

表2 比较大菱鲆Tlr3与人类及其他硬骨鱼氨基酸序列
2.2 进化分析
使用MEGA6软件将大菱鲆和其他物种的TLR基因构建进化树(图2),以便于更进一步地对大菱鲆tlr3基因鉴定和进化关系验证。系统发育分析进一步验证本研究的假设:在同一分支内,大菱鲆Tlr3与其他脊椎动物TLR3聚合在一起(图1)。TLR3进一步分为两个亚组,如图2所示,第一亚组为鱼类,第二亚组为高级脊椎动物。在第一亚组中,大菱鲆Tlr3与牙鲆Tlr3关系最近,完全相似匹配分数为98%~100%,然后是半滑舌鳎,它们与其他直系同源分支的斑点叉尾鮰Tlr3和斑马鱼Tlr3形成这一亚组。第二个亚组包括人类和小鼠、鸡和龟、蛙。
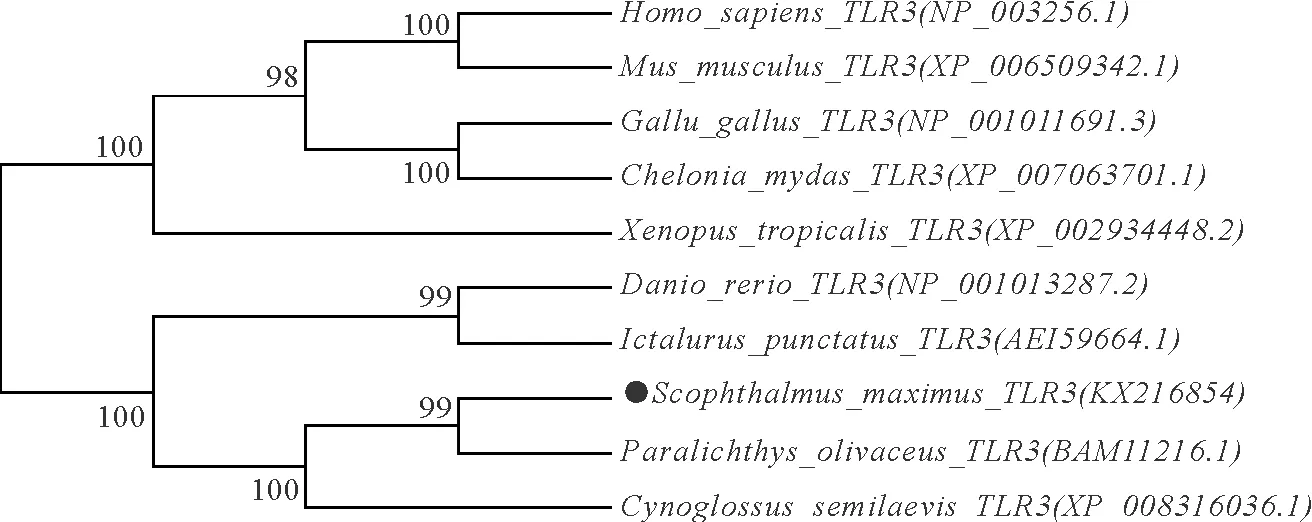
图2 Toll样受体TLR3的系统进化树
进化树分析与进化关系一致,并且所有的分支节点都有高的自展值。因此,进化分析显示出与其他物种相应基因明确的直系同源关系。除了先前的观察,大菱鲆Tlr3与牙鲆基因关系更近,可以预测大菱鲆和牙鲆之间Tlr3的功能相似性。TLR3的系统发育分析表明,大菱鲆的Tlr3与其对应物可能在配体识别和对刺激的免疫应答中具有类似的作用,在大菱鲆养殖中对促进大菱鲆稳定发育具有不可替代的作用。
2.3 大菱鲆tlr3基因在基体组织中的分布
在包括血液、肝脏、脾脏、鳃、皮肤、肠、头肾和脑在内的8个大菱鲆健康组织中,用实时荧光定量PCR进行大菱鲆tlr3基因的组织分布分析(图3)。总体而言,在所有检测组织中大菱鲆tlr3均有表达,但表达水平不同。如图3所示,tlr3基因在肠中的表达水平最高,随后是血液、脑、头肾、鳃、脾脏、皮肤,在肝脏中的表达水平最低。大菱鲆tlr3在体内不同发育阶段、体外不同水生环境下对刺激的黏膜免疫反应的作用仍需要进一步研究来详细阐明。

图3 Toll样受体基因tlr3在大菱鲆不同的健康组织中的基因表达分析
2.4 鳗弧菌感染后tlr3基因的表达图谱
在不同病原菌感染后的黏膜组织中表征tlr3的表达模式,可以阐明tlr3基因在大菱鲆黏膜免疫中的作用,因此选取宿主与病菌相互作用的第一场所——黏膜组织(鳃、皮肤和肠)作为研究对象。在感染单一革兰氏阴性菌鳗弧菌和单一革兰氏阳性菌海豚链球菌之后,及时采集黏膜组织,这些采集时间点是细菌附着和入侵的关键时间。本文首次在黏膜组织中对病菌浸泡感染后的大菱鲆tlr3基因进行立体表达分析。
感染鳗弧菌后,大菱鲆tlr3在不同组织中显示不同的表达模式(图4)。在肠中,大菱鲆tlr3在感染后12 h上调53.5倍,24 h后上调99.10倍;在皮肤感染2 h、6 h和12 h后,大菱鲆tlr3的表达没有出现明显变化,直到感染24 h后,才上调2.4倍;在鳃中,大菱鲆tlr3的表达是散发性的,仅在感染后2 h(上调3.8倍)和24 h(上调7.8倍)检测到诱导变化。

图4 受鳗弧菌感染后大菱鲆黏膜组织中的tlr3表达的实时qPCR分析
2.5 海豚链球菌感染后TLR基因的表达图谱
与鳗弧菌感染不同,海豚链球菌感染后,大菱鲆tlr3表现出明显不同的表达模式。在肠中,大菱鲆tlr3的诱导最强,在所有采集时间点均表达上调,与鳗弧菌感染相似,且在感染后4 h达到峰值(约157.5倍)。与鳗弧菌感染的诱导不同,受海豚链球菌攻击并感染2 h后,大菱鲆tlr3上调且表达更强、更早。在皮肤中,感染4 h后大菱鲆tlr3上调23.1倍,12 h时最高,为43.9倍。感染4 h、8 h和12 h后,在鳃中均发现类似于皮肤中的大菱鲆tlr3上调表达。但受海豚链球菌感染后,皮肤和鳃中诱导大菱鲆tlr3的表达倍数和时间与受鳗弧菌感染后的表达不同(图5)。

图5 受海豚链球菌感染后大菱鲆黏膜组织tlr3表达的实时qPCR分析
3 讨论
本研究确定了在大菱鲆转录组中单个完整的tlr3基因,鉴别了其基因结构、组织表达分布,并通过革兰氏阳性细菌和革兰氏阴性细菌攻毒诱导该基因表达。
3.1 TLR3的比较结构域和功能
在哺乳动物中,已经证明TLR3对双链RNA的应答作用[46-49]。首个被研究的具有三维结构特征的TLR是人的TLR3[50-51],小鼠TLR3是第一个与配体结合并与46 bp的dsRNA结合的核酸感应结构[52]。TLR3与TLR7、TLR8、TLR9类似,不是由位于表面或外/内膜上的微生物组分诱导,而是受来自细菌或病毒的核酸诱导[53]。TLR3通过TRIF依赖的TLR受体信号通路激活诱导细胞因子IRF3和NF-κB,从而诱导I型干扰素及促炎细胞因子的产生[54]。在哺乳动物的有效抗病毒免疫中,TLR3不是必须存在的[55],然而,在TLR3缺陷小鼠的脾脏中发现病毒载量增加了1 000倍[56],人类的TLR3缺陷显示与单纯疱疹病毒的易感性有关[57]。在鱼中,Tlr3主要位于内质网(ER)中,并且负责识别相对短的dsRNA,从而在抗病毒免疫中发挥重要作用[58]。在大菱鲆中,Tlr3检测主要分布在上皮细胞中,并且可以更多地识别配体而不是dsRNA[45]。在鱼类中,已有多个证据表明,dsRNA病毒、polyI:C和细菌病原体可刺激Tlr3上调表达[59,63]。
使用ExPASy服务器可预测翻译后修饰的不同数目的糖基化和磷酸化位点。本研究在大菱鲆Tlr3中预测了9个N-糖基化位点。在之前的研究中,发现了大菱鲆Tlr2的5个N-糖基化位点[28]。在人体中,有4个TLR2糖基化位点被预测,其中之一对分泌效果有显著影响[64]。因此推测TLR3分子中的糖基化可能对受体表面表达、运输、模式识别和突变有影响。预测的N-糖基化位点参与将组织蛋白酶转运到溶酶体的过程,根据组织蛋白酶S的抗原呈递作用,可以推测不同数量的N-糖基化位点可能与其免疫功能有关[65]。在不同物种中鉴定出的不同数目的N-糖基化位点也可以反映其在抵抗病原体入侵时的差异作用。
由于大菱鲆Tlr3的氨基酸序列更类似于牙鲆,对其进行了结构域结构比较(图1)。LRR结构域是富含疏水氨基酸亮氨酸的20~30个AA长序列的阵列[66]。Gong等[66]发现所有LRR序列都存在高度保守片段,具有共有序列LxxLxLxxN(Cx)xL,并且包含多种二级结构的可变片段LxxLxxxxLxxLxL。此外还观察到了其他类型的LRR,例如典型的T型(xxLxxxxLxxLxx)和细菌S型[xxLPx(x)LPxx][67]。这些符号中的字母L代表形成疏水核心的氨基酸,如亮氨酸、异亮氨酸、缬氨酸和苯丙氨酸,N表示天冬酰胺、苏氨酸、丝氨酸或半胱氨酸,x是任意氨基酸[66]。一般来说,尽管一些LRR的精确定位对于每个物种都是不同的,大菱鲆Tlr3的LRRs与在牙鲆和其他物种中鉴定的LRR分布相似(图1)。不同的TLR在不同物种中的LRR数目存在差异,而在人类TLR3肽中达到18个(表2)。在数量上,LRR这种对病原体识别的变化赋予TLR参与宿主抵抗病原体感染的特异性[11]。
3.2 TLR3在细菌感染反应中的表达
一般来说,不同的细菌感染后有不同的表达模式。尽管大菱鲆tlr3在海豚链球菌感染中的表达强度和时间比在鳗弧菌感染中更强、更早,但在肠道中是一致的,这也说明海豚链球菌在肠、皮肤和鳃中引起比鳗弧菌感染更急性的反应。因此,大菱鲆tlr3在肠中以时间依赖性和细菌依赖性方式诱导。
研究发现,海豚链球菌的数量主要在肾脏和脾脏中增加,Nguyen等[68]在肠道、鳃、皮肤黏液和其他组织中也发现了大量的海豚链球菌。TLR3在人和小鼠的体外dsRNA(来自病毒)存在下发生上调表达[23],但在体内的功能作用仍然不清楚[55,69]。之前的研究表明,TLR3对于dsRNA病毒在体内和体外用polyI:C刺激诱导具有应答反应[59,62],这与受革兰氏阴性细菌病原体Edwardsiellaictaluri感染时,斑点叉尾鮰和斑马鱼中tlr3mRNA上调具有一致的证据[59,63],我们目前已发现,受革兰氏阴性细菌鳗弧菌和革兰氏阳性细菌海豚链球菌感染后,大菱鲆tlr3一般呈上调表达趋势。对病原体感染的这种非特异性应答需要进一步研究,以揭示特定的细菌配体识别和宿主与微生物之间的相互作用。
4 结论
本研究鉴定了大菱鲆中的tlr3基因,该基因是其他鱼类和高级脊椎动物的直系同源基因,其基因结构、氨基酸序列和预测的结构域在物种间是保守的。本研究首次检测了大菱鲆tlr3基因受不同细菌感染后在大菱鲆的黏膜组织中的表达模式。tlr3基因的黏膜表达说明tlr3在防止病原体附着和进入大菱鲆宿主防御系统中起到第一道防线的关键作用。之后应进一步进行功能研究以更好地表征TLR及其家族基因,并确定不同黏膜组织在病原性不利环境中对硬骨鱼类的保护作用。免疫组化技术有助于调查在受感染后不同时间点,细菌进入的位点和鱼的感染过程。TLRs与先天免疫和适应性免疫功能密切相关,在早期发育阶段研究检测其基因和蛋白质的表达水平,不仅有助于辨别先天和适应性免疫应答的关系,而且还能对病原致病机制有所探究。深入研究TLR和先天免疫反应对健康发展大菱鲆育种计划有深远而重要的意义。
