生物信息学分析PDGFRB在胃癌中的表达及其临床意义
2021-06-28史维俊吴华彰
苏 旭,史维俊,王 静,吴华彰
(蚌埠医学院 1.生命科学学院癌症转化医学安徽省重点实验室;2.第一附属医院胃肠外科,安徽 蚌埠 233030)
胃癌是常见的消化道恶性肿瘤之一。据世界癌症报告统计,2015年全球胃癌的发病率与死亡率分别居于所有恶性肿瘤中的第5位和第3位[1]。我国更是胃癌的高发地区,全球胃癌患者约有42%来自于中国[2]。近年来,虽通过手术治疗、放化疗、靶向药物等多种治疗方法,患者生存期延长,生活质量有所提高。然而,早期胃癌的检出率很低,大多数患者在诊断时已是晚期阶段,失去了治疗的最佳时机[3]。因此,识别胃癌进展和预后新的分子靶点[4],开展有效的胃癌靶向治疗是迫切需要的。随着基因组学、表观基因组学的发展,现代医学已经进入精准医学时代,通过对胃癌的生物信息学分析,已有大量关于胃癌发生发展、转移、耐药及预后相关分子标记物的研究报道,并且研究开发出分子靶向药物改变了胃癌的临床治疗模式,如靶向表皮生长因子(epidermal growth factor receptor,EGFR)药物、PI3K信号通路抑制剂、PARP抑制剂等[5-8]。由此可见,基因组学大数据深度挖掘是寻找胃癌诊断、治疗及相关靶点的新型研究手段。在此项研究中,利用生物信息学技术分析胃癌中PDGFRB的表达情况[9],探究PDGFRB与胃癌患者临床病理特征和预后的关联,聚焦其在胃癌中可能参与的生物学过程,了解PDGFRB对胃癌发生发展和预后的影响及临床价值,以期为胃癌的早期诊断和靶向治疗提供理论依据。
1 资料与方法
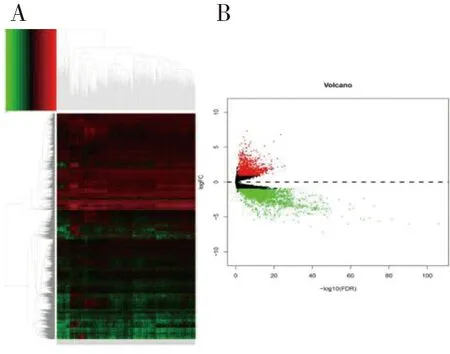
1.1 数据下载与整理TCGA是美国国家癌症研究所(National Cancer Institute)和美国人类基因组研究所(National Human Genome Research Institute)共同监督的一个项目,目前已经收录了60多种的肿瘤类型。本研究从TCGA数据库中下载RNA-seq数据和临床数据(https://www.cancer.gov/),包括正常胃粘膜上皮样本32例及胃癌患者样本375例。利用Perl软件进行基因ID的转换和临床数据信息的提取,构建基因表达矩阵,随后采用R语言根据log(FC)>1,FDR<0.05[10]的筛选标准初步筛选胃癌组织中的差异表达基因,并对差异基因进行热图和火山图的绘制。
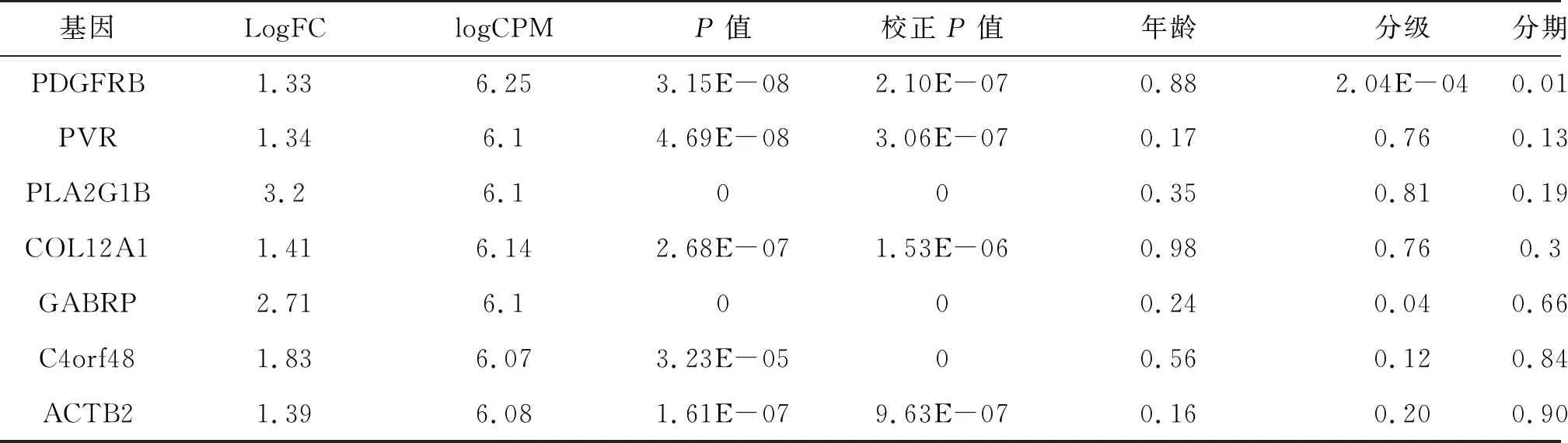
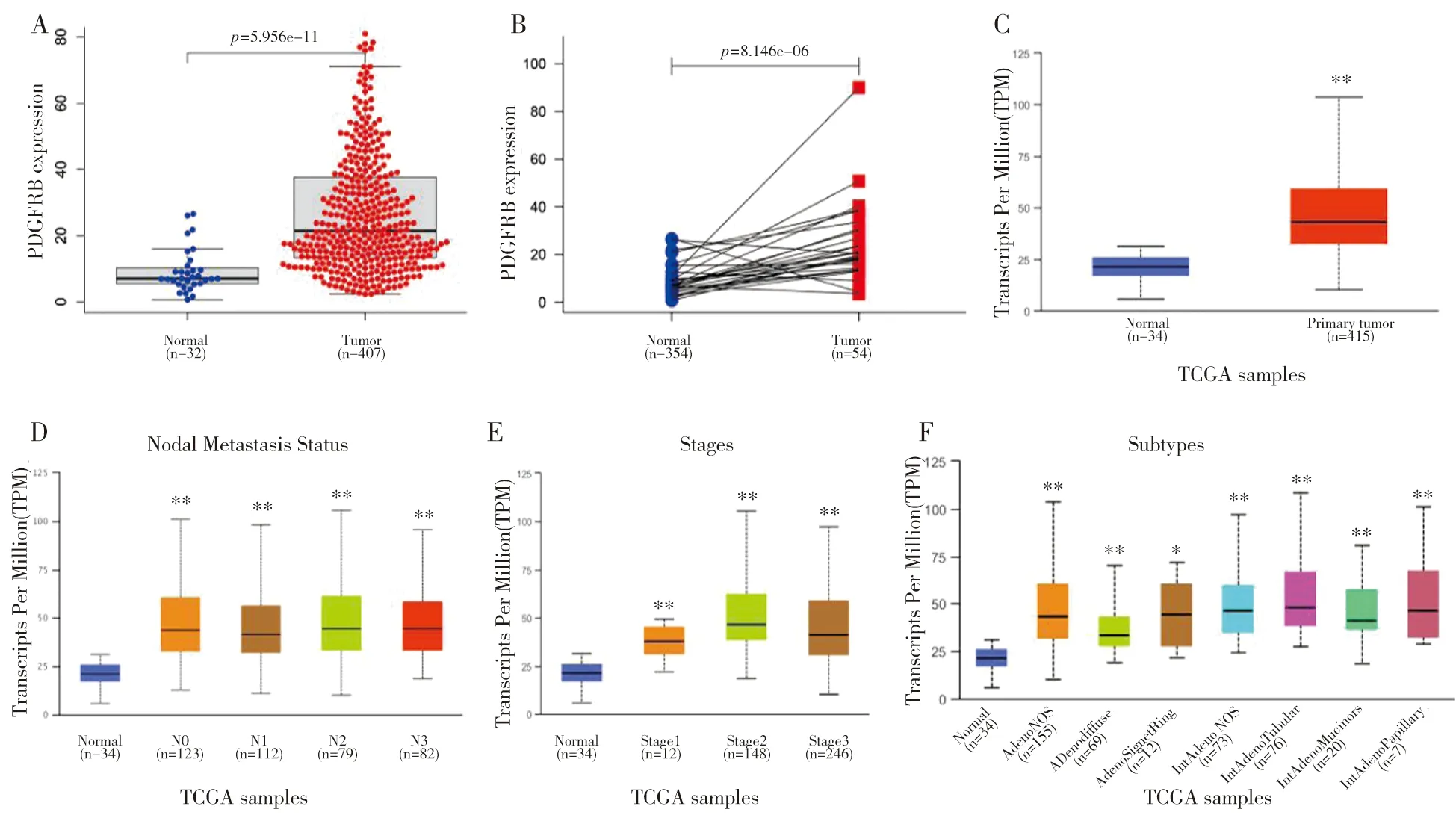
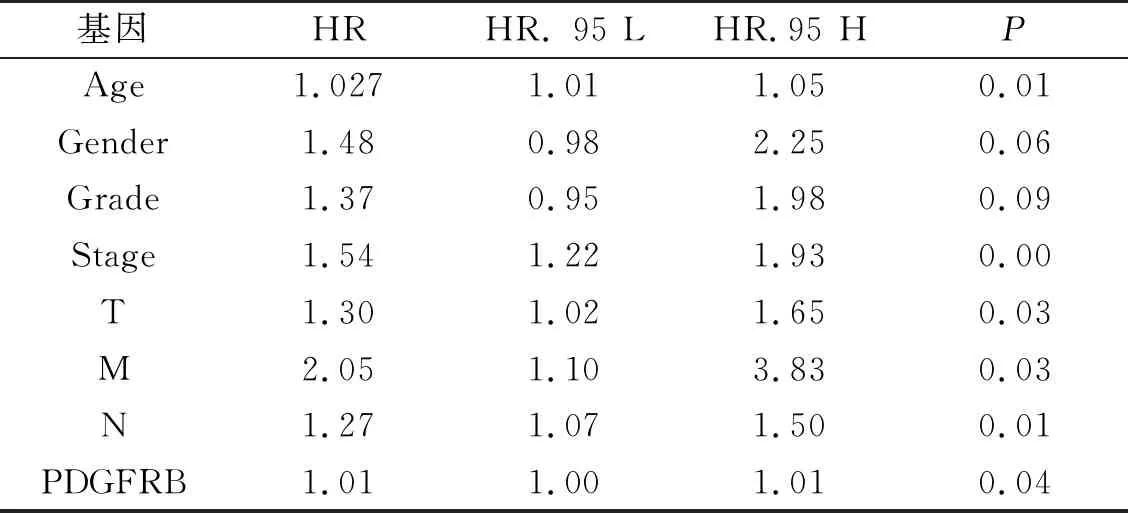
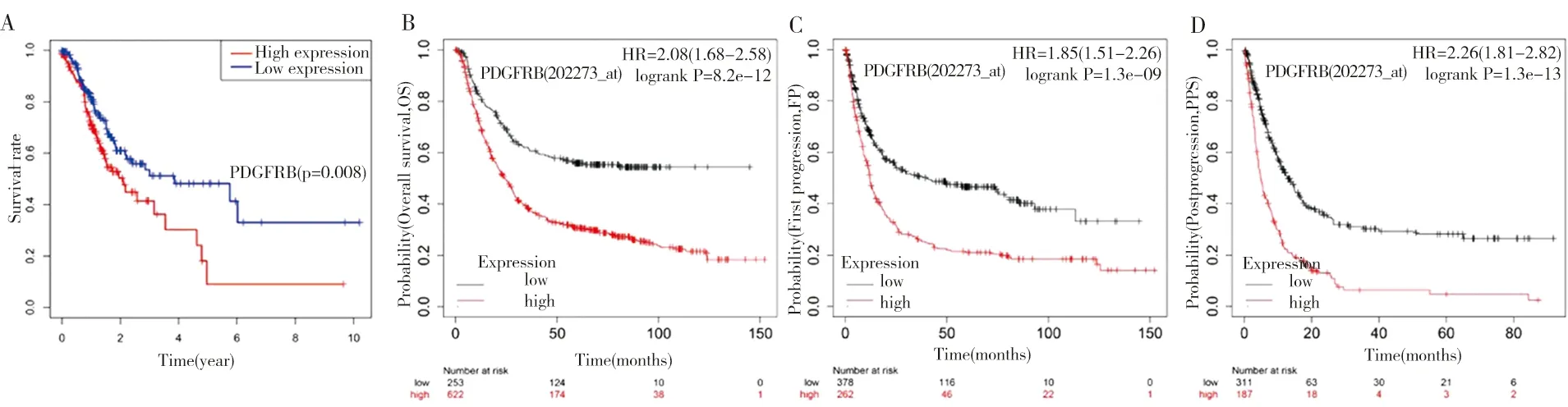
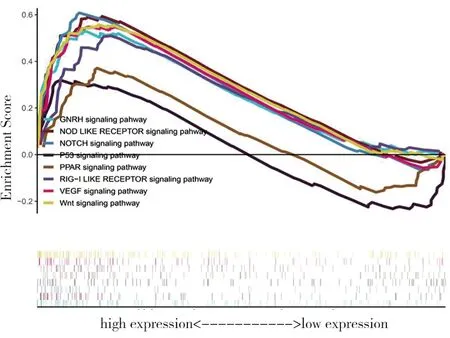
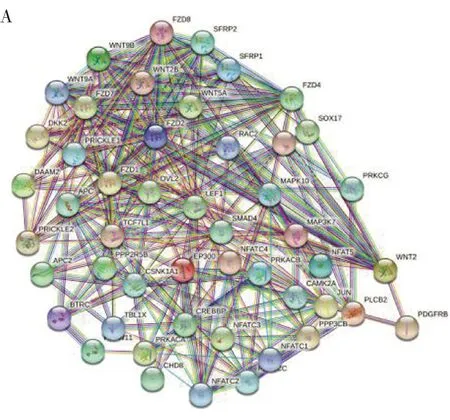
1.2 目的基因的筛选为研究差异表达的基因对胃癌患者的影响,对表达上调的差异基因进一步筛选分析,设置筛选标准:log(FC)>1.3,6 1.3 PDGFRB基因的差异表达及临床病理特征的相关性分析为分析PDGFRB基因表达水平和临床病理特征的相关性,使用Perl软件将目的基因表达数据和临床病理数据相结合,构建成包含目的基因信息和临床信息的表达矩阵。随后,利用R软件获得目的基因在胃癌组织和正常胃黏膜组织中的差异表达,通过在线分析软件UALCAN(http://ualcan.path.uab.edu/analysis.html)分析PDGFRB表达水平与与胃癌患者的淋巴结转移、分期和组织亚型等临床病理特征的相关性。 1.4 独立预后分析将临床数据表达矩阵导入R软件,利用survival程序包进行Cox回归分析PDGFRB基因表达水平与胃癌患者生存率、年龄、分期和TMN分期等预后因素的相关性。利用在线分析网站Kaplan-Meier Plotter(https://kmplot.com/)分析PDGFRB基因分别在高表达和低表达胃癌患者中的生存情况。 1.5 PDGFRB基因的富集分析利用Perl软件输出应用于目的基因富集分析的表达数据集文件(.gct)和表型数据文件(.cls),进行GSEA软件的下载安装(https://www.gsea-msigdb.org/)和Java 8运行环境[12],将表达数据集文件和表型数据文件导入GSEA,对PDGFRB基因的信号通路进行KEGG富集分析,筛选富集分析标准为(P<0.005,FDR<0.005)。 1.6 PDGFRB基因与潜在靶基因的相关性分析将目的基因PDGFRB输入STRING数据库进行分析,并选择Homo sapiens类别的蛋白质相互作用网络,构建目的基因蛋白相互作用网络。将目的基因与潜在靶基因的基因数据导入R软件,分析目的基因与潜在靶基因的相关性。 2.1 胃癌组织中差异表达基因的筛选对375例胃癌和32例胃粘膜样本中基因的差异表达情况进行筛选分析,初步筛选出胃癌样本中差异化表达基因2676个,其中在胃癌组织中高表达和低表达的基因分别为1110和1566个,见图1。 图1 胃癌组织中基因差异表达分析 2.2 胃癌组织中目的基因PDGFRB的筛选胃癌是多基因异常导致的复杂性疾病,为研究差异表达基因对胃癌患者的影响,根据筛选标准:log(FC)>1.3,6 表1 候选基因与胃癌患者临床病理特征相关性分析 2.3 PDGFRB基因在胃癌中的差异表达及临床病理相关性分析为进一步探讨PDGFRB基因是否可能参与了胃癌的发生发展,通过R语言中的limma[13]和beeswarm[14]程序包对PDGFRB基因的表达矩阵进行差异化分析显示,相对于正常胃粘膜组织,PDGFRB基因在胃癌组织中显著高表达(图2A)(P<0.001)。对32例配对胃癌及癌旁组织分析发现,PDGFRB基因表达水平在胃癌组织中显著增高(图2B)(P<0.001)。UALCAN在线分析发现,PDGFRB基因在胃癌组织中显著高表达(图2C),且表达水平与淋巴结转移、分期及亚型显著相关(图2D、2E、2F)(P<0.001)。以上结果表明,PDGFRB基因可能作为重要的潜在癌基因参与胃癌的发生发展。 图2 PDGFRB基因在胃癌中的差异表达及临床病理相关性分析 2.4 PDGFRB基因与胃癌患者的独立预后分析为进一步确定PDGFRB基因可以作为调控胃癌发生和影响预后的潜在靶基因,Cox回归分析显示,PDGFRB基因表达水平与胃癌患者年龄、分期和TMN分期等独立预后因素显著相关(P<0.05),见表2)。另外,K-M Plot在线分析发现,PDGFRB基因表达水平与胃癌患者总生存率(OS)(图3A、3B)、首次进展期生存率(FP)(图3C)、后进展期生存率(PPS)(图3D)显著相关(P<0.01)。 表2 PDGFRB基因与胃癌患者的独立预后分析 图3 PDGFRB基因表达与胃癌患者预后显著相关 2.5 PDGFRB基因多GSEA富集分析为探索PDGFRB基因调控胃癌恶性进展的分子机制,利用GSEA富集分析软件对PDGFRB基因进行信号通路富集分析。结果显示,在富集的相关信号通路中,PDGFRB基因在Wnt信号通路中富集相关性居于首位且富集分数最高(P<0.01)(表3、图4)。 表3 PDGFRB基因GSEA富集分析结果 图4 PDGFRB基因通路富集分析 2.6 PDGFRB基因与潜在靶基因的相关性分析为进一步分析PDGFRB基因通过调控Wnt信号通路影响胃癌发生发展的分子机制,利用STRING在线分析网站构建PDGFRB基因蛋白相互作用网络,蛋白质之间的连接线越多,相互关联性越强,分析显示PDGFRB基因与Wnt2、PLCB2之间具有强关联性(图5A),且PDGFRB基因与Wnt2、PLCB2的表达呈正相关(P<0.001)(图5B、5C)。 图5 PDGFRB基因相关的蛋白互作及相关通路中的分子相关性分析 胃癌是世界范围内发病率和致死率最高的癌症之一,近半数的胃癌发生于东亚地区,尤其是中国、韩国、日本及蒙古国[15]。胃癌的发生也是多因子、多步骤相互作用的复杂生物学过程,了解胃癌的发生发展对于其诊断和治疗非常重要[16]。随着基因组学、表观基因组学的发展,通过对胃癌的生物信息学分析,越来越多的胃癌潜在基因治疗靶点被研究发现,为诊断和治疗胃癌提供了一个新的方向[17]。 本研究通过对TCGA数据库中胃癌差异化表达基因的生存、预后及临床病理相关性分析,通过UALCAN在线分析发现,PDGFRB基因在胃癌组织中显著高表达,其表达水平与胃癌患者的淋巴结转移、分期和组织亚型显著相关。采用Cox回归分析显示,PDGFRB基因可能参与胃癌发生发展,且可能是胃癌诊断、治疗和预后潜在的生物标志物。为进一步分析PDGFRB基因在调节胃癌发生发展中的分子机制,通过GSEA单基因富集分析发现,PDGFRB基因在Wnt信号通路上显著富集[18-20]。利用在线网站STRING构建PDGFRB蛋白互作网络,该蛋白互作网络可以更好地理解PDGFRB与其它蛋白之间的功能关联,结果发现共有60个预测的功能蛋白与PDGFRB相互作用,其中两种蛋白基因被证实与胃癌的发生发展和预后有关。于是通过R语言对PDGFRB与这两种基因进行相关性分析,得出PDGFRB基因与Wnt2[21]、PLCB2[22]表达呈正相关,这提示PDGFRB基因可能通过促进Wnt2/PLCB2信号网络,激活Wnt信号通路,促进细胞恶性增殖从而促进胃癌的发生发展。研究表明,Wnt信号通路与人类肿瘤相关,如肺癌[23]、肝癌[24]、结肠癌[25]等。Wnt信号通路激活,促进细胞增殖以及存活而不进入分化,持续性的激活Wnt信号通路使得信号通路中的其他信号分子有可能发生突变,例如永久性失活APC复合物,即“遗传调控门控基因”缺失,进一步激活Wnt信号通路,从而促进肿瘤的发展[26-30]。 综上所述,PDGFRB基因在胃癌组织中高表达提示其可能作为重要的潜在癌基因参与胃癌的发生发展;PDGFRB基因与临床病理特征显著相关,可作为评价胃癌预后的独立危险因素;PDGFRB基因可能通过促进Wnt2/FZD1信号网络,激活Wnt信号通路,促进细胞恶性增殖从而促进胃癌的发生发展。本研究采用生物信息学方法对PDGFRB基因进行了深入分析,分析结果可为胃癌的实验研究提供一个重要的研究方向,由于生物信息学分析方法利用了网络芯片数据和在线数据分析网站,缺乏相应的实验数据作为支持验证,存在一定的局限性。因此,PDGFRB在胃癌发生发展中的具体功能及其作用机制仍有待进一步实验研究验证。2 结果

3 讨论
