柑桔叶斑驳病毒江西分离物全基因组序列测定与分析
2021-06-23钟家美黄爱军
丁 敏,王 莹,钟家美,黄爱军
(赣南师范大学生命科学学院/国家脐橙工程技术研究中心,江西赣州,341000)
柑桔叶斑驳病毒(Citrusleafblotchvirus, CLBV)属于乙型线形病毒科柑桔病毒属,是一种正义单链RNA病毒,在多数柑桔品种上不引起明显症状[1],但可引起德威特桔橙(Dweet tangor,Citrusreticulate×C.sinensis)斑驳花叶症状和在香橼(‘Etrog’ citron,C.medica)上引起茎陷点症状[2-3],还可能与以枳橙或枳柚做砧木时的砧穗不亲和症状有关[4]。目前CLBV已在美国、西班牙、澳大利亚等多个国家报道,寄主范围包括柑桔、甜樱桃、猕猴桃和牡丹[5-7]。我国于2017年首次在柠檬上检测出该病毒[8]。CLBV在自然条件下通过病穗嫁接和带毒工具农事操作进行传播,同时有证据显示,CLBV具有一定的种传概率[9-10]。虽然目前未见CLBV在柑桔产业上引起显著危害的报道,但是不能排除该病毒对特定柑桔品种或者与其他病毒有协同作用产生严重危害。
目前报道的所有CLBV基因组结构均相同,基因组5’和3’端各有一段非编码区(untranslated regions, UTRs),5’端有甲基化帽子结构,3’端有ploy A尾巴;其余包含有3个开放阅读框(open reading frame, ORF)。ORF1编码一个227 kDa的多聚蛋白,这个多聚蛋白经酶切割后,得到不同功能的蛋白,包括甲基转移酶、AlkB蛋白、木瓜蛋白酶样蛋白酶、解旋酶和RNA依赖的RNA聚合酶。ORF2编码约40 kDa的细胞间运动蛋白。ORF3编码约41 kDa的外壳蛋白[11-13]。Li等[14]测定分析了中国4个不同省份柑桔样品上的CLBV全长序列,核酸序列一致度在98.9%~99.8%间,表现出较小变异程度;而项周等[15]对来自湖北和四川的14个CLBV的CP基因分析结果显示,CP基因核苷酸和氨基酸序列差异分别可达18%和12%,表现出较大的变异程度。目前,尚未有江西CLBV全长序列的报道。本研究拟通过对CLBV全基因序列测定,对其序列特征、系统发育关系等进行分析。
1 材料与方法
1.1 供试材料感染CLBV的纽荷尔脐橙(C.sinensisOsbeck)样品于2017年采集于江西省万安县窑头镇通津村柑桔园,样品叶片上并未表现出斑驳黄化症状。采集感病植株的枝条进行嫁接,保存于国家脐橙技术研究中心温网室,并采集叶片低温保湿带回实验室进行RNA抽提。抽提RNA于-80 ℃冰箱保存备用。
1.2 主要试剂植物总RNA提取试剂RNAiso Plus,PrimeScript Ⅱ 1stStrand cDNA Synthesis Kit试剂盒,SMARTer 5'/3' kit、PrimeSTAR GXL DNA 聚合酶、DNA凝胶回收试剂盒为宝生物工程公司产品。pEASY-Blunt Zero Cloning Kit,Trans1 -T1 Phage Resistant化学感受态细胞为北京全式金生物技术有限公司产品。所有操作均参照试剂盒说明书进行。所得植物总RNA采用Nanodrop 2000(Thermo Scientific,美国)检测其浓度和纯度。CLBV基因组5’和3’端RACE扩增引物,根据GenBank中已经登录的基因组序列的保守区设计特异性引物,所有引物均由金斯瑞生物科技有限公司合成。所用引物的相关信息见表1。
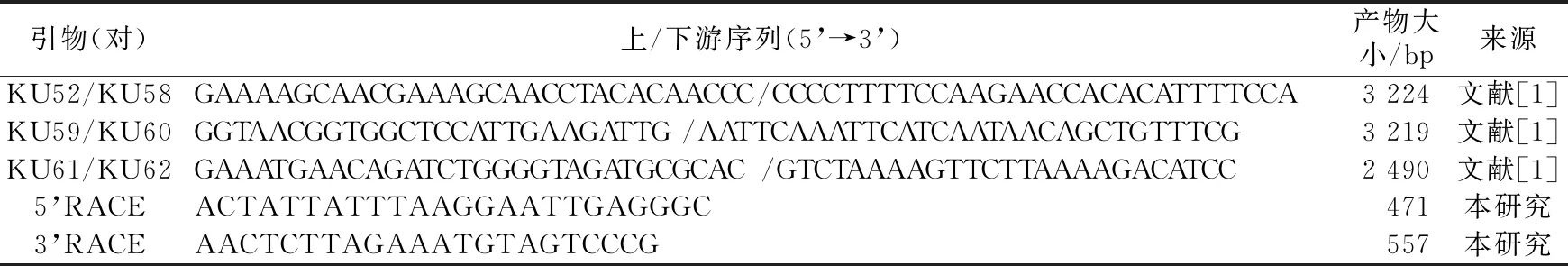
表1 柑桔叶斑驳病毒全基因组扩增引物相关信息
1.3 试验方法CLBV基因组全长扩增、克隆及测序:采用RNAiso试剂从采集的感病叶片样品中提取总RNA,具体步骤参照说明书。以总RNA为模板,参照反转录试剂盒说明书进行反转录。反应体系(20 μL):随机引物(Random 6 mers)1.0 μL,10 m mol/L的dNTP溶液1.0 μL,RNase Free 单蒸水6.0 μL,RNA模板2.0 μL,以上反应液置于PCR仪上65 ℃解链5 min,然后取出立即置于冰上冷却约2 min,再依次加入5× PrimeScript Buffer 4.0 μL,RNase Inhibitor(40 U/μL) 0.5 μL,PrimeScript RTase(200 U/μL)1.0 μL,RNase Free 单蒸水4.5 μL,30 ℃反应10 min,42 ℃反应50 min,95 ℃反应5 min酶失活后,冰上冷却。 PCR扩增反应体系(25 μL):5× PrimeSTAR GXL Buffer 5.0 μL,2.5 m mol/L的dNTP溶液2.0 μL,10 μ mol/L正向引物/反向引物0.5 μL,PrimeSTAR GXL DNA Polymerase 0.5 μL,反转录产物为模板1.0 μL,灭菌水15.5 μL。扩增反应步聚为:98 ℃变性10 s,60 ℃退火15 s,68 ℃延伸时间视片段长度而定(按1 min / kb计算)。PCR反应结束后,取5 μL产物用1.5%琼脂糖凝胶电泳进行条带检测。目的条带的胶回收纯化参照试剂盒说明进行。经纯化产物与全式金pEASY-Blunt Zero载体连接后,转化Trans1 -T1 Phage Resistant化学感受态细胞。每个条带至少挑选3个以上阳性克隆进行测序。病毒5’/3’端扩增采用SMARTer 5’/3’试剂盒,步骤按照说明书进行。扩增所得目的条带克隆、测序步骤同上。
序列分析:测序所得结果应用Megalign软件(DNASTAR)进行序列拼接,得到病毒序列全长。分析所用CLBV分离物序列均下载自GenBank,利用在线软件Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)进行序列一致性分析。序列比对及进化树构建采用MEGA 6.06软件,以邻接法(Neighbor-joining)构建系统进化树,Bootstrap值设置为1 000次。
2 结果与分析
2.1 CLBV江西分离物的全基因组序列特征利用3对CLBV特异性引物通过RT-PCR扩增得到3条片段大小为3 224、3 219和2 490 bp的目的条带;利用RACE技术和巢式PCR扩增获得5’端和3’端序列,片段大小分别为471和557 bp。分离物全基因组分段扩增结果如图1所示,各片段结果均符合预期扩增片段大小。
通过测序、拼接获得了CLBV分离物基因组全长为8 747 nt,将其命名为CLBV-JX,提交GenBank后序列号为MT863785。该分离物全长序列包含3个ORF,ORF1(74-5 962)编码多聚蛋白,ORF2(5 962-7 050)编码移动蛋白,ORF3(7 115-8 206)编码外壳蛋白,5’端和3’端非翻译区分别为73和541 nt,ORF2 和ORF3之间有一段64 bp的间隔区(见图1)。
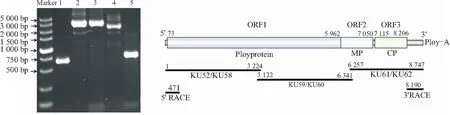
注:在电泳图中,1为5’端RACE,2为KU52/KU58,3为KU59/KU60,4为KU61/KU62,5为3’端RACE扩增产物。
2.2 CLBV江西分离物与其他来源分离物的序列一致性序列一致性分析显示,在全基因组水平上,CLBV-JX与其他6个CLBV分离物的一致度为74.3%~97.2%,与柑桔来源的分离物一致度高(96.8%~97.2%),与猕猴桃来源的分离物一致度最低,仅为74.3%。在5’和3’端非编码区上,核苷酸一致度分别为77.9%~98.6%和62.7%~98.9%。在3’端非编码区表现出更大的变异性,CLBV-JX与来源于甜樱桃的分离物(KR023647)一致度仅为62.7%。3个编码框区域,核苷酸一致度分别为72.7%~97.1%、78.2%~96.4%和79.0%~98.8%,氨基酸一致度分别为79.4%~98.5%、96.1%~100%和88.0%~98.8%。当CLBV分离物来源寄主是柑桔时,核苷酸和氨基酸水平变异小,当寄主是甜樱桃和猕猴桃时,核苷酸和氨基酸水平变异大(见表2)。这可能与病毒适应不同寄主有关。
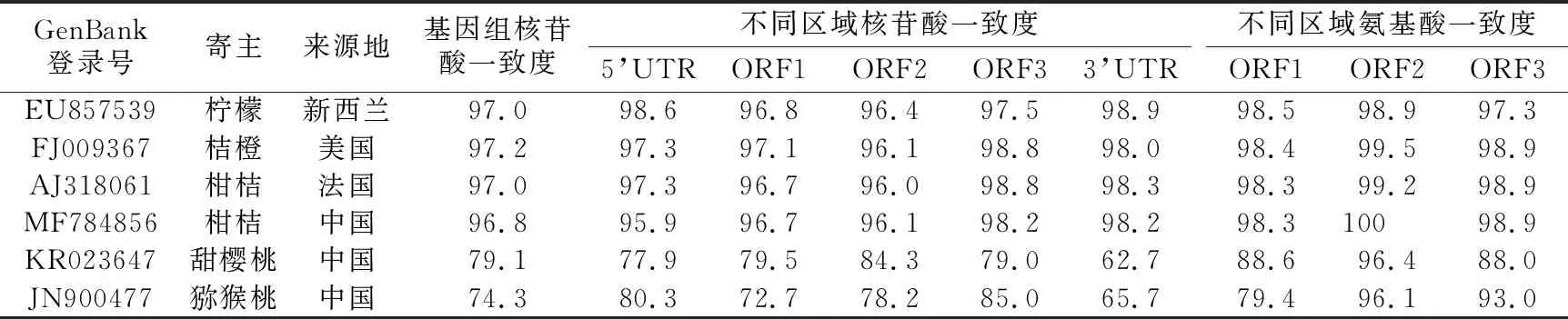
表2 CLBV-JX9分离物(MT863785)与其他来源CLBV分离物的核苷酸和氨基酸序列一致性 %
2.3 CLBV江西分离物基因组序列的系统发育以不同来源CLBV分离物的核苷酸序列进行系统发育分析的结果表明,所有来源于柑桔的CLBV分离物紧密聚为一支,表现出较近的遗传发育关系。来源于甜樱桃的CLBV分离物与来源柑桔的CLBV分离物聚为一个大支,遗传发育关系变远。来源于猕猴桃的3个CLBV分离物形成了两个单独分支,与以上分离物表现出较远的遗传距离(见图2)。以上结果说明,CLBV在进化上可能与对寄主的适应性存在一定相关性,在柑桔上目前遗传变异还较小,而在猕猴桃上变异较大。
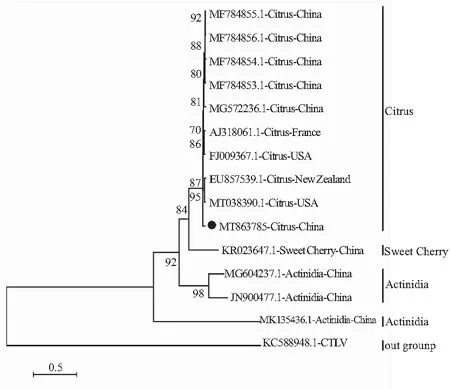
图2 不同来源CLBV分离物基因组序列的系统发育树
3 小结与讨论
本研究利用克隆、测序等技术获得了CLBV-JX分离物的全基因组序列,并对CLBV-JX与其他寄主来源CLBV分离物的序列进行了一致性和系统发育分析。CLBV-JX全长8 747 nt,编码3个ORF。序列一致度比较结果显示,CLBV-JX与来源于柑桔的分离物序列一致度高,核苷酸和氨基酸序列一致度均高于95.9%,这与Vives等[16]所得结果相同。CLBV-JX与来源于甜樱桃和猕猴桃的分离物的一致性低,ORF1和ORF3编码的氨基酸序列一致度最低,分别为79.4%和88.0%,特别是在3’端非编码区,核酸一致度仅为62.7%,显示出该病毒在不同寄主上的高度变异特性。项周等[15]和朱晨熹等[17]针对中国不同CLBV分离物的CP序列分析,发现其在不同寄主间也表现出较大的分子变异。本试验构建的CLBV不同分离物全基因组的核苷酸序列的系统进化树显示,所有柑桔来源CLBV分离物紧密聚为一支,并没有明显的地理差异,但与其他寄主来源分离物表现出较远的系统发育距离。目前,对不同地区其他寄主来源的CLBV分离物报道不多。例如:猕猴桃在我国南方地区也有一定种植面积,但对寄主为南方猕猴桃的CLBV分离物研究相对较少;另外以芍药为寄主的CLBV全基因序列还未有报道。因此,无法确定其他寄主来源CLBV分离物是否存在地理差异。不同寄主间核苷酸序列一致度低在类似病毒的相关研究中也有发现。如:赵磊[18]对已报道的所有苹果茎沟病毒(Apple stem grooving virus, ASGV)的CP基因进行系统进化分析,发现其在以苹果、梨树和柑桔为寄主的分离物间的核苷酸序列存在较大差异,但不同地域间的序列一致度没有明显差异;王利平等[19]发现,苹果褪绿斑病毒(Apple chlorotic leaf spot virus, ACLSV)群体具有遗传多样性,且不同分离物间亲缘关系呈现一定的地域相关性。本研究表明,不同寄主来源CLBV分离物间的核苷酸和氨基酸序列差异较大,可能是因为CLBV需要适应不同寄主环境而发生了变异,此推测有待进一步验证。同时,对于CLBV全基因序列的地理差异,还需通过对其他寄主来源进行深入研究。
