黄曲霉菌的差异挥发性代谢产物及其代谢途径分析
2021-06-21黄海游李淑军钟读波胥志祥孙磊业韩丰霞
黄海游,胡 昊,李淑军,钟读波,胥志祥,孙磊业,韩丰霞
(1.昆明理工大学 环境科学与工程学院,云南 昆明650500;2.云南大学 科技咨询发展中心,云南 昆明650000;3.云南云测质量检验有限公司,云南昆明650000;4.昆明理工大学创新发展研究院,云南昆明650000)
世界卫生组织 (World Health Organization,WHO)公告指出每年发生的食源性疾病达数10亿例;发达国家每年至少有三分之一的人群患食源性疾病,其中约有170万15岁以下的儿童因食源性微生物污染引起腹泻而死亡[1]。据调查,我国2010—2014年,食源性疾病暴发案例有2 000多起,发病人数约有63 000人,其中死亡人数约有970人,而微生物引起食源性疾病暴发案例有41.1%,其中微生物病原菌是引起食源性疾病暴发的主要原因[2]。黄曲霉的污染不仅对人的健康造成巨大伤害,而且对农业经济造成巨大损失[3]。据联合国粮食及农业组织报道,全球每年约有2%的农作物因为受霉菌污染而失去利用价值[4]。
代谢组学以小分子生物标志物为研究对象,相较于大分子物质(蛋白质、脂质和核酸),其研究更快速和准确,因此在不同学科领域作为快速检测方法被加以开发和应用。其中,环境中物质的变化可通过介导微生物代谢使微生物对环境变化产生持续反应[5]。同样地,不同食品中微生物代谢物的变化也能反映出微生物处于在不同环境(食品)中的变化。黄光琦等[6]研究指出,黄曲霉毒素B1(aflatoxin B1)通过与tRNA结合形成加成物(B-tRNA),降低tRNA与某些氨基酸结合的活性,从而抑制必需氨基酸(如赖氨酸,亮氨酸,精氨酸和甘氨酸)与tRNA的结合,最终在翻译水平上干扰了蛋白质生物合成,影响了细胞的正常代谢。Lee等[7]在2015年通过代谢组学分析方法发现了黄曲霉中新的四肽和抗炎代谢产物。谢华里等[8]利用非靶标代谢组学研究了温度对黄曲霉代谢的影响,发现温度影响多个代谢途径,且曲酸和黄曲霉毒素的累积变化规律相似。然而,大分子物质检测的滞后性导致不能提前预防黄曲霉菌产生的潜在健康问题。基于此,作者从小分子代谢物入手,通过寻找食物污染早期的差异性代谢物,为食品安全预防和检测提供技术支撑。
1 材料与方法
1.1 材料与试剂
豆腐干作为一种常见的休闲零食和佐餐佳品,其食品原材料和加工过程都极易被微生物污染[9],因此从豆腐干入手研究黄曲霉菌在豆腐干中的差异性代谢物。
黄曲霉菌(CGMCC 3.6431):购自中国普通微生物菌种保藏管理中心(China General Microbiological Culture Collection Center,CGMCC);即食豆腐干:购自某超市(主要成分为大豆,水,植物油,食用盐,酿造酱油,红糖,芝麻,香辛料,谷氨酸钠,食用香精,双乙酸钠,乙基麦芽酚,5’呈味核苷酸二钠,氯化镁)。乙腈(色谱纯)、甲醇(色谱纯):美国西格玛公司产品;二氯甲烷(色谱纯):美国迈瑞达公司产品;液体察氏培养基:百思生物公司产品。
1.2 仪器与设备
气相色谱质谱联用仪(7890N-5975C):美国安捷伦公司产品,并配自动进样器(A7683B)、MSD化学工作站和NIST 14.0数据库;固相微萃取头(75μm CAR-PDMS):美国色谱科公司产品;高速冷冻离心机(Allegra X-15R):美国贝克曼公司产品;冷冻干燥机(FDU-1200):日本东京理化公司产品;试管分散机(UTTD):德国艾卡公司产品;灭菌锅(BXM-30R):上海博讯公司产品;恒温培养箱(MJ-70-I):上海一恒公司产品。
1.3 实验方法
1.3.1 菌体准备 将-80℃保藏的1 mL菌液加入200 mL察氏培养基中,在30℃培养箱内振荡(150 r/min)培养5 d。将豆腐干和超纯水以质量比1∶10的比例加入灭菌搅拌机里,打碎10 min,制作成豆腐干液体培养基。取培养好的1 mL菌液加入200 mL豆腐干培养基中,在37℃培养箱内培养黄曲霉菌5 d。
1.3.2 代谢物提取 取上述样品10 g于50 mL离心管中,迅速加入10 mL的-40℃冷甲醇溶液(体积分数60%)[10-12],振荡混匀后置于-80℃冰箱中冷冻12 h进行淬灭。
将淬灭后的冰冻样品放于冷冻干燥机中进行冻干,冻干后的样品再加入10 mL乙腈、甲醇和水的混合溶液(体积比为2∶2∶1)[13-15],振荡后将溶液倒入带有3种规格玻璃球的液体均质器中破碎10 min。将破碎后的样品置于37℃培养箱中2 h,进行代谢物的提取。
将反应后的溶液离心15 min(-4℃、10 000 r/min),取出上清液,置于-80℃冰箱中冻藏12 h,再将冰冻样品进行冷冻干燥。在GC-MS应用中,一种是测定生物体自然产生的挥发性代谢物,一种是需要衍生化的代谢物。为了保存更多更原始的物质,选择测定其自然状态下的挥发性代谢物,不作衍生化处理。进样前,加入2 mL二氯甲烷溶解,混匀,然后上机测样。
1.3.3 顶空固相微萃取(HS-SPME)挥发性代谢物取10 mL培养5 d后的豆腐干混合液加入到顶空瓶中,用密封橡胶垫封死,待萃取。
将75μm CAR PDMS固相微萃取头刺入顶空瓶中,在离液面2 cm处进行30 min(40℃)萃取。萃取结束后立即手动上机检测。
1.3.4 GC-MS条件 DB-624毛细管色谱柱(60 m×320μm×1.80μm);载气:高纯氦气;流速:2.0 mL/min;柱温升温程序:初始温度40℃,保持6 min,然后以10℃/min升温至230℃,保持10 min。自动进样1.0μL;采用不分流模式;进样口温度230℃。
质谱条件:离子源温度230℃;接口温度230℃;全扫描模式,质量扫描范围29~350m/z。
1.4 真菌基因组测序
利用目前使用最广泛的二代测序平台Illumina Hiseq×10平台,对质检合格的DNA样品构建插入片段为约400 bp的片段,进行PE150(pair-end)测序,即双端测序,单端测序读长150 bp,每个样品提供不低于基因组100×覆盖深度的原始测序数据量;真菌基因组精细图,采用“二代+三代”的测序模式,对同一样品分别提供≥100倍PE150数据和≥80倍的三代数据,基于所获得的测序数据进行一系列分析。
1.5 数据处理
原始数据经过去噪和归一化后整理成Excel表,通过NIST 14.0质谱数据库比对确认各物质。利用峰面积通过SIMCA 14.1进行主成分分析和正交偏最小二乘判别分析,选取VIP值大于1的物质作为潜在的差异物,根据分子量小、相对产量高、代谢重要性强的原则选取深入分析的代谢物。基因组序列信息通过KEGG genes和KEGG LIGAND数据库比对,进行分类和整理,使用Gephi软件构建代谢网络图。
2 结果与分析
2.1 GC-MS测定结果
图1从下往上分别为6个豆腐干培养基样品总离子流(TIC)图和12个接种黄曲霉菌的豆腐干培养基总离子流图,只有在保留时间25.194 min时有明显的差异,是接种黄曲霉菌的样品中(Z,Z)-9,12-十八碳二烯酸甲酯独有的峰。

图1 豆腐干培养基(CBDB)和接种黄曲霉菌的豆腐干培养基(CBDHQ)总离子流叠放图Fig.1 Total ion chromatogram stacking map of dried bean curd medium and dried bean curd medium inoculated with Aspergillus flavus
图2是顶空固相微萃取实验的总离子流叠放图,从下往上分别为3个豆腐干培养基样品总离子流(TIC)图和3个接种黄曲霉菌的豆腐干培养基总离子流图。分析可知,乙醇、2-甲基-1-丙醇、2-甲基-丁醛、1-戊烯-3-醇、3-甲基-1-丁醇、7-[2-(4-甲氧基苯基)乙烯基]-2-甲基-3-苯基吡唑并[1,5-a]嘧啶是接种了黄曲霉的样品中独有的挥发性物质。需要通过判别模型结合自然挥发特征物质来整理差异代谢物的代谢途径。
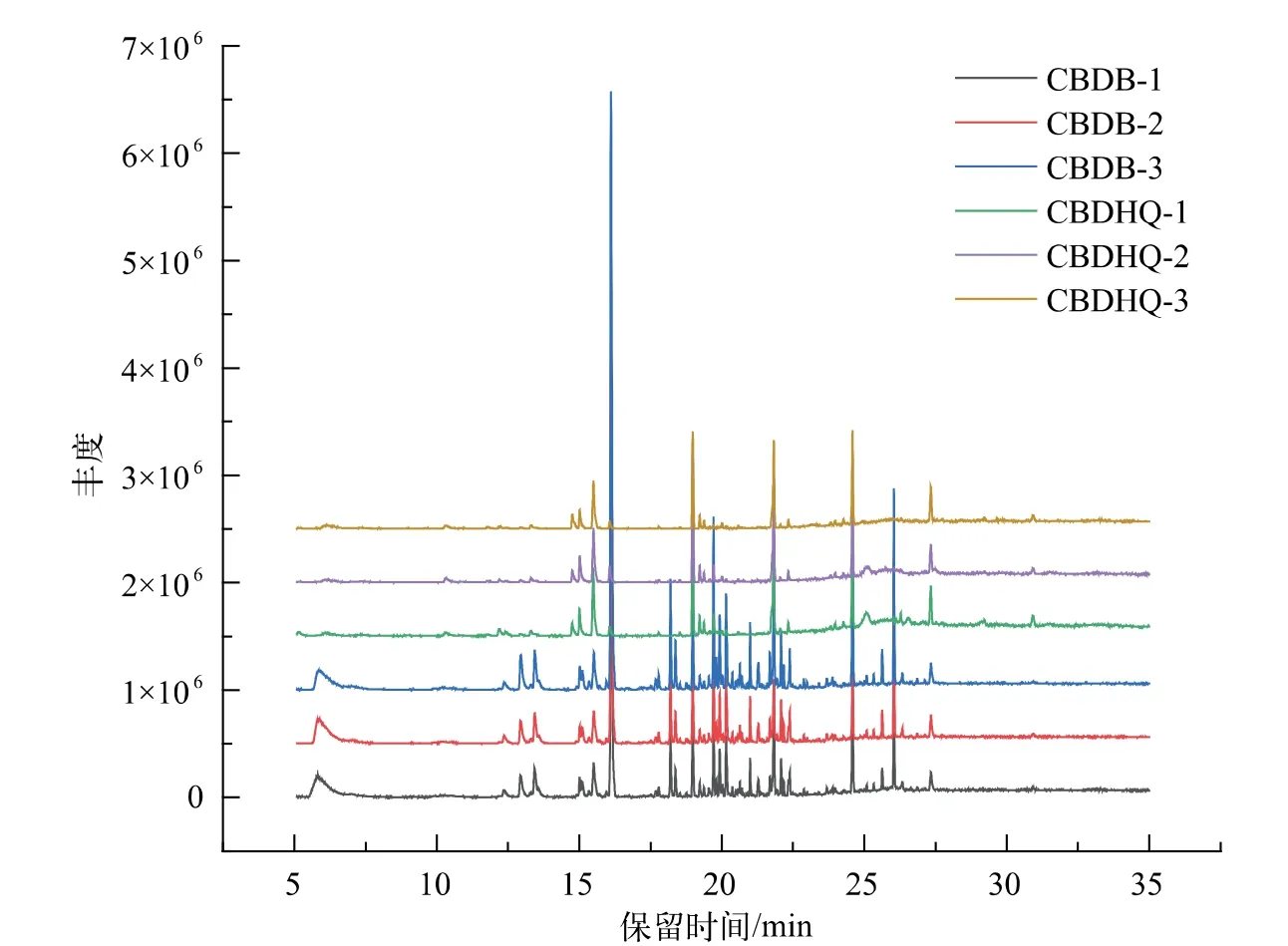
图2 顶空固相微萃取实验的豆腐干培养基(CBDB)和接种黄曲霉菌的豆腐干培养基(CBDHQ)总离子流叠放图Fig.2 Total ion chromatogram stacking map of dried bean curd medium and dried bean curd medium inoculated with Aspergillus flavus in headspace solid phase microextraction
利用检测所得的各物质的归一化峰面积建立模型,主成分分析(PCA)使得由m个样品和m×n个物质峰面积组成的n维矩阵降维到一个平面空间内,将最具差异的影响因子方向作为主成分。主成分分析也是代谢组学中最常用的分析方法[16]。从图3(a)可知,豆腐干培养基(CBDB)和接种黄曲霉菌5 d的豆腐干培养基(CBDHQ)在主成分分析模型上有非常明显的区别,说明两组样品的检出物有着明显的差异。
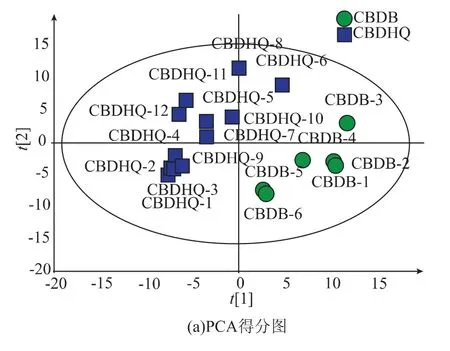
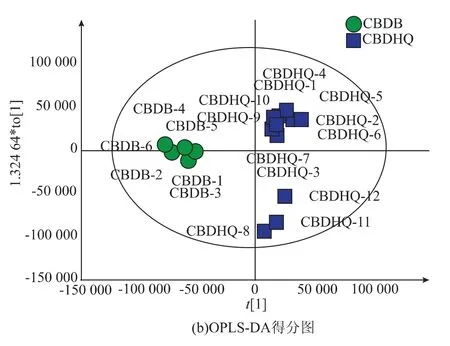
图3 豆腐干培养基(CBDB)和接种了黄曲霉菌的豆腐干培养基(CBDHQ)代谢物主成分分析得分图与正交偏最小二乘判别得分图Fig.3 Principal component analysis score map and orthogonal partial least squares discriminant score map of dried bean curd medium and dried bean curd medium inoculated with Aspergillus flavus
偏最小二乘判别方法(PLS-DA)是一种有监督的分类程序,其中类成员信息用于建立判别模型,因此可用于识别最能鉴别样品组的化合物。作为代谢组学常用的分析方法,2002年Trygg和Wold[17]把PLS算法升级为正交偏最小二乘法(OPLS),OPLS更易于看出两组之间的差异[18]。SIMCA将OPLS-DA也作为了软件14.1版的分析功能。VIP值(投影的可变重要性)总结了变量在解释X和Y相关时的重要性,大于1的VIP值表示重要的X变量,低于0.5的值表示不重要的X变量。1到0.5之间的间隔是灰色区域,其中重要性级别取决于数据集的大小。本研究的OPLS-DA模型见图3(b),模型中R2Y=0.954,Q2=0.755,说明模型有很好的稳定性和预测性。VIP预测值大于1的物质有1,2,3-三甲基-苯、均三甲苯、乙醇、十六烷酸甲酯、2,4-二叔丁基苯酚、乙酸、三氯甲烷、1-丁醇、1-甲基-3-(1-甲基乙基)-苯。
2.2 黄曲霉菌基因组测定结果
基因组全长37 687 868 bp,GC含量为48.2%。共编码13 897个基因,包含10 949个功能基因、272个tRNA基因、43个rRNA基因和2 633个未知基因。在KEGG基因分类系统中,具体的信号通路相关基因有576个;代谢相关基因有2 514个;人类疾病相关基因有787个;遗传信息处理相关基因有788个;组织系统相关基因有555个;环境信息处理相关基因有337个。
为了更清晰地表达黄曲霉菌的代谢信息,利用Gephi软件建立黄曲霉菌的代谢途径关系网络图,见图4,箭头所指方向为代谢正向,图中每个点代表一个代谢途径,点的大小代表与该代谢途径相关的代谢途径的数量。对于黄曲霉菌,大部分的基因表达是关于基础代谢(占代谢途径总数的35.47%)的,且大部分其它代谢途径受基础代谢的影响。其中,代表MAPK信号通路的点在非基础代谢点中最大,表明该通路影响和被影响的代谢途径很多。
乙酸的代谢途径见图5。乙酸在牛磺酸和亚牛磺酸代谢途径中由乙酰磷酸酯经乙酸激酶(ACK)产生。它在丙酮酸代谢中由乙酰辅酶A在乙酰辅酶A合成酶(ACS)作用下产生,也能在乙酰辅酶A合成酶(ACS)或乙醛脱氢酶ALDH(NAD+)作用下由乙醛产生;由乙酰磷酸酯在乙酸激酶(ACK)或酰基磷酸酶(ACYP)作用下产生。乙酸在甲烷代谢途径中由乙酰辅酶A在乙酰辅酶A合成酶(ACS)作用下产生;由乙酰磷酸酯经乙酸激酶(ACK)产生。乙酸主要参与了丙酮酸代谢、糖酵解过程、甲烷代谢过程,它作为乙酰辅酶A的中间产物,极大地影响了黄曲霉毒素的合成[19]。糖酵解过程主要影响了三羧酸循环(TCA cycle),淀粉和蔗糖代谢,戊糖和葡萄糖醛酸相互转化的过程;牛磺酸和亚牛磺酸代谢途径主要影响了氰基氨基酸代谢和谷胱甘肽代谢;丙酮酸代谢主要影响了甘氨酸、丝氨酸、苏氨酸合成和代谢,缬氨酸、亮氨酸、异亮氨酸生物合成,糖酵解和糖异生过程,三羧酸循环,丁酸代谢,酮体的合成与降解,赖氨酸的合成,脂肪酸的合成和烯二炔类抗生素的生物合成;乙酸参与的乙醛酸和二羧酸代谢影响了丙酮酸代谢,三羧酸循环和糖酵解过程;甲烷代谢过程影响了硫代谢,甘氨酸、丝氨酸、苏氨酸代谢,戊糖磷酸途径和乙醛酸、二羧酸代谢。
乙醇是在糖酵解途径中由丙酮酸转化为乙酸,乙酸转化为乙醛,乙醛再经乙醇脱氢酶(ADH1)产生乙醇的代谢途径产生,其主要影响三羧酸循环,淀粉和蔗糖代谢,戊糖和葡萄糖醛酸的相互转化过程。1-丁醇只由丁醛在丁醇脱氢酶(BDH)的作用下生成,影响丙酮酸代谢,三羧酸循环和维生素B6的代谢。2-甲基-1-丙醇、1-戊烯-3-醇可能是在丙酸代谢中衍生出来的产物,与NAD+的脱氢作用有关;2-甲基-丁醛和3-甲基-1-丁醇猜测与丁酸代谢中丁醇脱氢酶(BDH)的作用有关;1,2,3-三甲基-苯、均三甲苯、2,4-二叔丁基苯酚、1-甲基-3-(1-甲基乙基)-苯猜测可能与芳香族化合物代谢有关。其中,乙醇、2-甲基-1-丙醇、2-甲基-丁醛、1-戊烯-3-醇、3-甲基-1-丁醇可作为今后判断黄曲霉污染的潜在特征气味物质。
3 结 语
通过代谢组学研究方法更清晰地区分被黄曲霉污染的豆腐干,并通过模型计算出差异性代谢物1,2,3-三甲基-苯、均三甲苯、乙醇、十六烷酸甲酯、2,4-二叔丁基苯酚、乙酸、三氯甲烷、1-丁醇、1-甲基-3-(1-甲基乙基)-苯。作者结合代谢组学和基因组信息,通过对代谢网络的构建,明确了乙酸、乙醇和1-丁醇在黄曲霉菌代谢网络中的代谢过程和作用。乙醇、2-甲基-1-丙醇、2-甲基-丁醛、1-戊烯-3-醇、3-甲基-1-丁醇也可作为黄曲霉菌的特征性挥发物质。此研究对今后建立快速检测黄曲霉污染的方法有着积极的意义。
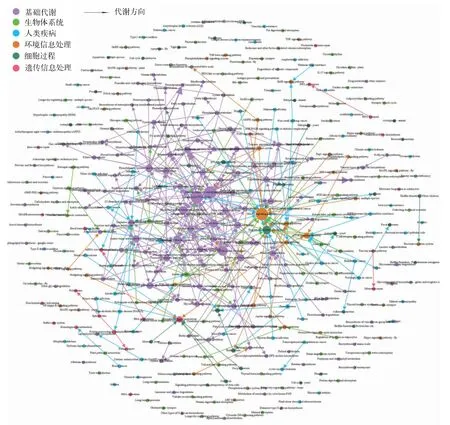
图4 黄曲霉菌代谢网络图Fig.4 Metabolic network map of Aspergillus flavus
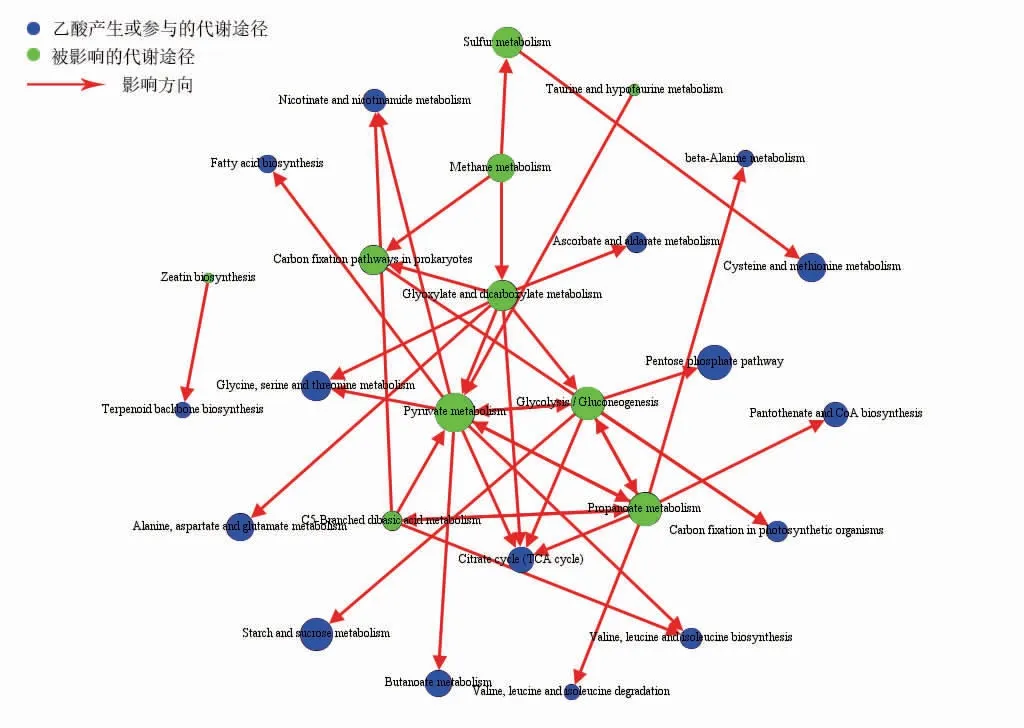
图5 黄曲霉菌中乙酸参与的代谢途径网络Fig.5 Metabolic pathway network of acetic acid in Aspergillus flavus
