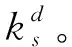基于K-shell的超网络关键节点识别方法
2021-06-19周丽娜李发旭巩云超
周丽娜,李发旭,巩云超,胡 枫
(青海师范大学 a.计算机学院;b.青海省藏文信息处理与机器翻译重点实验室;c.藏语智能信息处理及应用国家重点实验室,西宁 810008)
0 引言



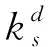
1 相关工作
1.1 超网络相关概念
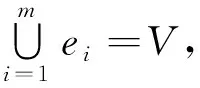
1.2 超网络的拓扑指标
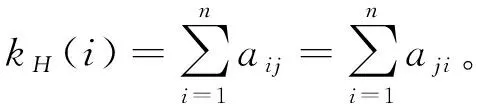
1.3 K-shell算法
K-shell算法是图算法中的一种经典算法,用以计算每个节点的核数。该算法将网络划分为从核心到边缘的不同层次,具体划分过程为:首先,将网络中度为1的节点及其连边删除;其次,删除后网络中将出现新的度为1的节点,继续删除新出现的度为1节点及其连边;最后,重复上述操作直到网络中不再新出现度为1的节点为止。此时所有被删除的节点构成第一层,即1-shell,节点的ks值为1。以此类推,进一步得到更高的壳,直至网络中的所有节点都被赋予ks值。图1为3-shell分解过程示例图。
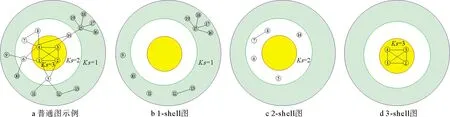
图1 3-shell分解示例图
2 基于超网络的K-shell算法
2.1 K-shell算法在超网络中的基本思想及分解过程
在超网络中,节点超度表示包含该节点的超边数,通常认为超度大的节点重要性高,但超度仅是衡量节点重要性的局部性指标,忽略了超网络全局信息对节点重要性的影响。本文将基于复杂网络位置思想的K-shell指标扩展到超网络中,提出超网络的K-shell分解算法。算法步骤为:1)删除超网络中超度为1的所有节点,删除超度为1的节点后若网络中存在超边超度为1的超边,则删除此超边;重复此过程,直到网络中不存在超度为1的节点及超边,所有被删除的节点ks值为1;2)重复上述操作,删除超网络中所有超度值不大于2的节点和超度为1的超边,此时,所有被删除节点的ks值为2;3)以此类推,直到超网络中所有节点都被赋予ks值。
如图2所示超网络的3-shell分解过程示意图,图2b~图2d为1-shell、2-shell和3-shell分解图。
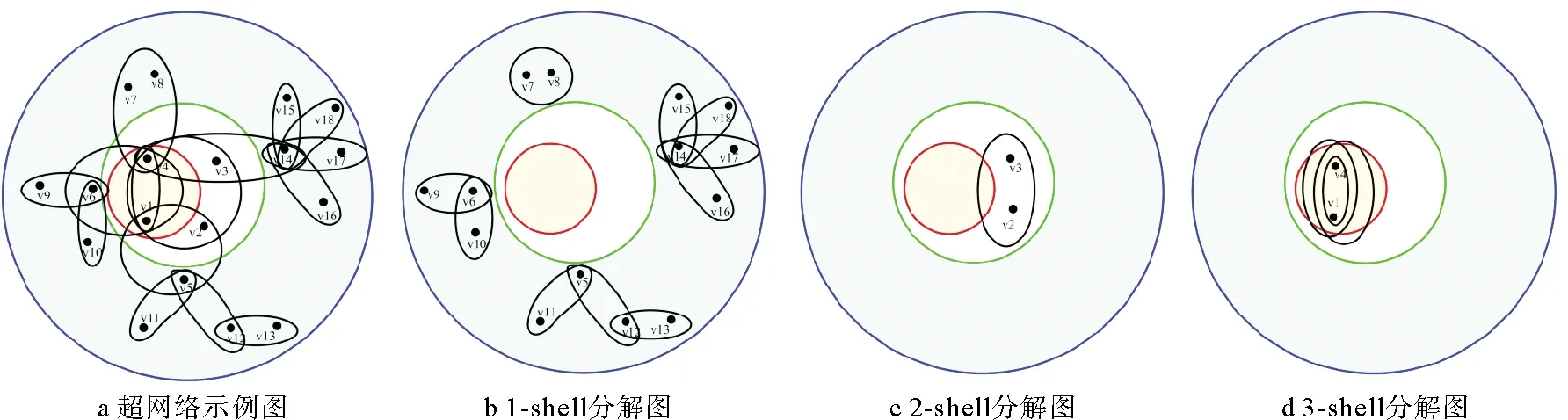
图2 超网络的3-shell分解过程示例图
图2a为图1a中添加超边以后的超网络图,在图1a中删除ks值为1的节点后,其余节点的度值均大于1。继续删除度值为2的节点,即删除节点5以及与它相连的两条边,则节点1与节点2的度值均变为3。与复杂网络不同,如图2a所示,删除超网络中节点7和节点8后,所在超边的超度变为1,依据超网络的K-shell算法删除这条超边。删除节点9和节点10及其所在的超边后,继续删除网络中超度变为1的节点6,删除该节点后,超边的超度为2,因此保留该超边。综上所述,复杂网络中删除一个节点时与之相连的边同时删除;而超网络中,只有当超边中只剩一个节点,即超边超度为1时,才删除此超边。
分析图2a中,超度最大的节点4、节点14,其超度值为5,由K-shell算法得到节点4的ks值为3,节点14的ks值为1。表明节点14位于网络的边缘位置,虽然超度值最大但并不是重要节点。因此,利用超度刻画超网络中的重要节点并不完全准确。但由K-shell算法最终分解得到多个重要节点,导致节点的排序结果过于粗糙,如图2d所示。为了解决此问题,本文进行了改进。
2.2 改进的超网络K-shell算法
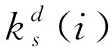
(1)
其中,dH(i)表示节点i的超度,ks(i)表示节点i的ks值。
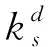

表1 不同指标对示例超网络的排序结果
3 实证分析
3.1 示例超网络分析
为了验证本文算法的有效性和正确性。对图2示例网络进行分析,通过删除节点形成的子网络数目及子图规模大小反映算法的正确性。当删除节点后生成的子图数量越多、子图最大规模越小时,关键节点识别算法的准确性越高。表2为按照节点重要性排序逐一删除节点后得到的子图数及子图最大规模。图3以删除节点数为横坐标,以删除节点后得到的子图数与子图规模为纵坐标,展示不同指标之间的差异。
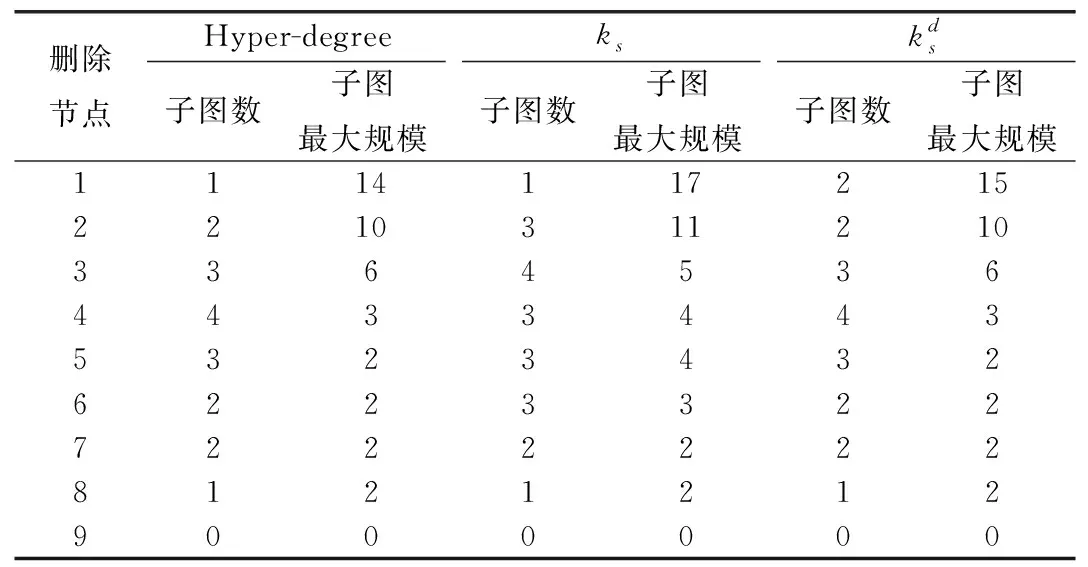
表2 节点被删除后的子图数及子图最大规模
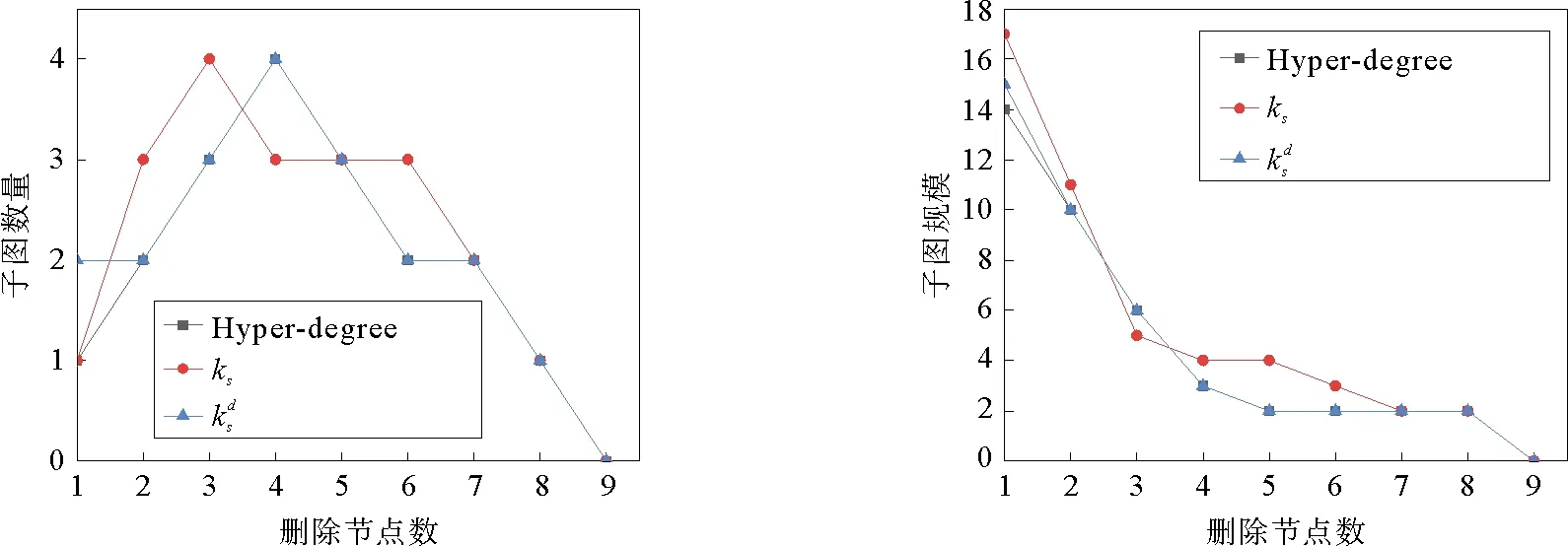
图3 节点被删除后子图数与子图规模变化情况
3.2 数据来源
蛋白复合物数据来自数据库CORUM(Comprehensive Resource of Mammalian Protein Complexes)[33]。该数据库包含2 314个人类蛋白质节点以及它们之间相互作用形成的1 342个复合物超边。这些复合物大多由3~4个蛋白质组成。此外,最大的复合物包含143个蛋白质,而最小的复合物仅有一个蛋白质。在蛋白质复合物超网络中,节点的超度表示包含该蛋白质的复合物的个数,也是判断蛋白质是否是关键蛋白的重要依据。
3.3 实验结果分析
将超网络中的K-shell算法应用于蛋白复合物超网络,从而得到所有蛋白质节点的K-shell值与超度的关系,结果如图4所示。
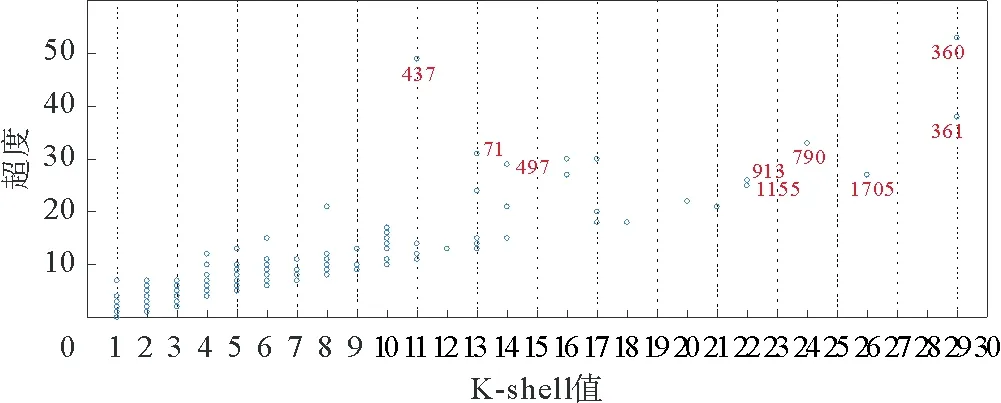
图4 超度与K-shell值的散点图
图4中横轴表示K-shell值,处于同一竖线上的蛋白质具有相同的ks值。纵轴的超度表示该蛋白质复合物的数目。此超网络中,超度值小于10的节点,ks值大部分小于5,占总节点的80%左右,说明大多数蛋白质构成的复合物数量较少。节点360与节点361的ks值为29,处于网络的中心位置,是网络的关键节点。文献[34]以介数和节点度为中心测度分析蛋白质在网络中的位置,提出关键蛋白倾向于位于网络的中心位置。ks值越高代表节点越趋于网络的中心位置。蛋白复合物超网络中节点360与361的ks值最大、处于网络的中心位置,为蛋白复合物超网络中的关键蛋白。由CORUM数据库可知,这两个蛋白质结合在一起参与生成的复合物数量占大多数。在生物学中,HDAC蛋白对细胞染色体的结构修饰和基因表达调控发挥着重要的作用,是具有条件依赖性的必需基因,而节点360的蛋白ID是HDAC1,节点361的蛋白ID是HDAC2。由此可知,超网络的K-shell算法能较为准确地识别网络中的关键节点。
由图4知,ks值小的节点与其超度值呈线性分布,剩余节点分布则较为分散。例如,节点71的超度值大于节点497,但节点71的ks值却小于节点497的ks值。节点437的超度值仅次于节点360,但是它们的ks值相差甚大;节点437的ks值为11,位于超网络拓扑结构的中间位置,因此,蛋白复合物超网络中不存在节点超度值大,却处于网络边缘位置的节点。节点792,920和921的超度值与ks值均为21,但比较超度与ks值的节点排名相差较大,如节点792,根据超度值排名位于第14位,根据ks值排名位于第6位。若将上述蛋白质移除,至少有21种复合物无法形成,从而影响细胞的生物学功能,甚至使生物体无法存活,因此这些蛋白质是构成此类复合物的必需蛋白质。
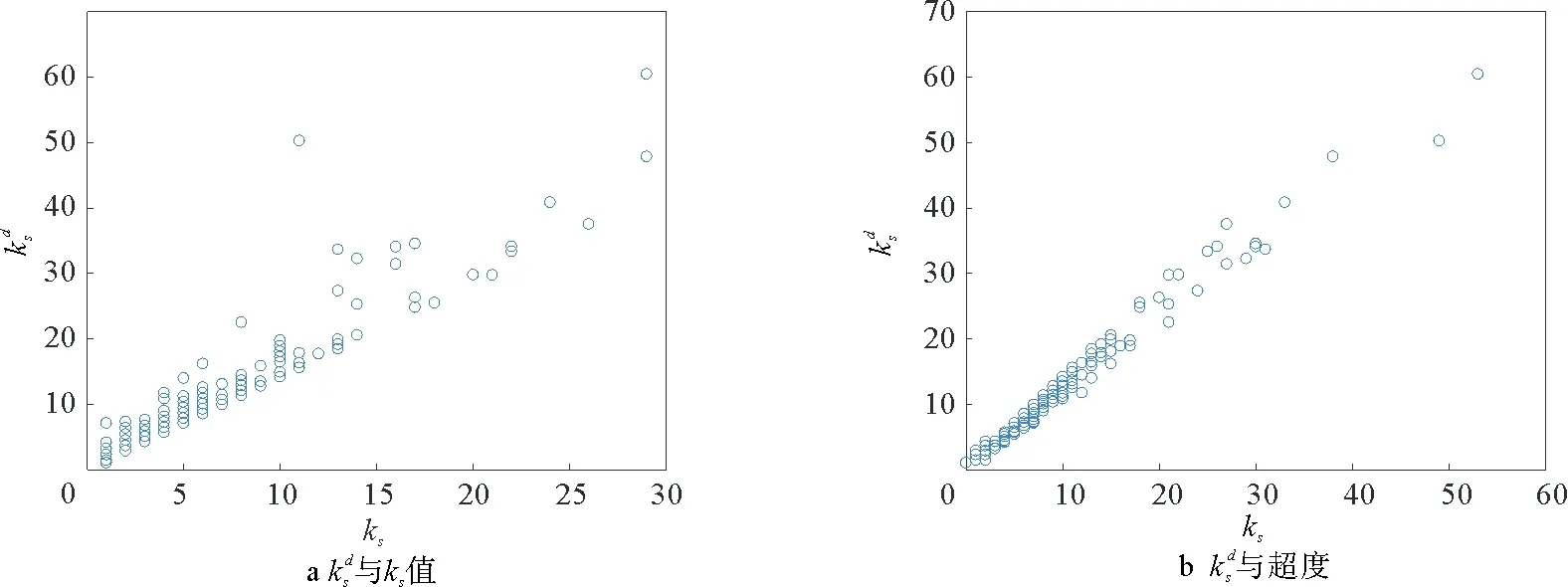
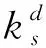

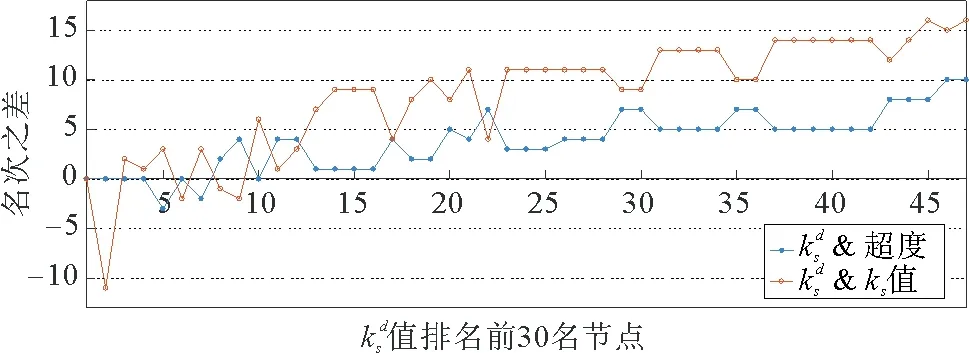
图6 ks值与超度与排名之差
4 结论