过敏者外周血DNA甲基化差异
2021-06-17刘明哲郭华林冯彦李建国李鹏飞高彩荣郭相杰
刘明哲,郭华林,冯彦,李建国,李鹏飞,高彩荣,郭相杰
1.山西医科大学法医学院,山西 晋中 030600;2.山西医科大学第一医院,山西 太原 030000;3.中国辐射防护研究院,山西 太原030000
近年来,过敏反应的发病率逐年升高,已成为影响人类健康的全球性疾病[1]。虽然过敏与遗传因素有关[2],但传统的遗传易感模型不能完全解释近年来过敏反应快速增加的现象。研究表明,过敏反应可能与早期环境暴露有关[3],环境因素可能通过影响DNA 甲基化水平改变基因表达,进而影响过敏的发生、发展[4]。
在我国,严重药物过敏反应导致的死亡也时有发生,法医学实践中常涉及药物过敏性休克死亡的鉴定,其中以头孢类药物过敏最为多见[5]。本研究通过对头孢类药物致过敏性休克死亡者外周血有核细胞DNA 进行甲基化芯片检测,运用生物信息学和随机森林算法进行分析,对DNA 甲基化与头孢类药物过敏的相关性进行研究,为过敏性休克死亡提供新的鉴定方法。
1 材料与方法
1.1 仪器与试剂
2100 生物分析仪(美国Agilent 公司),甲基化芯片Infinium MethylationEPIC BeadChip(简称“850K芯片”),HiSeqTM2000 高通量测序仪(美国Illumina公司)。
磁珠法基因组DNA 提取试剂盒[生工生物工程(上海)股份有限公司],Agencourt AMPure XP 核酸纯化试剂盒(美国Beckman Coulter 公司),EZ DNA Methylation-GoldTM试剂盒(美国Zymo Research公司)。
1.2 研究对象
收集头孢类药物过敏性休克死亡者5 例为实验组,免疫正常者5 例为对照组,分别采集外周静脉血,采用磁珠法提取DNA 样本以备检测。本实验已通过山西医科大学科学研究伦理审查委员会审批(审批号2018LL016)。
头孢类药物过敏性休克死亡者样本从山西医科大学司法鉴定中心获得,均为山西汉族人,年龄20~60 岁,其中男性3 人、女性2 人。入选标准:(1)有明确的头孢类药物输注史或血液(或可疑输注部位皮肤)检出头孢类药物成分;(2)输注头孢类药物后出现休克症状,并在短时间内死亡;(3)咽喉、肺及胃肠组织免疫组织化学染色结果显示类胰蛋白酶、类糜蛋白酶多量阳性表达;(4)排除其他原因(疾病、中毒)死亡的可能;(5)尸体未发生腐败,血清总IgE>200 IU/mL。
对照组为身体健康的山西汉族人,年龄20~25岁,其中男性2 人、女性3 人,无过敏史、既往无手术史,实验室皮肤点刺实验均为阴性,血清总IgE<100 IU/mL,中国人群常见过敏原特异性IgE 均为阴性,头孢类及青霉素类药物皮试阴性。
1.3 构建DNA文库
(1)样本DNA 片段化处理后连接甲基化接头并纯化;(2)加接头的DNA 片段经重亚硫酸盐处理,未发生甲基化的C 碱基转化为U 碱基;(3)利用PCR 选择性富集、扩增加接头的DNA 片段,构建DNA 文库;(4)使用定量聚合酶链反应(quantitative polymerase chain reaction,qPCR)进行DNA 文库定量;(5)使用2100 生物分析仪对PCR 富集片段进行质量控制,验证DNA 文库的片段大小及分布。
1.4 DNA甲基化检测
基于二代测序平台(HiSeqTM2000高通量测序仪),使用850K 芯片对头孢类药物过敏性休克死亡者与免疫正常者进行DNA 甲基化检测。
1.5 过敏相关DNA甲基化位点的筛选
对数据进行质量控制和过滤(过滤低质量位点、SNP 干扰位点等)后,使用R 语言methylkit 包分析测序数据、R 语言ChAMP 包分析芯片数据,得到头孢类药物过敏性休克死亡者与免疫正常者的DNA 甲基化差异位点,与人类参考基因组37(Genome Reference Consortium human 37,GRCh37;https://grch37.ensembl.org)比对并进行注释。使用R 语言随机森林包(randomForest)基于所得DNA 甲基化差异位点进行有监督的随机森林类别回归(classification)分析,并评估DNA 甲基化差异位点重要性[6]。在R 语言的随机森林算法(randomForest 包)中,使用MeanDecreaseAccuracy(变量被替换后的准确率下降平均值)与MeanDecreaseGini(变量被替换后Gini 指数下降平均值)参数对变量的重要性进行评估,数值越大,重要性越高,即对模型准确分型的贡献越高,则该变量(DNA 甲基化差异位点)的差异更为显著。对两种参数获得DNA 甲基化差异位点的可靠性进行评估时,认为分布梯度(即不同差异位点之间横坐标指数差异)越明显,该参数获得的DNA 甲基化差异位点越可信[6]。随机森林函数关键参数:ntree=500,mtry=2,importance=TURE;余为默认值。
2 结果
对头孢类药物过敏性休克死亡者与免疫正常者外周血DNA 进行甲基化检测后,分析甲基化差异位点,得到约10000个甲基化差异位点(P<0.05)。使用R语言randomForest 包进行有监督的随机森林类别回归分析,评估DNA 甲基化差异位点的重要性,得到前30 个位点(图1)。由图1 可见,在评估头孢类药物过敏相关DNA 甲基化位点的可靠性时,MeanDecrease-Accuracy 的梯度分布更优,MeanDecreaseAccuracy 前30 个位点的详细信息见表1,其中头孢类药物过敏相关前30 个甲基化差异位点对应在ETS1、PRR23B、GNAS等27 个基因上。
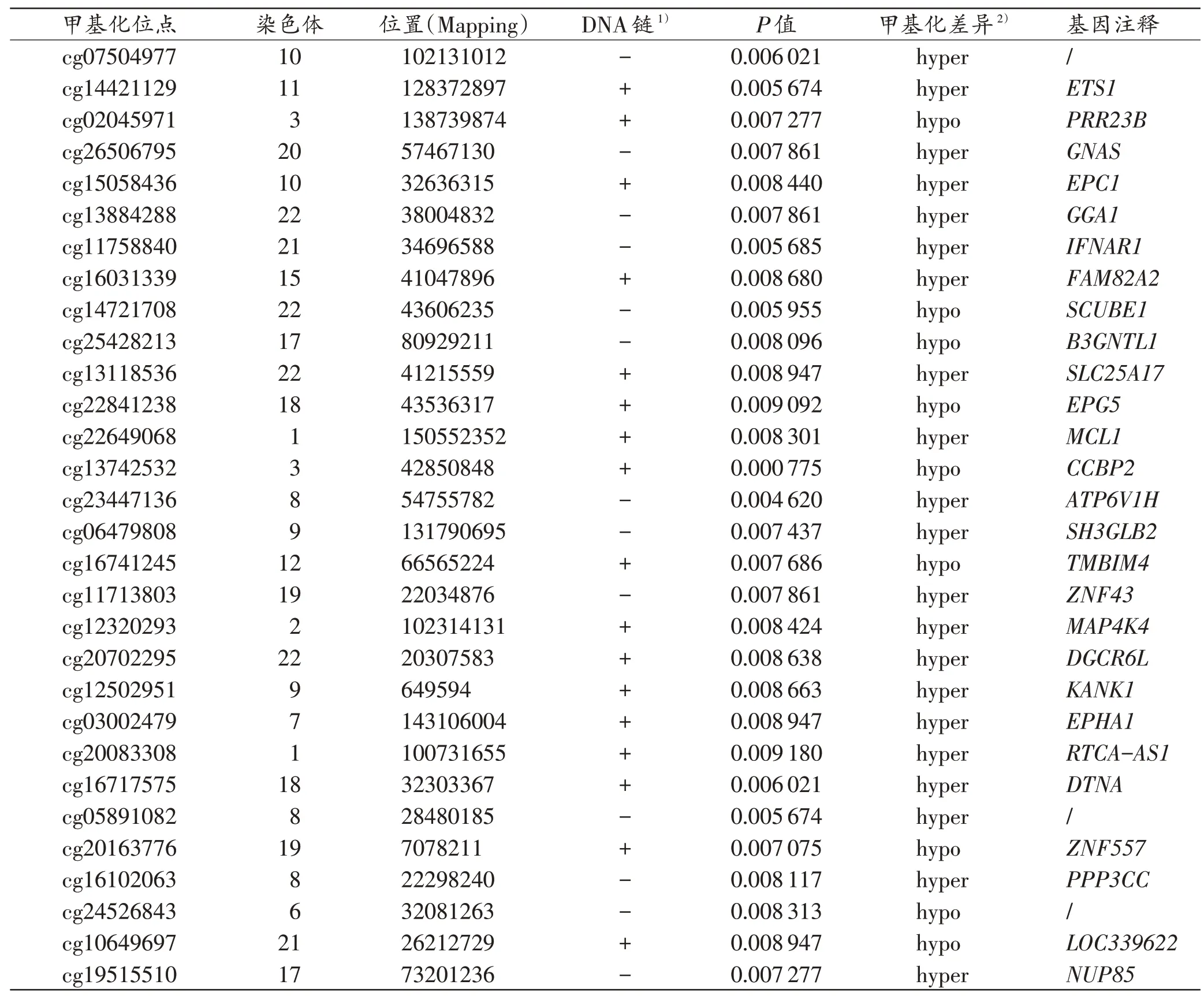
表1 头孢类药物过敏相关DNA甲基化差异位点信息(MeanDecreaseAccuracy前30个)Tab.1 Information of the DNA methylation differential sites associated with cephalosporin drugs anaphylaxis(top 30 of MeanDecreaseAccuracy)

图1 头孢类药物过敏相关DNA甲基化差异位点的重要性(前30个)Fig.1 Importance of DNA methylation differential sites associated with cephalosporin drug anaphylaxis(top 30)
3 讨论
药物过敏性休克死亡的死因判定是法医病理学鉴定的一大难题。目前,涉及过敏性休克死亡的鉴定,是在结合案情、发病经过、临床症状、尸体检验结果和实验室检查结果的基础上,综合分析得出的排除性结论[7]。因此,寻找准确、客观的实验室诊断指标是药物过敏性休克死亡法医学鉴定亟待解决的问题。
已有研究[3-4]表明,DNA 甲基化水平的改变与过敏反应的发生、发展有关。但是,目前过敏与DNA 甲基化的相关研究大都局限于过敏性哮喘[8-9]、过敏性紫癜等过敏性疾病中。本研究对头孢类药物过敏性休克死亡者外周血DNA 甲基化进行检测、分析,获得10 000 个甲基化差异位点,其中前30 个位点对应ETS1、PRR23B、GNAS等27 个基因。HE 等[10]研究认为,ETS1基因甲基化通过调节T 细胞参与了过敏性疾病的发生,说明头孢类药物过敏与DNA 甲基化有关,通过寻找DNA 甲基化差异可能为药物过敏性休克死亡的法医学鉴定提供辅助诊断依据。本实验筛选获得的DNA 甲基化差异位点,可将头孢类药物过敏性休克死亡者与正常人群区分开来,说明其有望应用于过敏性休克死亡案例的鉴定诊断。但由于本实验样本量相对较小,在实际应用中还存在一定的局限,下一步实验还需进一步扩大样本量进行药物过敏与DNA 甲基化差异研究。
由于不同过敏原的致敏机制不同[11-12],致敏的发生也与环境影响因素有关[13-14],因此,法医学实践中运用DNA 甲基化差异进行药物过敏性休克死亡法医学辅助诊断时,应考虑到药物种类不同其相关的DNA甲基化差异位点(基因)也不同。
综上所述,头孢类药物过敏与DNA 甲基化相关,通过寻找DNA 甲基化差异可能为药物过敏性休克死亡的法医学鉴定提供辅助诊断依据。
